Дизайн системы олигонуклеотидов и оптимизация условий амплификации ECF-генов бактерий рода Salinicola семейства Halomonadaсеае
Автор: Ананьина Л.Н., Шестакова Е.А., Пьянкова А.А., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 3, 2017 года.
Бесплатный доступ
Осуществлен поиск нуклеотидных последовательностей ес/-оперона бактерий сем. Halomonadaceae в публичной базе данных Национального центра биотехнологической информации (США) (bttps://www.ncbi.rdm.nrhgov). Анализ выравненных 33 нуклеотидных последовательностей представителей родов Salinicola, Halomonas, Chromohalobacter, Kushneria, Cobetia и Halotalea позволил обнаружить консервативные области. К ним была разработана система олигонуклеотидных праймеров для амплификации фрагмента ес/-оперона, включающего гены: ес/А, кодирующий Ь-2,4-диаминобутир-атацетилтрансферазу, и ectB, детерминирующий Ь-2,4-диаминобутиратаминотрансферазу. Экспериментально подобраны условия полимеразной цепной реакции и состав ПЦР-смеси для амплификации участка ес/-оперона на ДНК матрице бактерий рода Salinicola.
Олигонуклеотиды, ес/-гены, полимеразная цепная реакция
Короткий адрес: https://sciup.org/147204838
IDR: 147204838
Текст научной статьи Дизайн системы олигонуклеотидов и оптимизация условий амплификации ECF-генов бактерий рода Salinicola семейства Halomonadaсеае
Осуществлен поиск нуклеотидных последовательностей ect-оперона бактерий сем. Halomonadaceae в публичной базе данных Национального центра биотехнологической информации (США) . Анализ выравненных 33 нуклеотидных последовательностей представителей родов Salinicola, Halomonas, Chromohalobacter, Kushneria, Cobetia и Halotalea позволил обнаружить консервативные области. К ним была разработана система олигонуклеотидных праймеров для амплификации фрагмента ect-оперона, включающего гены: ectA, кодирующий L-2,4-диаминобутир-атацетилтрансферазу, и ectB, детерминирующий L-2,4-диаминобутиратаминотрансферазу. Экспериментально подобраны условия полимеразной цепной реакции и состав ПЦР-смеси для амплификации участка ect-оперона на ДНК матрице бактерий рода Salinicola.
морских солеварнях, морях и океане [Ананьина и др., 2007; Aguilera et al., 2007; Kim et al., 2007; Huo et al., 2013; Raju et al., 2016].
Известно, что для выживания в условиях засоления бактерии поддерживают осмотический баланс, как правило, за счет синтеза de novo «совместимых веществ», основным из которых для эубактерий является эктоин. В настоящее время охарактеризованы генетические и биохимические системы биосинтеза эктоина бактерий родов Chromohalobacter и Halomonas семейства Halomo-nadaceae [Ono et al., 1999; Calderon et al., 2004; Cai et al., 2011; Schwibbert et al., 2011; Pastor et al., 2013], в то время как бактерии рода Salinicola остаются менее изученными [Olsson et al., 2017]. Синтез эктоина начинается реакцией трансаминирования L-аспартат-β-полуальдегида с последующим ацетилированием образующегося диаминобутирата (ДАБ) и завершается циклизацией Nγ-ацетил-L-2,4-диаминобутирата в эктоин. У бактерий семейства Halomonadaceae ген ect A, кодирующий L-2,4-ДАБацетилтрансферазу, и гены ect B и ect C, детерминирующие L-2,4-ДАБамино-трансферазу и эктоинсинтазу, соответственно, расположены в перечисленной последовательности и организованы в единый оперон ect АBС [Ono et al., 1999; Calderon et al., 2004].
В настоящее время в публичной базе данных Национального центра биотехнологической информации США (NCBI) представлены нуклеотидные последовательности ect-оперона четырех штаммов бактерий рода Salinicola, из них типовым штаммом валидно описанного вида является лишь Salinicola socius SMB35T. Таким образом, имеющаяся информация не дает представления о разнообразии ect-генов бактерий рода Salinicola. Ввиду этого представляется актуальным амплификация и последующее определение нуклеотидной последовательности ect-генов типовых штаммов рода Salinicola.
Из литературных источников известны пары вырожденных праймеров для амплификации ect- генов бактерий семейства Halomonadaceae [Kuhlmann, Bremer, 2002; Okamoto et al., 2004; Ананьина, Плотникова, 2011]. Проведенный нами анализ данных праймеров in silico показал, что представленные в базе данных NCBI нуклеотидные последовательности ect -генов бактерий рода Salinicola имеют несовпадения в 3’-области праймеров, что, в свою очередь, может затруднить амплификацию. Отсюда следует очевидная потребность в новой эффективной системе олигонуклеотидов для амплификации и секвенирования интересующих нас генов.
Целью данной работы явилась разработка и оптимизация системы вырожденных праймеров для амплификации и секвенирования ect-генов у типо- вых штаммов валидных видов рода Salinicola семейства Halomonadaceae.
Материалы и методы исследования
Объекты исследования
Объектами исследования были типовые штаммы узаконенных видов S. salarius DSM 1844T, S. peritrichatus JCM 18795T, S. acroporae JCM 30412T, S. halophilus CECT 5903T, S. socius SMB35T.
Среды и условия культивирования
Минеральная среда Раймонда (г/л деионизированной воды): NH 4 NO 3 – 2.0, MgSO 4 x7H 2 O – 0.2, KH 2 PO 4 – 2.0, Na 2 HPO 4 – 3, CaCl 2 x6H 2 O – 0.01, Na 2 CO 3 – 0.1, pH – 7.0 [Raymond, 1961].
Агаризованная богатая среда Раймонда следующего состава (г/л среды Раймонда): триптон – 5, дрожжевой экстракт – 2.5, NaCl – 30, агар – 15.
Культивирование бактерий проводили на ага-ризованной богатой среде Раймонда в термостатируемом шкафу ТС-1/80 СПУ (Россия) при температуре 28оС.
Молекулярно-генетические и биоинформационные методы исследования
Геномную ДНК выделяли методом щелочного лизиса целых клеток 1 колонии бактериальной культуры, которую вносили в эппендорф, содержащий 100 мкл 0.05 М раствора NaOH, перемешивали и далее выдерживали 15 мин. при 95 ° С, после этого замораживали при -20 ° С в течение 15 мин. Процедуру последовательного замораживания и оттаивания повторяли 3 раза [Versalovic et al., 1994].
Амплификацию ect -генов на матрице ДНК исследуемых штаммов проводили на приборе С1000 Touch («Bio-Rad», США).
ПЦР выполняли в 25 мкл смеси, состоящей из 1х буфера для Taq -полимеразы («Синтол», Россия), 0.25 мМ дНTФ, 1.5 мМ MgCl 2 , 0.004 мг БСА, 10 пкмоль каждого праймера, 2 ед. акт. Taq- полимеразы («Синтол», Россия) и 20 нг геномной ДНК. Кроме того, была использована ПЦР-смесь того же состава, в которой количество каждого праймера было увеличено до 50 пкмоль.
Процедура ПЦР включала начальную денатурацию при 95°С, которая длилась 5 мин. Далее следовали 30 циклов, состоящих из последовательно повторяющихся шагов: денатурации при 94°С в течение 30 сек., отжига праймеров при температурах 38, 39, 42, 46, 50, 54, 57 или 58°С на протяжении 30 сек., каждый цикл завершала элонгация при 72°С длительностью 1 мин. 20 сек. После 30-го цикла следовала терминальная элонгация при 72°С продолжительностью 5 мин. Условия последующих реакций были теми же, но температура отжига праймеров составляла 50°С.
Продукты амплификации разделяли электрофорезом в 1%-ном агарозном геле в 0.5х ТВЕ-буфере в напряжении электрического поля 5.0 V/см в течение 40 мин. ДНК была визуализирована после окрашивания бромистым этидием (0.5 мгк/мл) в проходящем УФ-свете и документирована системой Gel Doc XR («Bio-Rad», США) для преобразования сигналов в цифровые данные. Отображаемая на дисплее электрофореграмма, состояла из пикселей. Каждый пиксель имел координаты X и Y, предоставляющие информацию о горизонтальном и вертикальном положении на электрофореграмме, и значение Z - интенсивность сигнала пикселя. Полученные электрофореграммы обрабатывали с
Секвени ро вание генов проводили с помощью разработанных праймеров, Big Dye Terminator Ready Reaction Kit v3.1 («Thermo Fisher Scientific», США) на приборе Genetic Analyzer 3500xl («Thermo Fisher Scientific», США), следуя инструкциям фирмы-производителя.
Биоинформационное обеспечение. Публичная база данных Национального центра биотехнологической информации США (NCBI) . Пакет программ BioEdit ( edu), позволяющий выравнивать нуклеотидные последовательности. Пакет программ OligoAnalyzer 3.1 для определения термодинамических и структурных характеристик праймеров. Подбор праймеров осуществляли, следуя рекомендациям [Патрушев, 2004; Designing PCR primers and probes decoded-articles/pipet-tips/decoded/2013/10/21/ designing-pcr-primers-and-probes].
Результаты и их обсуждение
Конструирование праймеров
Поиск генетических локусов, кодирующих ферменты синтеза эктоина, осуществляли в геномах умеренных галофильных бактерий сем. Halomonadaceae, депонированных в публичной базе данных NCBI (США). В анализ были включены нуклеотидные последовательности 33 штаммов бактерий представителей родов Halomonas , Chromohalobacter , Cobetia, Kushneria, Halotalea и Salinicola .
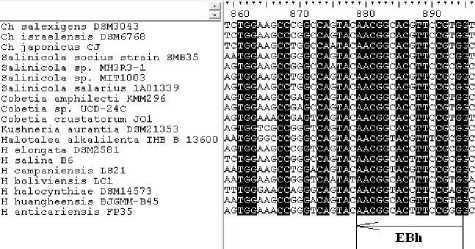
С применением пакета программ BioEdit нуклеотидные последовательности генов ect-оперона были выравнены (рис. 1). Далее осуществляли поиск консервативных областей длиной от 16 (минимально допустимая длина праймера) до 20 нуклеотидов, у которых на 3'-конце предполагаемого праймера первые два нуклеотида были одинаковыми для всех последовательностей, а третий нуклеотид имел не более 2 замен. На 5'-конце предполагаемого праймера допускали наличие позиций, имеющих до 4 нуклеотидных замен.
а
б
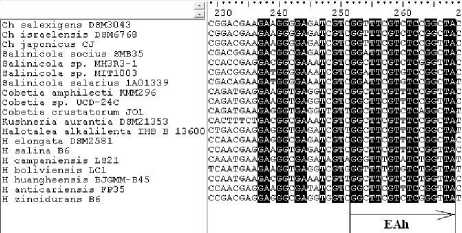
Рис. 1. Множественное выравнивание нуклеотидных последовательностей ectA- (а) и ectB-генов (б) представителей родов Halomonas, Chromohalobacter, Cobetia, Kushneria, Halotalea и Salinicola семейства Halomonadaceae:
консервативные нуклеотиды выделены цветом, участки, комплементарные праймерам, ограничены линиями и стрелками, указывающими направление амплификации
Было обнаружено две области, соответствующие перечисленным параметрам: позиции 253— 269 ect А-гена и позиции 880—895 ect В - гена согласно нумерации Chromohalobacter salexigens DSM 3043T (AJ011103) (рис. 1). Принимая во внимание в выбранных регионах вариабельные нуклеотидные позиции, были сконструированы следующие праймеры:
EAh 5' GGITTYGTITCIGGYTA 3' и
EBh 5' CICGRAAIGTRCCGTT 3'. Пара праймеров имела допустимую разницу в содержании ГЦ-пар (до 6%). Отличие между минимальными и максимальными значениями температур плавления праймеров составило 3.4 и 2.1оС, соответственно. Праймеры не образовывали тугоплавких шпилек со свободной энергией Гиббса менее -3 ккал•моль-1. Однако были выявлены гомо- и гетеродимеры, образующиеся за счет спаривания от 2 до 5 нуклеотидов, характеризующиеся ΔG более -9 ккал•моль-1. Биохимические, термодинамические и структурные характеристики подобранных праймеров представлены в таблице.
Характеристика праймеров
|
Параметр |
EAh |
EBh |
|
Длина, п.н. |
17 |
16 |
|
Степень вырожденности* |
4 |
4 |
|
Содержание ГЦ, % |
50 |
56.2 |
|
Температура плавления, оС мин. - макс. (средняя) |
42.2—57.6 (50.1) |
45.6—59.7 (52.5) |
|
Тугоплавкие шпильки ΔG < -3 ккал • моль-1 |
- |
- |
|
Количество гомодимеров, мин. – макс. значение ΔG (ккал • моль-1), в скобках указано количество спаренных оснований |
18, ΔG -0.96 ккал • моль-1 (2) — ΔG -8.19 ккал • моль-1 (4) |
17, ΔG -2.24 ккал • моль-1 (2) — ΔG -8.45 ккал • моль-1 (4) |
|
Количество гетеродимеров, значение ΔG (ккал • моль-1), в скобках указано количество спаренных оснований |
17, ΔG -1.46 ккал • моль-1 (2) — ΔG -9.97 ккал • моль-1 (5) |
|
|
Порядковый № нуклеотида от начала гена, соответствующего 5'-концу праймера согласно нумерации Ch. salexigens DSM 3043T (AJ011103), в скобках указано название гена |
253 ( ect A) |
895 ( ect B) |
* Степень вырожденности праймера рассчитывали, перемножая количество вариантов нуклеотидов в каждой позиции.
Апробирование праймеров
С целью оптимизации условий полимеразной цепной реакции провели несколько реакций, отличающихся значениями температуры на этапе отжига. Установлено, что наибольшая концентрация продукта ПЦР была получена при температуре отжига праймеров 50 ° С (рис. 2). Далее провели тестирование праймеров на ДНК матрице типовых штаммов валидных видов рода Salinicola в реакционной смеси того же состава при температуре отжига 50 ° С. Для штаммов S. salarius DSM 1844T, S. peritrichatus JCM 18795T, S. acroporae JCM 30412T получен один продукт длиной около 1200 п.н., соответствующий рассчитанной длине такового Ch. salexigens DSM 3043T. В то время как на ДНК матрице штамма S. socius SMB35T амплификация привела к синтезу одного неспецифического фрагмента длиной около 200 п.н., отличающегося от ожидаемой.
Для штамма S. halophilus CECT 5903T ПЦР-продукт не был синтезирован (рис. 3а). Используемые праймеры являются вырожденными, т.е. представляют смесь комбинаций нуклеотидных последовательностей в неизвестном соотношении. Вероятно, концентрация варианта олигонуклеотида, комплементарного нуклеотидным последовательностям генов-мишеней штаммов S. halophilus CECT 5903T и S. socius SMB35T, меньше других. Поэтому была увеличена концентрация олигонуклеотидов в ПЦР смеси с 10 пкмоль до 50 пкмоль, что привело к синтезу искомого фрагмента на матрице ДНК штаммов S. socius SMB35T и S. halophilus CECT 5903T (рис. 3б). Но на матрице ДНК штамма S. socius SMB35T были синтезированы неспецифические фрагменты размером около 800 п.н. и 200 п.н.
а
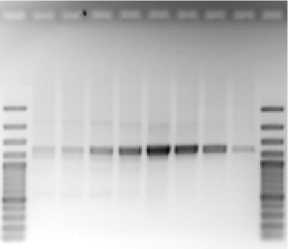
М 1 2 3 4 5 6 7 8 М
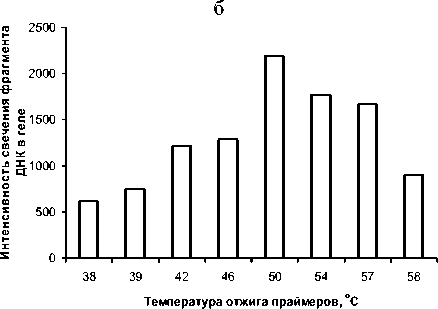
Рис. 2 . Электрофореграмма продуктов амплификации фрагмента ect -оперона штамма S. pe-ritrichatus JCM 18795T при разных температурах отжига праймеров (а) и концентрация ПЦР-продукта, оцененная по интенсивности свечения амлифицированного фрагмента ДНК (б):
1 — 38 ° С, 2 — 39 ° С, 3 — 42 ° С, 4 — 46 ° С, 5 — 50 ° С, 6 — 54 ° С, 7 — 57 ° С, 8 — 58 ° С; М — маркер молекулярных длин 100 bp Plus («Thermo Fisher Scientific, США»)
Для подтверждения амплификации фрагмента ect- оперона была определена нуклеотидная последовательность ПЦР-продуктов типовых штаммов S. salarius DSM 1844T, S. peritrichatus JCM 18795T,
S. acroporae JCM 30412T, S. halophilus CECT 5903T. Последующий сравнительный анализ полученных нуклеотидных последовательностей с депонированными в публичных базах ect-генами с помощью программы Blastn выявил их гомологию с ect-генами бактерий рода Salinicola семейства Halomonadaceae.
а
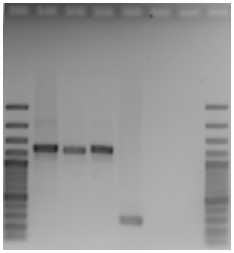
М 1 2 3 4 5 К М б
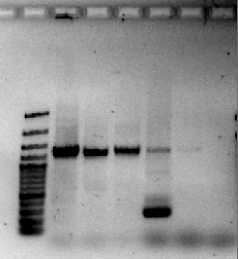
М 1 2 3 4 5 К
Рис. 3 . Электрофореграмма продуктов амплификации фрагмента ect -оперона штаммов рода
Salinicola при концентрации праймеров 10 пкмоль (а) и 50 пкмоль (б):
-
1 — S. peritrichatus JCM 18795T, 2 — S. acropo-rae JCM 30412T, 3 — S. salarius DSM 1844T, 4 — S. socius SMB35T, 5 — S. halophilus CECT 5903T,
M — маркер молекулярных длин 100 bp Plus («Thermo Fisher Scientific, США »), K — отрицательный контроль без ДНК
Заключение
Таким образом, предложенную систему праймеров можно использовать для амплификации фрагмента ect -оперона у бактерий рода Salinicola семейства Halomonadaceae.
Исследование выполнено при финансовой поддержке РФФИ и Министерства образования и науки Пермского края в рамках проекта № 17-44590178.
Список литературы Дизайн системы олигонуклеотидов и оптимизация условий амплификации ECF-генов бактерий рода Salinicola семейства Halomonadaсеае
- Ананьина Л.Н. и др. Salinicola socius gen. nov., sp. nov. -новая умеренно галофильная бактерия из ассоциации микроорганизмов, утилизирующей нафталин//Микробиология. 2007. Т. 76, № 3. С. 369-376.
- Ананьина Л.Н, Плотникова Е.Г. Новая система олигонуклеотидов для амплификации ес/-генов бактерий семейства Halomonadaceae II Вестник Уральской медицинской академической науки. 2011. Т. 4, № 1. С. 17.
- Патрушев Л.Н. Искусственные генетические системы. Т. 1: Генная белковая инженерия. М.: Наука, 2004. 526 с.
- Aguilera М. et al. Chromohalobacter salarius sp. nov., a moderately halophilic bacterium isolated from a solar saltern in Cabo de Gata, Almeria, southern Spain//Int. J. Syst. Evol. Microbiol. 2007. Vol. 57. P. 1238-1242.
- Cai L. et al. Comparative genomics study of polyhy-droxyalkanoates (PHA) and ectoine relevant genes from Halomonas sp. TD01 revealed extensive horizontal gene transfer events and co-evolutionary relationships//Microb. Cell Fact. 2011. Vol. 10. P. 1-15. URL: http://www.microbialcellfactories.eom/content/10/l/8 8.
- Calderon M.I. et al. Complex regulation of the synthesis of the compatible solute ectoine in the halophilic bacterium Chromohalobacter salexigens DSM 3043T//Microbiology. 2004. Vol. 150. P. 3051-3063.
- de la Haba R.R. et al. Taxonomic study of the genus Salinicola: transfer of Halomonas salaria and Chromohalobacter salarius to the genus Salinicola as Salinicola salarius comb. nov. and Salinicola halophilus nom. nov., respectively//Int. J. Syst. Evol. Microbiol. 2010. Vol. 60. P. 963-971.
- Designing PCR primers and probes URL: https://eu. idtdna. com/pages/decoded/decoded-articles/pipettips/decoded/2013/10/21/designing-pcr-primers-and-probes.
- Huo Y.-Y. et al. Salinicola peritrichatus sp. nov., isolated from deep-sea sediment//Antonie van Leeuwenhoek. 2013. Vol. 104. P. 55-62.
- Kim K.K. et al. Halomonas gomseomensis sp. nov., Halomonas janggokensis sp. nov., Halomonas salaria sp. nov. and Halomonas denitrificans sp. nov., moderately halophilic bacteria isolated from saline water//Int. J. Syst. Evol. Microbiol. 2007. Vol. 57. P. 675-681.
- Kuhlmann A.U., Bremer E. Osmotically regulated synthesis of the compatible solute ectoine in Bacillus pasteurii and related Bacillus spp.//Appl. Environ. Microbiol. 2002. Vol. 68, № 2. P. 772-783.
- Lepcha R.T. et al., Comparative 16S rRNA signatures and multilocus sequence analysis for the genus Salinicola and description of Salinicola acroporae sp. nov., isolated from coralAcropora digitiferall Antonie van Leeuwenhoek. 2015. Vol. 108. P. 59-73.
- Okamoto T. et al. Comparative phylogenetic analyses of Halomonas variabilis and related organisms based on 16S rRNA, gyrB and ectBC gene sequences//Syst. Appl. Microbiol. 2004. Vol. 27, № 3. P. 323-333.
- Olsson B.E. et al. Draft genome sequences of strains Salinicola socius SMB35T, Salinicola sp. MH3R3-1 and Chromohalobacter sp. SMB17 from the Verkhnekamsk potash mining region of Russia//Stand. Genomic. Sci. 2017. Vol. 19. P. 1-13. DOI 10.1186/s40793-017-0251-5.
- Ono H. et al. Characterization of biosynfhetic enzymes for ectoine as a compatible solute in a moderately halophilic eubacterium, Halomonas elongate II J. Bacteriol. 1999. Vol. 181. P. 91-99.
- Pastor J.M. et al. Role of central metabolism in the osmoadaptation of the halophilic bacterium Chromohalobacter salexigens II J. Biol. Chem. 2013. Vol. 288, № 24. P. 17769-17781.
- Raj и К. et al. Salinicola rhizosphaerae sp. nov., isolated from the rhizosphere of the mangrove Avicennia marina II Int. J. Syst. Evol. Microbiol. 2016. Vol. 66. P. 1074-1079.
- Raymond R.L. Microbial oxidation of n-paraffinic hydrocarbons//Develop. Ind. Microbiol. 1961. Vol. 2. P. 23-32.
- Schwibbert K. et al. A blueprint of ectoine metabolism from the genome of the industrial producer Halomonas elongata DSM 2581T//Environ. Microbiol. 2011. Vol. 13. P. 1973-1994.
- Versalovic J. et al. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction//Meth. Cell. Mol. Biol. 1994. Vol. 5. P. 25-40.


