Генетическая дифференциация на основании полиморфизма микросателлитных маркеров популяций тополя черного на Среднем и Южном Урале
Автор: Мартыненко Никита Александрович, Ахметов Артур Рамирович, Боронникова Светлана Витальевна, Нечаева Юлия Сергеевна, Пришнивская Яна Викторовна, Никоношина Наталья Алексеевна
Журнал: Известия Самарского научного центра Российской академии наук @izvestiya-ssc
Рубрика: Биологические ресурсы: флора
Статья в выпуске: 5-1 т.17, 2015 года.
Бесплатный доступ
Проанализированы основные методы определения генетической дифференциации популяций древесных видов растений на основании полиморфизма молекулярных маркеров, таких как SSR (Simple Sequence Repeat)-PCR маркеры. С использованием четырех методов установлено генетическое расстояние между четырьмя изученными популяциями Populus nigra L. на Урале. Наибольшее генетическое расстояние ( D =1,086), определенное по методам Дж. Рейнольдса, Б. Вейра и С. Кокерхэма, отмечено между популяцией P.nigra около г. Стерлитамак Республики Башкортостан и популяцией ООПТ «Спасская гора» Пермского края. Наименьшее генетическое расстояние ( D =0,036), определенное по методу Л. Кавалли-Сфорца и А. Эдвардса, установлено между популяциями около г. Стерлитамак и около г. Инзер Республики Башкортостан. При сравнении методов кластеризации, таких как метод невзвешенных парногрупповых средних (UPGMA), метод ближайшего соседа (Neighbor Joining Method)» и метод взвешенных наименьших квадратов по критерию Фитч-Марголиаша, выявлено, что для определения генетической дифференциации изученных популяций P. nigra с использованием полиморфизма кодоминантных маркеров, наиболее оптимален метод Л. Кавалли-Сфорца и А. Эдвардса. При кластеризация в программе STRUCTURE 2.3.4 изученные популяции P. nigra разделились на 2 кластера: популяции из Республики Башкортостан и популяция из Пермского края. Установлена высокая корреляция между генетической дифференциацией и географическим расстоянием изученных четырех популяций P. nigra на основании теста Мантела. Показана высокая корреляция между генетическим и географическим расстоянием у изученных популяций P. nigra, так как высок коэффициент детерминации (R 2) показателя генетической дифференциации (0,694 по методу С. Райта, 0,787 по методу М. Нея).
Популяция, микросателлит, кластеризация, генетическое расстояние, генетическое разнообразие
Короткий адрес: https://sciup.org/148204003
IDR: 148204003 | УДК: 575.174.015.3:
Текст научной статьи Генетическая дифференциация на основании полиморфизма микросателлитных маркеров популяций тополя черного на Среднем и Южном Урале
Никоношина Наталья Алексеевна, студентка разнообразия популяций древесных видов растений, к которым относятся тополь чёрный (Populus nigra L.), играющий важнейшую роль при сохранении пойменных экосистем и используемый как источник древесины в промышленности и строительстве. Кроме этого, это вид относится в роду Populus L., который является модельным родом для изучения геномов древесных видов растений [2].
Тополь чёрный (Populus nigra L., Salicaceae) – анемофилльное, двудомное растение. Его пыльца и семена разносятся ветром на большие расстояния. Этим определяется передача генов и значительное генетическое разнообразие этого вида. Следовательно, выявление взаимосвязи между генетической дистанцией и географическим расстояния между популяциями на примере P. nigra является одним из важнейших аспектов в молекулярно-генетическом анализе дифференциации популяций древесных растений. Эта взаимосвязь является предметом пристального внимания и обсуждения, тем более, что такая корреляция установлена в Пермском крае у природных популяций тополя дрожащего [3], но является спорной при обсуждении результатов молекулярно-генетического анализа популяций P. nigra [4], так и других видов рода Populus [5-7]. Возможно, самым универсальным показателем генетической дистанции является генетическое расстояние, определенное по методу М. Нея [8] и показатель генетической дифференциации популяций С. Райта [9], так как именно они чаще всего используются в программах для компьютерного анализа генетических данных. Но рядом авторов для микросателлитых маркеров [10, 11], рекомендуется использовать и другие методы, а именно показатели Дж. Рейнольдса, Б. Вейра, С. Кокерхама [12], Л. Кавалли-Сфорца и А. Эдвардса [13]. Показатели генетической дифференциации популяций могут варьировать в зависимости от подхода их определения. Важным моментов является перечень условий, которые учитываются при кластеризации популяций, например, какая именно модель избрана для построения филогенетического дерева.
Цель работы: выявление генетической дифференциации на основании полиморфизма микросателлитных маркеров между четырьмя популяциями P. nigra Среднего и Южного Урала, анализ четырех методов определения генетической дифференциации и взаимосвязи генетического и географического расстояния между изученными популяциями P. nigra .
Материал и методика исследования. Объектами исследований являлись четыре популяции P. nigra из разных ботанико-географи-ческих районов Среднего и Южного Урала: первая популяция (Sp_1 или Спасская гора), расположенная в историко-природном комплексе «Спасская гора» в зоне островной Кунгурской лесостепи южной тайги Пермского края (Средний Урал); вторая популяция (B_2 или Бирск,) находится около г. Бирск Республики Башкортостан в лесостепном районе Южного Урала; третья популяция (St_3 или Стерлитамак), расположенная около г. Стерлитамак Республики Башкортостан в степном районе Южного Урала; четвертая популяция (In_4 или Инзер,) находится на территории Южно-Уральского заповедника в горном районе Южного Урала. Минимальное географическое расстояние между популяциями составило около 137 км (между St_3 и In_4), а максимальное – около 380 км (между Sp_1 и St_3).
Для молекулярно-генетического анализа P. nigra были собраны листья с 30-32 случайно избранных деревьев на расстоянии не менее 50 м друг от друга . Для выделения ДНК использовали модифицированную методику S.O. Rogers [14], в которой использовали в качестве детергента СТАВ+PVPP (цетилтриметиламониум бромид + поливинилполипирролидон). При выделении ДНК из листьев брали навеску по 100 мг. Концентрацию ДНК определяли с помощью спектрофотометра NanoDrop 2000 («Thermo Scientific», USA) и для ПЦР выравнивали до 10 нг/мкл.
Наиболее соответствующими поставленной нами задаче генетическими маркерами являются высокополиморфные микросателлитные локусы. Кроме возможности оценки популяционногенетических параметров, анализ нескольких по-лиаллельных микросателлитных локусов даёт возможность составить уникальный генетический портрет каждого дерева. Нами было избрано семь микросателлитных (SSR) локусов (ORNL214, PMGC14, wpms09, wpms20, wpms17, wpms14, wpms15) из базы данных микросателлитов Международного Консорциума по геномам тополей IPGC (International Populus Genome Consortium) SSR Resource и из двух статей [15, 16]. Все праймеры были уже проверенны как на других популяциях P . nigra (в Европе), так и на других видах тополя – P . trichocarpa , P . tremuloides .
ПЦР, фрагментный анализ и компьютерный анализ данных проведен в Федеральном научноисследовательском центре лесов, опасных природных явлений и ландшафта (Federal Research and Training Centre for Forests, Natural Hazards and Landscape, Department of Genetics, Austria), в отделе исследований генома под руководством доктора Б. Хейнце (Вена, Австрия). Для амплификации SSR-методом мы использовали следующую программу: предварительная денатурация 98°C, 3 мин.; первые десять циклов 95°С, 30 сек.; t отж. 58°С, 30 сек., каждый цикл уменьшая температуру на 1°С; 72°С, 30 сек.; в последующих двадцати девяти циклах 95°С, 30 сек.; t отж. 53°С, 30 сек.; 72°С, 30 сек. Последний цикл элонгации длился 4 мин при 72ºС. Фрагментный анализ произведен на секвенаторе GenomeLab GeXP производства Beckman Coulter (USA). Компьютерный анализ полученных данных произведен с помощью программного обеспечения GenomeLab GeXP [17]. Тест Мантела, предназначенный для выявления связей между генетической дифференциацией популяций и географическим расстоянием [18], а также расчёт показателя генетической дифференциации популяций С. Райта (Fst) (первый метод) проводился с помощью программы GenAlЕx 6.5 [19]. На базе программы GENDIST пакета программ PHYLIP v 3.695 [20], были рассчитаны генетические дистанции М. Нея (второй метод), Дж. Рейнольдса (третий метод) и Л. Кавалли-Сфорца (четвёртый метод). Кластеризация по методу невзвешенных парногрупповых средних (UPGMA) и методу ближайшего соседа (Neighbor Joining Method)» проведена в модуле «Neighbor» того же пакета программ. Также в модулях «Kitsch» и «Fitch» пакета программ PHYLIP v. 3.695 проведена кластеризация по методу взвешенных наименьших квадратов по критерию В. Фитч-Марголиаша [21].
Визуализацию полученных дендрограмм проводили с помощью программы TREEVIEW [22]. Кластерный анализ исследуемых популяций P. nigra был проведен и с помощью программы STRUCTURE 2.3.4 [23]. Эта программа использует методы Монте Карло по схеме марковской цепи (Markov Chain Monte Carlo, MCMC), что позволяет минимизировать неравновесие Харди-Вайнберга и неравенство по сцеплению локусов в кластерном анализе индивидов [24]. Количество кластеров (К) находилось в диапазоне от 1 до 12. Для визуализации результатов, их математического подтверждения методами Evanno [25] была использована веб-программа STRUCTURE Harvester [26]. Данная web-программа была использована для детекции подходящего количества кластеров путём их индивидуального перебора.
Таблица 1. Матрицы генетических расстояний между четырьмя популяциями P. nigra
|
1 |
||||
|
B_2 |
0,000 |
|||
|
In_4 |
0,288 |
0,000 |
||
|
Sp_1 |
0,703 |
1,081 |
0,000 |
|
|
St_3 |
0,101 |
0,167 |
0,786 |
0,000 |
|
2 |
||||
|
B_2 |
0,000 |
|||
|
In_4 |
0,288 |
0,000 |
||
|
Sp_1 |
0,707 |
1,086 |
0,000 |
|
|
St_3 |
0,101 |
0,168 |
0,790 |
0,000 |
|
3 |
||||
|
B_2 |
0,000 |
|||
|
In_4 |
0,094 |
0,000 |
||
|
Sp_1 |
0,266 |
0,339 |
0,000 |
|
|
St_3 |
0,036 |
0,084 |
0,288 |
0,000 |
|
4 |
||||
|
B_2 |
0,000 |
|||
|
In_4 |
0,142 |
0,000 |
||
|
Sp_1 |
0,165 |
0,247 |
0,000 |
|
|
St_3 |
0,047 |
0,064 |
0,160 |
0,000 |
Примечание: обозначения: 1. – генетическое расстояние по М. Нею, 2 – генетическое расстояние по Дж. Рейнольдсу, Б. Вейру и С. Кокерхаму, 3 – генетическое расстояние по Л. Кавалли-Сфорцу и А. Эдвардсу, 4 – по показателю генетической дифференциацию С. Райту; обозначения популяций: B_2 – «Бирск», In_4 – «Инзер», Sp_1 – «Спасская гора», St_3 – «Стерлитамак»
Экспериментальная часть и обсуждение результатов. При использовании каждого из четырех анализируемых подходов наибольшее генетическое расстояние установлено между популяциями P. nigra In_4 и Sp_1. Значение этого показателя при определении методом М. Нея составило 1,081, Дж. Рейнольдса – 1,086, Л. Кавалли-Сфорца – 0,339, С. Райта – 0,247 (табл. 1). Следует отметить, географическое расстояние между популяциями In_4 и Sp_1 составляет 369 км, что меньше, чем между самыми отдаленными популяциями St_3 и Sp_1 (380 км). Самое низкое генетическое расстояние при каждом из четырех анализируемых методов (табл. 1) установлено между популяциями B_2 и St_3 – 0,047, 0,101, 0,101 и 0,036 и определено соответственно первым, вторым, третьим и четвёртым методом. Эти популяции менее дифференцированы, потому что они произрастают в пойме одной реки – Белой. Между ними происходит обмен генами – распространение семян и вегетативных частей вниз по течению реки. Такую вариабельность значений генетического расстояния можно объяснить применением различных формул.
При построении филогенетических деревьев с использованием четырех анализируемых методов у дендрограмм различалась топология получившихся ветвей. Она была одинаковой при построении дендрограмм методом ближайшего соседа модуля «Neighbor» и методом наименьших квадратов по критерию Фитч-Марголиаша модуля «Fitch», но она отличалась от двух других при построении дендрограмм методом невзвешенных парногрупповых средних (UPGMA) модуля «Neigh-bor» и методом наименьших квадратов по критерию Фитч-Марголиаша модуля «Kitsch» (рис. 1).
Полученные результаты можно объяснить различием в методах кластеризации. Метод ближайшего соседа модуля «Neighbor» и метод наименьших квадратов по критерию В. Фитч-Марголиаша модуля «Fitch» не используют так называемые «эволюционные часы» [27], то есть не учитывают скорость преобразования генотипа в эволюции вида, строят неукоренившееся филогенетическое дерево и используют аддитивную модель построения. Метод невзвешенных парногрупповых средних (UPGMA) модуля «Neighbor» и метод наименьших квадратов по критерию В. Фитч-Марголиаша модуля «Kitsch» наоборот используют «эволюционные часы», строят укоренившееся дерево и используют ультраметриче-скую модель построения. Интересен тот факт, что ближе всех друг к другу в филогенетическом дереве находились популяции St_3 и In_4. Их близость, возможно, обуславливает преобладающий на территории республики Башкортостан юго-западный ветер [28], способствующий переносу пыльцы.
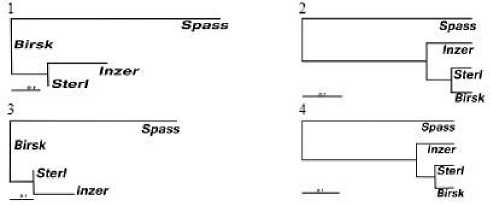
Рис. 1. Дендрограмма, построенная на матрице значений генетического расстояния:
по М. Нею с помощью четырёх методов: 1 – метод ближайшего соседа (Neighbor Joining Method)», 2 – метод невзвешенных парногрупповых средних (UPGMA), 3 – метод взвешенных наименьших квадратов по критерию В. Фитч-Марголиаша в модуле «Fitch», 4 – метод взвешенных наименьших квадратов по критерию В. Фитч-Марголиаша в модуле «Kitsch; обозначения популяций: «Spass» – «Спасская гора», «Inzer» – «Инзер», «Sterl» -Стерлитамак, «Birsk» – «Бирск»
Дендрограммы с одинаковой топологией имеют различный масштаб длин ветвей. В качестве примера можно привести дендро-граммы (рис. 2), полученные на основании генетического расстояния по второму и четвёртому методам с помощью метода невзвешенных парногрупповых средних (UPGMA). Метод наименьших квадратов по критерию В. Фитч-Марголиаша обоих модулей позволяет нам сравнить полученные дендрограммы по показателям суммы квадратов и среднего процента стандартного отклонения (табл. 2).
Наименьшие значения обоих показателей в обоих модулях, а, следовательно, и наименьший разброс полученных значений у параметра генетического расстояния отмечен в четвёртом методе, а наибольшие – в первом. Это свидетельствует о том, что топология и длина ветвей оптимальнее в случае с использованием параметра генетического расстояния, определенным четвёртым методом Л. Кавалли-Сфорца и А. Эдвардса. Во всех полученных дендрограммах наблюдалось обособление популяции Sp_1, иногда даже с очень длинной ветвью. Это также подтверждается результатом кластерного анализа, проведенного c помощью STRUCTURE 2.3.4 (рис. 3).

Рис. 2. Дендрограммы, полученные на основании генетического расстояния, определенного по методу М. Нея (1) и Л. Кавалли-Сфорца и А.Эдвардса (2) с помощью метода невзвешенных парногрупповых средних (UPGMA); обозначения популяций: «Spass» – «Спасская гора», «Inzer» – «Инзер», «Sterl» – «Стерлитамак», «Birsk» – «Бирск»
Таблица 2. Сумма квадратов и средний процент стандартного отклонения по изученным показателям генетической дифференциации в модулях «Fitch» и «Kitsch»
|
Модуль/ показатель |
Первый метод (по Райту) |
Второй метод (по М. Нею) |
Третий метод (по Дж. Рейнольдсу) |
Четвертый метод (по Л. Кавалли-Сфорцу) |
|
|
«Fitch» |
сумма квадратов |
0,07703 |
0,02386 |
0,02386 |
0,00045 |
|
средний процент стандартного отклонения |
8,77661 |
4,88504 |
4,88504 |
0,66916 |
|
|
«Kitsch» |
сумма квадратов |
0,68400 |
0,43100 |
0,43100 |
0,07200 |
|
средний процент стандартного отклонения |
26,15798 |
20,75619 |
20,75619 |
8,45951 |
|
При выявлении корреляции между генетическим и географическим расстоянием с помощью теста Мантела получены высокие коэффициенты детерминации (R2), но они различны при анализируемых четырех методах. Тест Мантела, проведенный для каждого параметра генетической дифференциации, показал высокую корреляцию между значениями географического и генетического расстояния. Очень тесная корреляция наблю- далась также и у изученных ранее уральских популяций P.tremula ( r =0,9 [3]). Так, при использовании первого, второго, третьего и четвёртого методов значения коэффициента детерминации составляли 0,694, 0,787, 0,703 и 0,780 соответственно (рис. 4). Исследования тополей в Европе показали низкие показатели R2 у таких видов как P. tomentosa (R2 =0,007 [6]), P.tremuloides (R2=0,202 [7]), P.euphratica (R2=0,10 [5]), так и P.nigra (R2=0,07 [4]).
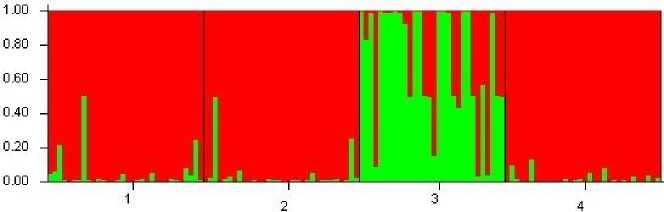
Примечание: по оси ординат – доля частот аллелей соответствующего кластера, по оси абсцисс – порядковый номер популяций, где 1– «Бирск», 2 – «Инзер», 3 – «Спасская гора», 4 – «Инзер»; тёмным (красным) цветом обозначен первый кластер (генотипы популяций из Республики Башкортостан), а светлым )зеле-ным) – второй (генотипы популяции Спасская гора из Пермского края)
Рис. 3. Кластерный анализ генотипов изученных популяций P. nigra Урала с помощью программы STRUCTURE
Выводы: установлено, что для определения генетической дифференциации изученных популяций P. nigra с использованием полиморфизма кодоминантных маркеров, таких как SSR-PCR маркеры, наиболее оптимальным является метод Л. Кавалли-Сфорца и А.Эдвардса . Тем не менее, общие закономерности генетической дифференциации сохраняются при каждом из четырех проанализированных методов. Доказана правильность кластеризации изученных популя-ций и высокая корреляция между генетическим и географическим расстоянием между популяциями, так как высок коэффициент детерминации (R2) показателя генетической дифференциации (0,694 по методу С.Райта, 0,787 по методу М.Нея). Эти данные совпадает с результатами, полученными при изучении семи популяций P. tremula Пермского края [3] на основании полиморфизма межмикросателлит-ных маркеров. При анализе в программе STRUCTURE 2.3.4 четыре популяции P.nigra разделились на 2 кластера: популяции из Республики Башкортостан и из Пермского края. Данные о генетической диф-ференциации популяций необходимы для оценки и сопоставления состояния генофондов древесных видов растений и создания программ сохранения генетических ресурсов.
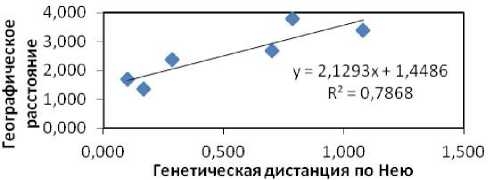
Рис. 4. Зависимость между генетическим расстоянием по методу М.Нея и географическим расстоянием изученных четырех популяций P.nigra
Выражаем искреннюю благодарность Федеральному научно-исследовательскому центру лесов, опасных природных явлений и ландшафта, отделу исследований генома, Вена, Австрия (Federal Research and Training Centre for Forests, Natural Hazards and Landscape, Department of Genetics, Austria) и руководителю отдела геномики – доктору Бертольду Хайнце и сотруднице отдела Ренате Слански.
Работа выполнена при финансовой поддержке задания 2014/153 государственных работ в сфере научной деятельности в рамках базовой части государственного задания Минобрнауки России (проект 144, № гос. рег.
Список литературы Генетическая дифференциация на основании полиморфизма микросателлитных маркеров популяций тополя черного на Среднем и Южном Урале
- Букварева, Е.Н. Принцип оптимального разнообразия биосистем и стратегия управления биоресурсами/Е.Н. Букварева, Г.М. Алещенко//Государственное управление в XXI веке: традиции и инновации. -М., 2006. С. 204-210.
- Brunner, A.M. Poplar genome sequence: functional genomics in an ecologically dominant plant species/A.M. Brunner, V.B. Busov, S.H. Strauss//Trends in plant science. 2004. V. 9, №. 1. P. 49-56.
- Svetlakova, T.N. Genetic diversity and differentiation in Ural populations of the aspen, Populus tremula L., as revealed by inter-simply sequence repeat (ISSR) markers/T.N. Svetlakova, S.V. Boronnikova, Y.A. Yanbaev//Silvae Genetica. 2014. V. 63, №. 1-2. P. 39-41.
- Imbert, E. Dispersal and gene flow of Populus nigra (Salicaceae) along a dynamic river system/E. Imbert, F. Lefèvre//Journal of Ecology. 2003. V. 91, №. 3. P. 447-456.
- Wang, J. Genetic variation within and between populations of a desert poplar (Populus euphratica) revealed by SSR markers/J. Wang et al.//Annals of forest science. 2011. V. 68, №. 6. P. 1143-1149.
- Du, Q. Genetic diversity and population structure of Chinese white poplar (Populus tomentosa) revealed by SSR markers/Q. Du et al.//Journal of Heredity. 2012. V. 103, №. 6. -С. 853-862.
- Callahan, C.M. Continental-scale assessment of genetic diversity and population structure in quaking aspen (Populus tremuloides)//Journal of biogeography. 2013. V. 40, №. 9. P. 1780-1791.
- Nei, M. Estimation of average heterozygosity and genetic distance from a small number of individuals//Genetics. 1978. V. 89, №. 3. P. 583-590.
- Wright, S. Isolation by distance//Genetics. 1943. V. 28, №. 2. P. 114.
- Takezaki, N. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA/N. Takezaki, M. Nei//Genetics. 1996. V. 144, №. 1. P. 389-399.
- Chapuis, M.P. Microsatellite null alleles and estimation of population differentiation/M.P. Chapuis, A. Estoup//Molecular biology and evolution. 2007. V. 24, №. 3. P. 621-631.
- Reynolds, J. Estimation of the coancestry coefficient: basis for a short-term genetic distance/J. Reynolds, B.S. Weir, C.C. Cockerham//Genetics. 1983. V. 105, №. 3. P. 767-779.
- Cavalli-Sforza, L.L. Phylogenetic analysis. Models and estimation procedures/L.L. Cavalli-Sforza, A.W.F. Edwards//American journal of human genetics. 1967. V. 19, №. 3 Pt. 1. P. 233.
- Rogers S.O. Phylogenetic and taxonomic information from herbarium and mummified DNA//Conservation of Plant Genes II: Utilization of Ancient and Modern DNA. 1994. P. 47-67.
- Van der Schoot, J. Development and characterization of microsatellite markers in black poplar (Populus nigra L.)/J. van der Schoot et al.//Theoretical and Applied Genetics. 2000. V. 101, №. 1-2. P. 317-322.
- Smulders, M.J.M. Trinucleotide repeat microsatellite markers for black poplar (Populus nigra L.)/M.J.M. Smulders et al.//Molecular Ecology Notes. 2001. V. 1, №. 3. P. 188-190.
- David, V.A. Automated DNA detection with fluorescent-based technologies/V.A. David, M. Menotti-Raymond//Molecular Genetic Analyses of Populations. 1998. P. 337-370.
- Mantel, N. The detection of disease clustering and a generalized regression approach//Cancer research. 1967. V. 27, №. 2, Part 1. P. 209-220.
- Peakall, R.O.D. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research/R.O.D. Peakall, P.E. Smouse//Molecular ecology notes. 2006. V. 6, №. 1. P. 288-295.
- Felsenstein, J. PHYLIP -Phylogeny Inference Package//Cladistics. 1989. № 5. P. 164-166.
- Fitch W.M. Evidence suggesting a non-random character to nucleotide replacements in naturally occurring mutations//Journal of molecular biology. 1967. V. 26, №. 3. P. 499-507.
- Page, R.D.M. TreeView -An application to display phylogenetic trees on personal computer//Comp. Appl. Biol. Sci. 1996. V. 12. P. 357-358.
- Falush, D. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies/D. Falush, M. Stephens, J.K. Pritchard//Genetics. 2003. V. 164, №. 4. P. 1567-1587.
- Smulders M.J.M. Structure of the genetic diversity in black poplar (Populus nigra L.) populations across European river systems: Consequences for conservation and restoration/M.J.M. Smulders et al.//Forest Ecology and Management. 2008. V. 255, №. 5. P. 1388-1399.
- Evanno, G. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study/G. Evanno, S. Regnaut, J. Goudet//Molecular ecology. 2005. V. 14. №. 8. P. 2611-2620.
- Earl, D.A. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method/D.A. Earl et al.//Conservation genetics resources. 2012. V. 4, №. 2. P. 359-361.
- Mangelsdorf, P.C. Evolution under domestication//American Naturalist. 1952. P. 65-77.
- Научно-прикладной справочник по климату СССР. -Л.: Гидрометеоиздат. Серия 3. Части 1-6, вып. 9. 1990. 557 с.


