Генетическая паспортизация российских сортов клевера лугового (Тrifolium pratense L.) на основе SSR- и SRAP-маркеров
Автор: Клименко И.А., Шамустакимова А.О., Душкин В.А., Мавлютов Ю.М., Антонов А.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Молекулярные маркеры
Статья в выпуске: 3 т.58, 2023 года.
Бесплатный доступ
Генетическая паспортизация - мощное дополнение к традиционным методам сортоиспытания и эффективный способ идентификации сортов сельскохозяйственных растений. В Российской Федерации, как и во всем мире, переходят к внедрению современных ДНК-технологий в селекционные программы, в систему регистрации сортов и коммерческого распространения семян, однако в работе с кормовыми травами преобладают подходы, основанные на фенотипической оценке. Это снижает эффективность отбора, увеличивает сроки и затраты на выведение новых сортов, их регистрацию и правовую защиту. В представленной работе впервые проведена оценка генетического полиморфизма коллекции российских и зарубежных сортов клевера лугового с помощью SSR- и SRAP-маркеров, выявлены сортоспецифичные ДНК-фрагменты для дифференциации изучаемого материала, уникальность которых подтверждена секвенированием. На основе полученных результатов для ряда российских сортов составлены эталонные генетические паспорта. Наша цель заключалась в разработке системы определения сортовой принадлежности и эталонных генетических паспортов на основе SSR- и SRAP-маркеров для российских сортов клевера лугового. Материалом служили семена 15 российских сортов клевера лугового ( Trifolium pratense L.), полученные в ЦКП Биологические коллекции кормовых растений (ФНЦ ВИК им. В.Р. Вильямса) и 6 образцов зарубежной селекции из Коллекции генетических ресурсов Всероссийского института генетических ресурсов растений им. Н.И. Вавилова. Геномную ДНК выделяли из суммарной навески части растительной ткани 30 7-суточных проростков от каждого сорта. Использовали базовый SDS-метод с модификациями. Количественные и качественные показатели полученных ДНК-проб определяли посредством электрофореза в агарозном геле, измерения концентрации и оценки чистоты. Финальную концентрацию образов перед использованием в ПЦР доводили до 30 нг/мкл. Генотипирование выполняли с помощью 35 микросателлитных маркеров (база данных Red Clover Marker Database, http://marker.kazusa.or.jp/Red_clover) и 40 комбинаций праймеров к SRAP-мар-керам. При анализе 12 сортов клевера лугового с использованием 35 SSR-маркеров было получено 476 продуктов амплификации. Определены 8 пар микросателлитных (SSR) локуcов, идентифицирующих сорта в изучаемой выборке. Из 40 испытанных комбинаций праймеров, разработанных к SRAP-маркерам, выбрали 18 для анализа на расширенной коллекции из 16 российских и зарубежных сортов. С их использованием было выявлено 812 продуктов ПЦР, включая 85 полиморфных, что составило 10,5 %. Набор из 7 SRAP-маркеров можно использовать для определения сортовой принадлежности. Для верификации результатов уникальные для соответствующих сортов фрагменты ДНК были секвенированы. Аннотированные нуклеотидные последовательности, идентифицирующие сорта Трифон, Марс, Топаз, Атлант, Тетраплоидный ВИК, Метеор, ВИК 77, депонированы в международной базе генетических ресурсов GenBank NCBI (https://www.ncbi.nlm.nih.gov/). На основе данных ДНК-фингерпринтинга составлены молекулярно-генетические формулы, отображающие аллельный состав микросателлитных локусов и полиморфизм интрон-экзонных участков генома, и разработаны 10 эталонных генетических паспортов для сортов клевера лугового российской селекции.
Кормовые культуры, генетическое разнообразие, ssr-маркеры, srap-маркеры, днк-полиморфизм, генетическая паспортизация
Короткий адрес: https://sciup.org/142238892
IDR: 142238892 | УДК: 633.32:577.21 | DOI: 10.15389/agrobiology.2023.3.494rus
Текст научной статьи Генетическая паспортизация российских сортов клевера лугового (Тrifolium pratense L.) на основе SSR- и SRAP-маркеров
Одно из важнейших направлений развития геномных биотехнологий в сельском хозяйстве Российской Федерации — разработка и применение методов ДНК-идентификации и генетической паспортизации селекционных достижений. Широкое использование этих подходов обеспечит повышение эффективности регистрации новых сортов и защиты авторских прав селекционеров, поможет в борьбе с фальсификатом на рынке семян. На текущий момент оценка сортов на соответствие критериям ООС-теста (от- личимость, однородность и стабильность) основана на описании морфологических признаков. Однако это трудоемкий и затратный по времени процесс, который требует соответствующего опыта, а в некоторых случаях — специальных условий для проведения испытаний (например, по яровизации ячменя и пшеницы) (1). Усложняет задачу и ограниченное число оценочных дескрипторов, которые к тому же подвержены влиянию условий окружающей среды. Это, в частности, признаки устойчивости к болезням, обязательные для оценки при сертификации некоторых сортов овощей (томаты) или время цветения для многих аутбридинговых видов (райграс) (2-4). Для ряда культур выявление признаков, определяющих оригинальность сорта, осложняется высокой степенью внутрипопуляционных вариаций или, напротив, межвидового морфологического сходства (виды-двойники) (5, 6). По мере роста числа сортов сокращается генетическая база, поскольку селекция, как правило, сосредоточена на некоторых наиболее важных агрономических признаках, что затрудняет выявление различий на морфологической основе (7). При этом у многолетних видов проявление ряда признаков требует длительного времени и соответствующей стадии развития.
Эффективность оценки может быть значительно повышена за счет интеграции в систему сортоиспытания методов, основанных на использовании молекулярных ДНК-маркеров. Практически неограниченное количество таких маркеров и высокая степень выявляемого полиморфизма (вне зависимости от исследуемой части растений и условий окружающей среды) позволяют дифференцировать даже трудноразличимые сорта. С помощью ДНК-маркеров можно выявлять скрытую изменчивость, что повышает точность результатов и разрешающую способность анализа (8-10).
Генетическая идентификация основана на определении комбинации аллелей конкретного гена, характерных для исследуемого организма. При наличии информативных и удобных методик полученные результаты можно легко документировать и разработать молекулярно-генетический паспорт сорта с указанием длины фрагментов ДНК в конкретных участках хромосом. Паспорт позволяет определить уникальность образца и уровень генетической изменчивости вида, оценить степень родства с известными сортами и соответствие стандарту, провести анализ однородности семян.
Методы, лежащие в основе создания генетических паспортов, успешно применяются не только в сортоиспытании, но и в селекции. Возможность выделить ценные генотипы на начальной стадии развития растения сокращает продолжительность испытаний и повышает эффективность селекционного процесса, особенно в случаях, когда отбор по фенотипу оказывается длительным и недостаточно надежным (11). Анализ полиморфизма ДНК обеспечивает селекционеров информацией, которую можно использовать для контроля результатов гибридизации, при подборе родительских форм для скрещиваний, для выявления источников генов, влияющих на хозяйственно ценные признаки.
Молекулярные маркеры, применяемые для оценки генетической изменчивости и идентификации линий, сортов и форм, должны отвечать определенным требованиям: быть высокополиморфными, воспроизводимыми, равномерно распределяться по геному с охватом разных его областей. Наиболее часто с этой целью используют RFLP- (restriction fragment length polymorphism), RAPD- (random amplified polymorphic DNA), AFLP-(amplified fragment length polymorphism), SSR- (simple sequence repeats) маркеры, которые представляют собой анонимные фрагменты ДНК, отражающие полиморфизм в случайно выбранных участках генома (14, 15).
Однако результаты оценки полиморфизма не всегда коррелируют с известными морфологическими признаками. Для целей прикладной генетики и селекции более рациональным считается анализ на основе функциональных молекулярных маркеров, идентифицирующих изменения в транскрибируемых кодирующих последовательностях ДНК — генах (16-18). Основным ресурсом для разработки таких маркеров служат библиотеки EST (Expressed Sequence Tags — экспрессирующиеся целевые сиквенсы), в которых содержатся секвенированные фрагменты ДНК длиной 500-700 п.н. Для многих сельскохозяйственных культур в настоящее время созданы базы данных c информацией по EST-SSR-маркерам, разработанным на основе комплементарной ДНК (кДНК) (19, 20). SSR-маркеры позволяют выявлять полиморфизм простых повторяющихся последовательностей ДНК (110 п.н.), которые различаются по длине у некоторых образцов растений (21). На вариабельности этих областей генома основан и метод идентификации, предполагающий, что генотипы одного и того же сорта (если он достаточно выравненный) должны содержать аллели одинаковой длины.
В последние годы для оценки генетического полиморфизма сортов и видов успешно применяется система SRAP-маркеров (sequence related amplified polymorphism), которые разработаны для амплификации фрагментов ДНК в интрон-экзонных участках гена (22). Преимущества маркеров этого типа — простота использования и статистической обработки, высокая информативность и стабильность результатов (значительно выше, чем при RAPD-маркировании). С помощью SRAP-метода легко обнаружить полиморфизм даже в близкородственном селекционном материале, поэтому он успешно используется для различения сортов разных культур (23-25).
В России, как и во всем мире, переходят к использованию современных методов ДНК-анализа в селекционных программах, в системе регистрации новых сортов и коммерческого распространения семян. Разработаны генетические паспорта для ряда важных сельскохозяйственных культур, в основном продовольственного назначения — риса, пшеницы, свеклы, картофеля и др. (26-29). Менее изученными остаются кормовые травы, в работе с которыми преобладают подходы, основанные на фенотипической оценке. Это существенно снижает эффективность отбора исходного и селекционного материала, увеличивает сроки создания новых сортов и затраты на их испытание и регистрацию. Решением проблемы станет разработка высокопроизводительной, относительно недорогой и не привязанной к морфологическим параметрам растения методики для анализа генетической изменчивости сортов и форм на уровне ДНК.
Из кормовых многолетних трав в регионах Нечерноземной зоны России наиболее распространен клевер луговой. Он используется не только в качестве ценного высокобелкового корма для животных, но и как отличный предшественник для других сельскохозяйственных культур, играет важную роль в биологизации земледелия (30). К настоящему времени усилиями российских селекционеров создано более 100 сортов этого вида. Применение современных методов молекулярного анализа расширяет возможности их надежной идентификации и правовой защиты, а также способствует ускорению процесса создания новых форм, высокоурожайных и устойчивых к болезням.
В представленной работе нами впервые проведена оценка генетического полиморфизма коллекции российских и зарубежных сортов клевера лугового с помощью SSR- и SRAP-маркеров, выявлены сортоспецифичные ДНК-фрагменты для дифференциации изучаемого материала, уникальность которых подтверждена секвенированием. На основе полученных результатов для ряда российских сортов составлены эталонные генетические паспорта.
Наша цель заключалась в разработке системы определения сортовой принадлежности и эталонных генетических паспортов на основе SSR- и SRAP-маркеров для российских сортов клевера лугового.
Методика. Исследования выполнены в 2019-2021 годах в ФГБНУ Федеральный научный центр кормопроизводства и агроэкологии им. В.Р. Вильямса. Материалом служили семена 15 российских сортов клевера лугового ( Trifolium pratense L.), полученные в ЦКП Биологические коллекции кормовых растений (ФНЦ ВИК им. В.Р. Вильямса) и 6 образцов зарубежной селекции из Коллекции генетических ресурсов ВИР (ФИЦ Всероссийский институт генетических ресурсов растений им. Н.И. Вавилова, г. Санкт-Петербург).
Геномную ДНК выделяли из суммарной навески части растительной ткани 7-суточных проростков, по 30 генотипов от каждого сорта (балк-образец). Использовали базовый SDS-метод с модификациями (31). Количественные и качественные показатели полученных ДНК-проб определяли посредством электрофореза в агарозном геле и измерения концентрации и чистоты на спектрофотометре UV-vis Nabi («MicroDigital Co., Ltd.», Корея). Финальную концентрацию образов перед использованием в ПЦР доводили до 30 нг/мкл. Генотипирование выполняли с помощью 35 микросателлит-ных маркеров, разработанных S. Sato с соавт. (32) при анализе структуры генома клевера лугового и размещенных в базе данных Red Clover Marker Database , и 40 комбинаций праймеров к SRAP-маркерам (22, 33).
В реакционной смеси для SSR-анализа объемом 15 мкл на 1 образец содержалось: 3 мкл 10½ ПЦР-буфера (Taq Turbo Buffer); 0,2 мкл полимеразы (Tag DNA Polymerase — 5 ед/мкл); 0,1 мкл каждого праймера (100 мкМ); 1 мкл 50½ дНТФ (dNTP mix 10 mM each) и 1 мкл образца ДНК (30 нг/мкл). Все реагенты производства компании ООО «Евроген Лаб» (Россия). Амплификацию проводили в термоциклере T100 («Bio-Rad», США) по модифицированной программе Touchdown PCR, включающей этап первоначальной денатурации 3 мин при 94 ° С и 3 этапа последовательного снижения температуры отжига праймеров на 2 ° С через каждые 3 цикла: 30 с при 94 ° С, 30 с при 68 ° С (3 цикла); 30 с при 94 ° С, 30 с при 66 ° С (3 цикла); 30 с при 94 ° С, 30 с при 64 ° С (3 цикла). В дальнейшем программа амплификации предусматривала этап элонгации цепи: 30 с при 94 ° С, 30 с при 62 ° С, 30 с при 72 ° С (3 цикла); 30 с при 94 ° С, 30 с при 60 ° С, 30 с при 72 ° С (3 цикла); 30 с при 94 ° С, 30 с при 58 ° С, 30 с при 72 ° С (3 цикла). После этого температура гибридизации праймеров снижалась до оптимального значения 55 ° С и сохранялась оставшиеся 30 циклов реакции: 30 с при 94 ° С, 30 с при 55 ° С, 30 с при 72 ° С. Заключительный этап элонгации цепи проходил 10 мин при 72 ° С (32, 34). Воспроизводимость результатов проверяли посредством 3-кратной повторности экспериментов, включая использование в качестве матрицы балк-образцов ДНК, полученных из разных выборок проростков.
Полученные ПЦР-продукты предварительно анализировали после электрофореза в 1,6 % агарозном геле (LE2, «Lonza», США) и детекции c помощью прибора GelDoc™ XR+ («Bio-Rad», США). Размер фрагментов определяли в сравнении с молекулярным маркером 100 bp GeneRuler DNA Ladder («Thermo Fisher Scientific», США) в программе ImageLab («Bio-Rad Lab., Inc.», США). Результаты анализа сводили в общую таблицу и выяв- ляли наличие сортоспецифичных аллелей по каждому маркеру.
Для валидации природы и размеров уникальных для сорта аллелей ПЦР-продукты были клонированы с использованием вектора pAL2-T (ЗАО «Евроген», Россия) и секвенированы на генетическом анализаторе ABI PRISM 3130XL («Applied Biosystems, Inc.», США) с использованием набора Big Dye terminator v.3.1 cycle sequencing kit («Applied Biosystems, Inc.», США). Данные секвенирования анализировали с помощью программы Ugene (35), а затем выравнивали в алгоритме BLAST (Basic Local Alignment Search Tool, . В качестве референса использовали EST/геномные последовательности из базы данных Red Clover Marker Database .
Для повышения точности определения длины всех фрагментов, выявленных в ДНК-профиле сортов-кандидатов на паспортизацию, разрабатывали контрольные маркеры. По результатам электрофореза в 10 % акриламидном геле (камера «BIO-RAD Tetra Cell», США) устанавливали размеры аллелей образцов в сравнении с контрольным маркером.
Состав компонентов реакционной смеси для большей части SRAP-маркеров соответствовал предложенному H.B. Rhouma с соавт. (23), однако для успешной амплификации некоторых комбинаций праймеров пришлось оптимизировать содержание реагентов и программу ПЦР. Модифицированная смесь объемом 20 мкл на 1 образец состояла из 3 мкл 10½ ПЦР-буфера (Taq Turbo Buffer); 0,5 мкл 50½ дНТФ; 0,1 мкл 100 мкМ праймера; 0,4 мкл полимеразы (Tag DNA Polymerase, 5 ед/мкл); 1,0 мкл образца ДНК (30 нг/мкл).
Амплификацию осуществляли на приборе T-100 Thermal Cycler («Bio-Rad», США) при следующих условиях: начальная денатурация 4 мин при 94 ° С; 1 мин при 94 ° С, 1 мин при 35 ° С, 1 мин при 72 ° С (10 циклов); 1 мин при 94 ° С, 1 мин при 50 ° С, 1 мин при 72 ° С (30 циклов); 5 мин при 72 ° С (финальная элонгация). Размер амплифицированных фрагментов ДНК определяли после горизонтального электрофореза в сравнении с молекулярным маркером 1 kb (ООО «Евроген Лаб», Россия). Целевые (сортоспецифичные) продукты ПЦР вырезали из агарозного геля и очищали на колонках, используя набор Сleanup Standart («Евроген», Россия), а затем очищенные ампликоны клонировали с помощью вектора pAL2-T («Евроген», Россия) согласно инструкции производителя и секвенировали для установления первичной последовательности нуклеотидов. Результаты анализа сиквенсов в программах Ugene и BLAST (35) использовали для отбора SRAP-маркеров, идентифицирующих отдельные сорта.
С помощью анализа главных координат (Principal Coordinate Analysis, PCoA) и программного пакета GenAlEx (версия 6.2) (36) визуализировали генетические взаимосвязи между исследуемыми образцами.
Результаты. Сорта клевера лугового, включенные в настоящее исследование, приведены в таблице 1.
1. Перечень сортов клевера лугового ( Trifolium pratense L.), включенных в исследование, и применяемая система маркирования
|
Сорт |
Оригинатор (страна происхождения/ката-ложный номер в коллекции ВИР) |
Год внесения в Госреестр |
Система маркирования |
|
|
Ранний 2 |
ФНЦ ВИК им. В.Р. Вильямса |
1995 |
SSR, |
SRAP |
|
Трифон |
ФАНЦ Северо-Востока им. Н.В. Рудницкого |
2014 |
SSR, |
SRAP |
|
Памяти Лисицына |
ФНЦ Зернобобовых и крупяных культур; |
2005 |
SSR, |
SRAP |
|
ФНЦ ВИК им. В.Р. Вильямса; Сибирский ФНЦ |
||||
|
агробиотехнологий РАН |
||||
|
Пеликан |
ФНЦ Лубяных культур |
1992 |
SSR, |
SRAP |
|
Продолжение таблицы 1 |
|||
|
Трио |
ФАНЦ Северо-Востока им. Н.В. Рудницкого; ФНЦ ВИК им. В.Р. Вильямса |
1995 |
SSR, SRAP |
|
Ветеран |
ФНЦ ВИК им. В.Р. Вильямса |
2011 |
SSR, SRAP |
|
Тетраплоидный ВИК ФНЦ ВИК им. В.Р. Вильямса |
1973 |
SSR, SRAP |
|
|
Марс |
ФНЦ ВИК им. В.Р. Вильямса |
1993 |
SSR |
|
ВИК 77 |
ФНЦ ВИК им. В.Р. Вильямса |
2006 |
SSR |
|
Метеор |
Сибирский ФНЦ Агробиотехнологий РАН; ФНЦ ВИК им. В.Р. Вильямса |
2007 |
SSR |
|
Топаз |
ФНЦ ВИК им. В.Р. Вильямса; ООО Агрокомплекс-Н |
2000 |
SSR |
|
Атлант |
ФИЦ Тюменский научный центр РАН; Сибирский ФНЦ Агробиотехнологий РАН |
2007 |
SSR |
|
Алтын |
ФНЦ ВИК им. В.Р. Вильямса; ОНО Моршанская селекционная станция |
1999 |
SRAP |
|
ВИК 84 |
ФНЦ ВИК им. В.Р. Вильямса |
1991 |
SRAP |
|
Воронежский |
ФНЦ ВИК им. В.Р. Вильямса |
2015 |
SRAP |
|
Marathon |
К-48013 (Франция) |
– |
SRAP |
|
Freedom |
К-51532 (США) |
– |
SRAP |
|
Ganymed |
К-53648 (Чехия) |
– |
SRAP |
|
Metis |
К-53792 (Дания) |
– |
SRAP |
|
Nemaro |
К-50958 (Германия) |
– |
SRAP |
|
Norlac |
К-51526 (Канада) |
– |
SRAP |
|
Примечание. |
Прочерки означают, что сорт не внесен в Государственный реестр РФ. |
||
Для оценки генетического полиморфизма сортов клевера лугового необходима репрезентативная выборка генотипов (не менее 30-50 на образец), чтобы аккумулировать максимальное число маркеров, характерных для перекрестноопыляемых популяций с высоким уровнем гетерогенности. Существенно снизить затраты труда, времени и финансовых средств в таких случаях позволяет использование для анализа суммарного образца ДНК отдельных генотипов (балк-стратегия) (37, 38). Ранее наши модификации SDS-метода выделения ДНК (39) позволили получить препараты хорошего качества, пригодные для использования в ПЦР с разными типами маркеров, из общей навески, сформированной из высокообводненной ткани 30 проростков каждого сорта (31).
Основными критериями отбора микросателлитных локусов для настоящей работы служили число выявляемых аллелей, охват всех групп сцепления по расположению на хромосомах и небольшая длина получаемых ПЦР-фрагментов (100-300 п.н.). Все SSR-маркеры были представлены дии три-нуклеотидными повторами. Для определения степени однородности материала случайные выборки проростков сортов Ранний 2, Марс, Тетрап-лоидный ВИК, Трифон (по 10 генотипов от каждого) были проанализированы с использованием микросателлитных локусов RCS1307, RCS5600 и RCS5208. Выявлена достаточно высокая генетическая выравненность материала, что не характерно для перекрестноопыляемых культур с высоким уровнем гаметофитной самонесовместимости. В других исследованиях также сообщается о значительной степени ДНК-полиморфизма, выявленного внутри популяций клевера лугового с маркерами разных типов (RAPD, AFLP, SSR) — от 67,5 до 83,6 % (17, 38, 40). Видимо, в нашем случае на результаты повлияла ограниченность генетического материала, использованного в селекционных схемах (включенные в анализ образцы имели общего оригинатора — ФНЦ ВИК им. В.Р. Вильямса).
Общая схема исследования межсортового генетического полиморфизма и разработки генетических паспортов клевера лугового представлена на рисунке 1.
Для изучения межсортовой генетической изменчивости и последующей паспортизации с использованием 35 пар SSR-праймеров на 12 сортах клевера лугового получили 476 продуктов амплификации, в среднем по 13,6 аллеля на локус. Эти данные сопоставимы с результатами других исследований. Так, I. Radinovic с соавт. (41) при изучении 46 генотипов клевера лугового различного эколого-географического происхождения выявили с помощью 14 SSR-маркеров 13,36 аллелей на локус в среднем. Исследователи из Бразилии (42) генотипировали 57 образцов клевера лугового из Северной Америки и Европы и определили, что на каждый из 7 испытанных SSR-локусов приходилось в среднем по 9 аллелей.
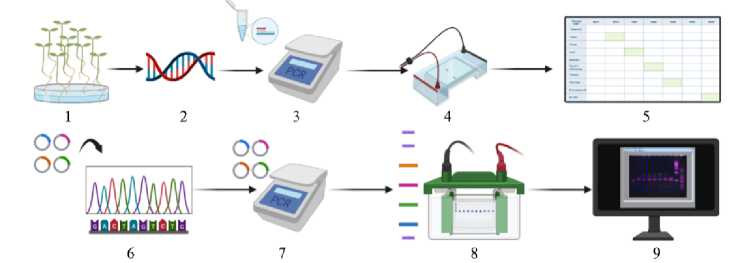
Рис 1. Схема ДНК-фингерпринтинга клевера лугового (Trifolium pratense L.) для генетической паспортизации: 1-9 — процедуры с применением SSR-маркеров, 1-6 — процедуры с использованием SRAP-маркеров; 1 — формирование суммарной навески проростков 30 генотипов каждого сорта, 2 — выделение ДНК, 3 — амплификация в термоциклере («Bio-Rad», США), 4 — предварительный электрофорез в агарозном геле, 5 — отбор сортоспецифичных маркеров, 6 — секвенирование по Сэнгеру и анализ результатов, 7 — ПЦР с плазмидной ДНК, 8 — разработка специфического (контрольного) маркера и вертикальный ПААГ-электрофорез, 9 — определение размеров продуктов в сравнении с контрольным маркером для составления молекулярно-генетической формулы сорта (схема создана в программе Biorender .
В нашей работе размер полученных ампликонов варьировал от 91 п.н. (с праймерной парой RCS5305) до 359 п.н. (RCS1535). Мы определили набор из 8 информативных микросателлитных локусов, которые генерировали воспроизводимые полиморфные продукты и были пригодны для различения сортов в исследуемой выборке. Так, с праймерами к маркеру RCS1307 были обнаружены уникальные аллели, выделявшие сорта Марс, Топаз и Трифон, а по локусу RCS0017 различались Метеор и Топаз.
В целом на основе SSR-анализа был выявлен невысокий уровень как внутрисортового, так и межсортового полиморфизма ДНК, что, вероятно, обусловлено происхождением и селекционной историей материала. Среднее значение генетической изменчивости между сортами равнялось 5,3 %. Это оказалось существенно ниже показателей, известных из работ других исследователей (43, 44).
Существенно повысить разрешающую способность анализа позволяет использование дополнительных маркерных систем, основанных на полиморфизме последовательностей в различных структурных элементах генома. С помощью набора из 40 комбинаций SRAP-праймеров мы геноти-пировали расширенную коллекцию образцов клевера лугового, составленную из сортов российской селекции, которые не удалось паспортизировать по панели SSR-маркеров, и зарубежных (для сравнительной оценки генетического полиморфизма).
Базовая программа ПЦР (23) оказалась неэффективной для комбинаций SRAP-маркеров F10-R9, Me4-R9, ME1-EM1. В результате оптимизации состава компонентов реакционной смеси, а также изменения температурных и временных параметров ПЦР с использованием 18 комбинаций праймеров мы получили отчетливые и воспроизводимые продукты (в общей сложности — 812). Наибольшее число амплифицированных фрагментов было выявлено с F13-R9 (62 ампликона), тогда как с ME1-EM2 этот показатель был минимальным (22 ампликона) при среднем значении 45,1 на каждую комбинацию. Полиморфными оказались 85 ампликонов, что составило 10,5 % от общего количества. Уникальные для отдельных сортов фрагменты ДНК (до 10) обнаружили с использованием F9-R9, F10-R8 и F10-R9 и всего по 1 сортоспецифичному фрагменту с F9-R14 и Me4-EM1.
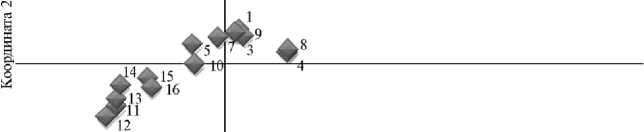
На основании полученной информации мы оценили генетические взаимосвязи между изучаемыми образцами и визуализировали вариации их распределения на координатной плоскости методом PCoA-анализа (рис. 2).
ф 2
Координата 1
Рис. 2. Результаты кластеризации российских и зарубежных сортов клевера лугового ( Trifolium pratense L.) методом главных координат (PCoA-анализ) по данным SRAP-анализа: 1 — Ранний 2, 2 — Трифон, 3 — Памяти Лисицына, 4 — Пеликан, 5 — Трио, 6 — Ветеран, 7 — Алтын, 8 — ВИК 84, 9 — Тетраплоидный ВИК, 10 — Воронежский, 11 — Norlac, 12 — Freedom, 13 — Ganymed, 14 — Metis, 15 — Nemaro, 16 — Marathon.
На диаграмме выделились две относительно компактные группы, одна из которых объединяла сорта зарубежной селекции, а в другой оказались разработки селекционеров ФНЦ ВИК им. В.Р. Вильямса — ультраран-неспелые диплоидные сорта Трио, Алтын, Воронежский, Ранний 2, а также тетраплоиды Памяти Лисицына и Тетраплоидный ВИК. Образцы в первой группе значительно перекрывались, что указывает на большое генетическое сходство между ними, возможно, в результате интенсивного потока генов. Наиболее генетически близкими оказались Norlac (Канада) и Freedom (США) а также европейские сорта Ganymed и Metis. На небольшой генетической дистанции от них расположились сорта из Западной Европы — Nemaro (Германия) и Marathon (Франция).
Выявленное сходство между сортами второго кластера, вероятно, было следствием их генетического родства и общей селекционной истории. В то же время тетраплоидные сорта ВИК 84 и Ветеран селекции ФНЦ ВИК им. В.Р. Вильямса расположились на координатной плоскости обособленно от остальных сортов своей группы. Сорт Ветеран выделялся среди других образцов высокой фотосинтетической и симбиотической активностью, долголетием в агрофитоценозах. Сорту ВИК 84 были свойственны повышенная устойчивость к поражению клеверным раком, антракнозом и фузариозом, высокая зимостойкость и засухоустойчивость. Он кластеризовался совместно с сортом Пеликан (Пензенский НИИСХ), выведенным методом сложногибридной популяции из нескольких сортов раннеспелого клевера, в том числе селекции ФНЦ ВИК им. В.Р. Вильямса. На значительной дистанции от других расположился диплоидный одноукосный сорт Трифон (НИИСХ
Северо-Востока им. Н.В. Рудницкого), полученный методом многократного рекуррентного отбора на основе сорта Krano (Дания). Клевер луговой сорта Трифон — зимостойкий и устойчивый к склеротиниозу и фузариозу, поскольку биотипический отбор проводился на искусственном фоне с повышенной нагрузкой к этим патогенам (45). О высокой степени генетического сходства свидетельствовало близкое расположение на диаграмме образцов Воронежский и Трио. Сорт Воронежский создавался в рамках программы экологической селекции на Воронежской опытной станции посредством селекционной проработки в различных условиях лучших сортообразцов ВНИИ кормов им. В.Р. Вильямса. Результаты генотипирования позволяют предположить, что в формировании нового сорта участвовали и растения сорта Трио. В настоящее время сорт Воронежский считается источником высокой зимостойкости, засухоустойчивости и семенной продуктивности. Благодаря хорошему адаптивному потенциалу, он широко востребован на рынке семян.
-
2. Результаты выравнивания нуклеотидных последовательностей клевера лугового, полученных с помощью SSR-анализа, относительно референса из базы данных Red Clover Kazusa ( http://marker.kazusa.or.jp/Red_clover )
|
Сорт |
Праймер |
Номер клона/длина продукта, п.н. |
Совпадение с референсом |
|
|
идентичность |
индели |
|||
|
Марс |
RCS1307 |
1.3/155 |
149/155 (96 %) |
1/155 (0 %) |
|
RCS3666 |
1.4/216 |
210/216 (97 %) |
4/216 (1 %) |
|
|
1.5/226 |
28/28 (100 %)a |
0/28 (0 %)a |
||
|
179/182 (98 %)a |
0/182 (0 %)a |
|||
|
Топаз |
RCS1307 |
2.2/151 |
140/161 (87 %) |
16/161 (9 %) |
|
RCS0017 |
9.1/150 |
100/101 (99 %)a |
0/101 (0 %)a |
|
|
24/24 (100 %)a |
0/24 (0 %)a |
|||
|
9.2/144 |
100/101 (99 %)a |
0/101 (0 %)a |
||
|
44/44(100 %)a |
0/44 (0 %)a |
|||
|
Трифон |
RCS1307 |
3.3/152 |
149/155 (96 %) |
3/155 (1 %) |
|
3.5/157 |
152/157 (97 %) |
2/157 (1 %) |
||
|
Памяти Лисицына |
RCS1307 |
4.5/169 |
154/169 (91 %) |
14/169 (8 %) |
|
Атлант |
RCS1307 |
5.5/141 |
136/155 (88 %) |
15/155 (9 %) |
|
5.6/137 |
135/155 (87 %) |
19/155(12 %) |
||
|
RCS2199 |
7.4/198 |
190/198 (96 %) |
8/198 (4 %) |
|
|
7.6/200 |
190/200 (95 %) |
10/200 (5 %) |
||
|
RCS4797 |
8.2/175 |
36/36 (100 %)a |
0/36 (0 %)a |
|
|
141/143 (99 %)a |
1/143 (0 %)a |
|||
|
8.3/158 |
116/116 (100 %)a |
0/116 (0 %)a |
||
|
44/44 (100 %)a |
0/44 (0 %)a |
|||
|
Тетраплоидный |
RCS1307 |
6.2/158 |
152/157 (97 %) |
2/157 (1 %) |
|
ВИК |
6.6/155 |
151/155 (97 %) |
1/155 (0 %) |
|
|
RCS5781 |
9.4/205 |
187/208 (90 %) |
12/208 (5 %) |
|
|
9.6/201 |
184/203 (91 %) |
6/203 (2 %) |
||
|
RCS4532 |
10.2/235 |
234/237 (99 %) |
2/237 (0 %) |
|
|
Метеор |
RCS0017 |
8.4/166 |
164/166 (99 %) |
2/166 (1 %) |
|
8.6/172 |
164/172 (95 %) |
8/172 (4 %) |
||
|
RCS3070 |
4.1/333 |
136/137 (99 %)a |
1/137 (0 %)a |
|
|
123/125 (98 %)a |
0/125 (0 %)a |
|||
|
4.2/323 |
136/137 (99 %)a |
1/137 (0 %)a |
||
|
125/125 (100 %)a |
0/125 (0 %)a |
|||
|
RCS3510 |
5.5/254 |
252/271 (93 %) |
17/271 (6 %) |
|
|
5.6/257 |
255/271 (94 %) |
14/271 (5 %) |
||
|
Трио |
RCS0017 |
10.5/164 |
164/164 (100 %) |
0/164 (0 %) |
|
Ветеран |
RCS0017 |
11.2/164 |
149/164 (91 %) |
0/164 (0 %) |
|
RCS4797 |
6.2/180 |
180/190 (95 %) |
10/190 (5 %) |
|
|
ВИК 77 |
RCS3510 |
2.1/249 |
243/271 (90 %) |
22/271 (8 %) |
|
RCS7228 |
3.5/182 |
181/185 (98 %) |
3/185 (1 %) |
|
|
3.6/181 |
180/185 (97 %) |
4/185 (2 %) |
||
Примечани е. a — нуклеотидные последовательности, имеющие крупную вставку или делецию, по сравнению с референсом из базы данных.
Для валидации результатов анализа было проведено секвенирование по Сэнгеру выявленных уникальных фрагментов амплификации (21 — с использованием SSR-маркеров, 5 — SRAP). Анализ полученных данных в поисковой системе ВLAST выявил высокую степень гомологии сиквенсов в сравнении с нуклеотидными последовательностями из референсной базы данных (табл. 2).
На рисунке 3 в качестве примера представлены результаты выравнивания нуклеотидной последовательности ПЦР-фрагмента, полученного с использованием комбинации SRAP-маркеров ME1-EM1.
> tripr.scaffold_37024 Length=549
Score = 398 bits (160), Expect = 8e-83 Identities = 164/166 (9956), Gaps = 0/166 (056) Strand=Plus/Plus
Query 5 [iGAGTCCAAACCGGAT^ACACACATAATTCAATCGCATACATCATCACATAAACATAGATTAA 64 llllll 111111111 И 11111111111 Ш 11111111111111 IM 1111 [ 11111
Sbjct 61 TCCAAATAGGATACACACACATAATTCAATCGCATACATCATCACATAAACATAGATTAA120
Query 65 ATCATGTTACCATCACATTCTTAATATACTTTTTTATTAAAATAACATAATTCTAATATT124
IIIIIIIIIIIIIIIIIHIIIIIIIIIIIIlllllllllllllllllHIIIIIIIIII
Sbjct 121 ATCATGTTACCATCACATTCTTAATATACTTTTTTATTAAAATAACATAATTCTAATATT180
Query 125
Sbjct 181
CTTCTTTATTAGGAGAATCCAATTCCTAAAATTCTACATTCATAAG 170
Hllllllllllllllllllllllllllllllllllllllllllll
CTTCTTTATTAGGAGAATCCAATTCCTAAAATTCTACATTCATAAG 226
> tripr.scaffold_38235
Length=527
Score = 466 bits (211), Expect = 6e-112
Identities = 213/214 (9956), Gaps = 0/214 (0%)
Strand=Plus/Minus
Query 171 TGAAATTCTGGTACACAGTAGACAAQTQTTQTCCACGATGTGCCGACACTTGTCGCAACA IIIIIIIIIIIIIIIIIIIIHHIIIIIIIIIIIHIIIIIIIIIIIIIIIIIIIIIII
Sbjct 461 TGAAATTCTGGTACACAGTAGACAAGTGTTGTCCACGATGTGCCGACACTTGTCGCAACA
Query 231 CTTGTCTTAGCAGTAAGAACAATAAAACAGAGCATTAAAAACATATACTGAAAAGCATAA I I I I I I I I I I I I I I I I I I I I И I I I I I I I I I I I I 1 I I I I I I I I I I I I I I I I I I I I I I I I I
Sbjct 401 CTTGTCTTAGCAGTAAGAACAATAAAACAGAGCATTAAAAACATATACTGAAAAGCATAA
Query 291 ACGACACACATAATTGTTAACCCAGTTCAGCCTAACAGCCTAATCTGGGGGATACCAATC IIIHIIIIIIIIIIIIIIIHHIIIIIIIIIIIIIIHIIIIIIIIIIIHIIIHII
Sbjct 341 ACGACACACATAATTGTTAACCCAGTTCAGCCTAACAGCCTAATCTGGGGGATACCAATC
Query 351 CAGGAGGAAATTCACTATCAGTAGTATtAaTICGTACGCAGTC I 384 и1111111111111111111 iiiiii hull -----------
Sbjct 281 CAGGAGGAAATTCACTATCAGCAGTATTAATTCG 248
Рис. 3. Выравнивание последовательности уникального ПЦР-фрагмента для клевера лугового ( Trifolium pratense L.) сорта Трио, амплифицированного с использованием SRAP-комбинации ME1-EM1 (query) относительного генома T. pratense (sbjct). Отмечены последовательности праймеров.
Аннотированные нуклеотидные последовательности, идентифицирующие сорта Трифон, Марс, Топаз, Атлант, Тетраплоидный ВИК, Метеор, ВИК 77, включены в международную базу генетических ресурсов GenBank NCBI под индивидуальными номерами доступа: MW520170; OP493602 и OP493613; OP493603 и OP493612; OP493606 и OP493607; OP493608 и OP493609; OP493610 и OP493611; OP546652 и OP546653. Эта информация может быть использована для фундаментальных и прикладных исследований по клеверу луговому, включая создание новых методов и технологий. Чтобы повысить точность определения размеров аллелей, не являющихся уникальными для сорта в исследуемой выборке, но выявленных в составе ДНК-профиля (по 8 локусам), мы создали специфические контрольные маркеры, представляющие собой ПЦР-продукты, полученные с использованием плазмид.
Коммерческие маркеры-стандарты не годились для этих целей, поскольку большой диапазон в размерах делений маркерной лестницы не позволял установить незначительную разницу в несколько нуклеотидов между ампликонами. В нашем исследовании размер аллелей в ДНК-профилях изучаемых образцов сопоставляли с контрольным маркером после электрофореза в 10 % ПААГ (рис. 4).
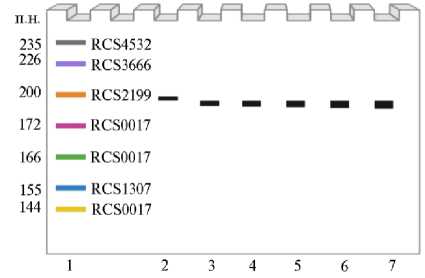
Рис. 4. Схема электрофореза с использованием контрольного маркера для верификации размеров аллелей в ДНК-профилях сортов клевера лугового ( Trifolium pratense L.), паспортизируемых на основе SSR-маркеров: 1 — контрольный маркер, составленный из ампликонов, наработанных с клонов 9.2 (144 п.н.), 1.3 (155 п.н.), 8.4 (166 п.н.), 8.6 (172 п.н.), 7.6 (200 п.н.), 1.5 (226 п.н.), 10.2 (235 п.н.), 2 — ампликон с клона 7.4 паспортизируемого сорта Атлант (SSR-маркер RCS 2199, 198 п.н.), 3-7 — ПЦР-продукты с геномной ДНК сортов Марс, ВИК 77, Метеор, Топаз, Тетраплоидный ВИК (SSR-маркер RCS 2199).
Для изученной коллекции определены идентификационные ДНК-маркеры: восемь пар SSR-локуcов и семь SRAP-комбинаций, которые выявляли уникальность аллельного состава не менее чем у двух российских сортов (табл. 3).
-
3. SSR- и SRAP-маркеры для ДНК-идентификации российских сортов клевера лугового ( Trifolium pratense L.)
Маркер (ссылка)
Последовательность нуклеотидов 5´-3´
Размер ПЦР-фрагментов, п.н.
Код маркера
SSR-маркеры
RCS3666 (32)
CATGGCTGCCTGAGGTTAAT/ TCTGTTTCTTGTCTCGGCCT
216-230
A
RCS3510 (32)
TTCACAAGTTTTTCGGGTGA/ GCCAAAGGGAAGGTTCAATC
249-257
B
RCS7228 (32)
TCAACAATGTGGCTTCTCCT/ AAGGTTCCCAACCCAATTTC
179-187
C
RCS4797 (32)
GCCCGTCTACCTTTTGTTCA/ GCGCCATAAGCAACTGTGTA
155-180
D
RCS2199 (32)
AAAAAGAAAGCGTTAAAGGGG/ GCATTGCCTTTTGCTTCTTC
178-200
E
RCS5781 (32)
GATCGATCCGAAAACCAAAA/ TGCCATCGAGAGAGAAGGTT
165-210
F
RCS1307 (32)
CCCTTCTAGCCTAGCAACCA/ GCGGAAAAGATTCAGCCTAA
137-158
G
RCS0017 (32)
GCGGAAAAGATTCAGCCTAA/
GGACTTCTCTGATATTGAACTGAATG
144-177
H
SRAP-маркеры
F10-R9 (23)
GTAGCACAAGCCGGAAG/ GACTGCGTACGAATTTCA
116-1442
A
Me4-R9 (22)
CGAATCTTAGCCGGAAT/ GACTGCGTACGAATTTCA
165-519
B
ME1-EM1 (22)
TGAGTCCAAACCGGATA/ GACTGCGTACGAATTAAT
125-700
С
F13-Em2 (23)
CGAATCTTAGCCGGCAC/ GACTGCGTACGAATTCGG
234-763
D
F13-R9 (23)
CGAATCTTAGCCGGCAC/ GACTGCGTACGAATTTCA
115-1315
E
F11-R7 (23)
CGAATCTTAGCCGGATA/
GACTGCGTACGAATTGAG
232-1762
F
Me4-EM5 (22, 23)
CGAATCTTAGCCGGAAT/ GACTGCGTACGAATTAAC
139-748
G
-
4. Молекулярно-генетические формулы изученных российских сортов клевера лугового ( Trifolium pratense L.), разработанные на основе SSR- и SRAP-маркеров
Сорт
Формула
SSR-маркирование
Марс
ВИК 77 Метеор Топаз Атлант Тетраплоидный ВИК
А 216/226 B 252/252 C 180/180 D 155/169 E 189/194 F 192/208 G 155/155 H 165/177
A 219/219 B 249/249 C 182/182 D 165/174 E 180/180 F 193/207 G 156/156 H 144/144
A 219/219 B 254/257 C 187/187 D 170/179 E 178/189 F 165/209 G 142/142 H 166/172
A 220/230 B 252/252 C 179/179 D 157/164 E 181/190 F 194/209 G 151/151 H 144 /150
A 221/221 B 252/252 C 179/184 D 158/175 E 198/200 F 195/210 G 137/141 H 145/145
A 218/230 B 252/252 C 179/179 D 170/180 E 181/193 F 201/205 G 155/ 158 H 144/144
SRAP-маркирование
ВИК 84
Трио Воронежский Пеликан
А 331 В 286 С 446 ,356 D 700,447,290,234 E 1070,565,180,115 F 1055,685,534,313,232 G 748,638,523,359
А 400,305 В 286 С 393 D 700,234 E 1070, 300,115 F 685,313,232 G 609,359,139
А 900,800,585,400,317,168 В 129 D 700,270,234 E 1070,300,115 F 800,685,534,313,232 G 748,638,204,139
А 338 ,175 В 286 С 356,234 D 700,234 E 1070,115 F 800,685,313,232 G 748,638,359
Информацию, полученную в результате секвенирования уникальных для сорта ДНК-фрагментов и верификации размеров остальных аллелей в его ДНК-профиле (в сравнении с контрольным маркером), использовали для составления индивидуальной молекулярно-генетической формулы. Прописными буквами латинского алфавита в формулах обозначали исследуемые локусы (код маркера, см. табл. 3), нижним цифровым индексом — размер выявленных аллелей в парах нуклеотидов (табл. 4).
Пр им еч ан и е. Буквы латинского алфавита — код маркера, цифровой индекс — размер фрагментов амплификации (п.н.); жирным шрифтом выделены уникальные продукты, подтвержденные секвенированием.
Молекулярные формулы послужили основой для создания генетических паспортов клевера лугового по двум системам маркирования (шесть — по SSR-локусам, четыре — по SRAP-маркерам). Дополнительно в паспорт была включена информация по происхождению сорта, регионам возделывания, основным морфобиологическим признакам и хозяйственно ценным свойствам (рис. 5).

Рис. 5. Молекулярно-генетические паспорта клевера лугового ( Trifolium pratense L.) сорта Марс на основе системы SSR-маркеров (слева) и сорта Трио на основе системы SRAP-маркеров (справа) .
Таким образом, в результате анализа с использованием SSR- и SRAP-маркеров была проведена оценка генетического полиморфизма сортов клевера лугового. Оптимизированы технологии генотипирования с учетом специфики изучаемой культуры, включая модификации к способу выделения ДНК из суммарной навески проростков и к условиям амплификации с применением SRAP-маркеров. По каждой системе маркирования получены уникальные ДНК-профили и определены маркеры для генетической идентификации. Размеры сортоспецифичных фрагментов ДНК, идентифицирующих семи коммерчески значимых российских сортов клевера лугового, подтверждены секвенированием. Аннотированные нуклеотидные последовательности депонированы в мировой базе генетических ресурсов GeneBank NCBI. Составлены молекулярно-генетические формулы сортов, которые отображают аллельный состав микросателлитных локусов и полиморфизм интрон-экзонных участков генома. Эти данные легли в основу разработки 10 эталонных генетических паспортов (шесть — по SSR-локусам, четыре — по SRAP-маркерам) для российских сортов клевера лугового. По эталонному паспорту можно идентифицировать сорт, провести анализ его однородности и генетической чистоты, установить соответствие анонимного образца известному стандарту. Предлагаемый метод паспортизации клевера лугового прошел тройную систему контроля, включающую 3-кратную повторность экспериментов, клонирование и секвенирование основных фрагментов, использование контрольного маркера для повышения точности определения длин амплифицированных продуктов. Эти приемы позволяют получить точные аллельные профили сортов, снижают вероятность ошибок генотипирования, служат подтверждением достоверности полученных результатов и могут использоваться при разработке генетических паспортов разных видов кормовых культур.
Список литературы Генетическая паспортизация российских сортов клевера лугового (Тrifolium pratense L.) на основе SSR- и SRAP-маркеров
- Reid A., Kerr E.M. A rapid simple sequence repeat (SSR)-based identification method for potato cultivars. Plant Genetic Resources, 2007, 5(1): 7-13 (doi: 10.1017/S1479262107192133).
- Cooke R.J., Reeves J.C. Plant genetic resources and molecular markers: variety registration in a new era. Plant Genetic Resources, 2003, 1(2-3): 81-87 (doi: 10.1079/PGR200312).
- Arens P., Mansilla C., Deinum D., Cavellini L., Moretti A., Rolland S., Schoot H., Calvache D., Ponz F., Collonnier C., Mathis R., Smilde D., Caranta C., Vosman B. Development and evaluation of robust molecular markers linked to disease resistance in tomato for distinctness, uniformity and stability testing. Theoretical and Applied Genetics, 2010, 120: 655-664 (doi: 10.1007/s00122-009-1183-2).
- Wang J., Cogan N.O.I., Forster J.W. Prospects for applications of genomic tools in registration testing and seed certification of ryegrass varieties. Plant Breed, 2016, 135(4): 405-412 (doi: 10.1111/pbr.12388).
- Bonow S., Von Pinho E.V.R., Vieira M.G.C., Vosman B. Microsatellite markers in and around rice genes: applications in variety identification and DUS testing. Crop Science, 2009, 49(3): 880-886 (doi: 10.2135/cropsci2008.06.0380).
- Матвеева Т.В., Павлова О.А., Богомаз Д.И., Демкович А.Е., Лутова Л.А. Молекулярные маркеры для видоидентификации и филогенетики растений. Экологическая генетика, 2011, IX(1): 32-44.
- Korir N.K., Han J., Shangguan L., Wang Ch., Kayesh E., Shang Y., Fang J. Plant variety and cultivar identification: advances and prospects. Critical Reviews in Biotechnology, 2012, 33(2): 111-125 (doi: 10.3109/07388551.2012.675314).
- Tommasini L., Batley J., Arnold G.M., Cooke R.J., Donini P., Lee D., Law J.R., Lowe C., Moule C., Trick M., Edwards K.J. The development of multiplex simple sequence repeat (SSR) markers to complement distinctness, uniformity and stability testing of rape (Brassica napus L.) varieties. Theoretical and Applied Genetics, 2003, 106(6): 1091-1101 (doi: 10.1007/s00122-002-1125-8).
- Bhandary H.R., Bhanu A.N., Srivastava K., Singh M.N., Shreya, Hemantaranjan A. Assesment of genetic diversity in crop plants — an overview. Adv. Plants Agric. Res., 2017, 7(3): 279-286 (doi: 10.15406/apar.2017.07.00255).
- Jamali S.H., Cockram J., Hickey L.T. Insights into deployment of DNA markers in plant variety protection and registration. Theoretical and Applied Genetics, 2019, 132: 1911-1929 (doi: 10.1007/s00122-019-03348-7).
- Schlegel R. Hybrid breeding boosted molecular genetics in rye. Vavilov Journal of Genetics and Breeding, 2015, 19(5): 589-603 (doi: 10.18699/VJ15.076).
- Kolliker R., Enkerli J., Widmer F. Characterization of novel microsatellite loci for red clover (Trifolium pratense L.) from enriched genomic libraries. Molecular Ecology Notes, 2006, 6: 50-53 (doi: 10.1111/j.1471-8286.2005.01133.x).
- Noli E., Teriaca M.S., Sanguineti M.C., Conti S. Utilization of SSR and AFLP markers for the assessment of distinctness in durum wheat. Molecular Breeding, 2008, 22: 301-313 (doi: 10.1007/s11032-008-9176-4).
- Schulman A.H. Molecular markers to assess genetic diversity. Euphytica, 2007, 158: 313-321 (doi: 10.1007/s10681-006-9282-5).
- Сухарева А.С., Кулуев Б.Р. ДНК-маркеры для генетического анализа сортов культурных растений. Биомика, 2018, 10(1): 69-84 (doi: 10.31301/2221-6197.bmcs.2018-15).
- Varshney R.K., Graner A., Sorrels M.E. Genetic microsatellite markers in plants: features and applications. Trends in Biotechnology, 2005, 23(1): 48-55 (doi: 10.1016/j.tibtech.2004.11.005).
- Dias P.M.B., Julier B., Sampoux J.P., Barre P., Dall’Agnol M. Genetic diversity in red clover (Trifolium pratense L.) revealed by morphological and microsatellite (SSR) markers. Euphytica, 2007, 160: 189-205 (doi: 10.1007/s10681-007-9534-z).
- Andersen J.R., Lübberstedt T. Functional markers in plants. Trends in Plant Science, 2003, 8(11): 554-560 (doi: 10.1016/j.tplants.2003.09.010).
- Gupta P.K., Rustgi S. Molecular markers from the transcribed expressed region of the genome in higher plants. Functional & Integrative Genomics, 2004, 4(3): 139-162 (doi: 10.1007/s10142-004-0107-0).
- Ronning C.M., Stegalkina S.S., Ascenzi R.A., Bougri O., Hart A.L., Utterbach T.R., Vanaken S.E., Riedmuller S.B., White J.A., Cho J., Pertea G.M., Lee Y., Karamycheva S., Sul-tana R., Tsai J., Quackenbush J., Griffiths H.M., Restrepo S., Smart C.D., Fry W.E., van der Hoeven R., Tanksley S., Zhang P., Jin H., Yamamoto M.L., Baker B.J., Buell C.R. Comparative analysis of potato expressed sequence tag libraries. Plant Physiology, 2003, 131(2): 419-429 (doi: 10.1104/pp.013581).
- Powell W., Machray G.C., Provan J. Polymorphism revealed by simple sequence repeats. Trends in Plant Science, 1996, 1(7): 215-222 (doi: 10.1016/1360-1385(96)86898-1).
- Li G., Quiros C.F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theo-retical and Applied Genetics, 2001, 103: 455-461 (doi: 10.1007/s001220100570).
- Rhouma H.B., Taski-Adukovic K., Zitouna N., Sdouga D., Milis D., and Trifi-Farah N. Assess-ment of the genetic variation in alfalfa genotypes using SRAP markers for breeding purposes. Chilean Journal of Agricultural Research, 2017, 77(4): 332-339 (doi: 10.4067/S0718-58392017000400332).
- Alghamdi S., Al-Faifi S., Migdadi H., Khan M., El-Harty E., Ammar M. Molecular diversity as-sessment using sequence related amplified polymorphism (SRAP) markers in Vicia faba L. Interna-tional Journal of Molecular Sciences, 2012, 13(12): 16457-16471 (doi: 10.3390/ijms131216457).
- Yousefi S., Saeidi H., Assadi M. Genetic diversity analysis of red clover (Trifolium pratense L.) in Iran using sequence related amplified polymorphism (SRAP) markers. Journal of Agricultural Sci-ence and Technology, 2018, 20(2): 373-386.
- Цветков И.А., Иванов А.Н., Глазко В.И. Генетическая дифференциация сортов риса по IRAP-маркерам. Известия ТСХА, 2006, 4: 155-159.
- Вдовиченко Л.Д., Глазко В.И. Генетическая паспортизация сортов пшеницы с использованием ISSR-PCR маркеров. Сельскохозяйственная биология, 2007, 3: 33-37.
- Федулова Т.П., Федорин Д.Н., Богомолов М.А., Голева Г.Г. ДНК-фингерпринтинг в изучении генетического разнообразия Beta vulgaris L. Вестник Воронежского государственного аграрного университета, 2018, 3(58): 46-53 (doi: 10.17238/issn2071-2243.2018.3.46).
- Колобова О.С., Малюченко О.П., Шалаева Т.В., Шанина Е.П., Шилов И.А., Алексеев Я.И., Велишаева Н.С. Генетическая паспортизация картофеля на основе мультиплексного анализа 10 микросателлитных маркеров. Вавиловский журнал генетики и селекции, 2017, 21(1): 124-127 (doi: 10.18699/VJ17.230).
- Новоселов М.Ю. Клевер луговой (Trifolium pratense L.). В кн.: Основные виды и сорта кормовых культур. М., 2015, 26-30.
- Клименко И.А., Антонов А.А., Душкин В.А., Шамустакимова А.О., Мавлютов Ю.М. Эффективный способ выделения ДНК для ПЦР-анализа из «балк-образцов» проростков. Адаптивное кормопроизводство, 2021, 3(47): 29-48 (doi: 10.33814/AFP-2222-5366-2021-3-29-48).
- Sato S., Isobe S., Asamizu E., Ohmido N., Kataoka R., Nakamura Y., Kaneko T., Sakurai N., Okumura K., Klimenko I., Sasamoto S., Wada T., Watanabe A., Kothari M., Fujishiro T., Tabata S. Comprehensive structural analysis of the genome of red clover (Trifolium pratense L.). DNA Research, 2005, 12(5): 301-364 (doi: 10.1093/dnares/dsi018).
- Aneja B., Yadav N.R., Chawla V., Yadav R.C. Sequence related amplified polymorphism (SRAP) molecular marker system and its applications in crop improvement. Mol. Breeding, 2012, 30: 1635-1648 (doi: 10.1007/s11032-012-9747-2).
- Don R.H., Cox P.T., Wainwright B.J., Baker K., Mattick J.S. «Touchdown» PCR to circumvent spurious during gene amplification». Nucleic Acids Res., 1991, 19(14): 4008 (doi: 10.1093/nar/19.14.4008).
- Okonechnikov K., Golosova O., Fursov M., the UGENE team. Unipro UGENE: a unified bio-informatics toolkit. Bioinformatics, 2012, 28(8): 1166-1167 (doi: 10.1093/bioinformatics/bts091).
- Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 2012, 28(19): 2537-2539 (doi: 10.1093/bioinfor-matics/bts460).
- Kraft T., Säll T. An evaluation of the use of pooled samples in studies of genetic variation. He-redity, 1999, 82: 488-94 (doi: 10.1038/sj.hdy.6885120).
- Herrmann D., Boller B., Widmer F., Kölliker R. Optimization of bulked AFLP analysis and its application for exploring diversity of natural and cultivated populations of red clover. Genome, 2005, 48(3): 474-486 (doi: 10.1139/g05-011).
- Dellaporta S.L., Wood J., Hicks J.B. A plant DNA mini preparation: Version II. Plant Molecular Biology Reporter, 1983; 1(4): 19-21 (doi: 10.1007/BF02712670).
- Ulloa O., Ortega F., Campos H. Analysis of genetic diversity in red clover (Trifolium pratense L.) breeding populations as revealed by RAPD genetic markers. Genome, 2003, 46(4): 529-535 (doi: 10.1139/g03-030).
- Radinovic I., Vasiljevic S., Brankovic G., Ahsyee R.S., Momirovic U., Perovic D., Surlan-Mo-mirovic G. Molecular characterization of red clover genotypes utilizing microsatellite markers. Chilean Journal of Agricultural Research, 2017, 77(1): 41-47.
- Dias P.M.B., Julier B., Sampoux J.P., Barre P., Dall’Agnol M. Genetic diversity in red clover (Trifolium pratense L.) revealed by morphological and microsatellite (SSR) markers. Euphytica, 2008, 160: 189-205 (doi: 10.1007/s10681-007-9534-z).
- Doris H., Beat B., Bruno S., Franco W., Roland K. QTL analysis of seed yield components in red clover (Trifolium pratense L.). Theor. Appl. Genet., 2006, 112: 536-545 (doi: 10.1007/s00122-005-0158-1).
- Kölliker R., Enkerli J., Widmer F. Characterization of novel microsatellite loci for red clover (Trifolium pratense L.) from enriched genomic libraries. Molecular Ecology Notes, 2006, 6: 50-53 (doi: 10.1111/j.1471-8286.2005.01133.x).
- Грипась М.Н., Арзамасова Е.Г., Попова Е.В., Онучина О.Л. Новый высокопродуктивный, толерантный к болезням сорт клевера лугового Трифон. Аграрная наука Евро-Северо-Во-стока, 2013, 2(33): 19-23.


