Генетические маркеры в козоводстве (обзор)
Автор: Селионова М.И., Трухачев В.И., Айбазов а-М.М., Столповский Ю.А., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Обзоры, проблемы
Статья в выпуске: 6 т.56, 2021 года.
Бесплатный доступ
По данным международной продовольственной организации (ФАО), биоразнообразие коз представлено 635 породами, которых разводят в 170 странах (https://www.fao.org/dad-is). Высокая адаптивность к различным климатическим условиям и уникальность получаемой продукции определили широкое географическое распространение и существенный рост мирового поголовья этих животных в последние десятилетия (И.Н. Скидан с соавт., 2015; А.И. Ерохин с соавт., 2020). Микросателлитные маркеры ДНК (microsatellites, STR, short tandem repeats) используются для изучения генетической дифференциации пород и популяций коз (C. Wei с соавт., 2014; G. Mekuriaw с соавт., 2016). Установлены незначительные генетические расстояния (FST 0,033-0,069) между породами, разводимыми в Европе, что может служить подтверждением частого обмена генетическим материалом между ними. Для пород Восточной и Юго-Восточной Азии выявлена более существенная генетическая дифференциация (FST 0,134-0,183), которая предположительно обусловлена эколого-географическими особенностями ареалов животных, в частности их удаленностью друг от друга (K. Nomura с соавт., 2012; G. Wang с соавт., 2017; P. Azhar с соавт., 2018). В молочном козоводстве наибольший интерес представляет полиморфизм SNP (single nucleotide polymorphism) в генах казеина ( CSN1S1 , CSN1S2 , CSN2 ) и b-лактоглобулина ( BLG ) (N. Silanikove с соавт., 2010; Ворожко И.В. с соавт., 2016) . Для CSN1S1 описано 18 аллельных вариантов, для CSN2 - 8, для CSN3 - 16 (S. Ollier с соавт., 2008; T.G. Devold с соавт., 2010). Установлено, что генотип CSN1S1AA ассоциирован с большим количеством белка в молоке и меньшим содержанием липидов и среднецепочечных жирных кислот (Y. Chilliard с соавт., 2006; D. Marletta с соавт., 2007). Козы с генотипом BLGАВ характеризовались более продолжительным периодом лактации, большим количеством молока, жира и белка (А.С. Шувариков с соавт., 2019). Секвенирование генома козы в рамках проекта AdaptMap и последующая разработка чипа 52К SNP BeadChipGoat позволили расширить область поиска участков генома, вовлеченных в селекционный процесс (G. Tosser-Klopp с соавт., 2014; A. Stella с соавт., 2018). Получены данные о связи генов некоторых транскрипционных факторов ( RARA , STAT ), цитокинов ( PTX3 , IL6 , IL8 ), ферментов ( DGAT1 ) с показателями молочной продуктивности (P. Martin с соавт., 2018; D. Ilie с соавт., 2018). Продемонстрирована связь генов репептора-1 меланокортина ( MC1R ), его антагониста - сигнального белка агути ( ASIP ) и рецептора цитокинов KIT ( KIT ) с окрасом шерстных волокон; генов фактора роста фибробластов 5 ( FGF5 ), фактора активируемого гипоксией 1 ( EPAS1 ) и регулирующего энергетический обмен фермента NOXA1 ( NOXA1 ) - с шерстной продуктивностью коз и их адаптацией к высоте местности (X. Wang с соавт., 2016; S. Song с соавт., 2016; J. Guo с соавт., 2018). Таким образом, оценка генетических взаимоотношений между породами, поиск маркеров генов, ассоциированных с хозяйственно ценными признаками, перспективны для использования в селекционных программах и дальнейшего развития козоводства (L.F. Brito с соавт., 2016; S. Desire, 2016; A. Molina с соавт., 2018; Т.Е. Денискова с соавт., 2020). Однако, несмотря на определенные достижения, гены и их молекулярные маркеры, связанные с экономически важными признаками у коз, такими как особенности размножения, пуховая, шерстная и молочная продуктивность, а также определяющие устойчивость к болезням, остаются в значительной степени неизвестными.
Козы, микросателлиты, генетическая дифференциация пород, генетические маркеры продуктивности, gwas
Короткий адрес: https://sciup.org/142231899
IDR: 142231899 | УДК: 636.39:575.17 | DOI: 10.15389/agrobiology.2021.6.1031rus
Текст обзорной статьи Генетические маркеры в козоводстве (обзор)
Козоводство — одна из динамично развивающихся отраслей животноводства. По данным ФАО (Food and Agriculture Organization of the United Nations, FAO), за 30 лет мировое поголовье коз увеличилось почти вдвое: с 589 млн в 1991 году до 1 млрд 200 млн к началу 2020 года. Сегодня в мире насчитывается 635 пород коз, которых разводят в 170 странах, при этом к трансграничным отнесены лишь 38 пород (DAD-IS, информационная система по разнообразию домашних животных, .
Цель нашего обзора — обобщить и проанализировать данные о со- временных генетических маркерах для исследования биоразнообразия, генетической структуры, определения степени инбридинга, чистоты пород и популяций коз, полногеномного поиска ассоциаций (genome-wide association studies, GWAS) с целью выявления генов, связанных с экономически важными признаками продуктивности.
Домашняя коза ( Capra hircus ) широко распространена в мире и представлена большим породным разнообразием благодаря своим биологическим особенностям, в частности высокой адаптивности к различным климатическим условиям содержания. Коз разводят в горных, высокогорных, степных и полупустынных зонах, которые характеризуются скудной травяной растительностью. На столь ограниченном кормовом фоне другие виды животных (крупный рогатый скот, лошади, овцы) не могут восполнить потребности в питательных веществах и энергии. Повсеместное разведение коз и рост их численности связаны и с общемировой тенденцией увеличения спроса на продукцию с уникальными свойствами, к которой относятся козий пух, могер, козье молоко, козлятина (1).
Козий пух издревле использовался для изготовления теплых изделий, обладающих особой легкостью, мягкостью и эластичностью, что актуально и в настоящее время. Стада пуховых коз широко распространены в Турции, Индии, Монголии, Китае, Афганистане, Киргизии, Узбекистане и России .
Производство козьего молока в странах Азии, Африки, Северной и Южной Америки за последние 10 лет увеличилось в среднем на 21,3; 18,4 и 9,5 %. Во Франции, Греции, Италии, Испании и Голландии доля потребления козьего молока (с учетом изготовления сыров) составляет 15-20 % от общего объема молочного производства (2). Козье молоко все чаще рассматривается как сырье для продуктов с высокой биологической, а в отдельных случаях и терапевтической ценностью и для детского питания. Одна из особенностей козьего молока заключается в значительно большей дисперсии жировых шариков в сравнении с коровьим (средний диаметр 3,19 мкм, общая площадь — 21,78 см2/мл, против 3,51 мкм и 17,11 см2/мл), что обеспечивает его высокую переваримость за счет доступности для липолитических ферментов. В молоке коз на 54,6-80,2 % больше ненасыщенных короткоцепочечных жирных кислот (C 4:0 -С 10:0 ) (3). Кроме того, высокое содержание β -казеина и ничтожно малое количество (практически отсутствие) α s1 -казеина, который вызывает аллергические реакции, приближают козье молоко по своему составу к женскому грудному молоку. Молоко коз также отличается физико-химическими свойствами мицелл казеина, которые содержат больше кальция и неорганического фосфора, менее сольватированы и более устойчивы к нагреванию, поэтому по сравнению с молоком других видов животных легче теряют казеин, что определяет высокую сыропригодность (4).
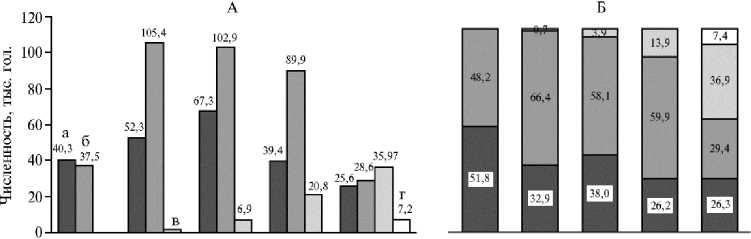
В России в настоящее время разводят 10 пород и популяций коз различного направления продуктивности. Поголовье на конец 2019 года составило 97370 животных (табл. 1).
Следует отметить, что за последние 20 лет наблюдалось существенное изменение породного состава коз по направлениям продуктивности. Начиная с 2015 года, отмечается значительное снижение численности коз шерстного направления продуктивности и, соответственно, их доли в общем поголовье с 59,9 до 29,4 %. При этом заметно выросло число коз молочного направления. Так, в 2005 году козы молочного направления продуктивности отсутствовали в структуре российского козоводства, а к концу 2019 года на их долю приходилось уже 36,9 % (рис. 1).
-
1. Численность (гол.) коз ( Capra hircus ) различных пород и популяций, разводимых в Российской Федерации, в разные годы (5-7)
|
Порода (популяция) |
Год |
||||
|
2000 |
2005 |
2010 |
2015 |
2019 |
|
|
Алтайская белая пуховая |
8300 |
||||
|
Альпийская (Alpine) |
900 |
5230 |
|||
|
Горноалтайская пуховая |
15700 |
11300 |
27300 |
22200 |
10800 |
|
Дагестанская пуховая |
5700 |
16600 |
19500 |
н.д. |
5000а |
|
Дагестанская шерстная |
5800 |
16700 |
19600 |
н.д. |
11000а |
|
Донская (придонская) |
2000 |
1600 |
н.д. |
н.д. |
н.д. |
|
Зааненская (Saanen) |
1100 |
6900 |
19900 |
29770 |
|
|
Карачаевская |
н.д. |
н.д. |
н.д. |
н.д. |
8000а |
|
Мурсиано гранадина (Murciano-Granadina) |
470 |
||||
|
Нубийская (Nubian) |
330 |
||||
|
Оренбургская |
16900 |
22800 |
20500 |
17200 |
6500 |
|
Русская белая |
н.д. |
н.д. |
н.д. |
н.д. |
170а |
|
Советская шерстная |
31700 |
88700 |
83300 |
89900 |
28600 |
|
Тувинская грубошерстная |
н.д. |
н.д. |
н.д. |
н.д. |
7200а |
|
Итого |
77800 |
158800 |
177100 |
150100 |
97370 |
|
Не идентифицированные |
2800 |
28500 |
7100 |
63200 |
41130 |
Примечание. Алтайская белая пуховая порода была официально утверждена в 2016 году. Козы заанен-ской породы завезены в Российскую Федерацию в 2001 году, пород альпийская, мурсиано гранадина и нубийская — в 2015-2018 годах. Общая численность рассчитана на основании данных, официально представленных организациями по племенному животноводству. н.д. — нет данных, а — численность определена на основании информации, представленной в книгах учета администраций поселков, где находятся фермерские хозяйства (согласно которым осуществляется ветеринарный контроль).
2000 2005 2010 2015 2019 2000 2005 2010 2015 2019
Год
Численность коз ( Capra hircus ) различных направлений продуктивности (А) и распределение коз по направлениям продуктивности (%) (Б) на территории Российской Федерации с 2005 по 2019 год: а — пуховые, б — шерстные, в — молочные, г — грубошерстные (5-7).
В настоящее время для ускоренного развития козоводства недостаточно использовать только традиционные методы, в связи с чем возрастает необходимость интеграции в селекционный процесс современных ДНК-технологий, поскольку они позволяют повысить эффективность селекции посредством отбора носителей аллелей, ассоциированных с хозяйственно ценными признаками (8, 9). Можно выделить следующие типы ДНК-маркеров, получившие наибольшее распространение в исследовании геномов животных, включая коз: RFLP (restriction fragment length polymorphism), RAPD (randomly amplified polymorphic DNA), AFLP (amplified fragment length polymorphism), MS (microsatellites, STR, short tandem repasts), SNP (single nucleotide polymorphism), CNV (copy number variation). Наибольшее распространение в исследованиях генома коз получили микросателлиты, известные также под названием STR-маркеров, и маркеры на основе однонуклеотидных полиморфизмов (SNP).
Микросателлиты (STR-маркеры). В силу доступности, невысокой стоимости и информативности микросателлиты остаются одними из наиболее распространенных маркеров в филогенетических и таксономи- ческих исследованиях и используются в программах сохранения генетических ресурсов сельскохозяйственных животных. Это особенно актуально для аборигенного животноводства вообще и козоводства в частности, поскольку в мире насчитывается около 600 аборигенных пород коз (10, 11).
При исследовании генетических процессов в популяциях наиболее часто применяют F-статистики Райта, или индексы фиксации, которые характеризуют индивидуальный (Fis), субпопуляционный (Fst) и популяционный (Fit) уровни генетической структуры популяции: Fis = (Hs - Hi)/Hs, Fst = (Ht - Hs)/Hт, Fit = (Ht - Hi)/Hт, где Hi — наблюдаемая гетерозиготность, Hs — ожидаемая гетерозиготность в субпопуляциях, Ht — ожидаемая гетерозиготность во всей популяции при панмиксии. Fis указывает на снижение гетерозиготности из-за неслучайного спаривания, Fit — на степень инбридинга особей в целом по популяции. При Fis, it > 0 наблюдается дефицит гетерозиготных особей, при Fis, it < 0 — избыток. Fst указывает на снижение гетерозиготности из-за ограничения потока генов и генетического дрейфа между субпопуляциями. Fst для двух популяций служит значением генетической дистанции. При Fst < 0,05 дифференциация популяций незначительная, при Fst > 0,25 — значимая (12). M. Nei выразил индексы фиксации через аллельные частоты, наблюдаемую и ожидаемую гетерозиготность для любых популяций и предложил использование генетических дистанций (13, 14).
С развитием генетических методов возрастало число микросателлит-ных локусов, которые использовали в исследовании биоразнообразия коз шерстного и пухового направлений продуктивности. Так, по 11 локусам микросателлитов изучили шесть популяций кашмирских коз из Китая, которые образовали три отдельных кластера: тибетский (Tibetan) (тибетские козы плоскогорного и долинного типов, Tibetan goat of Plateau type and Tibetan goat of Valley type), сычуаньский (Sichuan) (черные козы, black goat; мэйгу, Meigu; цзяньчан, Jianchang; байюй, Baiyu) и синьцзянский (Xinjiang) (синьцзянские козы, Xinjiang goat) (15). В другой работе использовали 14 микросателлитных локусов при изучении генетической дифференциации девяти кашмирских пород из Китая. Полученные значения Fst указывали на их высокую генетическую обособленность, при этом наибольшую удаленность демонстрировала порода хегу (Hegu), разводимая в Тибете (16).
В сравнительных исследованиях, выполненных В.Р. Харзиновой с соавт. (17), дана генетическая характеристика советской шерстной, таджикской шерстной, оренбургской пуховой, альпийской и зааненской молочных пород коз с использованием 10 локусов микросателлитов. Показано, что каждая из изученных пород имеет свою популяционно-генетическую структуру, определена степень генетической дифференциации пород.
В работе M.I. Selionova с соавт. (18) проведена оценка генетического разнообразия и генетических дистанций между шерстными и пуховыми породами коз, разводимыми на Северном Кавказе (карачаевская, дагестанская пуховая, дагестанская шерстная), в Сибири (советская шерстная) и на Южном Урале (оренбургская), а также между тремя видами горных козлов — сибирским козерогом (C. sibirica), безоаровым козлом (C. aegagrus) и туром (C. caucasica) с использованием 16 локусов микросателлитов. Наибольшим генетическим разнообразием характеризовались карачаевские козы — среднее число аллелей на локус составило 9,1, тогда как в других породах — 6,57,5. Выявлено формирование трех кластеров: первый образовали подвиды кавказского тура, второй — сибирского козерога, третий — породы домашних коз. В корне третьего кластера были локализованы группы безоарового козла, что служит косвенным подтверждением их участия как предковой формы домашних коз (18).
С использованием микросателлитных маркеров исследовано генетическое разнообразие пяти популяций аборигенных монгольских коз (гурван эгч, Gurvan egch; дархатская, Darhatskaya; бурах завхан, Burakh zavkhan; уль-гий уулан, Ulgiy uulan; алтай уулан, Altay uulan), двух популяций местных тувинских коз, и трех пород — советской, таджикской шерстной и оренбургской пуховой. Выявлены две основные группы, в одну были объединены преимущественно монгольские аборигенные популяции, в другую — среднеазиатские породы коз. Популяции тувинской местной козы разделились между соответствующими группами. При этом монгольские козы характеризовались высоким внутрипопуляционным разнообразием и низкой степенью генетических различий между популяциями (19).
Ряд исследований посвящен изучению генетического разнообразия молочных коз. В работе G. Wang с соавт. (20) использовались 15 микроса-теллитных маркеров для изучения пород, разводимых в Китае: собственной селекции (гуаньчжун, Guanzhong; лаошань, Laoshan; вэнденг, Wendeng), выведенной с использованием зааненской породы — синьонг (Xinong Saanen) и завезенной из Европы — нубийской (Nubian). Среднее число аллелей на локус составило 4,9, значения Fis находились в диапазоне от 0,09 до - 0,08, Fst равнялся 0,08. Между породами венденг (Wendeng) и лаошань (Laoshan) а также гуаньчжун (Guanzhong) и синьонг (Xinong) выявили наиболее тесные генетические связи, которые отражали историю формирования и географию разведения. Установлено, что все четыре китайские породы имели общего предка — зааненскую породу, которая была импортирована в Китай из Европы в XVIII веке (20).
A.M. Ara u jo с соавт. (21) с использованием 11 микросателлитных локусов провели сравнение местной молочной породы — мохото (Moxoto) с альпийской (Alpine) и зааненской (Saanen). Значение Fst между мохото и завезенными породами составило 0,08, тогда как между последними — 0,03, что указывало на их большее генетическое сходство (21).
Генетическую дифференциацию молочных пород коз, разводимых в Таиланде (джамунапари, Jamunapari; альпийская, Alpine; нубийская, Nubian; зааненская, Saanen; тоггенбургская, Toggenburg), изучали с помощью 12 микросателлитных маркеров. Альпийская, зааненская и тоггенбургская породы вошли в один филогенетический кластер, джамунапари и нубийская — в два других. При этом среднее число аллелей на популяцию на один мик-росателлитный локус составляло 7,4. Значения Fis находились в диапазоне от 0,18 до 0,04, Fst составлял 0,07 (22).
Микросателлитные маркеры применяли для идентификации породы коз с сохраняемым статусом при производстве молочных продуктов. Так, джиргентанскую породу (Girgentana) разводят на острове Сицилия, ее отличительная особенность — уникальное качество молока, но вследствие малой численности джиргентанские козы находятся под угрозой исчезновения, поэтому принимаются меры по сохранению породы (23). Панель из 20 микросателлитных маркеров была использована для генетической идентификации коз пород джиргентанская (Girgentana), мальтезская (Maltese) и деривата ди сириа (Derivata di Siria). Восемь аллелей микросателлитных маркеров присутствовали у пород джиргентанской (Girgentana) и деривата ди сирия (Derivata di Siria), но отсутствовали у мальтезских коз. Три мик-росателлитных маркера (FCB20, SRCRSP5, TGLA122), признанные наиболее информативными, были предложены для использования в системе генетического мониторинга молочных продуктов, получаемых от коз джир- гентанской породы и при смешивании с молоком от животных других пород (24).
В масштабном исследовании, выполненном в Китае с использованием 30 микросателлитных локусов и охватывающем более 2 тыс. коз 40 пород и популяций различного направления продуктивности, было установлено, что их генетическая структура обусловлена главным образом географическим происхождением и периодами миграции человека по территории страны. Более отчетливо генетическая дифференциация коз прослеживалась в Западном Китае, для популяций которого установлено два кластера — югозападный и северо-западный. Эти кластеры совпадали с разделением естественными природными барьерами (горные хребты, бассейны рек) (25).
Генетическое разнообразие и взаимосвязь между 20 породами, разводимыми в Индии, S.P. Dixit с соавт. (26) изучили на основе 25 микроса-теллитных маркеров. Большинство локусов были гетерозиготными, значения Fis колебались от 0,61 до 0,73. Наибольшим разнообразием характеризовалась порода канниаду (Kanniadu), наименьшим — османабади (Osmanabadi). Общее значение Fst составило 0,183, при этом 83,5 % генетической изменчивости оказалось обусловлено различиями между особями внутри породы и только 16,5 % — между породами. Наименьшее генетическое расстояние определено между породами ганджам (Ganjam) и малабари (Malabari) (0,22), наибольшее — между канниаду (Kanniadu) и малабари (0,83) (26).
При исследовании 18 аборигенных пород и популяций коз из семи стран Восточной Азии было применено 26 микросателлитных локусов. Среднее число аллелей на локус колебалось от 2,5 до 7,6 и в среднем по изученным породам составило 5,8, при этом наблюдался дефицит гетерозигот и общий инбридинг (Fis = 0,054, Fit = 0,181, р < 0,01). В Монголии и Бангладеш отмечалось большее генетическое разнообразие в популяциях коз, чем в Японии, Корее и Индонезии. Породы разделились на три кластера — восточноазиатский, юго-восточноазиатский и монгольский, что соотносилось с направлением продуктивности, географическим происхождением и путями миграции (27).
В работе J. Ca no n с соавт. (28) использовали 30 микросателлитных маркеров для генотипирования 45 пород коз, разводимых в 15 европейских и ближневосточных странах. Во всех породах выявлен дефицит гетерозиготности (Fis = 0,10) и отмечена средняя генетическая дифференциация между ними. Многомерный анализ частот аллелей выявил четыре кластера: первый составили породы Восточного Средиземноморья (Ближний Восток) (Fst = 0,033), второй — Центрального Средиземноморья (Fst = 0,040), третий — Западного Средиземноморья (Fst = 0,051), четвертый — Северной и Центральной Европы (Fst = 0,069). Снижение генетического разнообразия коз с юго-востока на северо-запад сопровождалось повышением дифференциации на уровне породы. Около 41 % генетической изменчивости было связано с географическим происхождением пород. Полученные данные рассматривались авторами как подтверждение гипотезы о том, что домашний скот мигрировал с Ближнего Востока в Западную и Северную Европу, при этом формирование пород было более систематическим в Северной и Центральной Европе, чем на Ближнем Востоке.
В ряде источников предложено рассматривать значение Fst, равное или превышающее 0,25 как значимое генетическое расстояние между породами, от 0,05 до 0,25 — как среднее, менее 0,05 — как незначительное (12, 13, 29, 30). Анализ приведенных данных позволяет заключить, что в целом между породами и популяциями коз, разводимыми в Европе, установлены небольшие генетические расстояния (Fst 0,033-0,069), что может рассматриваться как подтверждение частого обмена генами в результате скрещиваний для улучшения продуктивности. Для пород и популяций коз, обитающих в Восточной и Юго-Восточной Азии, генетическая дифференциация более существенная (Fst 0,134-0,183), что, по-видимому, обусловлено эколого-географическими особенностями и удаленностью ареалов.
Изучение истории происхождения коз, направлений их миграции, генетической дифференциации и особенностей генетической структуры как отражение приспособительных процессов к среде разведения не теряет актуальности. Для получения новых данных в настоящее время все шире используют однонуклеотидные полиморфизмы (SNP) (31-33).
Однонуклеотидные полиморфизмы (SNP-маркеры) . SNP представляют собой наиболее часто встречающийся тип полиморфизма как ядерной, так и митохондриальной ДНК. Главное преимущество использования SNP в качестве маркеров по сравнению с микросателлитами — их широкое распространение в геноме, ясный мутационный механизм с низкой гомоплазией и мутабельностью.
Кроме того, SNP у коз, в отличие от мультиаллельных микросателлитов, представлены в виде бивалентных вариантов. К методическим преимуществам анализа SNP относится отсутствие особых требований к качеству ДНК (анализ SNP проводится, как правило, посредством получения коротких фрагментов длиной менее 100 п.н.), более низкая степень ошибочного генотипирования, возможности автоматизации процесса и стандартизации получаемых данных. Исследование SNP получило широкое распространение еще на ранних этапах развития ДНК-диагностики сельскохозяйственных животных, так как именно этот тип изменчивости лежит в основе полиморфизма генов, ассоциированных с хозяйственно ценными признаками. Развитие высокопроизводительных технологий генотипирования сделало SNP доминирующим типом ДНК-маркеров в исследовании геномов сельскохозяйственных животных.
В настоящее время SNP считаются предпочтительным типом маркеров для геномной оценки, включая полногеномные ассоциативные исследования, для установления родства между индивидуумами, определения степени инбридинга и гибридизации, генетического картирования высокого разрешения и более полной характеристики генетических ресурсов (34).
Полиморфизм генов продуктивности коз. Наряду с филогенетическими исследованиями для селекционного улучшения продуктивности коз важное значение имеет установление генов и их аллельных вариантов, ассоциированных с хозяйственно ценными признаками. Для молочных коз это в первую очередь показатели, характеризующие количественные параметры удоя, — жирность молока и его белковомолочность (35). Основная часть белков молока — это казеин, содержащий четыре фракции ( α s1 -, α s2 -, β - и k-казеин), и сывороточные белки ( β -лактоглобулин, α -лак-тальбумин) (36, 37). Всесторонне изучено влияние этих белков на технологические свойства молока и возможность получения продуктов с заданными параметрами качества, что определило интерес к изучению генов, контролирующих их синтез (38, 39).
Наиболее изучен ген α s1 -казеина ( CSN1S1 ), полиморфизм которого определяется набором аллельных вариантов. Они определены как сильные для содержания α s1 казеина ∼ 3,5 г/л ( A , A' , B 1 , B 2 , B 3 , B 4 , C , H , L ), средние — ∼ 1,1 г/л ( E , I ), слабые — ∼ 0,45 г/л ( D , F , G ) и ноль-вариант ( O 1 , O 2 , N ) ( α s1 -казеин в молоке отсутствует) (37-39). Так называемые сильные аллели чаще встречаются у пород, разводимых в Испании, Италии, Франции,
Греции, средние и слабые широко представлены у коз в Новой Зеландии и Бразилии (43, 44). В гене CSN1S2 ( α s2 -казеин) описано 9 аллельных вариантов ( A , B , C , D , E , F , 0 , sub A и sub E ), в гене CSN2 ( β -казеин) — 8 ( A , A1 , O’ , O , B , C , D , E ), в гене CSN3 ( κ -казеин) — 16 ( A , B , B’ , B’’ , C , C’ , D , E , F , G , H , I , J , K , L , M ) (37, 40). Основные типы казеинов кодируются генами, локализованными на 6-й хромосоме и тесно сцепленными в едином кластере, протяженность которого составляет 250-350 тыс. п.н. (45).
Ряд работ посвящен влиянию полиморфизма генов основных белков молока на коагуляционные свойства, показатели пищевой ценности, формирование продуктивности коз. Установлено, что в продуктах из молока коз с генотипом AA по гену CSN1S1 содержание белка было на 4,5 % выше, чем из молока животных с генотипом FF , что обосновывает целесообразность отбора носителей аллеля À (40).
У коз, продуцирующих молоко с низким содержанием α s1 -казеина, отмечено достоверное снижение количества общих липидов и среднецепочечных жирных кислот С 8 -С 12 (каприловой, каприновой, лауриновой), а также пальмитиновой, стеариновой, линолевой и конъюгированной линолевой кислот. То есть полиморфизм гена CSN1S1 влияет на интенсивность липогенеза в секреторных клетках молочной железы (46, 47).
В работе, проведенной на пяти породах коз китайской селекции (более 4 тыс. животных), включая наиболее распространенную породу — белую кашемировую шаанбэй (Shaanbei White Cashmere), при секвенировании гена α s1 -казеина была выявлена лишь одна индел-мутация размером 11 п.н., обозначенная как генотип II , которая ассоциировалась с числом козлят при первом окоте. Особи с генотипом II имели достоверно большее количество потомков по сравнению с генотипами ID и DD , что позволило авторам рекомендовать эту индел-мутацию для включения в селекционные программы по увеличению многоплодия (48).
Ряд исследований посвящен влиянию гена β -лактоглобулина ( BLG ) на продуктивность коз. Так, А.С. Шуварикова с соавт. (49) установили, что козы зааненской породы с генотипом ÀÂ характеризовались более продолжительной лактацией, при этом от них было получено в среднем на 110,2 кг (р < 0,01) больше молока и на 3,7 кг и 3,5 кг (р < 0,05) больше соответственно молочного жира и белка по сравнению с генотипами AA и BB . Аналогичные данные получили А.Г. Фатихов с соавт. (50). В то же время лучшие показатели питательности и биологической ценности йогурта и творога отмечали для вариантов, изготовленных из молока коз нубийской и альпийской пород с генотипом ÂÂ (49).
О.А. Кравцова с соавт. (51) пришли к выводу, что в селекционные программы по улучшению молочных коз желательно включать генотипирование по генам weaver , BLG и гену гипофизарного транскрипционного фактора ( POU1F1 ). Установлено, что особи — носители комплексных генотипов T2T2 / S1S2 / D1D2 и T2T2 / S2S2 / D1D1 по генам weaver/BLG/POU1F1 имели более высокое содержание жира и белка в молоке (5,64 и 3,63 %), чем козы других генотипов (4,08 и 3,32 %). В работе Г.М. Гончаренко с соавт. (52) живая масса коз белой пуховой породы, гетерозиготных по BLG , была на 0,30-0,61 кг (p < 0,05) выше в сравнении с козами других генотипов.
Исследование геномов с использованием ДНК-чипов и секвенирования. Развитие методов генетического анализа, основанного на исследовании полных геномов по сотням или тысячам однонуклеотидных полиморфизмов, распределенных по всему геному, значительно расширило возможности идентификации участков генома, контролирую-1038
щих физиолого-биохимические процессы, которые определяют фенотипические различия животных (53, 54).
В настоящее время во многих странах мира проводятся исследования по поиску полногеномных ассоциаций (GWAS) с признаками продуктивности у коз (55). Применению GWAS предшествовала широкомасштабная работа Международного консорциума по изучению генома козы (The International Goat Genome Consortium, IGGC; по реализации нескольких исследовательских проектов, связанных с полным секвенированием генома этих животных. В рамках проекта AdaptMap были генотипированы 4653 животных в 148 популяциях из 35 стран с пяти континентов (56). Разработанный вариант SNP-панели основывался на анализе различий по 12 млн вариантов SNP, выявленных в геномах коз пород заа-ненская (Saanen), альпийская (Alpine), креольская (Creole), бурская (Boer), какангжанская (Katjang) и саванская (Savanna). Дальнейшую валидацию распределения SNP провели на 10 других породах коз. В результате отобрали 52295 SNP, которые были успешно использованы для изготовления чипа 52К SNP BeadChipGoat («Illumina, Inc.», США) (57). Полногеномное секвенирование геномов птицы и свиней из разных стран позволило предположить, что интенсивный искусственный отбор способствовал быстрой фенотипической эволюции у домашних животных (58, 59). Разработка ДНК-чипов существенно расширила возможности по идентификации локусов, находящихся под давлением отбора у свиней и крупного рогатого скота (6062), а также овец (63, 64). Эти результаты продемонстрировали, каким образом положительный отбор, действующий на признаки, изменил геном домашних животных. Однако следует отметить, что определенные ограничения в численности особей для генотипирования по SNP на чипах могут привести к изменению частотных распределений аллелей, что влияет на точность популяционно-генетического анализа (65). Например, практически все SNP, которые включены в чип GoatSNP50 BeadChip («Illumina, Inc.», США), были отобраны в шести зааненских, семи альпийских и трех креольских популяциях коз. При этом оказалось, что плотность распределения выявляемых SNP на геномной ДНК была недостаточной для получения точного результата при оценке локусов, находящихся под давлением отбора (57).
При проведении GWAS у британских молочных коз учитывали удой, содержание в молоке жира и белка, количество соматических клеток, а также показатели экстерьера — глубину вымени, место его прикрепления, форму соска, угол крепления соска, размер и форму передних и задних ног, крепость передних и задних копыт. Общая база фенотипических данных включала 137235 записей для обследованных 4563 коз. Анализ ассоциаций позволил выявить на 19-й хромосоме SNP, достоверно связанные с количеством молока. Кроме того, были обнаружены еще несколько SNP на 4-й, 8-й, 14-й и 29-й хромосомах, связь которых с молочной продуктивностью оказалась менее существенной. Три SNP, идентифицированные на 19-й хромосоме, были связаны с местом прикрепления и глубиной вымени и признаками, характеризующими строение передних ног. SNP с меньшей статистической связью обнаружены на хромосомах 4-6-й, 10-18-й, 21-й, 23-й и 27-й. Однако доля влияния на общую дисперсию признака, связанная со значимыми SNP, была низкой и варьировала от 0,4 до 7,0 % для количества молока и от 0,1 до 13,8 % для показателей экстерьера, что подтверждает их полигенную природу (66). Аналогичное заключение было сделано C. Wasike с соавт. (67) по результатам GWAS, выполненного на молочных козах в США.
Методом GWAS был проведен поиск генов, ассоциированных с количеством соматических клеток (somatic cell count, SCC), выбранным в качестве признака устойчивости к маститу. Фенотипические данные включали SCC для 1941 альпийских и зааненских коз, разводимых во Франции. В зааненской породе достоверную связь с SCC показал SNP, идентифицированный на 19-й хромосоме в области длиной от 33 до 42 Mbp, которая включала гены-кандидаты, связанные с реакцией на инфекции, вызванные интрамаммарными штаммами, — ген рецептора ретиноевой кислоты α (RARA) и гены транскрипционных факторов STAT (STAT3, STAT5A, STAT5B). Однако для альпийской породы этих ассоциаций не обнаружили (68).
При изучении популяций коз в Восточной Европе выявили 10 генов, влияющих на устойчивость к маститу и желудочно-кишечным инфекциям. Это были гены пентраксина 3 ( PTX3 ), интерлейкина-6 ( IL6 ), члена семейства лектиновых доменов C-типа 4 ( CLEC4E ), интерлейкина-8 ( IL8 ), антагониста рецепторов интерлейкина-1 ( IL1RN ), субъединицы рецептора ин-терлейкина-15альфа ( IL15RA ), члена суперсемейства фактора некроза опухолей 13 ( TNFSF13 ), супрессора передачи сигналов цитокинов 3 ( SOCS3 ), фактора некроза опухолей ( TNF ) и толл-подобного рецептора 3 ( TLR3 ) (69).
В другом исследовании с использованием GWAS, выполненном во Франции, предприняли попытку выявить гены, ассоциированные с появлением у коз дополнительных долей и сосков на вымени. Выборка включали 810 зааненских и 1185 альпийских коз, однако достоверных связей между SNP и указанными признаками обнаружить не удалось (70).
S. Desire с соавт. (71) применили GWAS для оценки селекционной значимости (genomic breeding value estimator, GEBV) и выявления SNP, связанных с удоем молока и приростом живой массы. Фенотипические данные охватывали период одного года для 320 особей. Полученное значение точности GEBV оказалось низким — 0,28 для обоих показателей. Тем не менее авторы считают, что при увеличении числа животных, периода исследований и общего числа наблюдений точность геномной оценки повысится (71).
P. Martin с соавт. (72) секвенировали ген DGAT1 и выявили 29 полиморфизмов, из которых R251L и R396W не были ранее описаны и ассоциировались со снижением количества жира в молоке. Частота встречаемости мутации R396W у зааненских и альпийских коз составила соответственно 13,0 и 7,0 %, частота R251L для обеих пород — 3,5 % (72).
При использовании одношагового подхода в сочетании с геномным линейным несмещенным прогнозом (genomic best linear unbiased prediction, GBLUP), точность оценки племенной ценности коз альпийской и заанен-ской пород (825 особей), составляющих племенное ядро на фермах Франции, была увеличена с 22 до 37 % по сравнению с двухэтапным методом и оказалась выше традиционной оценки по родословной (73). В другой работе применили несколько оценок прогноза: на основе родословной (best linear unbiased prediction, BLUP), обычного невзвешенного анализа (single-step genomic best linear unbiased prediction, ssGBLUP) и трех взвешенных анализов (weighted single-step genomic best linear unbiased prediction, WssGBLUP; single-step genomic best linear unbiased prediction with the maximum weight of SNPs included in the chromosomal region, WssGBLUPMax; single-step genomic best linear unbiased prediction with the sum of the weights of the SNPs included in the chromosomal region, WssGBLUPSum), рассчитанных для SNP в соответствии с их влиянием на содержание белка в молоке. Точность GEBV с ssGBLUP повысилась на 5-7 % по сравнению с традиционной моделью BLUP. WssGBLUP с большей точностью выявлял SNP, связанные с содержанием αs1-казеина, и оказался более эффективным для прогноза геномных значений селекции, чем невзвешенный ssGBLUP. Кроме того, авторы ука- зывают, что при использовании WssGBLUP было несколько проще проводить вычисления, что ускоряло геномный анализ (74).
В исследовании, выполненном в Испании и включающем 50649 записей молочной продуктивности 19067 коз флоридской породы, было установлено, что метод ssGBLUP повышал среднюю точность оценок племенной ценности на 1,06 % по сравнению с классическим BLUP. Корреляция между матрицей A (родословная) и матрицей G (геном) составляла 0,826. Корреляция между EBV (breeding value estimator) и GEBV (genomic breeding value estimator) составила 0,989, но при сравнении только EBV-генотипиро-ванных животных корреляция между этими оценками снижалась до 0,952, а средняя точность увеличивалась на 5,86 % (75).
С целью уменьшения стоимости генотипирования при контроле происхождения А. Talenti с соавт. (76) на основании анализа 109 коз альпийской породы предложили две панели низкой плотности, включающие 130 и 114 SNP с вероятностью случайных совпадений соответственно 1,51½10 - 57 и 2,94½10 - 34. Полученные результаты позволили с абсолютной точностью определить родственные связи. В дальнейшем была создана улучшенная панель, содержащая 195 SNP. Показано, что при сопоставимой стоимости чип 195 SNP может заменить микросателлитные маркеры, но с гораздо большей точностью (77).
Цвет шерсти у коз — полигенный признак, который часто определяется эпистатическим взаимодействием генов (78). К ним относятся гены меланокортинового рецептора первого типа (мelanocortin 1 receptor, MC1R ) и его эндогенного антагониста — сигнального белка агути (аgouti signaling protein, ASIP ). Ген MC1R играет ключевую роль в синтезе меланиновых пигментов и контроле количества эумеланина (черный/коричневый) или фео-меланина (красный/желтый). Это продемонстрировано в нескольких исследованиях, посвященных изучению действия MC1R на окрас у крупного рогатого скота и овец (79). Сходным образом мутации в гене MC1R связаны с цветом шерсти у коз пород джиргентанской (Girgentana), мальтезской (Maltese), деривата ди сирия (Derivata di Siria), мурсиано гранадина (Murciano-Granadina), камосита дель альпи (Camosciata delle Alpi) и зааненской (Saanen) (80). ASIP оказывает эпистатическое действие на ген MC1R и снижает активность MC1R, что приводит к усилению синтеза феомеланина. Желтая, или феомеланиновая, пигментация обусловлена действием доминантного аллеля в локусе ASIP , в то время как черная/коричневая, или эумеланино-вая, — действием рецессивного аллеля (81). У зааненских коз доминирующий аллель Awt (белый/рыжий), по-видимому, ответствен за белый цвет шерсти (82). Дупликация участков в гене ASIP приводит к формированию белого и черного окраса (83). Другой ген, влияющий на цвет шерсти коз, — ген протоонкогенного рецептора тирозинкиназы (proto-oncogene receptor tyrosine kinase, KIT ), который считается одним из ключевых в формировании окраса у многих видов животных (80, 84-86).
X. Wang с соавт. (87), основываясь на генотипировании с помощью секвенирования, выполненном на козах из восьми популяций, сообщили о нескольких генах, находящихся под положительным давлением отбора. С окраской шерсти был ассоциирован ген ASIP, с шерстной продуктивностью — ген фактора роста фибробластов 5 (fibroblast growth factor 5, FGF5), с адаптацией к условиям высотной гипоксии — ген активатора НАДФН-оксидазы 1 (NOXA1) (87). В дальнейшем редактирование гена FGF5 у эмбрионов козы привело к увеличению числа вторичных волосяных фолликулов и длины волокна, что подтверждает положительную ассоциацию гена с кашемировой продуктивностью и целесообразность его включения в про- граммы селекции пуховых коз (88).
-
J. Guo с соавт. (89) выполнили полногеномное секвенирование 38 коз трех китайских пород наньцзян коричневый (Nanjiang Yellow), цзиньтан черный (Jintang Black) и тибетской кашемировой (Tibetan cashmere goats) и сопоставили с геномом 30 коз пяти других пород, а также с геномами 21 безоарового козла из базы данных AdaptMap. В результате в 5 ′ -UTR районе гена FGF5 был определен новый SNP (c.-253G>A), ассоциированный с пуховой продуктивностью и адаптацией к низким температурам у тибетских кашемировых коз. Также установлена большая частота встречаемости AGG -аллеля в 12-м экзоне гена десмоглеина 3 (desmoglein 3, DSG3 ), который обусловливает клеточную адгезию и экспрессируется главным образом в коже (89). Сравнение геномов кашемировых коз разных пород показало, что локусы, находящиеся под давлением отбора, связаны с окрасом ( IRF4, EXOC2, RALY, EIF2S2, KITLG ), воспроизводством ( KHDRBS2 ) и адаптацией к высоте местности ( EPAS1 ) (90). Давление отбора для гена эндотелиального белка 1, содержащего домен PAS (endothelial PAS domain-containing protein 1, EPAS1 ) было установлено в исследованиях S. Song с соавт. (90) при секвенировании экзома 330 тибетских кашемировых коз, хорошо адаптированных к содержанию в горной местности (90).
2. Участки гомозиготности
(runs of homozygosity, ROHs)
, встречающиеся в геноме у более 50 % карачаевских коз (
Capra hircus
)
Мы исследовали находящиеся под давлением отбора локусы в популяции аборигенных карачаевских коз ( n = 37), анализируя участки гомозиготности (runs of homozygosity, ROH). Всего были идентифицированы 17 участков ROH размеров более 0,1 Mb, которые встречались в геноме у более чем 50 % карачаевских коз (в том числе 6 участков ROH, выявленные более чем у 60 % животных) (табл. 2). Для подтверждения этих данных и выбора позиционных генов-кандидатов требуется исследовать большее поголовье коз карачаевской породы.
|
Хромосома |
Число SNP |
Позиция начальная конечная |
Длина, Mb |
Позиционные гены-кандидаты |
Таким образом, мировое козоводство представлено множеством пород и популяций коз разного направления продуктивности, подавляющее число которых относится к аборигенным. Биоразнообразие коз достаточно хорошо изучено с использованием микросателлитных локусов ДНК. В то же время для получения новых фундаментальных знаний о происхождении коз, дрейфа генов, генетических взаимоотношений между домашними козами и их дикими предками в последнее время широко используются полногеномный анализ и сканирование генома с использованием ДНК-чипов. Получены данные о связи генов CSN1S1 , CSN1S2 , CSN2 , BLG , RARA , STAT , PTX3 , IL6 , IL8 , DGAT1 с молочной продуктивностью коз и качеством молока. Продемонстрирована ассоциация генов MC1R , ASIP и KIT с цветом шерстных (пуховых) волокон, FGF5 , EPAS1 и NOXA1 — с шерстной продуктивностью и адаптацией к высотной гипоксии. В настоящее время достигнуты определенные успехи в понимании формирования биоразнообразия коз, перспективности геномного подхода в селекции шерстных и молочных пород. Однако локусы, связанные с экономически важными признаками коз (воспроизводство, пуховая и шерстная продуктивность, окраска шерстных волокон, количество молока и содержание в нем белка, жира, соматических клеток и других компонентов), а также ассоциированные с приспособительными качествами и устойчивостью к болезням, все еще мало изучены. В перспективе усилия должны быть сосредоточены на решении этих вопросов и поиске генов-кандидатов на основе геномных и омиксных технологий.
Список литературы Генетические маркеры в козоводстве (обзор)
- Skapetas B., Bampidis V. Goat production in the world: present situation and trends. Livestock Research for Rural Development, 2016, 28(11): 200
- Ерохин А.И., Карасев Е.А., Ерохин С.А. Динамика поголовья коз и производства козьего молока и мяса в мире и в России. Овцы, козы, шерстяное дело, 2020, 4: 22-25 (doi: 10.26897/2074-0840-2020-4-22-25).
- Скидан И.Н., Гуляев А.Е., Казначеев К.С. Жировые глобулы как детерминанты пищевой и биологической ценности козьего молока. Вопросы питания, 2015, 84(2): 81-95.
- Шувариков А.С., Канина К.А., Робкова Т.О., Юрова Е.А. Состав и свойства овечьего, козьего и коровьего молока. Фермер. Поволжье, 2019, 7(84): 92-93.
- Григорян Л.Н., Хататаев С.А., Сверчкова С.В. Состояние козоводства Российской Федерации и его племенной базы. В сб.: Ежегодник по племенной работе в овцеводстве и козоводстве в хозяйствах Российской Федерации (2005 год). М., 2006: 312-313.
- Дунин И.М., Амерханов Х.А., Сафина Г.Ф., Григорян Л.Н., Хататаев С.А., Хмелевская Г.Н. Козоводство России и его племенные ресурсы. Ежегодник по племенной работе в овцеводстве и козоводстве в хозяйствах Российской Федерации (2019 год). M., 2020: 323-325.
- Новопашина С.И., Санников М.Ю., Хататаев С.А., Кузьмина Т.Н., Хмелевская Г.Н., Степанова Н.Г., Тихомиров А.И., Маринченко Т.Е. Состояние и перспективные направления улучшения генетического потенциала мелкого рогатого скота: научный и аналитический обзор. М., 2019.
- Денискова Т.Е., Доцев А.В., Форнара М.С., Сермягин А.А., Reyer H., Wimmers K., Brem G., Зиновьева Н.А. Геномная архитектура российской популяции зааненских коз в аспекте генофонда породы из пяти стран мира. Сельскохозяйственная биология, 2020, 55(2): 285-294 (doi: 10.15389/agrobiology.2020.2.285rus).
- Кошкина О.А., Денискова Т.Е., Зиновьева Н.А. Вариация числа копий (CNV) как перспективный генетический маркер: распространение, методы валидации и гены-кандидаты в геномах сельскохозяйственных животных (обзор). Аграрная наука Евро-Северо-Востока, 2020, 21(4): 355-368 (doi: 10.30766/2072-9081.2020.21.4.355-368).
- Mekuriaw G., Gizaw S., Dessie T., Mwai O., Djikeng A., Tesfaye K. A review on current knowledge of genetic diversity of domestic goats (Capra hircus) identified by microsatellite loci: how those efforts are strong to support the breeding programs? Journal of Life Science and Biomedicine, 2016, 6(2): 22-32.
- Azhar P., Chakraborty D., Iqbal Z., Malik A., Ajaz qaudir, Asfar А., Bhat I.A. Microsatellite markers as a tool for characterization of small ruminants: a review. International Journal of Current Microbiology and Applied Sciences, 2018, 7(1): 1330-1342 (doi: 10.20546/ijcmas.2018.701.162).
- Wright S. The genetical structure of populations. Ann. Eugenics, 1951, 15: 323-354.
- Nei M. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences, 1973, 70(12): 3321-3323 (doi: 10.1073/pnas.70.12.3321).
- Nei M. Genetic distance between populations. The American Naturalist, 1972, 106(949): 283-392 (doi: 10.1086/282771).
- Wang Y., Wang J., Zi X.-D., Huatai C.-R., Ouyang X., Liu L.-S. Genetic diversity of Tibetan goats of Plateau type using microsatellite markers. Archives Animal Breeding, 2011, 54(2): 188-197 (doi: 10.5194/aab-54-188-2011).
- Di R., Farhad Vahidi S.M., Ma Y.H., He X.H., Zhao Q.J., Han J.L., Guan W.J., Chu M.X., Sun W., Pu Y.P. Microsatellite analysis revealed genetic diversity and population structure among Chinese cashmere goats. Animal Genetics, 2011, 42(4): 428-431 (doi: 10.1111/j.1365-2052.2010.02072.x).
- Харзинова В.Р., Петров С.Н., Доцев А.В., Безбородова Н.А., Зиновьева Н.А. Популяционно-генетическая характеристика некоторых пород коз на основании анализа микроса-теллитов. Овцы, козы, шерстяное дело, 2019, 3: 7-12.
- Selionova M.I., Aibazov M.M., Mamontova T.V., Petrov S.N., Kharzinova V.R., Dotsev A.V. Zinovieva N. A. Genetic differentiation of Russian goats and wild relatives based on microsatellite loci. Journal of Animal Science, 2020, 98(4): 19-20 (doi: 10.1093/jas/skaa278.037).
- Бекетов С.В., Пискунов А.К., Воронкова В.Н., Петров С.Н., Харзинова В.Р., Доцев А.В., Зиновьева Н.А., Селионова М.И., Столповский Ю.А. Генетическое разнообразие и филогения пуховых коз Центральной и Средней Азии. Генетика, 2021, 57(7): 810-819 (doi: 10.31857/S0016675821070031).
- Wang G.Z., Chen S.S., Chao T.L., Ji Z.B., Hou L., Qin Z.J., Wang J.M. Analysis of genetic diversity of Chinese dairy goats via microsatellite markers. Journal of Animal Science, 2017, 95(5): 2304-2313 (doi: 10.2527/jas.2016.1029).
- Araújo A.M., Guimarães S.E.F., Machado T.M.M., Lopes P.S., Pereira C.S., Silva F.L.R., Ro-drigues M.T., Columbiano V.S., da Fonseca C.G. Genetic diversity between herds of Alpine and Saanen dairy goats and the naturalized Brazilian Moxotó breed. Genetics and Molecular Biology, 2006, 29(1): 67-74 (doi: 10.1590/S1415-47572006000100014).
- Seilsuth S., Seo J.H., Kong H.S., Jeon G.J. Microsatellite analysis of the genetic diversity and population structure in dairy goats in Thailand. Anim. Biosci., 2016, 29(3): 327-332 (doi: 10.5713/ajas.15.0270).
- Mastrangelo S., Tolone M., Montalbano M., Tortorici L., Gerlando R., Sardina M.T., Porto-lano B. Population genetic structure and milk production traits in Girgentana goat breed. Animal Production Science, 2016, 57(3): 430-440 (doi: 10.1071/AN15431).
- Sardina M., Tortorici L., Mastrangelo S., Di Gerlando R., Tolone M., Portolano B. Application of microsatellite markers as potential tools for traceability of Girgentana goat breed dairy prod-ucts. Food Research International, 2015, 74: 115-122 (doi: 10.1016/j.foodres.2015.04.038).
- Wei C., Lu J., Xu L., Liu G., Wang Z., Zhang L., Zhao F., Han X., Du L., Liu C. Genetic structure of Chinese indigenous goats and the special geographical structure in the Southwest China as a geographic barrier driving the fragmentation of a large population. PLoS ONE, 2014, 9(4): e94435 (doi: 10.1371/journal.pone.0094435).
- Dixit S.P., Verma N.K., Aggarwal R.A.K., Vyas M.K., Rana J., Sharma A., Tyagi P., Arya P., Ulmek B.R. Genetic diversity and relationship among Indian goat breeds based on microsatellite markers. Small Ruminant Research, 2010, 91(2): 153-159 (doi: 10.1016/j.smallrum-res.2010.02.015).
- Nomura K., Ishii K., Dadi H., Takahashi Y., Minezawa M., Cho C.Y., Sutopo, Faruque M.O., Nyamsamba D., Amano T. Microsatellite DNA markers indicate three genetic lineages in East Asian indigenous goat populations. Animal Genetics, 2012, 43(6): 760-767 (doi: 10.1111/j.1365-2052.2012.02334.x).
- Cañón J., García D., García-Atance M.A., Obexer-Ruff G., Lenstra J.A., Ajmone-Marsan P., Dunner S., The ECONOGENE Consortium. Geographical partitioning of goat diversity in Eu-rope and the Middle East. Animal Genetics, 2006, 37(4): 327-334 (doi: 10.1111/j.1365-2052.2006.01461.x).
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evo-lution, 1984, 38(6): 1358-1370 (doi: 10.1111/j.1558-5646.1984.tb05657.x).
- Kalinowski S.T. Counting alleles with rarefaction: private alleles and hierarchical sampling de-signs. Conservation Genetics, 2004, 5(4): 539-543 (doi: 10.1023/B:COGE.0000041021.91777.1a).
- Nicoloso L., Bomba L., Colli L., Negrini R., Milanesi M., Mazza R., Sechi T., Frattini S., Talenti A., Coizet B., Chessa S., Marletta D., D'Andrea M., Bordonaro S., Ptak G., Carta A., Pagnacco G., Valentini A., Pilla F., Ajmone-Marsan P., Crepaldi P., the Italian Goat Consor-tium. Genetic diversity of Italian goat breeds assessed with a medium-density SNP chip. Genetics, Selection, Evolution, 2015, 47(1): 62 (doi: 10.1186/s12711-015-0140-6).
- Mdladla K., Dzomba E.F., Huson H.J., Muchadeyi F.C. Population genomic structure and link-age disequilibrium analysis of South African goat breeds using genome-wide SNP data. Animal Genetics, 2016, 47(4): 471-482 (doi: 10.1111/age.12442).
- Brito L.F., Kijas J.W., Ventura R.V., Sargolzaei M., Porto-Neto L.R., Cánovas A., Feng Z., Jafarikia M., Schenkel F.S. Genetic diversity and signatures of selection in various goat breeds revealed by genome-wide SNP markers. BMC Genomics, 2017, 18: 229 (doi: 10.1186/s12864-017-3610-0).
- Zhang B., Chang L., Lan Y.X., Nadeem A., Guan F.L., Fu K.D., Li B., Yan X.C., Zhang B.H., Zhang Y.X., Huang Z.A., Chen H., Yu J., Li B.S. Genome-wide definition of selective sweeps reveals molecular evidence of trait-driven domestication among elite goat (Capra species) breeds for the production of dairy, cashmere, and meat. GigaScience, 2018, 7(12): giy105 (doi: 10.1093/gigascience/giy105).
- Zonaed Siddiki A.M.A.M., Miah G., Islam M.S., Kumkum M., Rumi M.H., Baten A., Hoss-ain M.A. Goat genomic resources: the search for genes associated with its economic traits. Inter-national Journal of Genomics, 2020, 2020(1): 5940205 (doi: 10.1155/2020/5940205).
- Хаертдинов Р.Р., Гафиатуллин Ф.И., Афанасьев М.П. Особенности белкового состава молока у основных видов сельскохозяйственных животных. Сельскохозяйственная биология, 2011, 2: 81-85.
- Ворожко И.В., Скидан И.Н., Черняк О.О., Гуляев А.Е. Современные тренды изучения полиморфизма генов, кодирующих белки козьего молока. Вопросы питания, 2016, 85 (5): 13-21.
- Barillet F. Genetic improvement for dairy production in sheep and goats. Small Ruminant Re-search, 2007, 70(1): 60-75 (doi: 10.1016/j.smallrumres.2007.01.004).
- Dodds K.G., McEwan J.C., Davis G.H. Integration of molecular and quantitative information in sheep and goat industry breeding programmes. Small Ruminant Research, 2007, 70(1): 32-41 (doi: 10.1016/j.smallrumres.2007.01.010).
- Marletta D., Criscione А., Bordonaro S., Guastella А.М., D’Urso G. Casein polymorphism in goat’s milk. Lait, 2008, 87(6): 491-504 (doi: 10.1051/lait:2007034).
- Devold T.G., Nordbø R., Langsrud T., Svenning C., Brovold M.J., Sørensen E.S., Christensen B., Ådnøy T., Vegarud G.E. Extreme frequencies of the αs1-casein ‘null’ variant in milk from Nor-wegian dairy goats – implications for milk composition, micellar size and renneting properties. Dairy Science and Technology, 2010, 91(1): 39-51 (doi: 10.1051/DST/2010033).
- Ollier S., Chauvet S., Martin P., Chilliard Y., Leroux C. Goat’s αs1-casein polymorphism affects gene expression profile of lactating mammary gland. Animal, 2008, 2(4): 566-573 (doi: 10.1017/S1751731108001584).
- Jordana J., Amills M., Diaz E., Angulo C., Serradilla J.M., Sanchez A. Gene frequencies of caprine αs1-casein polymorphism in Spanish goat breeds. Small Ruminant Research, 1996, 20(3): 215-221 (doi: 10.1016/0921-4488(95)00813-6).
- Enne G., Feligini M., Greppi G.F., Iametti S., Pagani S. Gene frequencies of caprine αs1-casein polymorphism in dairy goats, IDF Seminar «Milk Protein Polymorphism II». Palmerston North, 1997: 275-279.
- Küpper J., Chessa S., Rignanese D., Caroli A., Erhardt G. Divergence at the casein haplotypes in dairy and meat goat breeds. Journal Dairy Research, 2010, 77(1): 56-62 (doi: 10.1017/S0022029909990343).
- Chilliard Y., Rouel J., Leroux C. Goat’s alpha-s1 casein genotype influences its milk fatty acid composition and delta-9 desaturation ratios. Animal Feed Science and Technology, 2006, 131(3-4): 474-487 (doi: 10.1016/j.anifeedsci.2006.05.025).
- Silanikove N., Leitner G., Merin U., Prosser C.G. Recent advances in exploiting goat’s milk: quality, safety and production aspects. Small Ruminant Research, 2010, 89(2): 110-124 (doi: 10.1016/j.smallrumres.2009.12.033).
- Wang K., Hailong Y., Xu H., Yang Q., Zhang S., Pan C., Chen H., Zhu H., Liu J., Qu L., Lan X. A novel indel within goat casein alpha S1 gene is significantly associated with litter size. Gene, 2018, 671: 161-169 (doi: 10.1016/j.gene.2018.05.119).
- Шувариков А.С., Пастух О.Н., Жукова Е.В., Жижин Н.А. Оценка молочной продуктивности и качества молока коз в зависимости от породы и генотипа по гену BLG (бета-лактоглобу-лина). Известия ТСХА, 2019, 3: 130-148 (doi: 10.34677/0021-342X-2019-3-130-148).
- Фатихов А.Г., Хаертдинов Р.А., Камалдинов И.Н. Белковый состав и технологические свойства молока у зааненских коз в зависимости от их генотипа по бета-лактоглобулину. Молочнохозяйственный вестник, 2017, 1(25): 64-69.
- Кравцова О.А., Спиридонова С.В., Фаизов Т.Х. Способ генетического отбора молочных коз. Патент RU 2620977. Заявка № 2015140586 от 24.09.2015. Опубл. 30.05.2017 г. Бюл. № 16.
- Гончаренко Г.М., Гришина Н.Б., Хорошилова Т.С., Романчук И.В., Каргачакова Т.Б., Подкорытов Н.А. Влияние групп крови, генов CAST, BLG на продуктивность овец и коз Республики Алтай. Сибирский вестник сельскохозяйственной науки, 2018, 48(4): 63-71 (doi: 10.26898/0370-8799-2018-4-9).
- Visscher P.M., Wray N.R., Zhang Q., Sklar P., McCarthy M.I., Brown M.A., Yang J. 10 Years of GWAS discovery: biology, function, and translation. American Journal of Human Genetics, 2017, 101(1): 5-22 (doi: 10.1016/j.ajhg.2017.06.005).
- Meuwissen T., Hayes B., Goddard M. Genomic selection: a paradigm shift in animal breeding. Animai Frontiers, 2016, 6(1): 6-14 (doi: 10.2527/af.2016-0002).
- Ibtisham F. Zhang L., Xiao M., An L., Ramzan M.B., Nawab A., Zhao Y., Li G., Xu Y. Genomic selection and its application in animal breeding. Thai Journal of Veterinary Medicine, 2017, 47(3): 301-310.
- Stella A., Nicolazzi E.L., Tassell C., Rothschild M., Colli L., Rosen B., Sonstegard T., Crepaldi P., Tosser-Klopp G., Joost S., the AdaptMap Consortium. AdaptMap: exploring goat diversity and adaptation. Genetics, Selection, Evolution, 2018, 50: 61 (doi: 10.1186/s12711-018-0427-5).
- Tosser-Klopp G., Bardou F., Bouchez O., Cabau C., Crooijmans R., Dong Y., Donnadieu-Tonon C., Eggen A., Heuven H.C.M., Jamli S., Jiken A.J., Klopp C., Lawley C.T., McEwan J., Martin P., Moreno C.R., Mulsant P., Nabihoudine I., Pailhoux E., Palhiere I., Rupp R., Sarry J., Sayre B.L., Tircazes A., Wang J., Wang W., Zhang W., the International Goat Genome Consor-tium. Design and characterization of a 52K SNP chip for goats. PLoS ONE, 2014, 9(1): e86227 (doi: 10.1371/journal.pone.0086227).
- Rubin C.J., Megens H.-J., Barrio A.M., Maqboo K., Sayyab S., Schwochow D., Wang C., Carl-borgd Ö., Jerna P., Jørgensene C.B., Archibald A.L., Fredholm M., Groenen M.A.M., Anders-son L. Strong signatures of selection in the domestic pig genome. Proceedings of the National Academy of Sciences, 2012, 109(48): 19529-19536 (doi: 10.1073/pnas.1217149109).
- Rubin C.-J., Zody M.C., Eriksson J., Meadows J.R.S., Sherwood E., Webster M.T., Jiang L., Ingman M., Sharpe T., Ka S., Hallböök F., Besnier F., Carlborg O., Bed’hom B., Tixier-Boich-ard M., Jensen P., Siegel P., Lindblad-Toh K., Andersson L. Whole-genome resequencing reveals loci under selection during chicken domestication. Nature, 2010, 464(7288): 587-591 (doi: 10.1038/nature08832).
- Kemper K.E., Saxton S.J., Bolormaa S., Hayes B. J., Goddard M.E. Selection for complex traits leaves little or no classic signatures of selection. BMC Genomics, 2014, 15: 246 (doi: 10.1186/1471-2164-15-246).
- Zhao F., McParland S., Kearney F., Du L., Berry D.P. Detection of selection signatures in dairy and beef cattle using high-density genomic information. Genetics, Selection, Evolution, 2015, 47: 49 (doi: 10.1186/s12711-015-0127-3).
- Xu L., Bickhart D.M., Cole J.B., Schroeder S.G., Song J., Tassell C.P., Sonstegard T.S., Liu G.E. Genomic signatures reveal new evidences for selection of important traits in domestic cattle. Molecular Biology and Evolution, 2015, 32(3): 711-725 (doi: 10.1093/molbev/msu333).
- Kijas J.W., Lenstra J.A., Hayes B., Boitard S., Neto L.P., Cristobal M.S., Servin B., McCul-loch R., Whan V., Gietzen K., Paiva S., Barendse W., Ciani E., Raadsma H., McEwan J., Dal-rymple B. other members of the International Sheep Genomics Consortium. Genome-wide anal-ysis of the world’s sheep breeds reveals high levels of historic mixture and strong recent selection. PLoS Biology, 2012, 10(2): e1001258 (doi: 10.1371/journal.pbio.1001258).
- Kim E.-S., Elbeltagy A.R., Aboul-Naga A.M., Rischkowsky B., Sayre B., Mwacharo J.M., Roth-schild M.F. Multiple genomic signatures of selection in goats and sheep indigenous to a hot arid environment. Heredity, 2015, 116(3): 255-264 (doi: 10.1038/hdy.2015.94).
- Lachance J., Tishkoff S.A. SNP ascertainment bias in population genetic analyses: Why it is important, and how to correct it. BioEssays, 2013, 35(9): 780-786 (doi: 10.1002/bies.201300014).
- Mucha S., Mrode R., Coffey M., Kizilaslan M., Desire S., Conington J. Genome-wide associa-tion study of conformation and milk yield in mixed-breed dairy goats. Journal Dairy Science, 2018, 101(3): 2213-2225 (doi: 10.3168/jds.2017-12919).
- Wasike C.B., Rolf M., Silva N.C.D., Puchala R., Sahlu T., Goetsch A.L., Gipson T.A. 1683 Genome-wide association analysis of residual feed intake and milk yield in dairy goats. Journal of Animal Science, 2016, 94(5): 820 (doi: 10.2527/jam2016-1683).
- Martin P., Palhière I., Maroteau C., Clément V., David I., Tosser Klopp G., Rupp R. Genome-wide association mapping for type and mammary health traits in French dairy goats identifies a pleiotropic region on chromosome 19 in the Saanen breed. Journal Dairy Science, 2018, 101(6): 5214-5226 (doi: 10.3168/jds.2017-13625).
- Ilie D.E., Kusza S., Sauer M., Gavojdian D. Genetic characterization of indigenous goat breeds in Romania and Hungary with a special focus on genetic resistance to mastitis and gastrointestinal parasitism based on 40 SNPs. PLoS ONE, 2018, 13(5): e0197051 (doi: 10.1371/jour-nal.pone.0197051).
- Martin P., Palhière I., Tosser-Klopp G., Rupp R. Heritability and genome-wide association map-ping for supernumerary teats in French Alpine and Saanen dairy goats. Journal Dairy Science, 2016, 99(11): 8891-8900 (doi: 10.3168/jds.2016-11210).
- Desire S., Mucha S., Coffey M., Mrode R., Broadbent J., Conington J. Deriving genomic breed-ing values for feed intake and body weight in dairy goats. Proceedings of the World Congress on Genetics Applied to Livestock Production, 2016, 11: 818.
- Martin P., Palhière I., Maroteau C., Bardou P., Canale-Tabet K., Sarry J., Woloszyn F., Bertrand-Michel J., Racke I., Besir H., Rupp R., Tosser-Klopp G. A genome scan for milk produc-tion traits in dairy goats reveals two new mutations in DGAT1 reducing milk fat content. Scientific Reports, 2017, 7: 1872 (doi: 10.1038/s41598-017-02052-0).
- Cérillier C., Larroque H., Robert-Granié C. Comparison of joint versus purebred genomic eval-uation in the French multi-breed dairy goat population. Genetics, Selection, Evolution, 2014, 46: 67 (doi: 10.1186/s12711-014-0067-3).
- Teissier M., Larroque H., Robert-Granié C. Weighted single-step genomic BLUP improves accuracy of genomic breeding values for protein content in French dairy goats: A quantitative trait influenced by a major gene. Genetics, Selection, Evolution, 2018, 50: 31 (doi: 10.1186/s12711-018-0400-3).
- Molina A., Muñoz E., Díaz C., Menéndez-Buxadera A., Ramón M., Sánchez M., Carabaño M.J., Serradilla J.M. Goat genomic selection: impact of the integration of genomic information in the genetic evaluations of the Spanish Florida goats. Small Ruminant Research, 2018, 163: 72-75 (doi: 10.1016/j.smallrumres.2017.12.010).
- Talenti A., Nicolazzi E.L., Chessa S., Frattini S., Moretti R., Coizet B., Nicoloso L., Colli L., Pagnacco G., Stella A., Ajmone-Marsan P., Ptak G., Crepaldi P. A method for single nucleotide polymorphism selection for parentage assessment in goats. Journal Dairy Science, 2016, 99(5): 3646-3653 (doi: 10.3168/jds.2015-10077).
- Talenti A., Palhière I., Tortereau F., Pagnacco G., Stella A., Nicolazzi E.L., Crepaldi P., Tosser-Klopp G. AdaptMap Consortium. Functional SNP panel for parentage assessment and assignment in worldwide goat breeds. Genetics, Selection, Evolution, 2018, 50: 55 (doi: 10.1186/s12711-018-0423-9).
- Sturm R.A., Teasdale R.D., Box N.F. Human pigmentation genes: identification, structure and consequences of polymorphic variation. Gene, 2001, 277(1-2): 49-62 (doi: 10.1016/S0378-1119(01)00694-1).
- Switonski M., Mankowska M., Salamon S. Family of melanocortin receptor (MCR) genes in mammals — mutations, polymorphisms and phenotypic effects. Journal Applied Genetics, 2013, 54: 461-472 (doi: 10.1007/s13353-013-0163-z).
- Fontanesi L., Beretti F., Riggio V., Dall’Olio S., González E.G., Finocchiaro R., Davoli R., Russo V., Portolano B. Missense and nonsense mutations in melanocortin 1 receptor (MC1R) gene of different goat breeds: association with red and black coat colour phenotypes but with unexpected evidences. BMC Genetics, 2009, 10: 47 (doi: 10.1186/1471-2156-10-47).
- Adalsteinsson S., Sponenberg D.P., Alexieva S., Russel A.J.F. Inheritance of goat coat colors. Journal of Heredity, 1994, 85(4): 267-272 (doi: 10.1093/oxfordjournals.jhered.a111454).
- Martin P.M., Palhière I., Ricard A., Tosser-Klopp G., Rupp R. Genome wide association study identifies new loci associated with undesired coat color phenotypes in Saanen goats. PLoS ONE, 2016, 11(3): e0152426 (doi: 10.1371/journal.pone.0152426).
- Norris B.J., Whan V.A. A gene duplication affecting expression of the ovine ASIP gene is responsible for white and black sheep. Genome Research, 2008, 18(8): 1282-1293 (doi: 10.1101/gr.072090.107).
- David V.A., Menotti-Raymond M., Wallace A.C., Roelke M., Kehler J., Leighty R., Eizirik E., Hannah S.S., Nelson G., Schäffer A.A., Connelly C.J., O'Brien S.J., Ryugo D.K. Endogenous retrovirus insertion in the KIT oncogene determines white and white spotting in domestic cats. G3 Genes|Genomes|Genetics, 2014, 4(10): 1881-1891 (doi: 10.1534/g3.114.013425).
- Dürig N., Jude R., Holl H., Brooks S.A., Lafayette C., Jagannathan V., Leeb T. Whole genome sequencing reveals a novel deletion variant in the KIT gene in horses with white spotted coat colour phenotypes. Animal Genetics, 2017, 48(4): 483-485 (doi: 10.1111/age.12556).
- Holl H., Isaza R., Mohamoud Y., Ahmed A., Almathen F., Youcef C., Gaouar S.B.S., Antczak D.F., Brooks S.A. A frameshift mutation in KIT is associated with white spotting in the Arabian camel. Genes, 2017, 8(3): 102 (doi: 10.3390/genes8030102).
- Wang X., Liu J., Zhou G., Guo J., Yan H., Niu Y., Li Y., Yuan C., Geng R., Lan X., An X., Tian X., Zhou H., Song J., Jiang Y., Chen Y. Whole-genome sequencing of eight goat populations for the detection of selection signatures underlying production and adaptive traits. Scientific Re-ports, 2016, 6: 38932 (doi: 10.1038/srep38932).
- Wang X., Cai B., Zhou J., Zhu H., Niu Y., Ma B., Yu H., Lei A., Yan H., Shen Y., Shi L., Zhao X., Hua J., Huang X., Qu L., Chen Y. Disruption of FGF5 in cashmere goats using CRISPR/Cas9 results in more secondary hair follicles and longer fibers. PLoS ONE, 2016, 11(10): e0164640 (doi: 10.1371/journal.pone.0164640).
- Guo J., Tao H., Li P., Li L., Zhong T., Wang L., Ma J., Chen X., Song T., Zhang H. Whole-genome sequencing reveals selection signatures associated with important traits in six goats’ breeds. Scientific Reports, 2018, 8: 10405 (doi: 10.1038/s41598-018-28719-w).
- Song S., Yao N., Yang M., Liu X., Dong K., Zhao Q., Pu Y., He X., Guan W., Yang N., Ma Y., Jiang L. Exome sequencing reveals genetic differentiation due to high-altitude adaptation in the Tibetan cashmere goat (Capra hircus). BMC Genomics, 2016, 17: 122 (doi: 10.1186/s12864-016-2449-0).


