Геномная архитектура российской популяции зааненских коз в аспекте генофонда породы из пяти стран мира
Автор: Денискова Т.Е., Доцев А.В., Форнара М.С., Сермягин А.А., Reyer H., Wimmers K., Brem G., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Современные достижения и проблемы генетики и биотехнологии в животноводстве
Статья в выпуске: 2 т.55, 2020 года.
Бесплатный доступ
Зааненская порода коз ценится за высокую молочную продуктивность и хорошие адаптационные качества, которые способствовали ее широкому распространению в мире за пределами Швейцарии. В России зааненская порода официально рекомендована к разведению и имеет племенной статус. Разведение в локальных условиях окружающей среды, а также региональные особенности используемых селекционных стратегий могут приводить к существенному изменению аллелофонда пород, в связи с чем актуально проведение геномных исследований национальных популяций мировых пород с целью установления их современного генетического статуса. В настоящей работе впервые представлены данные полногеномного анализа российской популяции коз зааненской породы, для которой была проведена сравнительная оценка с оригинальным (Швейцария) и мировым генофондом зааненской породы, представленным четырьмя странами. Нашей целью было рассмотрение генетического разнообразия и установление структуры российской популяции зааненской породы в аспекте генофонда коз этой породы из пяти различных стран (Швейцария, Франция, Италия, Аргентина и Танзания), полногеномные SNP-профили которых были получены из базы данных проекта AdaptMap. Исследования проводили на козах ( Capra hircus ) зааненской породы ( n = 21, RUS), разводимых в одном из племенных репродукторов на территории Российской Федерации, в 2019-2020 годах. ДНК выделяли из отобранных фрагментов ушной раковины с помощью наборов ДНК-Экстран-2 (ЗАО «Синтол», Россия). Генотипирование осуществляли с использованием ДНК-чипа GoatSNP50 BeadChip («Illumina, Inc.», США), содержащего 53347 SNPs и обеспечивающего покрытие среднего интервала между SNP в размере 40 кб. Для оценки генетического разнообразия и сравнительного анализа российской популяции коз с генофондом коз этой породы из пяти различных стран использовали SNP-профили зааненских коз, разводимых в Швейцарии (SWI, n = 38), Италии (ITA, n = 22), Франции (FRA, n = 55), Аргентине (ARG, n = 11) и Танзании (TNZ, n = 8), которые были получены из публично доступного хранилища цифровых данных Dryad и сгенерированы в рамках проекта AdaptMap. В качестве образца оригинального генофонда была выбрана швейцарская популяция зааненской породы. Биоинформационную обработку и визуализацию данных полногеномного генотипирования проводили в программах PLINK 1.90, Admixture 1.3, SplitsTree 4.14.5, в пакетах R «diveRsity» и «pophelper». Наблюдаемая гетерозиготность варьировала от 0,381 у SWI до 0,423 у FRA и была высокой у RUS (Ho = 0,418). В популяциях SWI, ITA, FRA значения коэффициента инбридинга оказались близки к нулю; у RUS, ARG и TNZ был отмечен дефицит гетерозигот - соответственно 1,5; 8,9 и 6,0 %. Аллельное разнообразие было максимальным у ARG, RUS и FRA (Ar ³ 1,979) и минимальным - у SWI (Ar = 1,934). Анализ главных компонент и структура филогенетического дерева показали четкую дифференциацию между национальными и оригинальной популяциями зааненской породы. Анализ популяционной структуры подтвердил сохранение генетической составляющей кластера SWI у коз группы RUS. У RUS наблюдались наименьшие генетические дистанции с FRA (FST = 0,02; RST = 0,189) и ITA (FST = 0,023; RST = 0,215); наибольшая дифференциация RUS была выявлена с TNZ (FST = 0,054; RST = 0,311) и SWI (FST = 0,06; RST = 0,276). Таким образом, различные стратегии селекции привели к генетическим различиям между национальными популяциями коз зааненской породы, при этом российская популяция коз сохраняет в себе геномные компоненты исходного генофонда.
Зааненская порода, домашние козы, snp-маркеры, днк-чипы, генетическое разнообразие
Короткий адрес: https://sciup.org/142226295
IDR: 142226295 | УДК: 636.393.9:575.174.5:577.21 | DOI: 10.15389/agrobiology.2020.2.285rus
Текст научной статьи Геномная архитектура российской популяции зааненских коз в аспекте генофонда породы из пяти стран мира
За историю существования домашнего козоводства в мире было создано более 50 пород коз молочного направления продуктивности (1), среди которых наиболее популярна зааненская порода. Свое название она получила от швейцарской долины Зааненталь, где была выведена методом длительной народной селекции в середине XIX века (2, 3). Среди коз заа-
При проведении исследований использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФНЦ животноводства — ВИЖ им. академика Л.К. Эрнста. Исследование проведено при поддержке РФФИ, проект ¹ 18-316-20006.
ненская порода — своеобразный аналог голштинской породы коров, характеризующийся высокой молочной продуктивностью (от 300 до 2000 кг за 150-300 сут лактации) (3). Козы этой породы легко акклиматизируются к различным условиям содержания и кормления без существенной потери молочности. Они были импортированы из Швейцарии в большинство стран Европы (3, 4), Северной (5) и Южной Америки (6) Австралии (7), Азии (8) и Африки (9), где использование их ценного генофонда значительно преобразовало национальную молочную индустрию.
Согласно официальным данным (10), племенное молочное козоводство в России представлено четырьмя иностранными породами: альпийская (с 2015 года), зааненская (с 1993 года), мурсиано гранадина (Murciano-Granadina) (с 2019 года) и нубиан (Nubian) (с 2018 года). Заа-ненская порода, по данным на 2018 год, была наиболее распространена (11). Племенное поголовье коз зааненской породы было сосредоточено на трех племенных заводах, в шести племенных репродукторах и одном ге-нофондном хозяйстве и составляло 12,3 тыс. гол. Удой по стаду варьировал от 822 кг за 305 сут лактации в племрепродукторе ООО «КХ «Русь-1» (Ставропольский край, Буденновский р-н, с. Покойное) до 961 кг за 305 сут лактации в племрепродукторе ООО «Березка» (Курская обл., Курский р-н, д. Петровское) (4).
Помимо использования чистопородных зааненских коз для производства молочной продукции, перспективно их скрещивание с местными популяциями коз (1, 12). Так, козлы-производители зааненской породы — высококлассный улучшающий материал, позволяющий за короткие сроки преобразовать беспородные низкопродуктивные группы коз (с удоем 200250 кг за 305 сут лактации) в надежных продуцентов молока с удоем до 663 кг за 305 сут лактации (1, 12).
Геномные исследования домашних коз несколько отставали от других видов сельскохозяйственных животных (13). Только в 2013 году три крупные научные группы, ведущие проекты по поиску SNPs (single nucleotide polymorphism), объединились в Международный консорциум по геномике коз (International Goat Genome Consortium, IGGC) с целью создания ДНК-чипа (14). В результате отбора 53347 SNPs вошли в финальный состав чипа Goat SNP50 BeadChip («Illumina, Inc.», США).
Разработка ДНК-чипа обусловила повышенный интерес к исследованиям особенностей организации генома домашних коз. Например, L. Nicoloso с соавт. (15) исследовали генетическое разнообразие 14 итальянских пород коз на основе данных полногеномного сканирования. K. Mdladla с соавт. (16) продемонстрировали прикладную значимость полногеномного SNP анализа в исследованиях аборигенных пород коз ЮАР. Применив Goat SNP50 BeadChip, S.A. Rahmatalla с соавт. (17) идентифицировали гены, ассоциированные с ростом, развитием костей и формированием иммунной системы у суданских коз. L.F. Brito с соавт. (18) изучили генетическое разнообразие у более чем 1000 гол. из девяти популярных коммерческих пород коз.
Многочисленные исследования привели к генерации SNP-профилей пород коз разных стран во всем мире (3171 коз из 117 популяций), собранных в базу данных в рамках проекта AdaptMap (19, 20), которая была использована для выявления регионов гомозиготности (20), установления исторических путей миграции коз (21) и поиска локусов, находящихся под давлением селекции (22). Кроме того, создание публично доступных баз данных SNP-профилей позволяет изучать породы, импортированные в различные страны, с целью оценки их расхождения или сходства с ориги-286
нальным генофондом.
В настоящей работе впервые представлены данные полногеномного анализа российской популяции коз зааненской породы, для которой была проведена сравнительная оценка с оригинальным (Швейцария) и мировым генофондом зааненской породы, представленным четырьмя странами.
Нашей целью было рассмотрение генетического разнообразия и установление структуры российской популяции зааненской породы в аспекте генофонда коз этой породы из пяти различных стран (Швейцария, Франция, Италия, Аргентина и Танзания), полногеномные SNP-профили которых были получены из базы данных проекта AdaptMap.
Методика . Исследования проводили на козах ( Capra hircus ) заанен-ской породы ( n = 21, RUS), разводимых в одном из племенных репродукторов на территории Российской Федерации, в 2019-2020 годах. ДНК выделяли из отобранных фрагментов ушной раковины с помощью наборов ДНК-Экстран-2 (ЗАО «Синтол», Россия). Генотипирование осуществляли с использованием ДНК-чипа GoatSNP50 BeadChip («Illumina, Inc.», США), содержащего 53347 SNPs и обеспечивающего покрытие среднего интервала между SNP в размере 40 kb (14). Чтение ДНК-чипов проводили на приборе iScan Reader («Illumina, Inc.», США), по завершении необработанные данные загружали в программу GenomeStudio 2.0 («Il-lumina, Inc.», США) для предварительного анализа. Для параметров, характеризующих качество чтения (GenCall, GC) и кластеризацию SNP-маркеров (GenTrain, GT), были установлены предельные допустимые значения 0,5 (23). В программе PLINK 1.90 (24) для использования в последующих расчетах отбирали SNP-маркеры, имеющие частоту минорного аллеля (MAF) более 5 %, не отклоняющиеся от равновесия Харди-Вайнберга при p < 10-6, находящиеся в равновесии по сцеплению и расположенные только на аутосомах.
Для оценки генетического разнообразия и сравнительного анализа российской популяции коз с мировым генофондом использовали SNP-профили зааненских коз, разводимых в Швейцарии (SWI, n = 38), Италии (ITA, n = 22), Франции (FRA, n = 55), Аргентине (ARG, n = 11) и Танзании (TNZ, n = 8), которые были получены из публично доступного хранилища цифровых данных Dryad (25) и сгенерированы в рамках проекта AdaptMap (20, 21). В качестве образца оригинального генофонда была выбрана швейцарская популяция зааненской породы.
Наблюдаемая (Ho) гетерозиготность, несмещенная ожидаемая гетерозиготность (uHe), аллельное разнообразие (Аr), коэффициент инбридинга Fis (c доверительным интервалом 95 %), попарные значения показателей FST (26) и RST (дистанция Рейнольдса) (27) были рассчитаны в R пакете diveRsity (28). Анализ главных компонент (principal component analysis, PCA) выполняли в программе PLINK 1.9 с последующим построением графика в R пакете ggplot2 (29). Матрица попарных значений FST была визуализирована в виде групповой генетической сети Neighbor Net в программе SplitsTree 4.
Популяционная структура и генетическая однородность российской и других групп зааненских коз были установлены в программе Admixture 1.3 (31) с графическим представлением с помощью R пакета pophelper (32). 14.5 (30). Наиболее вероятное число предковых кластеров (К) определяли с помощью расчета значений ошибки кросс-валидации (CV error) для K от 1 до 7 в программе Admixture 1.3.
Биоинформационную обработку данных и работу с рисунками
(графиками) проводили с помощью программной среды R Project for Statistical Computing (33).
Результаты. Наблюдаемая гетерозитность варьировала от 0,381 у SWI до 0,423 у FRA (табл. 1). Значение показателя Ho у RUS (0,418) было ранжировано вторым вслед за максимальным. У трех популяций (SWI, ITA, FRA) коэффициент инбридинга оказался несущественен, что, вероятно, свидетельствует о состоянии, близком к генетическому равновесию. В группе RUS был отмечен небольшой дефицит гетерозигот (1,5 %). Группы ARG и TNZ характеризовались более значительным превышением числа гомозигот над гетерозиготами — соответственно 8,9 и 6,0 %.
1. Характеристика генетического разнообразия российской и пяти национальных популяций коз ( Capra hircus ) зааненской породы, оцененного с использованием ДНК-чипа GoatSNP50 BeadChip (2019-2020 годы)
|
Группа |
n |
1 Ho 1 |
uH e |
uF is |
Ar |
|
RUS |
21 |
0,418 |
0,424 |
0,015 (0,013; 0,017) |
1,979 |
|
SWI |
38 |
0,381 |
0,380 |
- 0,002 ( - 0,003; - 0,001) |
1,934 |
|
ITA |
22 |
0,417 |
0,418 |
0,002 (0; 0,004) |
1,975 |
|
FRA |
55 |
0,423 |
0,422 |
- 0,002 ( - 0,003; - 0,001) |
1,979 |
|
ARG |
11 |
0,386 |
0,426 |
0,089 (0,086; 0,092) |
1,980 |
|
TNZ |
8 |
0,388 |
0,414 |
0,06 (0,057; 0,063) |
1,970 |
|
Примечание. |
n — |
число особей в выборке, |
гол.; Ho — наблюдаемая гетерозиготность, uHe — несме- |
||
|
щенная ожидаемая гетерозиготность, uFis — коэффициент инбридинга, Ar |
рарифицированное аллель- |
||||
|
ное разнообразие. |
В |
скобках приведен |
размах |
изменчивости Fis при доверительном интервале 95 %. |
|
|
Ошибка средней арифметической для показателей Ho, uHe и Ar составляет |
±0,001. Группы коз заанен- |
||||
|
ской породы: RUS |
— |
российская, SWI — |
швейцарская, ITA — итальянская, |
FRA — французская, ARG — |
|
|
аргентинская, TNZ |
— |
танзанийская. |
|||
Наибольшее аллельное разнообразие было выявлено у популяций ARG (Ar = 1,980), RUS (Ar = 1,979) и FRA (Ar = 1,979). У группы SWI оно оказалось минимальным (Ar = 1,934).
Анализ главных компонент (рис. 1) продемонстрировал, что первая главная компонента, отвечающая за 7,76 % генетической изменчивости, обособляла группу SWI от остальных пяти популяций, включая RUS. Вторая главная компонента, соответствующая 4,28 % генетической вариабельности, отделяла SWI, ITA и частично FRA от популяций RUS (некоторые представители располагались практически на оси), ARG и TNZ (располагались в самом отдаленном секторе PCA-плота).
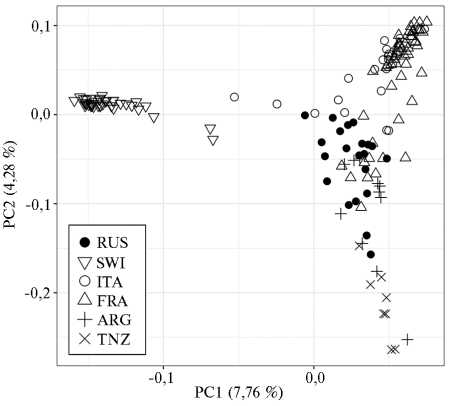
Рис. 1. Результаты PCA-анализа (principal component analysis), проведенного для российской и пяти национальных популяций коз ( Capra hircus ) зааненской породы на основе SNP-профилей, полученных с использованием ДНК-чипа GoatSNP50 BeadChip (2019-2020 годы) . Группы коз заанен-ской породы: RUS — российская, SWI — швейцарская, ITA — итальянская, FRA — французская, ARG — аргентинская, TNZ — танзанийская.
В структуре генетической сети, демонстрирующей взаимосвязи российской и пяти национальных популяций коз заанен-ской породы (рис. 2), было выделено два кластера. Первый включал длинную обособленную ветвь
SWI, примыкающий к ней подкластер ITA + FRA и более отдаленную короткую ветвь RUS (на стыке кластеров). Второй кластер был образован отдельными ветвями ARG и TNZ.
Для лучшего понимания степени генетической дифференциации между изучаемыми популяциями коз были рассчитаны показатели FST и R S T. Их значения (табл. 2) оказались максимальными между группами SWI и TNZ (F ST = 0,109; R ST = 0,374), а также между SWI и ARG (F ST = 0,078; R S T = 0,321). Минимальная дифференциация наблюдалась между ITA и
FRA (F ST = 0,008; R ST = 0,154).
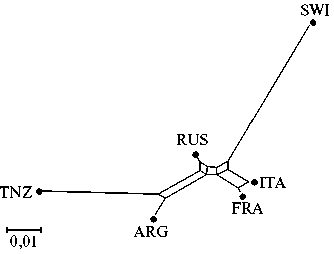
Рис. 2. Генетическая сеть Neighbor Net, построенная на основе попарных значений F ST и демонстрирующая взаимосвязи российской и пяти национальных популяций коз ( Capra hircus ) зааненской породы на основе SNP-профилей, полученных с использованием ДНК-чипа GoatSNP50 BeadChip (2019-2020 годы) . Группы коз зааненской породы: RUS — российская, SWI — швейцарская, ITA — итальянская, FRA — французская, ARG — аргентинская, TNZ — танзанийская.
При изучении генетических связей RUS с популяциями из других стран было установлено, что минимальные генетические дистанции наблюдались между RUS и группами FRA и ITA (F ST = 0,02; R ST = 0,189 и F ST = 0,023; R ST = 0,215), а наибольшая дифференциация — с группами TNZ и SWI (FST = 0,054; RST = 0,311 и F ST = 0,06; R ST = 0,276).
С целью анализа популяционной структуры изучаемых групп коз мы провели расчет наиболее вероятного числа кластеров (рис. 3, А). Наименьшая ошибка кросс-валидации была идентифицирована при К = 3 и составила 0,64445. При К = 2 группа SWI формировала свой собственный обособленный кластер (см. рис. 3, Б). При К = 3 группы ITA и FRA демонстрировали сходную популяционную структуру. Следует отметить, что популяция FRA состояла из неоднородных особей, большая часть которых была высоко консолидирована в своем кластере, а вторая часть (около 30 % животных) демонстрировала наличие иных геномных компонентов. Группа TNZ формировала свой кластер. Популяции RUS и ARG характеризовались присутствием всех трех выявленных генетических составляющих, при этом RUS сохраняла в своем генофонде наибольшую долю оригинального элемента (SWI).
2. Генетическая дифференциация между российской и пятью национальными популяциями коз ( Capra hircus ) зааненской породы, оцененная с помощью показателей FST и RST, рассчитанных на основе данных полногеномного генотипирования с использованием ДНК-чипа GoatSNP50 BeadChip (2019-2020 годы)
|
Группа |
RUS |
SWI |
ITA |
FRA |
ARG |
TNZ |
|
RUS |
0,276 |
0,215 |
0,189 |
0,254 |
0,311 |
|
|
SWI |
0,060 |
0,275 |
0,276 |
0,321 |
0,374 |
|
|
ITA |
0,023 |
0,059 |
0,154 |
0,263 |
0,327 |
|
|
FRA |
0,020 |
0,065 |
0,008 |
0,248 |
0,316 |
|
|
ARG |
0,029 |
0,078 |
0,035 |
0,035 |
0,318 |
|
|
TNZ |
0,054 |
0,109 |
0,066 |
0,065 |
0,044 |
П р и м е ч а н и е. Попарные значения FST представлены ниже диагонали, дистанции Рейнольдса RST — выше диагонали. Группы коз зааненской породы: RUS — российская, SWI — швейцарская, ITA — итальянская, FRA — французская, ARG — аргентинская, TNZ — танзанийская.
Высокопродуктивные, привлекательные для разведения породы сельскохозяйственных животных широко распространены за пределами места своего выведения. В скотоводстве к таким породам относятся голштинская и симментальская, в свиноводстве — ландрас, дюрок и крупная белая, в овцеводстве — рамбулье и ромни-марш, в козоводстве безусловный мировой лидер — зааненская порода. В странах-экспортерах начинается активная селекция завезенного генетического материала, в связи c чем оценка идентичности национальных популяций оригинальному генофонду, помимо генетического интереса, имеет прикладное значение, а именно способствует определению общего направления селекции и позволяет вносить возможные корректировки в ее цели (34). Так, сравнительный анализ SNP-профилей симментальского скота немецко-австрийской и российской селекции выявил геномные регионы с высокой частотой встречаемости идентичных гаплотипов, несмотря на определенную разницу в целях селекции (34).
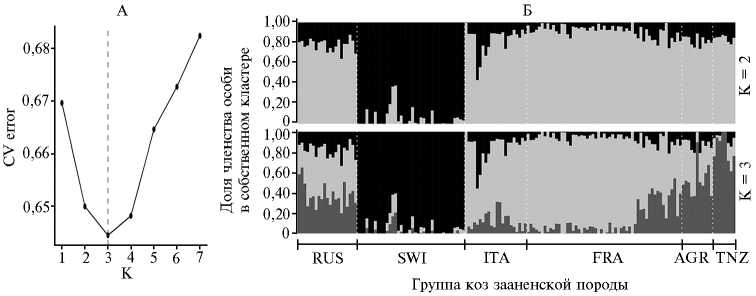
Рис. 3. Сопоставление популяционной структуры и анализ генетической однородности российской и пяти национальных популяций коз ( Capra hircus ) зааненской породы, проведенные с использованием ДНК-чипа GoatSNP50 BeadChip (2019-2020 годы) : А — график, построенный на основе значений ошибок кросс-валидации (CV error), рассчитанных при тестировании количества предковых кластеров (К) от 1 до 7 (пунктирной линией отмечено число кластеров, для которых ошибка кросс-валидации была наименьшей); Б — структура изучаемых популяций при числе кластеров, равном двум и трем. Группы коз зааненской породы: RUS — российская, SWI — швейцарская, ITA — итальянская, FRA — французская, ARG — аргентинская, TNZ — танзанийская.
Зааненская порода была вовлечена в разработку и тестирование ДНК-чипа GoatSNP50BeadChip, поэтому дифференциации между национальными популяциями зааненской породы будут полностью определяться их генетическими различиями и не зависеть от возможных погрешностей, вызванных внутривидовой разницей в полиморфных локусах. Например, ангорская порода коз не входила в инициальный список пород для разработки ДНК-чипа, поэтому S.F. Lashmar с соавт. (35) предварительно оценивали информативность GoatSNP50BeadChip для этой породы.
При сравнении показателей генетического разнообразия, рассчитанных в нашей работе для национальных популяций коз зааненской породы, были отмечены их высокие значения у всех групп, за исключением оригинальной швейцарской. A. Burren c соавт. (36) исследовали генетическое разнообразие у 10 локальных швейцарских пород коз, у которых наблюдаемая гетерозиготность варьировала от 0,369 (аппенцелльская и тоггенбургская) до 0,401 (гризон полосатый и павлин), а аллельное разнообразие — от 1,531 (стифельгесская) до 1,941 (цветная серна). Наблюдаемая гетерозиготность составила 0,385 у альпийской породы, 0,384 — у ламанчской, 0,338 — у нубийской, 0,379 — у зааненской и 0,353 — тогген-бургской породы канадской селекции. Кроме того, у всех перечисленных пород был зафиксирован незначительный недостаток гетерозигот (18). Следует отметить, что показатели Ho и Ar у локальных швейцарских коз и молочных пород коз канадской селекции скорее соответствовали значениям оригинальной группы SWI и несколько уступали аналогичным показателям у RUS, ITA, FRA, ARG и TNZ.
Подобно зааненской, ангорская порода, берущая начало из Турции, широко распространена во многих странах мира. C. Visser с соавт. (37) использовали Goat SNP50 BeadChip для изучения степени географической изоляции и генетической вариабельности в трех популяциях ангорской породы коз из Южной Африки, Франции и Аргентины. Среди национальных популяций ангорской породы была зафиксирована разница в ожидаемой и наблюдаемой гетерозитности (He = 0,371-0,397; Ho = 0,3650,414), сопоставимая с разницей, вычисленной в нашей работе (He = 0,3800,424; H o = 0,381-0,423).
Суммируя результаты PCA-анализа и структуры филогенетического дерева, мы выявили четкую дифференциацию между национальными (за исключением более близких ITA и FRA) и оригинальной популяциями коз зааненской породы. C. Visser с соавт. (37) тоже отмечали, что национальные популяции коз ангорской породы очень четко отделяются друг от друга. Значение показателя FST между ангорскими популяциями составило 0,120, тогда как в нашей работе максимальное значение показателя FST между TNZ и SWI было 0,109. Это может свидетельствовать о том, что популяции зааненской породы еще не так сильно отошли от исходной швейцарской, как ангорские козы.
Интересно, что в соответствии со значением FST наибольшая дифференциация была отмечена между RUS и SWI, тогда как по RST группа RUS оказалась наиболее обособлена от TNZ, что подтверждается положением соответствующих групп на графике PCA. По мнению G. Laval с со-авт. (38), вычисление дистанций Рейнольдса — это лучший метод оценки дивергенции между близкородственными группами, что согласуется с полученными нами данными. Кроме того, анализ популяционной структуры подтвердил сохранение геномных компонентов SWI у коз группы RUS.
Таким образом, проведенное полногеномное исследование российской популяции коз зааненской породы показало, что по генетическому и аллельному разнообразию эта группа превосходит оригинальную швейцарскую популяцию, но при этом соответствует группам французской и итальянской селекции. Результаты нашей работы подтвердили, что различные стратегии селекции привели к генетическим различиям между национальными популяциями зааненской породы, включая российскую. Тем не менее российская популяция коз зааненской породы сохраняет геномные компоненты, свойственные исходному генофонду, что, с одной стороны, создает необходимую вариабельность для отбора, с другой — оставляет возможность возвращения к оригинальному типу швейцарской селекции.
Список литературы Геномная архитектура российской популяции зааненских коз в аспекте генофонда породы из пяти стран мира
- Новопашина С.И., Санников М.Ю., Кизилова Е.И. Молочная продуктивность коз разных генотипов. Сб. науч. тр. Всероссийского научно-исследовательского института овцеводства и козоводства, 2017, 1(10): 225-229.
- Справочник пород и типов сельскохозяйственных животных, разводимых в Российской Федерации /Под ред. И.М. Дунина, А.Г. Данкверта. М., 2013.
- Devendra С., Haenlein G.F.W. Animals that produce dairy foods | Goat breeds. In: Encyclopedia of dairy sciences (second edition) /J.W. Fuquay (ed.). Academic Press, Amsterdam, 2011: 310-324 ( ). DOI: 10.1016/B978-0-12-374407-4.00035-2
- Новопашина С.И., Санников М.Ю., Хататаев С.А., Кузьмина Т.Н., Хмелевская Г.Н., Степанова Н.Г., Тихомиров А.И., Маринченко Т.Е. Состояние и перспективные направления улучшения генетического потенциала мелкого рогатого скота: научный аналитический обзор. М., 2019.
- Brito L.F., Jafarikia M., Grossi D.A., Kijas J.W., Porto-Neto L.R., Ventura R.V., Salgorzaei M., Schenkel F.S. Characterization of linkage disequilibrium, consistency of gametic phase and admixture in Australian and Canadian goats. BMC Genetics, 2015, 16: 67 ( ). DOI: 10.1186/s12863-015-0220-1
- da Silva T.G.F., Santos G.C.L., Duarte A.M.C., Turco S.H.N., da Cruz Neto J.F., da Rosa Ferraz Jardim A.M., dos Santos T.S. Black globe temperature from meteorological data and a bioclimatic analysis of the Brazilian northeast for Saanen goats. Journal of Thermal Biology, 2019, 85: 102408 ( ).
- DOI: 10.1016/j.jtherbio.2019.102408
- Zamuner F., DiGiacomo K., Cameron A.W.N., Leury B.J. Effects of month of kidding, parity number, and litter size on milk yield of commercial dairy goats in Australia. Journal of Dairy Science, 2020, 103(1): 954-964 ( ).
- DOI: 10.3168/jds.2019-17051
- Sun Y., Wang C., Sun X., Guo M. Proteomic analysis of whey proteins in the colostrum and mature milk of Xinong Saanen goats. Journal of Dairy Science, 2020, 103(2): 1164-1174 ( ).
- DOI: 10.3168/jds.2019-17159
- Makete G., Aiyegoro O.A., Thantsha M.S. Isolation, identification and screening of potential probiotic bacteria in milk from South African Saanen goats. Probiotics and Antimicrobial Proteins, 2017, 9(3): 246-254 ( ).
- DOI: 10.1007/s12602-016-9247-5
- Государственный реестр селекционных достижений, допущенных к использованию. Том 2 "Породы животных" (официальное издание). М., 2019.
- Ежегодник по племенной работе в овцеводстве и козоводстве в хозяйствах Российской Федерации (2018 год) /Под ред. Т.А. Мороз. М., 2019.
- Тощев В.К., Мустафина Г.Н. Использование зааненской породы в улучшении молочного козоводства. Аграрная наука, 2012, 5: 27-28.
- Ajmone-Marsan P., Colli L., Han J.L., Achilli A., Lancioni H., Joost S., Crepaldi P., Pilla F., Stella A., Taberlet P., Boettcher P., Negrini R., Lenstra J.A., Italian Goat Consortium, Econogene Consortium, Globaldiv Consortium. The characterization of goat genetic diversity: towards a genomic approach. Small Ruminant Research, 2014, 121(1): 58-72 ( ).
- DOI: 10.1016/j.smallrumres.2014.06.010
- Tosser-Klopp G., Bardou P., Bouchez O., Cabau C., Crooijmans R., Dong Y., Donnadieu-Tonon C., Eggen A., Heuven H.C.M., Jamli S., Jiken A.J., Klopp C., Lawley C.T., McEwan J., Martin P., Moreno C.R., Mulsant P., Nabihoudine I., Pailhoux E., Palhiere E., Rupp R., Sarry J., Sayre B.L., Tircazes A., Wang J., Wang W., Zhang W., the International Goat Genome Consortium. Design and characterization of a 52k SNP chip for goats. PLoS ONE, 2014, 9(1): e86227 ( ).
- DOI: 10.1371/journal.pone.0086227
- Nicoloso L., Bomba L., Colli L., Negrini R., Milanesi M., Mazza R., Sechi T., Frattini S., Talenti A., Coizet B., Chessa S., Marletta D., D'Andrea M., Bordonaro S., Ptak G., Carta A., Pagnacco G., Valentini A., Pilla F., Ajmone-Marsan P., Crepaldi P., the Italian Goat Consortium. Genetic diversity of Italian goat breeds assessed with a medium-density SNP chip. Genetics Selection Evolution, 2015, 47(1): 62 ( ).
- DOI: 10.1186/s12711-015-0140-6
- Mdladla K., Dzomba E.F., Huson H.J., Muchadeyi F.C. Population genomic structure and linkage disequilibrium analysis of South African goat breeds using genome-wide SNP data. Animal Genetics, 2016, 47(4): 471-482 ( ).
- DOI: 10.1111/age.12442
- Rahmatalla S.A., Arends D., Reissmann M., Ahmed A.S., Wimmers K., Reyer H., Brockmann G.A. Whole genome population genetics analysis of Sudanese goats identifies regions harboring genes associated with major traits. BMC Genetics, 2017, 18: 92 ( ).
- DOI: 10.1186/s12863-017-0553-z
- Brito L.F., Kijas J.W., Ventura R.V., Sargolzaei M., Porto-Neto L.R., Cánovas A., Feng Z., Jafarikia M., Schenkel F.S. Genetic diversity and signatures of selection in various goat breeds revealed by genome-wide SNP markers. BMC Genomics, 2017, 18: 229 ( ).
- DOI: 10.1186/s12864-017-3610-0
- Stella A., Nicolazzi E.L., Van Tassell C.P., Rothschild M.F., Colli L., Rosen B.D., Sonstegard T.S., Crepaldi P., Tosser-Klopp G., Joost S., the AdaptMap Consortium. AdaptMap: exploring goat diversity and adaptation. Genetics Selection Evolution, 2018, 50(1): 61 ( ).
- DOI: 10.1186/s12711-018-0427-5
- Bertolini F., Cardoso T.F., Marras G., Nicolazzi E.L., Rothschild M.F., Amills M., AdaptMap consortium. Genome-wide patterns of homozygosity provide clues about the population history and adaptation of goats. Genetics Selection Evolution, 2018, 50(1): 59 ( ).
- DOI: 10.1186/s12711-018-0424-8
- Colli L., Milanesi M., Talenti A. et al. Genome-wide SNP profiling of worldwide goat populations reveals strong partitioning of diversity and highlights post-domestication migration routes. Genetics Selection Evolution, 2018, 50(1): 58 ( ).
- DOI: 10.1186/s12711-018-0422-x
- Bertolini F., Servin B., Talenti A. et al. Signatures of selection and environmental adaptation across the goat genome post-domestication. Genetics Selection Evolution, 2018, 50(1): 57 ( ).
- DOI: 10.1186/s12711-018-0421-y
- Fan J.-B., Oliphant A., Shen R., Kermani B.G., Garcia F., Gunderson K.L., Hansen M., Steemers F., Butler S.L., Deloukas P., Galver L., Hunt S., Mcbride C., Bibikova M., Rubano T., Chen J., Wickham E., Doucet D., Chang W., Campbell D., Zhang B., Kruglyak S., Bentley D., Haas J., Rigault P., Zhou L., Stuelpnagel J., Chee M.S. Highly parallel SNP genotyping. Cold Spring Harb. Symp. Quant. Biol., 2003, 68: 69-78 ( ).
- DOI: 10.1101/sqb.2003.68.69
- Chang C.C., Chow C.C., Tellier L.C., Vattikuti S., Purcell S.M., Lee J.J. Second-generation PLINK: rising to the challenge of larger and richer datasets. GigaScience, 2015, 4: s13742-015-0047-8 ( ).
- DOI: 10.1186/s13742-015-0047-8
- Colli L., Milanesi M., Talenti A., Bertolini F., Chen M., Crisà A., Daly K., Del Corvo M., Guldbrandtsen B., Lenstra J.A., Rosen B.D., Vajana E., Catillo G., Joost S., Nicolazzi E.L., Rochat E., Rothschild M.F., Servin B., Sonstegard T.S., Steri R., Van Tassell C.P., Ajmone-Marsan P., Crepaldi P., Stella A., AdaptMap Consortium. Data from: Signatures of selection and environmental adaptation across the goat genome post-domestication, Dryad, Dataset, 2018 ( ).
- DOI: 10.5061/dryad.v8g21pt
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358-1370 ( ).
- DOI: 10.2307/2408641
- Reynolds J., Weir B.S., Cockerham C.C. Estimation of the coancestry coefficient: basis for a short-term genetic distance. Genetics, 1983, 105(3): 767-779.
- Keenan K., McGinnity P., Cross T.F., Crozier W.W., Prodöhl P.A. diveRsity: an R package for the estimation of population genetics parameters and their associated errors. Methods in Ecology and Evolution, 2013, 4(8): 782-788 ( ).
- DOI: 10.1111/2041-210X.12067
- Wickham H. ggplot2: elegant graphics for data analysis. Springer-Verlag, NY, 2009.
- Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution, 2006, 23(2): 254-267 ( ).
- DOI: 10.1093/molbev/msj030
- Alexander D.H., Novembre J., Lange K. Fast model-based estimation of ancestry in unrelated individuals. Genome Research, 2009, 19(9): 1655-1664 ( ).
- DOI: 10.1101/gr.094052.109
- Francis R.M. pophelper: An R package and web app to analyse and visualise population structure. Molecular Ecology Resources, 2017, 17(1): 27-32 ( ).
- DOI: 10.1111/1755-0998.12509
- R Core Team (2018). R: a language and environment for statistical computing. R foundation for statistical computing, Vienna, Austria. Режим доступа: https://www.R-project.org/. Без даты.
- Mészáros G., Fornara M.S., Reyer H., Wimmers K., Sölkner J., Brem G., Sermyagin A.A., Zinovieva N.A. Elevated haplotypes frequencies reveal similarities for selection signatures in Western and Russian Simmental populations. Journal of Central European Agriculture, 2019, 20(1): 1-11 ( ).
- DOI: 10.5513/JCEA01/20.1.2412
- Lashmar S.F., Visser C., Van Marle-Köster E. Validation of the 50k Illumina goat SNP chip in the South African Angora goat (short communication). South African Journal of Animal Science, 2015, 45(1): 56-59 ( ).
- DOI: 10.4314/sajas.v45i1.7
- Burren A., Neuditschko M., Signer-Hasler H., Frischknecht M., Reber I., Menzi F., Drögemüller C., Flury C. Genetic diversity analyses reveal first insights into breed-specific selection signatures within Swiss goat breeds. Animal Genetics, 2016, 47(6): 727-739 ( ).
- DOI: 10.1111/age.12476
- Visser C., Lashmar S.F., Van Marle-Köster E., Poli M.A., Allain D. Genetic diversity and population structure in South African, French and Argentinian Angora goats from genome-wide SNP data. PLoS ONE, 2016, 11(5): e0154353 ( ).
- DOI: 10.1371/journal.pone.0154353
- Laval G., SanCristobal M., Chevalet C. Measuring genetic distances between breeds: use of some distances in various short term evolution models. Genetics Selection Evolution, 2002, 34(4): 481-507 (doi: 10.1051/gse:2002019).


