Геномные источники разнообразия как драйверы доместикации (обзор)
Автор: Глазко В.И., Косовский Г.Ю., Глазко Т.Т.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Обзоры, проблемы, итоги
Статья в выпуске: 5 т.57, 2022 года.
Бесплатный доступ
Одомашнивание растений и животных - ключевое событие в истории человеческой цивилизации, его механизмы привлекают внимание многих исследователей, особенно в последние десятилетия в связи с сокращением биоразнообразия, в том числе у сельскохозяйственных видов. По определению, предложенному Мелиндой Зедер (М.А. Zeder, 2015), доместикация - это устойчивые мутуалистические отношения в ряду поколений, в которых доместикатор влияет на воспроизводство доместицируемых, оптимизируя их образ жизни для получения интересующего его ресурса, благодаря чему доместицируемые получают преимущества перед другими особями вида. Такие взаимоотношения сопровождаются межвидовой коэволюцией, они присутствуют не только у человека и домашних видов растений и животных, но и у представителей диких видов. В качестве универсальной черты домашних видов по сравнению с близкородственными дикими рассматривается высокое фенотипическое разнообразие, на которое обращал внимание еще Чарльз Дарвин (Ч. Дарвин, 1951). При попарных геномных сравнениях домашней собаки и волка, дикой и домашней кошки, доместицированного и дикого кролика обнаруживается относительно повышенная плотность ряда мобильных генетических элементов у доместицированных животных по сравнению с дикими. В последнее время мобильные генетические элементы, или транспозоны (ТЕs), рассматриваются как основные факторы геномных преобразований, генных, геномных дупликаций, геномных и генных реконструкций, а также горизонтальных обменов генетической информацией (K.R. Oliver, W.K. Greene, 2009). Число сравнительных геномных исследований TEs у доместицированных видов невелико, и роль таких элементов в доместикации, как правило, не обсуждается. Однако можно ожидать, что в основе всех эволюционных событий, в том числе в ответ на формирование новых условий вопроизводства при доместикации, лежат универсальные механизмы изменчивости геномов. Представленный обзор систематизирует такие механизмы. Рассматриваются TEs, обеспечивающие глубокие геномные преобразования, активные и пассивные формы их взаимодействий с геномом хозяина (K.R. Oliver с соавт., 2009). Описаны примеры возникновения новых генов на основе ТЕs, такие как ген синтицина (C. Herrera-Úbeda с соавт., 2021), ген-регулятор синаптической пластичности arc (Activity Regulated Cytoskeleton Associated Protein) (C. Herrera-Úbeda с соавт., 2021), семейство генов bex , кодирующих, в частности, рецептор фактора роста нейронов (E. Navas-Pérez с соавт., 2020; R.P. Cabeen с соавт., 2022). Обсуждаются конфликтные и кооперативные взаимодействия с геномом хозяина при перемещениях ретротранспозонов, разные механизмы их влияний на профили генной экспрессии. Рассматривается участие ТЕs в формировании и изменчивости сетей геномных регуляторных элементов, в частности микроРНК. Представлены примеры вовлеченности микроРНК в контроль и формирование хозяйственно ценных признаков у доместицированных растений и животных. Накопленные данные позволяют предполагать, что ведущим источником большой фенотипической изменчивости доместицированных видов служит относительно высокая насыщенность их геномов мобильными генетическими элементами и, как следствие, увеличение изменчивости геномных регуляторных сетей при формировании общей с человеком биоэкониши.
Доместикация, геномика, изменчивость, транспозоны, регуляторные сети, микрорнк
Короткий адрес: https://sciup.org/142236358
IDR: 142236358 | УДК: 636.012:575.174.015.3 | DOI: 10.15389/agrobiology.2022.5.832rus
Текст обзорной статьи Геномные источники разнообразия как драйверы доместикации (обзор)
Проблема поиска генетических основ доместикации как вхождения растений и животных в новые условия обитания и воспроизводства, направленно формируемые человеком, имеет теоретическую и практическую компоненты. Теоретическая связана с тем, что только процесс доместикации представляет собой прямую экспериментальную модель микро- и макроэволюции, доступной для изучения человеком (1), на что указывал еще Чарльз Дарвин (2). Практический аспект проблемы обусловлен тем, что непонимание механизмов доместикации не позволяет развивать методы управления генетическими потоками у доместицированных видов, что становится все более важным в связи с ростом населения Земли, сокращением плодородных земель и биоразнообразия (3).
К настоящему времени наименее противоречивое определение доместикации дано M.A. Zeder (4) в следующей форме: это устойчивые, многопоколенческие, мутуалистические отношения, в которых люди (домести-каторы) берут на себя значительную часть контроля над воспроизводством и уходом за растениями/животными (доместицируемыми) для обеспечения более предсказуемого запаса интересующего ресурса, посредством чего рас-тение/животное способно увеличить свой репродуктивный успех по сравнению с особями, не участвующими в таких отношениях; тем самым повышается приспособленность как людей, так и целевых доместицируемых видов. Сформировано представление о «синдроме доместикации», пересекающем таксономические различия. Например, у однолетних растений это касается признаков, ассоциированных с прорастанием и распространением семян, — изменениями в скорости прорастания, размере семян, их осыпании, толщине стенок, а также в сроках и морфологии механизмов распространения (5, 6). У животных к таким признакам относятся характеристики нервного гребня, с которыми связаны особенности поведения животных (в частности, социальная активность), адаптивный потенциал, включая показатели плодовитости, и изменчивость тех признаков, которые требуются до-местикатору и связаны с продуктивностью животных (7).
Необходимо отметить, что достаточно часто и у растений, и у животных характеристики, которые относят к синдромам доместикации, ассоциированы с полиморфизмом разных генов, которые при этом участвуют в сходных метаболических путях, что ожидаемо, поскольку большинство признаков синдрома доместикации полигенные (8).
Судя по накопленным данным, единственной общей чертой для всех доместицируемых видов является относительно повышенная фенотипическая изменчивость, которая служит источником ответа на факторы естественного и искусственного отбора, которые появляются при вовлечении представителей определенного вида в сферу человеческих интересов. В таком случае главным вопросом в изучении механизмов доместикации становится выяснение генетических основ такой большой изменчивости (9).
Мобильные генетические элементы (транспозоны, transposable elements — TEs). В последние годы принято считать мобильные генетические элементы, или транспозоны (ТЕs), основным фактором в геномных преобразованиях, в генных, геномных дупликациях, геномных и генных реконструкциях, а также в горизонтальных обменах генетической информацией (10-12). В некоторых исследованиях, учитывая филогенетическую и биологическую близость ТЕs к вирусам, предлагают совокупность таких последовательностей в геномах обозначать как эндовиром (13). Обнаружена определенная корреляция между плотностью геномного распределения, активностью ТЕs и интенсивностью видообразования (обилие видов) в разных таксонах (14). Однако возможность вовлечения TEs в повышенную фенотипическую изменчивость у доместицированных видов, как правило, не рассматривается.
Мобильные генетические элементы делятся на два основных класса — I и II. Класс I состоит из ретротранспозонов, в распространении которых по геному участвует РНК, при этом у автономных ретротранспозонов присутствует ген, кодирующий обратную транскриптазу. Класс II включает ДНК-транспозоны, не использующие РНК в качестве промежуточного звена для транспозиции. В каждом классе присутствуют автономные и неавтономные представители. В результате в настоящее время принята следующая основная классификация мобильных генетических элементов — транспозонов (МГЭ, ТЕs) (15). Класс I (объединяет ретротранспозоны) представлен автономными эндогенными ретровирусами (endogenous retroviruses, ERV), содержащими длинные концевые повторы (long terminal repeat, LTR), длинными диспергированными ядерными элементами (long interspersed nuclear elements, LINE), а также неавтономными элементами — короткими диспергированными ядерными элементами (short interspersed nuclear elements, SINE) и комплексными ретротранспозонами (продукты рекомбинации между ретротранспозонами и микросателлитами, composite retrotransposon SVA). Класс II (представлен ДНК-транспозонами) включает автономные элементы, несущие DDE-аминокислотный мотив, типичный для траспо-зазы/интегразы большинства семейств автономных ДНК-транспозонов, за исключением хелитронов (Helitrone, ДНК-транспозоны, реплицирующиеся по модели кольца и использующие для этого фермент с эндонуклеазным и геликазным доменом), а также неавтономный элемент — миниатюрный инвертированный повтор транспозируемого элемента (minature inverted repeat transposable element, MITE).
Мобильные генетические элементы называют драйверами эволюции, известен их существенный вклад в эволюцию, в частности, приматов (16). TEs были обнаружены в геномах разных таксономических групп — от бактерий до млекопитающих. Их повсеместное присутствие обусловлено выраженной тенденцией к распространению внутри генома, а также к колонизации других геномов. Некоторые ТЕs (например, SINE) могут возникать спонтанно из немобильных последовательностей ДНК в геноме, в то время как другие могут горизонтально передаваться между видами. TEs имеют древнее происхождение, восходящее к прокариотам. ДНК-ТЕs (класс II) связаны с последовательностями бактериальных вставок, ретро-ТЕs — с интронами второй группы (16, 17).
Некоторые TEs, по-видимому, присутствовали у эукариот с самого начала их существования, возможно, еще более миллиарда лет назад. TEs часто составляют большую, если не наибольшую часть эукариотического генома. Например, секвенированные геномы млекопитающих состоят из ТЕs, как минимум, на треть у неприматов и примерно наполовину — у приматов. TEs, по-видимому, являются важной детерминантой размера генома, причем организмы, обладающие очень большими геномами (например, растения), часто имеют гораздо более высокое содержание ТЕs (> 60 %) по сравнению с видами с относительно небольшими геномами (дрожжи, нематоды, насекомые и птицы), у которых доля ТЕs существенно ниже.
Благодаря современным возможностям полногеномного секвенирования и компьютерных программ анализа нуклеотидных последовательностей накоплены данные о присутствии ТЕs у разных таксонов (рис. 1) (18). Эти результаты свидетельствуют о широком распространении ТЕs и о том, что их число значительно различается между видами, внося очевидный вклад в различия размеров их гаплоидного генома. Существенную разницу можно обнаружить даже между близкородственными организмами. Например, в роде Entamoeba геном E. histolytica содержит ∼ 20 % TEs, в то время как у E. dispar эта доля составляет ∼ 10 %. Аналогичным образом в роде Oryza геном вида дикого риса O. australiensis более чем удвоился в размерах за 3 млн лет из-за амплификации TEs, в результате чего оказался почти в 3 раза больше геномов некоторых его ближайших родственников. Напротив, ряд таксономических групп демонстрируют лишь незначительные различия в содержании ТЕs, что может отражать определенные ограничения на размер генома или, наоборот, относительно высокую толерантность к расширению генома. Например, птицы имеют относительно консервативный размер генома (возможно, из-за метаболических затрат, связанных с активным полетом), несмотря на то, что TEs активны у большинства исследованных видов этой таксономической группы. У саламандр, наоборот, произошла экстремальная и независимая амплификация TEs, что привело к формированию гигантских геномов (см. рис. 1).
Интересно отметить, что наибольший размер генома и, соответственно, вклад ТЕs в него обнаружен у двоякодышащей рыбы и земноводного, то есть видов, у которых реализуется ароморфоз (выживание в двух средах) (рис. 1). Также обращают на себя внимание данные о неодинаковой представленности разных вариантов ТЕs у растений и животных: у растений эндогенные ретровирусы распространены больше, чем у животных, а у последних выше частота встречаемости SINE (18) (см. рис. 1).
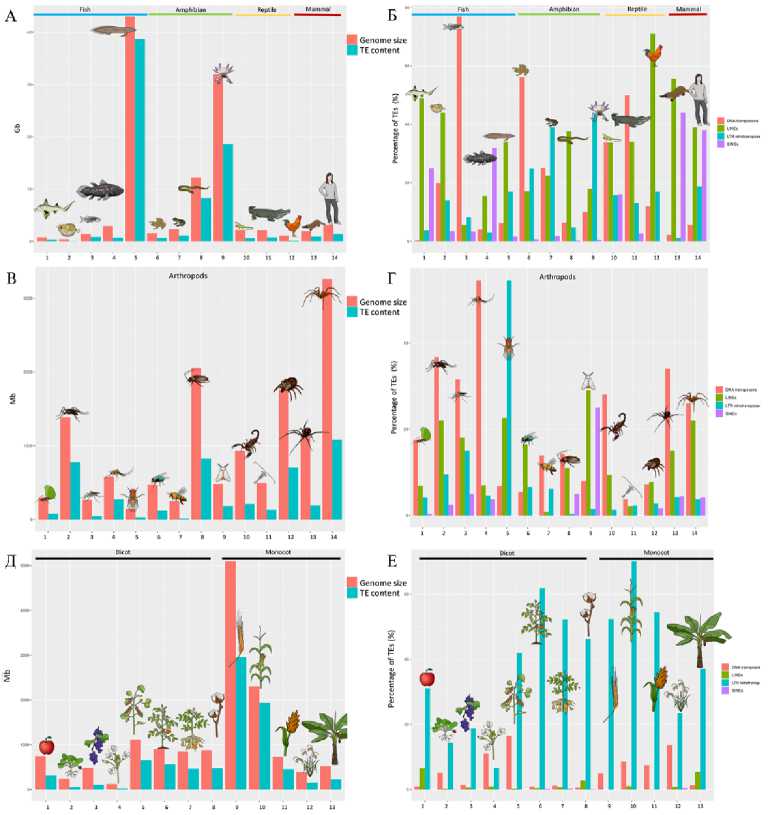
Рис. 1. Распространенность транспозонов ТЕs в геномах различных видов (18).
A. Размер генома (красный цвет) и содержащихся в нем ТЕs (голубой цвет) у позвоночных слева направо: 1 — Callorhinchus milii , 2 — Tetraodon nigroviridis, 3 — Danio rerio , 4 — Latimeria chalumnae , 5 — Neoceratodus forsteri , 6 — Xenopus tropicalis , 7 — Nanorana parkeri , 8 — Сhthyophis bannanicus , 9 — Ambystoma mexicanum , 10 — Anolis carolinensis , 11 — Alligator mississipiensis , 12 — Gallus gallus , 13 — Ornithorhynchus anatinus , 14 — Homo sapiens.
B. Доля (%) разных типов ТЕs: красный — ДНК-транспозоны, зеленый — LINE, сиреневый — SINE, голубой — эндогенные ретровирусы (ERV) позвоночных (виды см. A).
C. Размер генома (красный цвет) и содержащихся в нем ТЕs (голубой цвет) у артро-под: 1 — Acromyrmex echinatior , 2 — Aedes aegypti , 3 — Anopheles gambiae , 4 — Culex quinquefasciatus
(москиты), 5 — Drosophila melanogaster (плодовая мушка), 6 — Lucilia cuprina , 7 — Apis mellifera , 8 — Blattella germanica , 9 — Bombyx mori , 10 — Centruroides exilicauda , 11— Eurytemora affinis , 12 — Ixodes scapularis , 13 — Latrodectus hesperus , 14 — Loxosceles reclusa .
-
D. Доля (%) разных типов ТЕs у артропод: красный — ДНК-транспозоны, зеленый — LINE, сиреневый — SINE, голубой — эндогенные ретровирусы (ERV) позвоночных (виды см. C).
-
E. Размер генома (красный цвет) и содержащихся в нем ТЕs (голубой цвет) у растений: 1 — Malus domestica , 2 — Fragaria vesca , 3 — Vitus vinifera , 4 — Arabidopsis thaliana , 5 — Glycine max , 6 — Solanum lycopersicum , 7 — Solanum tuberosum , 8 — Gossypium raimondii , 9 — Hordeum vulgare , 10 — Zea mays , 11 — Sorghum bicolor , 12 — Oryza sativa , 13 — Musa acuminate.
-
F. Доля (%) разных типов ТЕs у растений: красный — ДНК-транспозоны, зеленый — LINE, сиреневый — SINE, голубой — эндогенные ретровирусы (ERV) позвоночных (виды см. E).
Активные и пассивные механизмы взаимодействия транспозонов с геномом хозяина, появление новых генов. ТЕs, как уже отмечалось, — мощные факторы эволюции генома (и, следовательно, фенотипического разнообразия), поскольку они способны вызывать генетические изменения большого масштаба. TEs могут менять работу геномов как активно, так и пассивно. Виды с активными TEs или с обильными однородными неактивными TEs, которые могут влиять на работу генома пассивно, вызывая эктопическую рекомбинацию, потенциально плодовиты и адаптируемы. И наоборот, таксоны с дефицитом TEs или обладающие гетерогенными популяциями неактивных TEs, часто оказываются хорошо приспособленными в своей нише, но имеют тенденцию к длительному застою и могут подвергаться риску исчезновения из-за отсутствия способности к адаптации при изменении условий или к диверсификации (17).
К механизмам влияния ТЕs на работу генома относят ряд геномных эффектов ТЕs, подразделенных на две группы — активных и пассивных воздействий (17).
Описано около 50 случаев неогенов, последовательности которых в значительной степени представляют собой производные ТЕs (17). К таким генам относятся TERT , CENPB , RAG1 / 2 . Эти неогены сделали возможными некоторые необычайно сложные эволюционные события, которые в противном случае могли не произойти, — например, формирование сигнальных последовательностей рекомбинаций, участвующих в перестройках рецептора V(D)J Ag. Они, как и сами гены рекомбиназы RAG1/2, по-види-мому, произошли от древнего ДНК-TE.
Экзоны ( частичные экзоны ) , белок-кодирующие области . TEs часто образуют независимые экзоны внутри генов (17).
Экстрагенные последовательности. TEs вносят вклад в формирование различных экстрагенных последовательностей, например центромер, теломер у дрозофилы и некоторых простейших, сайтов инициации репликации ДНК у дрожжей, областей, связанных с каркасом интерфазного ядра, матриксом хромосом у людей (17).
Прямой вклад в генную регуляцию. Полные и частичные промоторы, энхансеры, сайленсеры. Многие ТЕs управляют экспрессией генов, часто тканеспецифически (17). Помимо влияния на индивидуальные гены, ТЕs, по-видимому, оказывались мобильными носителями готовых промоторов (энхансеров) для широкого распространения дискретных регуляторных элементов по всему геному. Это обеспечивает регуляторную сеть, с помощью которой весь набор генов может совместно регулироваться для создания новых путей клеточного развития или совершенствования существующих.
Регуляторные (микро)РНК . Многие экзонизированные ТЕs кодируют микроРНК (миРНК, miRNA). Идентифицированы 55 генов микроРНК человека, сформированные из ТЕs, которые способны регулировать тысячи генов (17).
Косвенный путь регуляции: ретротранспозиция / трансдукция последовательностей генов, удвоение генов, перетасовка экзонов, расселение регулирующих элементов. Некоторые семейства ретро-ТЕs (например, элементы LINE и LTR) имеют склонность к трансдукции ДНК хозяина из-за их слабых сайтов терминации транскрипции. Дублирование генов также может происходить с помощью присвоения ретротранспозиционного аппарата TEs (гена обратной транскриптазы pol ) транскриптами мРНК хозяина. В организме человека насчитывается более 1000 возникших таким путем ретрогенов, у части из которых развились очень полезные функции, например ген GLUD2 ( Glutamate Dehydrogenase 2 ) важен для утилизации главного возбуждающего нейромедиатора глутамата при нейротрансмиссии (17).
Пассивные механизмы. Стимулирование дупликаций (или потерь) участков ДНК, дупликация генов, дупликация экзонов, сегментная дупликация. Простое присутствие в геноме большого числа неактивных элементов, похожих на ТЕs, создает множество высокогомологичных сайтов, что, как правило, вызывает эктопическую (неаллельную) рекомбинацию ДНК, обусловленную такой гомологией. Этим, вероятно, объясняется большинство продолжающихся эффектов TEs в организмах с низкой активностью транскрипции TEs при их высоком содержании TEs и низком разнообразии (17).
События дупликации ДНК особенно важны в эволюции, поскольку они создают функциональную избыточность и потенциал для усиления функции и/или экспрессии генов.
Стимулирование кариотипических изменений путем эктопической рекомбинации, внутри- и межхромосомные перестройки ДНК. TEs могут пассивно поддерживать основные хромосомные реорганизации, создавая высокогомологичные участки, рассеянные по всему геному, которые склонны к эктопической рекомбинации. Например, Alu-опосредованная транслокация t(11;22)(q23;q11) — наиболее частая конституциональная транслокация у человека (17).
ТЕs и новые гены хозяина. К активным влияниям ТЕs на эволюционные события относятся, в частности, примеры инкорпорации про-вирусных генов в геноме хозяина. Например, геном человека примерно на 8 % состоит из эндогенных ретровирусных последовательностей. В ходе эволюции большинство генов из этих последовательностей утратили свою функцию, но некоторые из них были захвачены и «одомашнены» в процессе так называемой экзаптации. Среди одомашненных вирусных генов есть группа, которая наиболее явно повлияла на эволюцию млекопитающих, — это гены синцитинов (19). Синцитины представляют собой захваченные вирусные белки — продукты гена оболочки наследственно эндогенизирован-ных ретровирусов. Гликопротеин оболочки (ген Env) имеет решающее значение в процессе проникновения вируса в клетку и индуцирует слияние оболочки вириона с плазматической мембраной клетки. В геноме человека обнаруживаются несколько генов Env, но только у двух из них, индуцирующих образование синцития, экспрессия плацентарноспецифическая. Поскольку именно наличие плаценты положено в основу выделения плацентарных млекопитающих в отдельную таксономическую группу, можно было бы ожидать, что синцитины, ответственные за развитие этого уникального органа, который формируется только на период беременности, будут ортологами у разных видов, но это не так. Синцитины приматов и мышей не являются ортологами, и есть свидетельства, указывающие на независимые события захвата соответствующих провиусных генов у предков каждой клады, а также у Scincidae рода Mabuya. Фактически у млекопитающих события захвата генов разных ретровирусов могут быть связаны с четырьмя основными типами плацентарных структур. Можно проследить, что различия между плацентой ящерицы из рода Mabuya и плацентой млекопитающего обусловлено разными событиями захвата генов Env (19). Важно подчеркнуть, что образование синцитиума с участием синцитинов может идти за счет разных синцитинов — обеспечивающих межклеточные слияния оболочечных белков многих ретровирусов (в том числе ВИЧ, вируса бычьего лейкоза), что необходимо для воспроизводства вируса (19).
Еще одним примером служит ген Arc , кодирующий белок Activity Regulated Cytoskeleton Associated Protein, который представляет особый интерес, поскольку считается главным регулятором синаптической пластичности (19). Белок Activity Regulated Cytoskeleton Associated Protein высвобождается из нейронов во внеклеточных пузырьках, которые опосредуют перенос мРНК Arc в новые клетки-мишени, где мРНК Arc может транслироваться. Обнаружено, что этот белок необходим для зависимых от его синтеза форм долговременной памяти и участвует в развитии депрессии у человека. Белок накапливается в слабых синапсах (вероятно, для предотвращения их нежелательного усиления), участвует в постсинаптической транспортировке и обработке бета-амилоида A4 (APP). В дополнение к своей роли в синапсах он вовлечен в регуляцию иммунной системы: специфически экспрессируется в мигрирующих дендритных клетках, тем самым участвуя в активации Т-клеток (19).
Авторы этих исследований (19) отмечают, что продукты генов arc опосредуют межклеточную коммуникацию и синаптическую пластичность через внеклеточные везикулы и во многом гомологичны полипротеинам группоспецифического ретровирусного антигена (Gag). У ретровирусов капсиды необходимы для клеточной инфекции, и их сборка в основном опосредуется Gag. Сходство между Arc и Gag не ограничивается аминокислотной последовательностью, поскольку Arc способен самопроизвольно собираться в структуру, напоминающую капсид. Фактически Arc не только формирует эти капсидоподобные структуры, но и инкапсулирует любую присутствующую при этом мРНК. Такие процессы обеспечивают перемещение молекул РНК между клетками нервной системы. Что касается захвата и эволюции этого вирусного белка, филогенетический анализ выявил, по крайней мере, два независимых события захвата, которые имели место у предков тетрапод и у шизофор. В обоих случаях кооптация arc приводила к сходным функциям белка транспортера РНК в нервной системе. В обеих линиях наиболее близким к предполагаемому исходному предковому варианту был ретротранспозон Ty3/gypsy, но по гомологии последовательностей Ty3/gypsy тетраподы кластеризовались с рыбами, в то время как мухи — с насекомыми, что, по всей видимости, указывает на независимое происхождение arc от ретротранспозонов Ty3/gypsy в каждой линии, несмотря на значительную гомологию его продукта с белком Gag (19).
К результатам доместикации транспозонов относится и образование нового кластера Bex / Tceal (состоит из 14 генов, локализован в Х-хромосоме предка плацентарных) после дивергенции клад сумчатых и плацентарных. Возникновение семейства генов bex , кодирующих, в частности, рецептор фактора роста нейронов, происходило посредством внедрения плацентарных ретротранспозонов HAL1b, L1ME-like и эгдогенного ретровируса Hnmph1 у предшественников плацентарных (20).
Анализ функциональной актиности генов Bex / Tceal был выполнен in vitro, а также на линиях мышей, мутантных по гену bex3 . Гомозиготные по мутации этого гена линии демонстрировали отклонения в морфологии 838
черепа, размеры мозжечка и головного мозга у них уменьшались, с чем могут быть связаны дефекты поведения, наблюдаемые у мышей-мутантов. У них обнаруживались нарушения в социальных взаимодействиях, построении гнезд, рабочей памяти и памяти распознавания объектов. Этот мутантный фенотип может означать, что подукт гена bex3 препятствует взаимодействию комплекса TSC1/2 (TSC Complex Subunit 1, подавляющий опухоль ген, кодирующий рост-ингибирующий белок хамартин) с мишенью рапа-мицина у мелкопитающих mTORС1/mTORC2, угнетая этот путь. mTORC2 принадлежит к семейству фосфатидилинозитол-киназ. Они опосредуют клеточные реакции на стрессы (в частности, в ответ на повреждения ДНК). Эта киназа представляет собой компонент двух разных комплексов — mTORC1, контролирующего общий синтез белка, рост и пролиферацию клеток, и mTORC2, который служит регулятором актинового цитоскелета и способствует выживанию клеток (21).
Ингибиторы mTOR используются при трансплантации органов в качестве иммунодепрессантов и оцениваются на предмет терапевтического потенциала при инфекциях SARS-CoV-2 (20). Мутации в гене белка mTOR связаны с синдромом Смита-Кингсмора (характеризуется макроцефалией, умственной отсталостью, судорогами) и соматической очаговой кортикальной дисплазией II типа (21). Ген ANGPTL7 (Angiopoietin Like 7, участвует в негативной регуляции развития карпиллярной сети) расположен в интроне mTOR . Высказано предположение, что нарушение регуляции ANGPTL7 под влиянием Bex3 связано с расстройством аутистического типа у людей.
Для описания этапов кооптации генов предложена следующая схема (19). На первом этапе мотив прото-BGW (Bex/GASP/Wex элемент, общий для трех генов bex , gasp , wex ) (21) присутствовал в Х-хромосоме предка плацентарных и сумчатых в положении выше по отношению к промотору гена gla альфа-галактозидазы ( p α ). На втором этапе в линии плацентарных ре-тротранскрибированный эндогенный ретровирус Hnrnph1 встраивался рядом с мотивом bgw в позиции выше gla , что привело к появлению ретрогена hnrnph2 . На третьем этапе область, содержащая кооптированные мотивы bgw и hnrnph2 , подверглась дупликации, рядом происходили вставки ретротранспозонов, подобных HAL1b и L1ME. На четвертом этапе bgw и открытая рамка считывания ORF, образовавшиеся при введении ретротранспозонов, соответствовали по нуклеотидным последовательностям прото- Bex / Tceal с сохраненным сайтом связывания YY1 из HAL1b. На следующем этапе мотив BGW и сайт связывания YY1 гена Bex / Tceal дублировались в положении выше ретрокопии гена armc10 , что привело к появлению наследуемого гена armcX (принадлежит к семейству сигнальных генов, локализованных в хромосоме Х, которые кодируют в белках повтор Армадилло и обладают опу-холь-подавляющей активностью) . На завершающем шестом этапе (еще до диверсификации плацентарной линии млекопитающих) семейства генов bex / tceal и armcx расширились, образуя кластер BGW.
Х-хромосома содержит непропорционально большое число генов, связанных с ментальными функциями, что подтверждается преобладанием случаев умственной отсталости у мужчин. Однако все девять сцепленных с Х-хромосомой генов, которые при мутации приводят к ментальным нарушениям, имеют ортологи у рыб или даже у более ранних эукариот. Оказалось также, что все гены bex , wex и gasp экспрессируются в головном мозге (21). Таким образом, эти специфичные для плацентарных видов гены могут рассматриваться как возможные кандидаты для участия в адаптивной эволюции неокортекса — уникальной для млекопитающих области переднего мозга (22).
Присутствие консервативного элемента BGW в пределах 5´-UTR генов bex , wex и gasp позволяет предполагать его участие в регуляции трансляции (21). Основанием служит то, что, как ранее было показано, на скорость трансляции влияют регуляторные районы, включающие консенсусную последовательность, вторичные структуры, предшествующие AUG (сайт ин-циации трансляции), внутренние сайты входа в рибосому и сайт распознавания последовательностей, специфичных для регуляторных факторов, таких как белок или РНК. Наличие трансляционного контроля для генов bex , wex и gasp может указывать на то, что кодируемые ими белки используются в определенных физиологических условиях, на определенных стадиях развития или в субклеточных компартментах. Еще одной возможной ролью элемента BGW может быть регуляция альтернативного сплайсинга (21).
Эти же авторы выявили гены с кодирующей последовательностью, перекрывающейся более чем на 50 % с аннотированными TEs и присутствующей более чем у одного вида (28 генов у человека и 9 генов у мыши) (21).
У растений описаны различные гены, мутации которых вовлекаются в искусственный отбор и обусловлены встраиванием ТЕs либо в кодирующие последовательности, либо в регуляторные мотивы (23-25). Такие же события обнаруживаются в генах у разных видов животных, в частности у кур (26, 27).
Ретротранспозоны LINE и SINE в геномных изменениях. Масштабность участия ретротранспозонов в геномных перестройках недавно была выявлена при сопоставлении результатов полногеномного секвенирования генома человека (28). В этой работе рассматривается трансдукция геномных элементов человека, связанная с такими ретротранспозонами, как L1 и SVA. При их транскрипции сигнал ее окончания на 3´-конце может игнорироваться РНК-полимеразой. В результате транскрипция завершается на геномном элементе хозяина, создавая химерный транскрипт. В работе проанализировали 3202 секвенированных генома у 26 групп населения из разных стран и идентифицировали 7103 полиморфных L1 и 3040 полиморфных SVA. Среди них обнаружили 268 и 162 варианта трансдукций 3´-участков длиной от 7 до 997 нуклеотидов с участием соответственно последовательностей, гомологичных L1 и SVA. В X-, 6-й и 7-й хромосомах выявлены специфические локусы с наиболее широко представленными L1 и SVA, которые, среди прочего, обусловливали наибольшее число трансдукций.
Особое значение имеют процессы, связанные с ТЕs, приводящие к изменению копийности генов (утрата или увеличение их числа) (29).
Взаимодействия между ТЕs и геномом хозяина имеют сложный характер и включают в каждом конкретном случае большое число различных механизмов (30). Множественные варианты встроек ТЕs в геном хозяина и взаимодействий с его различными геномными элементами может приводить к неодинаковым последствиям, включая как конфликтные, так и благоприятные взаимовыгодные отношения (31). К примерам конфликтных взаимоотношений относятся вставки TEs в экзоны, что приводит к мутациям со сдвигом рамки считывания и нарушениям структуры и функции белка. TEs также способны повышать геномную нестабильность, формируя структуры, создающие участки гомологии вдоль хромосом, что приводит к хромосомным перестройкам — дупликациям, делециям, инверсиям и транслокациям. При вставках TEs в регуляторные участки, например 5´-, 3´-обла-сти или интроны генов, происходят эпигенетические модификации, которые становятся причиной неадекватной активации или подавления экспрессии генов (31).
Примером кооперативных отношений (31) может быть использова- ние TEs геномом хозяина для генерирования новых регуляторных сигналов или кодирующих последовательностей. Такой процесс называют молекулярным одомашниванием (31). TEs способны вносить новые энхансерные последовательности для факторов транскрипции, изменяющие пространственно-временную регуляцию экспрессии генов. У дрозофилы после потери теломеразы автономные (лишенные LTR) ретротранспозоны HeT-A, TAHRE и TART активно участвуют в поддержании теломер. TEs могут способствовать поддержанию архитектуры генома, обеспечивая сайты связывания для белка CTCF, который отвечает за создание топологически ассоциированных доменов (TAD). Топологически ассоциированные домены непосредственно участвуют в сборке программ генной экспрессии (32), что существенно зависит от распределения и перемещений по геному различных ТЕs.
Следует отметить, что LINE широко представлены почти у всех эукариот (33). Длинные диспергированные ядерные элементы LINE1 — наиболее успешное семейство TEs у сухопутных млекопитающих. Длина LINE1 варьируется в пределах 6 тыс. п.н. LINE1 несут гены двух основных белков — ORF1 и ORF2 (аналогичные генам gag и pol экзогенных ретровирусов), участвующих в механизмах транспозиций. Продукт ORF1 представляет собой РНК‐связывающий белок, ORF2 обладает ферментативной активностью эндонуклеазы (EN) и обратной транскриптазы (RT). Несмотря на обилие, большинство элементов LINE1 не транспозируются из-за накопления мутаций. Из примерно 8 868 000 и 599 000 элементов LINE1 в геномах человека и мыши соответственно только 80-100 и около 2300 LINE1s транспозиционно компетентны (34).
Инсерция LINE1 происходит в основном через эндонуклеазозависимую обратную транскрипцию. РНК LINE1 формирует в цитоплазме комплекс с несколькими гомотримерами ORF1p и по крайней мере одним димером ORF2p с образованием рибонуклеопротеина (LINE1 RNP). РНП LINE1 проникает в клеточное ядро, где эндонуклеаза ORF2p освобождает 3´-гидроксильную группу. Свободная 3´-гидроксильная группа затем используется обратной транскриптазой ORF2p в качестве праймера для синтеза кДНК LINE1, начиная с хвоста polyA-мРНК LINE1. Хвост polyA-мРНК неавтономного короткого диспергированного геномного элемента (SINE) может конкурировать с polyA-мРНК LINE1 за обратную транскриптазу LINE1 ORF2p, таким образом захватывая механизм LINE1 для транспозиции. Кроме того, LINE1 ORF2p также может ретротранспозировать уникальные мРНК, кодирующие белки, и малые ядерные РНК.
Ретротранспозиции LINE1 зависят от других клеточных белков. Выявлен ряд положительных регуляторов транспозиций, к которым относятся, в частности, нуклеолин и гетерогенные ядерные рибонуклеопротеины (hnRNPs), митоген‐активированные протеинкиназы и циклин‐зави-симые киназы. Для завершения вставки LINE1 требуется механизм репарации ДНК. Имеется несколько механизмов ограничения транспозиции LINE1 в геномах млекопитающих на транскрипционном, посттранскрипционном и посттрансляционном уровнях. Транскрипция LINE1 подавляется с помощью метилирования CpG-участков ДНК и репрессивных модификаций гистонов на промоторе LINE1. Различные KRAB‐ZFPs (белки, несущие мотив взаимодействия с ДНК «цинковые пальцы») избирательно распознают ERV и LINE1 и привлекают KAP1 (белок гетерохроматина). Посттранскрипционное подавление мРНК LINE1 опосредуется через РНК-интерференцию малыми РНК. Ретротранспозиция LINE1 также может быть ограничена посттрансляционно участием ряда стимулируемых интерфероном генов (34).
Предполагаемая частота новых вставок LINE1 составляет примерно 1 на 100 рождений у человека и на 8 — у мышей. Возможности влияния LINE1 на работу генома множественны и включают, в частности, индукцию геномных инсерцией, управление ретротранспозицией элементов SINE/Al (которые сами выступают в роли основных модуляторов геномной измен-чивостиии). Эндонуклеазная активность LINE1 ORF2 является потенциальным индуктором мутагенных эффектов независимо от ретротранспозиции. Элементы LINE1 и SINE могут способствовать изменениям количества повторов в микросателлитных локусах, особенно богатых АТ, которых много в геномах.
Таким образом, благодаря распространенности в геномах элементы LINE1 и SINE могут индуцировать крупномасштабные геномные изменения, такие как дупликации, инверсии. Принято считать, что в среднем любые два гаплоидных генома человека различаются примерно на 1000 вставок TEs, главным образом из семейств LINE1 или Alu (34).
Эпигенетические эффекты транспозиций LINE и SINE. Присущее повторам L1 (LINE) и B1/Alu (SINE) свойство самосборки обеспечивает многочисленные точки для инициирования образования ядерных подобластей в интерфазном ядре. Повторяющиеся последовательности ДНК также служат якорными сайтами, необходимыми для функционирования механизмов транскрипции, для связывания регуляторных белков и РНК. ТЕs могут влиять на профили генной экспрессии за счет того, что их ДНК или РНК-транскрипты могут взаимодействовать с ДНК- и(или) РНК-свя-зывающими белками. Скопление молекул может приводить к агрегации содержащих один и тот же тип повтора, образуя в ядре отдельные компарт-мены и, таким образом, менять упаковку генома. Ядерная сегрегация ком-партментов, богатых L1 или B1/Alu, может дополнительно усиливаться связыванием последовательностей их ДНК с субъядерными структурами (соответственно с ядерными спеклами или ядрышком), которые служат каркасами для стабилизации ядерной архитектуры (32).
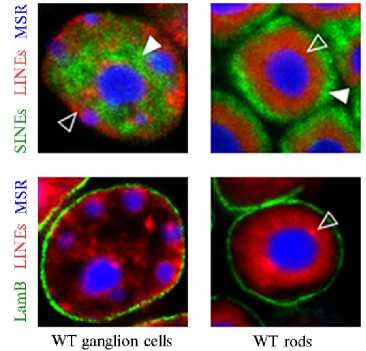
Рис. 2. Распределение маркеров гетерохроматина LINE (красный цвет) , эухроматина SINE (зеленый цвет) , микросателлитных последовательностей MSR (синий цвет) , LamB (белок ламины — белковой сети, подлежащей под ядерной оболочкой, зеленый цвет) в ядрах клеток нервных ганглиев (слева, WT ganglion cells) и в цилиндрических клетках фоторецепторов сетчатки глаза (справа, WT rods) (35).
По-видимому, наиболее наглядный пример участия тандемных и диспергиованных повторов в регуляции смены программ генной экспрессии через динамические изменения в архитектонике интерфазного ядра описан в работе, выполненной на интер фазных ядрах клеток цилиндрических фоторецепторов сетчатки ночных млекопитающих.
У них наблюдалась инвертированная картина локализации гетерохроматина и эухроматина по сравнению с другими ядрами: гетерохроматин размещался внутри, эухроматин — на периферии ядра под ламиной (рис. 2) (35). Авторы этого исследования приходят к выводу, что существенный вклад в такие раздичия могут вносить, в частности, контакты между гомологичными диспегированными повторами, локализованными в разных районах хроматина.
У группы близкородственных видов щетинистой крысы обнаружены выраженные межвидовые различия по хромосомному позиционированию ретротанспозонов LINE и SINE, что позволило высказать предположение об их вовлеченности в видообразование (36).
Интересно отметить, что вовлеченность взаимного позиционирования хромосом и их участков (доменов) в интерфазных ядрах в межвидовую дифференциацию обсуждалась достаточно давно В.Н. Стегнием (37) на примере политенных хромосом малярийного комара. Им же было выявлено участие мобильных генеических элементов, в частности LINE, в контактах хромосом с ламиной интерфазного ядра (38).
Широкое распространение элементов LINE1 в геномах млекопитающих делает их хорошими кандидатами на роль центров локальной организации хроматина и архитектуры хроматина более высокого порядка. Интересно, что элементы LINE1 и SINE, число которых в геноме зависит от активности LINE1 ORF2, имеют противоположное геномное распределение. LINE1 чаще встречаются в молчащих, почти не содержащих генов ге-терохроматизированных областях, богатых АТ-нуклеотидами, тогда как элементы SINE превалируют в богатых генами, экспрессирующихся эухрома-тических, богатых GC-нуклеотидами областях. Аналогичное общее подразделение геномных областей наблюдается между компартментами B и A (соответственно неэкспресирующиеся и экспресирующиеся районы генома) в интерфазном ядре. Предполагается, что наличие элементов LINE1 и SINE коррелирует с присутствием соответственно компартментов B и A, причем LINE1 и SINE могут прямо участвовать в формировании хромосомных областей этих двух компартментов. Высокая ассоциация РНК повторяющихся последовательностей, включая РНК LINE1, с хроматином, и доказательства того, что связанная с хроматином РНК способствует глобальной организации хроматина, свидетельствуют в пользу этого предположения (35).
Интересным фактом представляются данные, указывающие на то, что регуляторные эффекты встроек LINE в геном хозяина могут различаться в зависимости от функции генов (39). Сравнительный анализ локализации LINE1 и LINE2 в регуляторных последовательностях (промоторы, энхансеры) у генов, экспрессия которых тканеспецифична, и общих генов (генов домашнего хозяйства) позволил обнаружить относительно повышенную скорость эволюции тканеспецифичных генов, в чем активно участвует встраиваемый ретротранспозон LINE1. В регуляторных последовательностях генов домашнего хозяйства чаще присутствует более древний ретротранспозон LINE2 (39).
TEs, в частности LINE1 и различные ERV, глобально деметилируются и экспрессируются во время основных волн эпигенетического стирания, которые происходят на этапах предимплантации и развития зародышевой линии (34, 39). Аналогичным образом ослабление репрессии TEs в соматических клетках при деметилировании H3K9me2 происходит в искусственно индуцируемых плюрипотентных стволовых клетках (39). Многие семейства TEs действуют как важные цис‐элементы регуляции генов (39). Однако становится очевидным, что продукты генов TEs также играют существенную роль в раннем развитии организма. РНК, полученная при транскрипции элементов эндогенного ретровируса HERVH, важна для поддержания недифференцированного состояния эмбриональных стволовых клеток человека (возможно, действуя как длинная некодирующая РНК — lncRNAs, участвующая в регуляторных сетях генной экспрессии). Кодируемый эндогенным ретровирусом HERVK белок Rec экспрессируется на высоком уровне в эмбрионах человека, где он увеличивает противовирусную резистентность за счет усиления регуляции белков IFITM1 (индуцируемый интерфероном трансмембранный белок 1) (39). Rec также образует комплексы с подмножеством эндогенных РНК, регулируя таким образом их связывание с рибосомами и, как следствие, уровни экспрессии белков, что может быть важно для раннего эмбрионального развития.
Предполагается, что механизмы ограничения мутагенной активности ТЕs могут рассматриваться с точки зрения баланса между полезными и негативными функциями ТЕs, который обеспечивает воспроизводимое развитие организма до репродуктивного возраста, при этом сохраняя способность быстро генерировать генетическую новизну в изменяющейся среде. Однако такая метастабильность может в некоторых случаях привести к заболеванию в результате неблагоприятных событий ретротранспозиций. Другими словами, потенциальная реактивация ТЕs во взрослых соматических клетках и их связь с такими заболеваниями, как рак, может быть платой за значение ТЕs для развития и эволюционирования организмов (40).
Эндогенные ретровирусы. Эндогенные ретровирусы проявляют многие функции, влияющие на нормальную биологию клеток-хозяев (41). Некоторые из этих функций непосредственно связаны с взаимодействиями с экзогенными ретровирусами. Они могут включать интерференцию рецепторов, иммунную самотолерантность, рекомбинацию и одновременное действие ограничений и стимулирования экзогенной ретровирусной инфекции с использованием различных механизмов.
Эндогенные ретровирусы (ERV) делятся на разные классы и являются прямыми потомками экзогенных ретровирусов. Ретровирусы (экзогенные и эндогенные) подразделяют на три класса (I, II и III), каждый из которых соответствует расширенным группам ERV1, ERV2 и ERV3. Ретровирусы класса I — это эндогенные (ERV1) и экзогенные гамма- и эпсилон-вирусы, класса II — ERV2 и экзогенные альфа-, бета-, дельта-ретровирусы и лентивирусы, класса III — спумавирусы и ERV3 (ERV3/ERVL млекопитающих). Группа ERV4 в настоящее время включает ранее выявленный и описанный у крокодилов CrocERV, поскольку этот ERV отличается от ретровирусов любых других известных групп и, в то же время, со всеми имеет участки гомологии (42).
Геномы беспозвоночных эукариот также содержат ретровирусоподобные LTR-содержащие элементы — так называемые LTR-ретротранспозоны. Они делятся на три группы: группу Pseudoviridae (Copia/Ty1), присутствующую у растений и грибов, группу Metaviridae (Gypsy/Ty3), которая также встречается в растениях и грибах, и группу Semotivirus (Bel/Pao), выявленную у многоклеточных животных. Наиболее разнообразна группа Metaviridae , которая включает примерно 10 подгрупп. Одна из них (хромовирусы) имеет более широкий спектр хозяев (растения, грибы и позвоночные). Хромовирусы получили свое название потому, что их ген pol кодирует интегразу с хромодоменом (домен модификатора организации хроматина), нуклеосомсвязывающая часть которой может опосредовать встройку провируса, специфичную для последовательности генома хозяина (43).
Обнаруживаются участки гомологии между различными таксономически удаленными ретровирусами, в том числе патогенными, в частности с вирусами иммунодефцита человека, бычьего лейкоза, хромовирусами (в центромерах чаще у растений), ретровирусами полихет, что свидетельствует о широких масштабах рекомбинаций между этими вирусами (43).
Описан уникальный пример интеграции экзогенного ретровируса в геном коалы, которая происходила под наблюдением исследователей (44). Наглядно показано, как эндогенные ретровирусы возникают в результате инфекции клеток, формирующих гаметы. При ретровирусной инфекции РНК вирусного генома подвергается обратной транскрипции в провирус-ную ДНК, которая впоследствии интегрируется в геном хозяина. Иногда интеграция может происходить и в первичных эмбриональных клетках, что может приводить к образованию эмбриона и затем потомства с интегрированным провирусом во всех клетках. На протяжении многих поколений хозяина провирусная ДНК претерпевает существенные мутационные изменения (однонуклеотидные полиморфизмы, вставки и делеции), что обычно приводит к неспособности продуцировать инфекционный вирус (45).
Следует отметить, что ТЕs широко вовлечены в эпигеномную изменчивость, влючая изменения рисунка метилирования, модификацию гистонов, формирование микроРНК, трансгенерационное наследование (46, 47). Можно ожидать, что конструирование новых ниш, в которых участвуют человек и доместицируемые виды, способствует активации ТЕs и формированию новых регуляторных сетей на их основе (48-50).
Некодирующие РНК (ncRNA), микроРНК. Основные источники некодируюей РНК. Помимо функций информационных, рибосомных и транспортных РНК, многие другие РНК (некодирующие РНК — нкРНК, ncRNA) выполяют регуляторную роль у эукариот (51). Они действуют как регуляторы функциональной активности нуклеиновых кислот, распознавая у них специфические последовательности-мишени по гомологии, и участвуют в регуляции роста, развития и стрессовых реакций животных и растений. Регуляторные нкРНК, начиная от коротких и заканчивая длинными (lncRNAs), осуществляют контроль над широким спектром биологических процессов. В зависимости от способа биогенеза и функции нкРНК эволюционировали в различные формы, включают микроРНК (microРНК), малые интерферирующие РНК (siРНК), варианты микроРНК (isomiRNAs), длинные некодирующие РНК (lncRNAs), кольцевые РНК (circRNAs) и производные некодирующих РНК (51). Один из элементов регуляторных сетей — микроРНК, которая имеет шпилечную структуру и представляет собой производное ТЕ и других геномных элементов (51).
В эукариотических клетках экспрессия генов регулируется на нескольких уровнях. На посттранскрипционном уровне регуляция модулируется различными транс-действующими факторами, которые связываются со специфическими последовательностями в мРНК. Это влияет на различные процессы, такие как скорость деградации и эффективность трансляции мРНК, сплайсинг, локализация (52). МикроРНК в сочетании с ферментом Argonaute образуют комплекс сайленсинга, индуцируемый микроРНК (miRISC, RNA-induced silencing complex), который использует комплементарную нуклеотидную последовательность для подавления транскрипта-мишени. РНК-связывающие белки (RBP) способствуют посттранскрипционному контролю, влияя на стабильность и трансляцию мРНК при связывании с цис-элементами в транскрипте мРНК. RBP влияют на экспрессию генов через miRISC или его взаимодействие с мРНК-мишенью (52).
LINE 1 и эндогенные ретровирусы (ERV) индуцируют формирование дцРНК, образованных из конвергентных транскриптов или шпилечных структур в результате сквозной транскрипции элементов, расположенных голова к голове/хвост к хвосту. Элементы Alu (SINE) намного короче и образуют шпилечные структуры, а также открытые гибриды дцРНК (53).
МикроРНК в последние годы стали объектом глубоких исследований. Накоплен большой объем данных о вовлеченности разных микроРНК в регуляцию этапов развития и реакций на стресс у растений (51), а также в контроль оплаты кормов у некоторых основных видов сельскохозяйственных животных (54, 55).
Эпигенетические механизмы, такие как метилирование ДНК, модификация гистонов, экспрессия некодирующих РНК, представляются особенно важными при ответе многоклеточных организмов на стрессирующие факторы окружающей среды (54). Кроме того, абиотический стресс, например тепловой шок, может индуцировать подавление TEs, вызывая разрушение RISC под действием индуцируемого шаперона Hsp70, который направляет комплекс RISC в лизосомы.
В связи с вовлеченностью молекул микроРНК в эпигенетическую изменчивость были выполнены исследования их геномного распределения у ряда доместицированных видов млекопитающих (корова, собака, лошадь, свинья и кролик). Получены данные о преимущественной локализации микроРНК в интронах и межгенных пространствах (55). Обнаружено, что в процессе эволюции происходит появление новых микроРНК и утрата имевшихся (55).
Результаты сравнительного анализа так называемых молодых и старых ортогрупп (общих по происхождению) микроРНК в разных тканях у исследованных видов свидетельствовали о том, что экспрессия молодых групп имеет более выраженные тканеспецифичные особенности по сравнению со старыми группами (55). Локализация примерно 20 % новых ортогрупп ограничена головным мозгом, и их целевые мишени, по-видимому, обогащены геномными элементами для обеспечения активности нейронов и процессов их дифференцировки.
Изменения в регуляторной сети микроРНК обнаружены и при доместикации. Так, сравнение вымерших предшественников крупного рогатого скота Bos primigenius с представителями современного вида B. taurus показало, что некодирующие микроРНК стали ключевыми регуляторами пространственно-временной экспрессии генов-мишеней, контролирующих рост и развитие млекопитающих (56). В процессе одомашнивания отбор мутационных изменений в микроРНК и(или) сайтах связывания микроРНК мог бы обеспечить механизм для генерации некоторых признаков, которые отличают одомашненный крупный рогатый скот от их диких предшественников. Выполнен анализ последовательностей ДНК с открытой рамкой считывания для 19 994 пар ортологичных белок-кодирующих генов из существующих геномов Bos taurus и одного вымершего генома B. primigenius. Идентифицированы полиморфизмы сайтов связывания миРНК в 3´-UTR у 1620 этих ортологичных генов. Выявленные 1620 генов с сайтами связывания миРНК, которые различаются у B. taurus и диких предшественников, представляют собой гены-кандидаты, ассоциированные с доместикацией. Обнаружено, что эти 1620 генов-кандидатов участвуют в контроле пигментации, фертильности, нейробиологических процессов, метаболизма, иммунитета и характеристик продуктивности животных (включая качество молока и эффективность конверсии корма) (56). Полученные результаты свидетельствуют о том, что направленный отбор регуляторных вариантов миРНК был важен при одомашнивании и последующем искусственном отборе, который дал начало современному европейскому крупному рогатому скоту (56).
Показано, что доместицированные виды отличаются от близкородственных диких относительно повышенной частотой делеций (57), то есть мутаций, характер которых также тесно связан с ретротранспозонами (58).
Имеются и прямые результаты экспериментальных исследований, свидетельствующие о существенных различиях в распределении ТЕs у таких видов, как домашняя собака ( Canis lupus familiaris ), серый волк ( Canis lupus ) и красный волк ( Cuon alpinus ), которые связывают с процессами одомашнивания (59). Выявлены различия между геномами этих видов. Так, на долю TEs у собаки приходится 41,75 % нуклеотидных последовательностей генома, что выше, чем у серого волка (39,26 %) и красного волка (38,51 %). Наиболее дивергировавшие компоненты ТЕs в этих геномах — длинные диспергированные ядерные элементы LINE1 (L1) и микросателлиты, отличающие собаку от серого волка на 86,1 %, от красного волка — на 83,2 %.
Сравнение распределения мобильных генетических элементов между домашним кроликом ( Oryctolagus cuniculus domesticus ) и пищухой ( Ochotona princeps ) показало заметно большую частоту встречаемости у домашнего вида по сравнению с диким (60). Сходные различия выявлены и при сравнении домашней кошки ( Felis silvestris catus ) и близкородственного дикого вида ( Felis silvestris silvestris ) (61).
Таким образом, ответ на ключевой вопрос доместикации — откуда берется большой размах фенотипической изменчивости и почему одни виды поддаются одомашниванию, а другие нет, по нашему мнению, может заключаться в следующем. В процессе формирования новой многокомпо-нентой ниши человек (доместикатор)—растения/животные (доместицируе-мые) в геноме доместицируемого формируется повышенная плотность мобильных генетических элементов, основанная на пониженной резистентности геномов к интеграции транспозонов. В пользу этого предположения свидетельствуют вовлеченность транспозонов в эпигенетическую изменчивость (31, 51) и в организацию архитектоники интерфазного ядра (35), высокая скорость их эволюции (34), частоты рекомбинаций между ними (43, 58), разичия между домашними и дикими видами по распространенности некоторых транспозонов в геномах (56, 59-61). В наших предыдущих исследованиях мы отмечали, что основным нерешенным вопросом в поисках ведущих геномных особенностей у доместицированных видов при сравнении с близкородственными дикими является источник изменчивости, отличающий эти близкородственные формы (62, 63). Накопленные данные о ведущей роли транспозонов в эволюционных преобразованиях, в формировании и модуляциях регуляторных сетей, контролирующих профили генной экспрессии, дают основания полагать, что мобильные генетические элементы имеют существенное значение и для процессов доместикации.
Итак, по нашему мнению, высокая способность мобильных генетических элементов (ТЕs) генерировать изменчивость, относительно повышенная частота их встречаемости в геномах доместицированных видов по сравнению с близкородственными дикими, вовлеченность ТЕs в формирование регуляторных сетей профилей генной экспрессии позволяет предполагать, что формирование новых условий обитания и воспроизводства, контролируемых человеком, приводит к увеличенным контактам доместициру-емых форм с широким спектром новых для них экзогенных вирусов, вызывающих, в конечном итоге, повышенную изменчивость (в том числе и ре- гуляторных сетей), способствующую фенотипическому разнообразию и эффективности искусственного отбора. Такой или близкий к нему механизм, по-видимому, должен вовлекаться во все эволюционные процессы, связанные с появлением новых форм.
Авторы выражают искреннюю благодарность к.б.н. Ларисе Михайловне Федоровой за интерес, проявленный к работе, плодотворную дискуссию и полезные советы при подготовке статьи к опубликованию.
Список литературы Геномные источники разнообразия как драйверы доместикации (обзор)
- Andersson L., Purugganan M. Molecular genetic variation of animals and plants under domestication. Proc. Natl. Acad. Sci. USA, 2022, 119(30): e2122150119 (doi: 10.1073/pnas.2122150119).
- Дарвин Ч. Изменения домашних животных и культурных растений. Соч. Т. 4. М.-Л., 1951.
- Diamond J. Evolution, consequences and future of plant and animal domestication. Nature, 2002, 418(6898): 700-707 (doi: 10.1038/nature01019).
- Zeder M.A. Core questions in domestication research. Proc. Natl. Acad. Sci. USA, 2015, 112(11): 3191-3198 (doi: 10.1073/pnas.1501711112).
- Denham T., Barton H., Castillo C., Crowther A., Dotte-Sarout E., Florin S.A., Pritchard J., Barron A., Zhang Y., Fuller D.Q. The domestication syndrome in vegetatively propagated field crops. Ann. Bot., 2020, 125(4): 581-597 (doi: 10.1093/aob/mcz212).
- Burban E., Tenaillon M.I., Le Rouzic A. Gene network simulations provide testable predictions for the molecular domestication syndrome. Genetics, 2022, 220(2): iyab214 (doi: 10.1093/genet-ics/iyab214).
- Rubio A.O., Summers K. Neural crest cell genes and the domestication syndrome: a comparative analysis of selection. PLoS ONE, 2022, 17(2): e0263830 (doi: 10.1371/journal.pone.0263830).
- Glazko V.I., Kosovsky G.Yu., Glazko T.T. Human and domesticated species (review). Biogeosys-tem Technique, 2021, 8(1): 34-45 (doi: 10.13187/bgt.2021.1.34).
- Glazko V., Zybailov B., Glazko T. Asking the right question about the genetic basis of domestication: what is the source of genetic diversity of domesticated species? Adv. Genet. Eng., 2015, 4: 2 (doi: 10.4172/2169-0111.1000125).
- Shapiro J.A. Nothing in evolution makes sense except in the light of genomics: read-write genome evolution as an active biological process. Biology (Basel), 2016, 5(2): 27 (doi: 10.3390/biol-ogy5020027).
- Shapiro J.A. Living organisms author their read-write genomes in evolution. Biology (Basel), 2017, 6(4): 42 (doi: 10.3390/biology6040042).
- Shapiro J.A. What we have learned about evolutionary genome change in the past 7 decades. Biosystems, 2022, 215-216: 104669 (doi: 10.1016/j.biosystems.2022.104669).
- Trono D. Transposable elements, polydactyl proteins, and the genesis of human-specific transcription networks. Cold Spring Harb. Symp. Quant. Biol., 2015, 80: 281-288 (doi: 10.1101/sqb.2015.80.027573).
- Ricci M., Peona V., Guichard E., Taccioli C., Boattini A. Transposable elements activity is positively related to rate of speciation in mammals. J. Mol. Evol., 2018, 86(5): 303-310 (doi: 10.1007/s00239-018-9847-7).
- Rebollo R., Horard B., Hubert B., Vieira C. Jumping genes and epigenetics: towards new species. Gene, 2010, 454(1-2): 1-7 (doi: 10.1016/j.gene.2010.01.003).
- Oliver K.R., Greene W.K. Mobile DNA and the TE-Thrust hypothesis: supporting evidence from the primates. Mobile DNA, 2011, 2: 8 (doi: 10.1186/1759-8753-2-8).
- Oliver K.R., Greene W.K. Transposable elements: powerful facilitators of evolution. BioEssays, 2009, 31(7): 703-714 (doi: 10.1002/bies.200800219).
- Almojil D., Bourgeois Y., Falis M., Hariyani I., Wilcox J., Boissinot S. The structural, functional and evolutionary impact of transposable elements in eukaryotes. Genes (Basel), 2021, 12(6): 918 (doi: 10.3390/genes12060918).
- Herrera-Úbeda C., Garcia-Fernandez J. New genes born-in or invading vertebrate genomes. Front. Cell Dev. Biol., 2021, 9: 713918 (doi: 10.3389/fcell.2021.713918).
- Navas-Pérez E., Vicente-García C., Mirra S., Burguera D., Fernandez-Castillo N., Ferrán J.L., López-Mayorga M., Alaiz-Noya M., Suárez-Pereira I., Antón-Galindo E., Ulloa F., Herrera-Úbeda C., Cuscy P., Falcón-Moya R., Rodríguez-Moreno A., D'Aniello S., Cormand B., Marfany G., Soriano E., Carriyn Á.M., Carvajal J.J., Garcia-Fernandez J. Characterization of an eutherian gene cluster generated after transposon domestication identifies Bex3 as relevant for advanced neurological functions. Genome Biol, 2020, 21(1): 267 (doi: 10.1186/s13059-020-02172-3).
- Winter E.E., Ponting C.P. Mammalian BEX, WEX and GASP genes: coding and non-coding chimaerism sustained by gene conversion events. BMC Evol. Biol., 2005, 5: 54 (doi: 10.1186/14712148-5-54).
- Cabeen R.P., Toga A.W., Allman J.M. Mapping frontoinsular cortex from diffusion microstructure. Cerebral Cortex, 2022: bhac237 (doi: 10.1093/cercor/bhac237).
- Bhattacharyya M.K., Smith A.M., Ellis T.H., Hedley C., Martin C. The wrinkled-seed character of pea described by Mendel is caused by a transposon-like insertion in a gene encoding starch-branching enzyme. Cell, 1990, 60(1): 115-122 (doi: 10.1016/0092-8674(90)90721-p).
- Hazzouri K.M., Flowers J.M., Visser H.J., Khierallah H.S.M., Rosas U., Pham G.M., Meyer R.S., Johansen C.K., Fresquez Z.A., Masmoudi K., Haider N., El Kadri N., Idaghdour Y., Malek J.A., Thirkhill D., Markhand G.S., Krueger R.R., Zaid A., Purugganan M.D. Whole genome re-sequencing of date palms yields insights into diversification of a fruit tree crop. Nat. Commun., 2015, 6: 8824 (doi: 10.1038/ncomms9824).
- Feschotte C., Jiang N., Wessler S.R. Plant transposable elements: where genetics meets genomics. Nat. Rev. Genet., 2002, 3(5): 329-341 (doi: 10.1038/nrg793).
- Matsumine H., Herbst M.A., Ou S.H., Wilson J.D., McPhaul M.J. Aromatase mRNA in the extragonadal tissues of chickens with the henny-feathering trait is derived from a distinctive promoter structure that contains a segment of a retroviral long terminal repeat. Functional organization of the Sebright, Leghorn, and Campine aromatase genes. J. Biol. Chem, 1991, 266(30): 19900-19907.
- Li J., Davis B.W., Jern P., Dorshorst B.J., Siegel P.B., Andersson L. Characterization of the endogenous retrovirus insertion in CYP19A1 associated with henny feathering in chicken. Mobile DNA, 2019, 10: 38 (doi: 10.1186/s13100-019-0181-4).
- Halabian R., Makalowski W. A map of 3' DNA transduction variants mediated by non-LTR retroelements on 3202 human genomes. Biology, 2022, 11: 1032 (doi: 10.3390/biology11071032).
- Crow M., Suresh H., Lee J., Gillis J. Coexpression reveals conserved gene programs that co-vary with cell type across kingdoms. Nucleic Acids Research, 2022, 50(8): 4302-4314 (doi: 10.1093/nar/gkac276).
- Nicolau M., Picault N., Moissiard G. The evolutionary volte-face of transposable elements: from harmful jumping genes to major drivers of genetic innovation. Cells, 2021, 10(11): 2952 (doi: 10.3390/cells10112952).
- Colonna Romano N., Fanti L. Transposable elements: major players in shaping genomic and evolutionary patterns. Cells, 2022, 11(6): 1048 (doi: 10.3390/cells11061048).
- Lu J.Y., Chang L., Li T., Wang T., Yin Y., Zhan G., Han X., Zhang K., Tao Y., Percharde M., Wang L., Peng Q., Yan P., Zhang H., Bi X., Shao W., Hong Y., Wu Z., Ma R., Wang P., Li W., Zhang J., Chang Z., Hou Y., Zhu B., Ramalho-Santos M., Li P., Xie W., Na J., Sun Y., Shen X. Homotypic clustering of L1 and B1/Alu repeats compartmentalizes the 3D genome. Cell Research, 2021, 31(6): 613-630 (doi: 10.1038/s41422-020-00466-6).
- Ivancevic A.M., Kortschak R.D., Bertozzi T., Adelson D.L. LINEs between species: evolutionary dynamics of LINE-1 retrotransposons across the eukaryotic tree of life. Genome Biol Evol., 2016, 8(11): 3301-3322 (doi: 10.1093/gbe/evw243).
- Percharde M., Sultana T., Ramalho-Santos M. What doesn't kill you makes you stronger: transposons as dual players in chromatin regulation and genomic variation. BioEssays, 2020, 42(4): e1900232 (doi: 10.1002/bies.201900232).
- Falk M., Feodorova Y., Naumova N., Imakaev M., Lajoie B.R., Leonhardt H. Joffe B., Dek-ker J., Fudenberg G., Solovei I., Mirny L.A. Heterochromatin drives compartmentalization of inverted and conventional nuclei. Nature, 2019, 570(7761): 395-399 (doi: 10.1038/s41586-019-1275-3).
- Soares S.C., Eler E.S., E Silva C.E.F., da Silva M.N.F., Araujo N.P., Svartman M., Feldberg E. LINE-1 and SINE-B1 mapping and genome diversification in Proechimys species (Rodentia: Echimyidae). Life Science Alliance, 2022, 5(6): e202101104 (doi: 10.26508/lsa.202101104).
- Стегний В.Н. Популяционная генетика и эволюция малярийных комаров. Томск, 1991.
- Артемов Г.Н., Стегний В.Н. Молекулярно-генетический анализ районов прикрепления хромосом к оболочке ядра трофоцитов яичников малярийных комаров Anopheles комплекса maculipennis. Вестн. Том. гос. ун-та. Биология, 2010, 2(10): 123-131.
- Roller M., Stamper E., Villar D., Izuogu O., Martin F., Redmond A.M., Ramachanderan R., Harewood L., Odom D.T., Flicek P. LINE retrotransposons characterize mammalian tissue-specific and evolutionarily dynamic regulatory regions. Genome Biology, 2021, 22(1): 62 (doi: 10.1186/s13059-021-02260-y).
- Percharde M., Sultana T., Ramalho-Santos M. What doesn't kill you makes you stronger: transposons as dual players in chromatin regulation and genomic variation. BioEssays, 2020, 42(4): e1900232 (doi: 10.1002/bies.201900232).
- Chiu E.S., VandeWoude S. Endogenous retroviruses drive resistance and promotion of exogenous retroviral homologs. Annu. Rev. Anim. Biosci., 2021, 9: 225-248 (doi: 10.1146/annurev-animal-050620-101416).
- Chong A.Y., Kojima K.K., Jurka J., Ray D.A., Smit A.F., Isberg S.R., Gongora J. Evolution and gene capture in ancient endogenous retroviruses — insights from the crocodilian genomes. Retro-virology, 2014, 11: 71 (doi: 10.1186/s12977-014-0071-2).
- Benachenhou F., Sperber G.O., Bongcam-Rudloff E., Andersson G., Boeke J.D., Blomberg J. Conserved structure and inferred evolutionary history of long terminal repeats (LTRs). Mobile DNA, 2013, 4(1): 5 (doi: 10.1186/1759-8753-4-5).
- Chiu E.S., Gagne R.B. A natural laboratory to elucidate the evolution of endogenous-exogenous retroviral interactions. Mol. Ecol., 2021, 30(11): 2473-2476 (doi: 10.1111/mec.15850).
- Chiu E.S., VandeWoude S. Endogenous retroviruses drive resistance and promotion of exogenous retroviral homologs. Annu. Rev. Anim. Biosci., 2021, 9: 225-248 (doi: 10.1146/annurev-animal-050620-101416).
- Lanciano S., Mirouze M. Transposable elements: all mobile, all different, some stress responsive, some adaptive? Opinion in Genetics and Development, 2018, 49: 106-114 (doi: 10.1016/j.gde.2018.04.002).
- Hosaka A., Kakutani T. Transposable elements, genome evolution and transgenerational epige-netic variation. Curr. Opin. Genet. Dev., 2018, 49: 43-48 (doi: 10.1016/j.gde.2018.02.012).
- Cho J. Transposon-derived non-coding RNAs and their function in plants. Front. Plant Sci., 2018, 9: 600 (doi: 10.3389/fpls.2018.00600).;
- Shapiro J.A. Living organisms author their read-write genomes in evolution. Biology, 2017, 6(4): 42 (doi: 10.3390/biology6040042).
- Hosaka A., Kakutani T. Transposable elements, genome evolution and transgenerational epige-netic variation. Curr. Opin. Genet. Dev., 2018, 49: 43-48 (doi: 10.1016/j.gde.2018.02.012).
- Bhogireddy S., Mangrauthia S.K., Kumar R., Pandey A.K., Singh S., Jain A., Budak H., Varsh-ney R.K., Kudapa H. Regulatory non-coding RNAs: a new frontier in regulation of plant biology. Funct. Integr. Genomics, 2021, 21(3-4): 313-330 (doi: 10.1007/s10142-021-00787-8).
- Jungers C.F., Djuranovic S. Modulation of miRISC-mediated gene silencing in eukaryotes. Front. Mol. Biosci., 2022, 9: 832916 (doi: 10.3389/fmolb.2022.832916).
- Sadeq S., Al-Hashimi S., Cusack C.M., Werner A. Endogenous double-stranded RNA. Noncoding RNA, 2021, 7(1): 15 (doi: 10.3390/ncrna7010015).
- Hu G., Do D.N., Davoudi P., Miar Y. Emerging roles of non-coding RNAs in the feed efficiency of livestock species. Genes (Basel), 2022, 13(2): 297 (doi: 10.3390/genes13020297).
- Penso-Dolfin L., Moxon S., Haerty W., Di Palma F. The evolutionary dynamics of microRNAs in domestic mammals. Sci. Rep., 2018, 8(1): 17050 (doi: 10.1038/s41598-018-34243-8).
- Braud M., Magee D.A., Park S.D., Sonstegard T.S., Waters S.M., MacHugh D.E., Spillane C. Genome-wide microRNA binding site variation between extinct wild aurochs and modern cattle identifies candidate microRNA-regulated domestication genes. Front. Genet., 2017, 8: 3 (doi: 10.3389/fgene.2017.00003).
- Makino T., Rubin C.J., Carneiro M., Axelsson E., Andersson L., Webster M.T. Elevated proportions of deleterious genetic variation in domestic animals and plants. Genome Biol. Evol., 2018, 10(1): 276-290 (doi: 10.1093/gbe/evy004).
- Startek M., Szafranski P., Gambin T., Campbell I.M., Hixson P., Shaw C.A., Stankiewicz P., Gambin A.. Genome-wide analyses of LINE-LINE-mediated nonallelic homologous recombination. Nucleic Acids Research, 2015, 43(4): 2188-2198 (doi: 10.1093/nar/gku1394).
- Wang G.D., Shao X.J., Bai B., Wang J., Wang X., Cao X., Liu Y.H., Wang X., Yin T.T., Zhang S.J., Lu Y., Wang Z., Wang L., Zhao W., Zhang B., Ruan J., Zhang Y.P. Structural variation during dog domestication: insights from grey wolf and dhole genomes. National Science Review, 2019, 6(1): 110-122 (doi: 10.1093/nsr/nwy076).
- Yang N., Zhao B., Chen Y., D'Alessandro E., Chen C., Ji T., Wu X., Song C. Distinct re-trotransposon evolution profile in the genome of rabbit (Oryctolagus cuniculus). Genome Biol. Evol, 2021, 13(8): evab168 (doi: 10.1093/gbe/evab168).
- Ngo M.H., Arnal M., Sumi R., Kawasaki J., Miyake A., Grant C.K., Otoi T., Fernández de Luco D., Nishigaki K. Tracking the fate of endogenous retrovirus segregation in wild and domestic cats. J. Virol., 2019, 93(24): e01324-19 (doi: 10.1128/JVI.01324-19).
- Глазко В.И. Генные и геномные подписи доместикации. Сельскохозяйственная биология, 2018, 53(4): 659-672 (doi: 10.15389/agrobiology.2018.4.659rus).
- Glazko V., Zybailov B., Glazko T. Asking the right question about the genetic basis of domestication: what is the source of genetic diversity of domesticated species? Adv. Genet. Eng., 2015, 4: 2 (doi: 10.4172/2169-0111.1000125).


