Гибридные вычислительные кластеры для изучения структуры, функции и регуляции белков
Автор: Суплатов Дмитрий Андреевич, Попова Нина Николаевна, Копылов Кирилл Евгеньевич, Шегай Максим Викторович, Воеводин Владимир Валентинович, Швядас Витас Каятоно
Рубрика: Информатика, вычислительная техника и управление
Статья в выпуске: 4 т.6, 2017 года.
Бесплатный доступ
Изучение структуры, функции и регуляции белков с использованием биоинформатики и молекулярного моделирования является комплексной задачей, требующей сочетания различных методов и способов их исполнения. На практике, речь идет о конвейере из последовательных этапов, исполняемых различными программами, предъявляющими свои требования к вычислительным ресурсам. Гибридные вычислительные кластеры - системы, обладающие существенной мощностью и разнообразием аппаратных возможностей - необходимы для того, чтобы оптимально исполнить каждую отдельную стадию единого комплексного решения. При этом GPU-ускорители открывают новые возможности для поиска эффективных решений ресурсоемких задач биоинформатики и молекулярного моделирования.
Гибридные вычислительные кластеры, биоинформатика, молекулярное моделирование, последовательные этапы, кодизайн, gpu-ускорители
Короткий адрес: https://sciup.org/147160634
IDR: 147160634 | УДК: 004.021, | DOI: 10.14529/cmse170406
Текст научной статьи Гибридные вычислительные кластеры для изучения структуры, функции и регуляции белков
Взаимодействие белков с небольшими молекулами (лигандами) является одним из ключевых процессов в биологии. На настоящий момент наиболее хорошо изучены механизмы действия лигандов, которые связываются в активных центрах ферментов, однако роль других карманов и полостей на поверхности белков изучены недостаточно. В последние годы появляется все больше свидетельств явления аллостерии — регуляции функции белков посредством связывания низкомолекулярных лигандов в специализированных регуляторных центрах на поверхности [1]. На рис. 1 показана структура одного из ферментов гликолиза — важнейшего метаболического пути клетки. Поверхность этого большого и сложно устроенного белка покрыта большим количеством полостей — потенциальных сайтов связывания. Среди них можно увидеть как активный центр, выполняющий главную функцию этого фермента, так и известный аллостерический (регуляторный) сайт. Тем не менее, роль остальных участков связывания, а их большинство,
∗ Статья рекомендована к публикации программным комитетом Международной конференции «Суперкомпьютерные дни в России — 2017».
остается неизвестной. Как определить те центры связывания, которые важны для функции и регуляции фермента, а также научиться управлять его активностью при помощи низкомолекулярных синтетических и природных соединений?
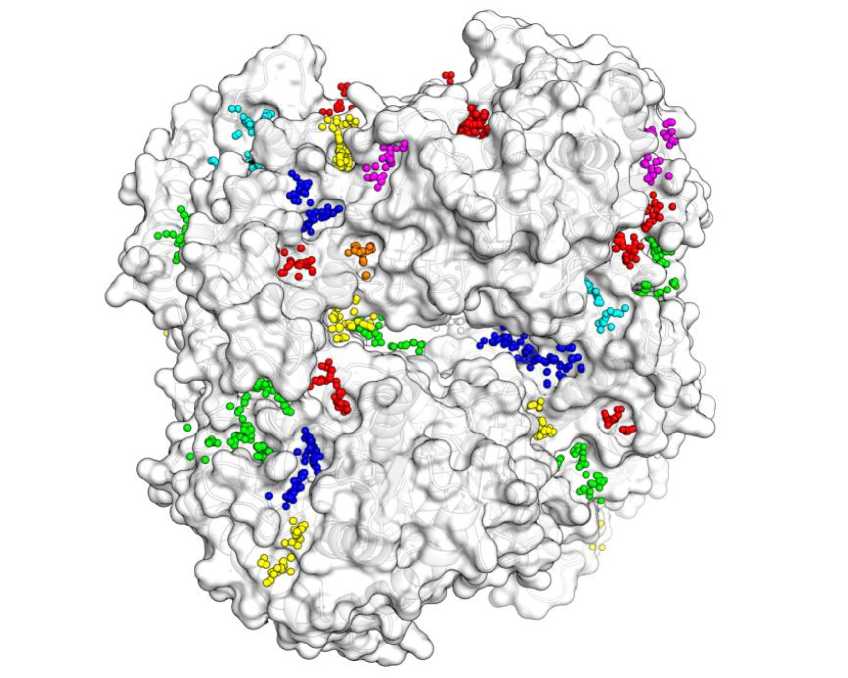
Рис. 1. Потенциальные сайты связывания (скопление шариков одного цвета) в структуре одного из ферментов гликолиза. Изображение подготовлено с помощью программы PyMol
Последние шестьдесят лет были периодом становления биоинформатики и молекулярного моделирования как ключевых подходов к изучению взаимосвязи между структурой и функцией в белках. Долгосрочные возможности вычислительных методов в биологии были осознаны в середине ХХ века после успеха пионерного исследования Фредерика Сэнгера по определению геномной последовательности белка инсулина. Основываясь на этом результате, Эмиль Цукеркандль и Лайнус Полинг в 1960-х годах высказали гипотезу о том, что аминокислотные последовательности можно «выравнивать» — т.е. сравнивать «побуквенно» и таким образом изучать эволюционные изменения в белках на молекулярном уровне — прообраз современного сравнительного анализа гомологичных (т.е. родственных) белков [2]. Работы Дэвида Филлипса по кристаллизации белков в конечном итоге привели к расшифровке первой трехмерной структуры лизоцима в 1965 году. В том же году увидел свет первый номер «Атласа последовательностей и структур белков» Маргарет Дэйхоф, а также ее первые программируемые компьютерные методы для изучения эволюции белков на основании выравнивания их аминокислотных последовательностей. Позже, в 1971 году, под руководством Вальтера Гамильтона был создан Protein Data Bank как хранилище трехмерных представлений био- логических макромолекул и первоначально содержал всего 7 структур. Публикация первой полной геномной последовательности в 1977 продолжилась созданием банка геномной информации GenBank в 1982 году, а затем и базы Swiss-Prot, основанной в 1986 году для того, чтобы дополнить геномную информацию о белках экспериментально проверенной функциональной аннотацией. За несколько лет до этого, в 1981 году, IBM представила миру новую аппаратную платформу — персональный компьютер — который использовал микропроцессор 4.77МГц от Intel. Через два года, в 1983 году, компания Apple представила Lisa — первый в своем роде компьютер с графическим пользовательским интерфейсом, а в 1984 году в комплекте Macintosh появилась мышь. Это было началом новой эры в науке, когда накопление экспериментальной информации о белках соседствовало с развитием персональных компьютеров. Доступность — первичных данных, программ и алгоритмов, а также вычислительной инфраструктуры — и потребность в новых методах анализа поступающей биологической информации создали условия для развития биоинформатики и вычислительной биологии и предопределили их роль в современной науке. Оборудование для хранения данных становилось дешевле, вычислительная мощность ЭВМ продолжала увеличиваться. Вычисления, которые казались ресурсозатратными в 1980-х, стали выполняться за мгновения после того, как вначале 2000 г. появился Pentium 4 — первый настольный процессор с гигагерцовой тактовой частотой.
Тот факт, что компьютеры становились все быстрее, позволил перенести акцент с программирования на биологию. Другими словами, стратегия заключалась в том, чтобы уменьшить время, затрачиваемое на написание и отладку кода и вкладывать больше усилий в разработку новых методов, осмысление и систематизацию результатов, понимание биологических закономерностей и механизмов изучаемых процессов. Такие языки как C/C++ позволяют писать программы, которые выполняются быстрее и требуют меньше памяти, но труднее в изучении и требуют бóльших усилий для отладки и оптимизации кода. Одним из ключевых аспектов новой философии стал выбор такой стратегии и языка программирования, которые максимально снижали продолжительность решения конкретной задачи, что подразумевает не столько скорость работы программы, сколько суммарное время работы коллектива от постановки научной проблемы до получения значимых результатов [3]. Наибольшее распространение в биоинформатике получили такие языки как Java, Perl и Python — они проще в изучении, требуют, как правило, меньше строк кода и используют автоматическое управление памятью. Эти удобства достигаются за счет издержек при обработке данных, которые уменьшают производительность приложения. Ускоренная разработка программ за счет экономии времени на обучении и применении «медленных» языков программирования стала возможной во многом благодаря тому, что большинство отдельных задач в компьютерной биологии могут быть решены за относительно небольшое время. Так, программа Modeller, которая применяется для предсказания трехмерной организации белков по гомологии с более изученными родственными белками, занимает одно ядро процессора на две минуты для того, чтобы построить модель структуры большого гомотетрамерного белка глице-ральдегид-3-фосфатдегидрогеназы, включающего более 1300 аминокислотных остатков. Выравнивание аминокислотных последовательностей нескольких сотен ДНК-зависимых РНК-полимераз, каждая из которых состоит из 1000 остатков (цепь в), можно построить с помощью программы Mafft за 3 секунды на одном ядре современного процессора.
Эксперимент по in silico докингу среднестатистического низкомолекулярного лиганда, включающего от нуля до десяти торсионных углов (степеней свободы), как правило, занимает от нескольких секунд до получаса с такими программами, как AutoDock и Leadfinder. Некоторые биоинформатические алгоритмы более требовательны к ресурсам, но могут быть значительно ускорены за счет эффективного использования современных многоядерных процессоров. В этом случае речь, как правило, идет о параллельном программировании в рамках общей памяти — OpenMP (например, выравнивание структур белков в программе MATT) или потоки в Java (например, поиск функционально важных остатков в больших суперсемействах ферментов с помощью программы Zebra). Только самые ресурсозатратные алгоритмы реализованы в виде параллельного кода с использованием MPI. Наиболее известными классами методов, имеющих оригинальные MPI реализации, являются филогенетический анализ, а также молекулярная динамика и связанные с ней подходы теоретической химии.
Анализ сложных биологических данных требует гораздо большего, чем запуск одной программы один раз — он подразумевает многократный запуск разных программ в определенной последовательности. При этом сложность представляет оптимизация не только основных приложений, но и скрепляющих их многочисленных подпрограмм, которые занимаются подготовкой ввода, анализом и систематизацией результатов. Например, сравнительный анализ родственных ферментов одного суперсемейства, объединяющего представителей с разными свойствами в рамках общей структурной организации, представляет огромный интерес как с точки зрения изучения структурнофункциональных взаимосвязей, так и для создания препаратов белков с улучшенными свойствами для практического применения, дизайна ингибиторов ферментов и лекарственных препаратов. Задача построения множественного выравнивания эволюционно удаленных белков подразумевает комбинированное использование как структурной, так и геномной информации. Можно сразу отметить, что подобные выравнивания сами по себе представляют коммерческую ценность [4, 5], что отражает сложность задачи с научной и технической точки зрения. На первом этапе происходит поиск эволюционно удаленных родственников по структурному сходству. Предполагается, что такие белки произошли от очень далекого общего предка, характеризуются широким функциональным разнообразием и могут сильно отличаться по аминокислотной последовательности. На втором этапе, каждая структура белка сопоставляется с базой данных известных аминокислотных последовательностей для выявления близких эволюционных родственников, которые обладают достаточным сходством по последовательностям и могут быть выравнены без использования структурной информации. Наконец, структуры удаленных гомологов выравниваются с использованием алгоритмов структурного сравнения, и соответствующие совмещения используются для выравнивания последовательностей гомологов без известной структуры. В те времена, когда объем информации в базах данных (в контексте рассматриваемого примера — количество известных структур и последовательностей представителей одного суперсемейства) был небольшим, подобные операции можно было выполнить относительно быстро в последовательном режиме, однако и польза от такого результата была ограничена. В последние годы бурное развитие методов геномики, протеомики, метаболомики и транскриптомики привело к увеличению объемов свободно доступных данных о структуре и функции ферментов. Первичные базы данных, собирающие информацию об аминокислотных последовательностях и струк- турах белков, растут в геометрической прогрессии и на сегодняшний день насчитывают 85 млн. записей в UniProtKB и 130 тыс. в PDB, соответственно. Активно развиваются вторичные базы данных, обобщающие структурную и функциональную информацию, классифицирующие белки/ферменты с разным строением, свойствами и происхождением. Анализ этой информации открывает новые возможности для понимания структурно -функциональных взаимосвязей в белках, однако требует новых программных и аппаратных решений, которые позволили бы обрабатывать данные такого объема и получать биологически осмысленный результат в разумные сроки. Решить проблему могут суперкомпьютеры. Но поставить на поток ре-имплементацию всех необходимых программ из оригинальных кодов, в том числе написанных на Java, Perl, Python и др., на код с поддержкой MPI не представляется возможным. Более важно то, что не всегда это представляется лучшим решением.
Таким образом, изучение свойств и строения ферментов с использованием биоинформатики и молекулярного моделирования является комплексной задачей, требующей сочетания различных методов и способов их исполнения. Фактически, речь идет о конвейере из последовательных этапов, исполняемых различными программами, предъявляющими свои требования к вычислительным ресурсам. Одни приложения требуют больше оперативной памяти, другие выигрывают от быстрых процессоров. Есть программы, которые критически зависят от скорости чтения с диска. Некоторые алгоритмы могут быть значительно ускорены на графических процессорах и т.д. Подобная структура создает основу для кодизайна, т.е. создания оптимального единого решения за счет взаимосвязанной разработки программного обеспечения и выбора аппаратной конфигурации для каждой стадии и отдельно взятой программы. В этой статье мы обсуждаем поиск новых путей регуляции конкретного фермента с использованием биоинформатики и молекулярного моделирования. Мы описываем последовательные этапы предложенного нами решения, обсуждаем программные и аппаратные аспекты каждой отдельной стадии, приводим описание оригинальных программных продуктов. Мы делаем вывод, что для изучения структуры, функции и регуляции ферментов необходимы гибридные кластеры — вычислительные системы, обладающие как существенной мощностью, так и разнообразием аппаратных возможностей — которые позволяют оптимально исполнить каждую отдельную стадию единого комплексного решения. Мы также заключаем, что GPU-ускорители открывают новые возможности для применения методов биоинформатики и молекулярного моделирования при решении задач естественных наук.
Структура решения и описание этапов
Глицеральдегид-3-фосфатдегидрогеназа (ГАФД) — фермент гликолиза, катализирующий центральную реакцию этого метаболического пути — гликолитическую окси-доредукцию. Интерес к ГАФД обусловлен тем, что блокирование этой реакции препятствует не только накоплению макроэргических фосфатов, но и восстановленного нико-тинамидадениндинуклеотида, используемого для получения энергии в дыхательной цепи. Помимо гликолиза ГАФД также принимает участие в других важных для клетки процессах. Иными словами, ГАФД как ключевой фермент метаболизма в клетках явля-
Д.А. Суплатов, Н.Н. Попова, К.Е. Копылов, М.В. Шегай, Вл.В. Воеводин и др. ется многообещающей мишенью для создания лекарств от различных бактериальных инфекций.
Для изучения структуры и функции ГАФД с целью поиска новых путей регуляции этого фермента нами предложено решение, основанное на последовательном и взаимосвязанном исполнении методов биоинформатического анализа родственных белков, молекулярного моделирования структуры фермента, подходов теоретической химии и статистического анализа.
Поиск новых регуляторных сайтов связывания
Первым этапом решения является поиск новых сайтов связывания регуляторных лигандов в ГАФД с использованием оригинального подхода биоинформатического анализа. В основе биоинформатического анализа лежит понятие «выравнивание» — сопоставление аминокислотных последовательностей и элементов структуры родственных белков. Проблема выравнивания белков заключается в том, что длительная эволюция привела к значительным отличиям их пространственных структур и аминокислотных последовательностей. В этой связи задача биоинформатического анализа разделяется на три подзадачи: (1) поиск родственных белков в базах данных на основании сходства структур и последовательностей, (2) построение множественного выравнивания и (3) анализ выравнивания и аннотация новый сайтов в структурах белков.
В настоящий момент в банке данных трехмерных моделей белков PDB насчитывается более 130 тыс. структур (более 350 тыс. структур отдельных цепей) и подзадача поиска структурного сходства предполагает исполнение процедуры парного сравнения с каждой из них, что представляет значительную вычислительную сложность. Для управления процедурой поиска родственных белков по структурному сходству по базе данных PDB нами разработано оригинальное приложение. Программа осуществляет форматирование базы данных белковых структур и их предварительный анализ. Это позволяет заранее отбросить наименее похожие структуры и сфокусировать поиск на заведомо более близких родственниках. Следующей важной особенностью программы является сохранение однажды посчитанных парных выравниваний в базе данных, организованной на основе PostgreSQL. Фокусирование области поиска за счет предварительного анализа данных и депонирование однажды посчитанных результатов в базе данных позволяет сократить время расчета как при повторном запуске одной и той же подзадачи (например, для уточнения параметров поиска), так и для независимых подзадач, в которых используются структурно похожие белки. Однако, не для всех белков известна их структура — для недавно открытых, а также малоизученных белков, как правило, известна только последовательность аминокислот, но не их расположение в пространстве. В таких случаях необходимо использовать поиск родственных белков в базах данных на основании сходства аминокислотных последовательностей. Количество известных последовательностей белков в базах данных Swiss-Prot и TrEMBL составляет на сегодняшний день в сумме более 85 млн. и подзадача поиска сходства предполагает исполнение процедуры парного сравнения с каждой из них. Для исполнения этой вычислительно сложной подзадачи мы используем GPU реализацию популярной программы BLAST [6, 7].
Подзадача построения множественного выравнивания родственных белков, выявленных в результате решения предыдущей подзадачи поиска в базах данных, предпола- гает использование двух типов алгоритмов — для выравнивания аминокислотных последовательностей и структур белков. Выравнивание последовательностей (фактически, сравнение строк текста) эволюционно близких белков не представляет существенной сложности и, как правило, может быть качественно исполнено на одном ядре современного процессора в пределах от нескольких секунд до минуты. Сопоставление структур, которое основано на пространственном совмещении координат атомов, напротив, представляет существенную вычислительную сложность. Для исполнения такого выравнивания нами была разработана оригинальная MPI ре-имплементация популярной программы MATT структурного выравнивания. Созданное приложение parMATT позволяет ускорить выравнивание структур белков в параллельном режиме с использованием MPI на кластере или суперкомпьютере. Программа полезна для выравнивания больших выборок, состоящих из сотен (и более) белков, которые характеризуются достаточным структурным сходством и наличием общего структурного ядра, а также предназначена для ускорения повторяющихся и часто исполняемых операций — пересчета выравнивания с уточненными параметрами, а также для работы публичных сервисов. Структурное выравнивание ГАФД из различных родственных организмов, в том числе человека и бактерий, приведено на рис. 2.
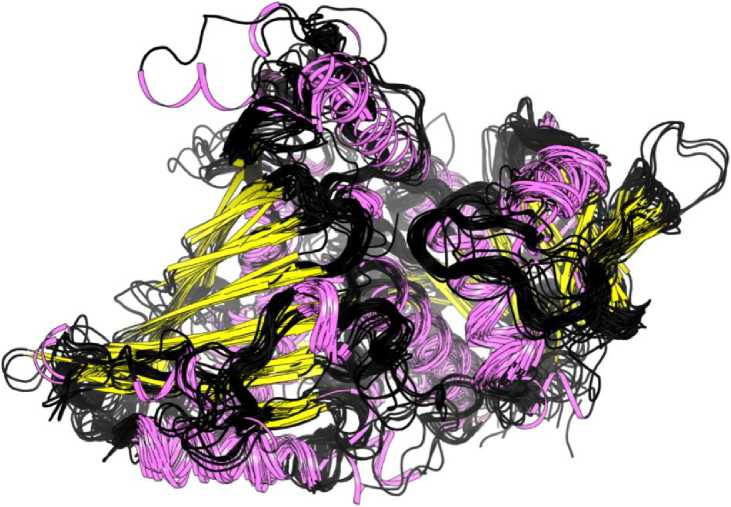
Рис. 2. Структурное выравнивание репрезентативной выборки ГАФД-подобных ферментов. Розовым и желтым цветом показаны участки с регулярной вторичной структурой — α-спирали и β-слои, соответственно. Черным показаны петли — участки без регулярной вторичной структуры. Изображение подготовлено с помощью программы PyMol
Последняя подзадача заключается в статистическом анализе полученного множественного выравнивания ГАФД из различных организмов, в том числе человека и бактерий, для выявления общих черт и особенностей организации активных и аллостерических центров. Суть подхода заключается в выявлении специфических позиций — таких участков структуры, которые отличают ферменты из разных организмов — и использо- вании их в качестве критерия для ранжирования полостей на поверхности белка по степени функциональной значимости (рис. 3). Вычисления исполняются с использованием оригинальных программ Zebra и pocketZebra [8–10]. Наиболее ресурсоемкой стадией этой подзадачи является расчет случайной модели, в процессе которого колонка выравнивания перетасовывает несколько тысяч раз. Основная часть расчета случайной модели производится независимо для каждой колонки в выравнивании, что позволяет выполнять эти вычисления параллельно.
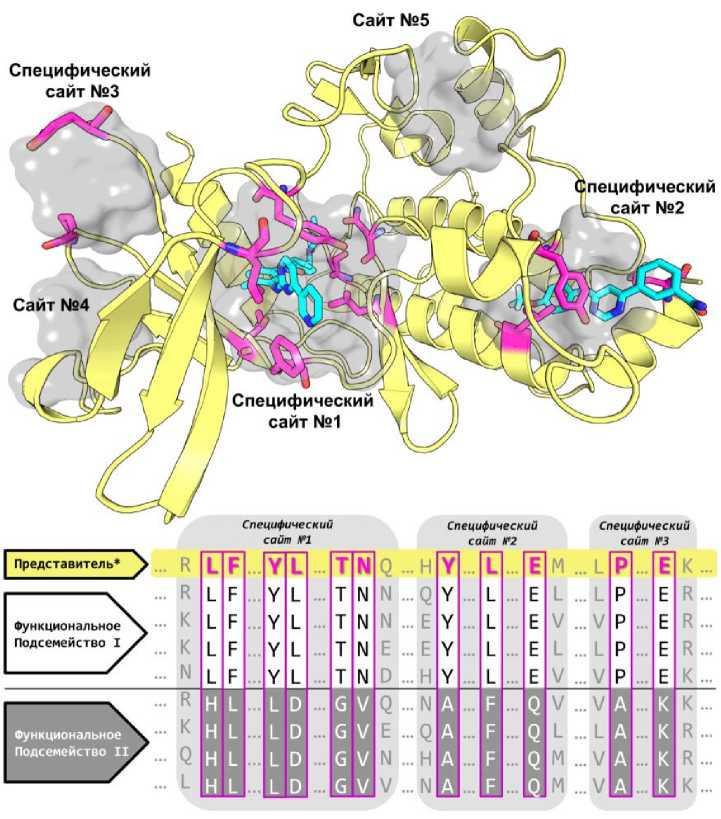
Рис. 3 . Схематическое представление поиска новых сайтов связывания лигандов (функциональных и регуляторных) в родственных белках [10]. Специфические позиции подсемейств показаны в виде палочек в структуре (сверху) и пурпурным цветом в колонках множественного выравнивания (снизу). Изображение подготовлено с помощью программы PyMol
С точки зрения аппаратной реализации, предложенное решение задачи биоинфор-матического анализа ориентировано на исполнение в рамках одного вычислительного узла, содержащего многоядерный процессор максимальной мощности, достаточное количество оперативной памяти, мощный графический ускоритель, а также SSD-накопитель. SSD-диск используется для хранения всех необходимых баз данных, что существенно ускоряет решение. При этом обновление наиболее крупных баз данных
(около 100 ГБ) допускается производить не чаще двух-трех раз в год, что позволяет экономить ресурс SSD-устройства. Видеоускоритель используется для исполнения процедуры поиска по сходству последовательностей с помощью GPU-BLAST. Многоядер-ность основного вычислителя способствует ускорению остальных параллельных операций, например, поиск по структурному сходству в базе данных, который предполагает выполнение большого количества независимых парных сравнений. Особый случай представляет подзадача построения множественного структурного выравнивания, которая, в случае необходимости, может быть исполнена оригинальной программой parMATT в режиме MPI на кластере или суперкомпьютере. Более подробно о задаче биоинформа-тического анализа можно узнать в недавних публикациях [11] и на сайте С оригинальной реализацией протокола построения множественных выравниваний функционально разнообразных семейств белков в рамках одного гибридного узла можно познакомиться по адресу
В результате выполнения текущего этапа исследования был обнаружен новый, ранее неизвестный сайт связывания лигандов в структуре ГАФД.
Скрининг ингибиторов
Вторым этапом решения является скрининг нового регуляторного сайта связывания лигандов в структуре ГАФД, обнаруженного в результате выполнения первого этапа работы, большой библиотекой потенциальных ингибиторов. Был разработан оригинальный комплекс программ для исполнения высокопроизводительного скрининга ингибиторов на суперкомпьютере, который включает в себя следующие компоненты: универсальный планировщик для запуска в параллельном режиме большого количества независимых подзадач (где каждая подзадача подразумевает докинг одного ингибитора в заданный сайт); программа для форматирования большой библиотеки лигандов для скрини-рования на суперкомпьютере в параллельном режиме; программа для подготовки структуры белка к скринированию ингибиторов в разные сайты на его поверхности (т.е. для подготовки «сеток» разных сайтов в структуре одного белка); программа для запуска собственно виртуального скрининга в параллельном режиме на суперкомпьютере; набор программ для обработки выходных данных, содержащих результаты скрининга. Подробное описание комплекса программ и принципа их работы приведены в недавней публикации [12]. Использование суперкомпьютера позволило проводить анализ больших библиотек ингибиторов с более высокой точностью, которая достигается за счет использования более аккуратного и ресурсозатратного алгоритма оценки энергии связывания лиганда в структуре белка. Наличие суперкомпьютерной системы позволило также использовать для докинга не единственную структуру белка, а ансамбль конформеров фермента, полученный при помощи молекулярной динамики, для того, чтобы учесть подвижность аминокислотных остатков и небольшие флуктуации в структурах сайта связывания.
Таким образом, с программной точки зрения задача скрининга рассматривается как совокупность большого числа подзадач докинга индивидуальных лигандов, которые могут быть исполнены параллельно и независимо, поскольку не обмениваются информацией в процессе расчета. С аппаратной точки зрения, одна подзадача докинга индивидуального лиганда выполняется на одном ядре классического CPU в интервале от нескольких секунд до нескольких минут, при этом не предъявляет существенных требований к остальным ресурсам.
В результате выполнения текущего этапа исследования с использованием суперкомпьютера ««Ломоносов-2» было выполнено в общей сложности около 30 млн. докингов, наиболее перспективные ингибиторы ГАФД из патогенных бактерий были отобраны для дальнейшего изучения.
Молекулярное моделирование комплексов фермент-ингибитор
На третьем этапе решения связывание в новом регуляторном сайте ГАФД наиболее перспективных ингибиторов, отобранных на предыдущем этапе, изучалось с использованием молекулярной динамики. Эта задача разделяется на три подзадачи — (1) подготовка моделей, (2) молекулярная динамика и (3) анализ результатов молекулярной динамики.
Подготовка моделей подразумевает построение полноразмерных молекулярных моделей белка и ингибитора, в том числе с учетом рКа ионизируемых групп и альтернативных степеней протонирования, а также параметризация этих моделей — т.е. приведение информации о них в соответствие с требованиями программы молекулярной динамики. Важной составляющей этой подзадачи является расчет зарядов на каждом атоме в структурах. Информация об атомах белка по умолчанию берется из стандартного силового поля. Однако для остальных молекул — например, кофакторов и ингибиторов — эти параметры необходимо вычислять. Процедура оценки зарядов атомов в низкомолекулярных соединениях включает, в том числе, методы, основанные на квантово-механических подходах теоретической химии, например, реализованных в популярном отечественном продукте Firefly (ранее известном как PC GAMESS). Особенностью этих методов, как правило, является интенсивное использование операций обращения к жесткому диску для записи и чтения промежуточных результатов. Суммарный объем перезаписываемой информации может достигать терабайта даже для небольших молекул (несколько десятков атомов), в этой связи использование SSD-накопителя ускорило бы процесс, однако быстро приведет к исчерпанию ресурса и, таким образом, экономически не оправдано. Представляется адекватным использовать для этой цели отдельные узлы с быстрыми классическими накопителями.
Полученные модели были использованы как стартовые конформации для метода молекулярной динамики. Использование суперкомпьютера позволило применить метод молекулярной динамики с более высокой точностью для изучения взаимодействия ингибиторов с аминокислотами нового регуляторного сайта связывания. Для этого температура была установлена на уровне 300К (27ºС), поскольку использование более высоких значений чаще приводит к возникновению артефактов; использована более сложная 4-сайтовая молекулярная модель воды TIP4P-Ew, которая более правильно воспроизводит физико-химические характеристики растворителя; использован адекватный отступ между поверхностью белка и краем молекулярной ячейки для исключения артефактов в периодической системе; параметры для учета электростатических взаимодействий были установлены на максимальные рекомендованные значения. Для каждой модели фермента в комплексе с ингибитором были вычислены по пять независимых траекторий для того, чтобы собрать больше информации для статистической обработки. Метод молекулярной динамики является вычислительно сложным. С программной точки зрения некоторые процедуры молекулярной динамики уже частично реализованы на GPU и получили широкое распространение благодаря существенно более высокой скорости расче- тов — например, популярный метод расчета PME электростатики в явно заданном растворителе и NVE/NVT/NPT ансамблях в пакете Amber версии 14 и выше [13–15]. С аппаратной точки зрения, стадии минимизации, нагрева и релаксации (10–20 коротких, последовательно запускаемых стадий) удобно исполнять на мощном локальном видеоускорителе, таком как GeForce GTX 980 Ti. Наиболее ресурсоемкая стадия свободной динамики в каждом случае исполнялась на четырех видеокартах Tesla K40 суперкомпьютера ««Ломоносов-2».
Последняя подзадача подразумевает анализ результатов молекулярной динамики. Эта стадия зависит не столько от мощности компьютера и организации вычислительного процесса, сколько от выбранной стратегии анализа, а также подготовленности и опытности эксперта, и по этой причине мы не будем подробно описывать ее здесь.
По результатам молекулярного моделирования наиболее перспективные лиганды были рекомендованы для экспериментальной проверки ингибирующей активности по отношению к ГАФД из разных организмов.
Заключение
Экспериментальная проверка лигандов, отобранных по результатам биоинформати-ческого анализа и компьютерного моделирования, показала наличие ингибирующей активности по отношению к бактериальной ГАФД и отсутствие влияния на родственный фермент в организме человека в изученных концентрациях. Последующая проверка наиболее эффективных ингибиторов бактериального ГАФД показала подавление роста культуры Mycobacterium tuberculosis . Таким образом, установлена противотуберкулезная активность предложенных соединений. Подходы, аналогичные использованным в этой работе, были ранее применены нами для изучения структуры и функции ферментов из других суперсемейств, поиска и характеристики новых центров связывания, а также получения препаратов ферментов с улучшенными свойствами [16-20].
Мы делаем вывод, что для изучения структуры, функции и регуляции ферментов необходимы вычислительные системы, обладающие как существенной мощностью, так и разнообразием аппаратных возможностей. Отдельные компоненты такого гибридного кластера должны быть ориентированы на конкретные методы компьютерной биологии, которые, являясь частью единого комплексного решения, используются последовательно и взаимосвязано при решении конкретных задач. Так, для исполнения методов биоинформатики в первую очередь необходимы отдельные независимые узлы с многоядерными процессорами максимальной мощности, GPU-ускорителями и SSD-накопителями для хранения баз данных. Для исполнения метода молекулярной динамики необходимы мощные GPU-ускорители. Для исполнения молекулярного скрининга необходимы быстрые CPU, при этом связь между ними не влияет на эффективность расчета, поскольку подзадачи докинга полностью независимы друг от друга.
Мы также хотим обратить внимание на то, что речь в статье идет именно о «вычислительных кластерах». Разумеется, когда речь заходит о больших организациях, таких как МГУ имени М.В. Ломоносова, масштабные вычислительные системы и суперкомпьютеры [21] необходимы для того, чтобы удовлетворить потребности всех пользователей в вычислительных ресурсах. Однако если отдельно рассматривать потребности небольших научных коллективов, то для их реализации будет достаточно и относительно скромных ресурсов, как по размерам и сложности устройства, так и по стоимости. Это обусловлено, прежде всего, развитием GPU-технологий. Еще недавно для запуска одной молекулярной динамики требовалось использование сотен ядер классических CPU, установленных на десятках отдельных узлов и связанных дорогой сетью. Сегодня аналогичную скорость расчета можно получить даже на одном игровом видеоускорителе, таком как GeForce GTX 980 Ti. Стоимость таких устройств несопоставима меньше затрат на суперкомпьютер. Важно то, что использование мощных GPU-ускорителей позволяет не столько анализировать биологические системы с бóльшей скоростью, сколько дает возможность увеличивать точность анализа за счет перенастройки параметров или усложнения соответствующих процедур. В последние годы были сделаны первые шаги в сторону создания GPU-реализаций некоторых биологически-ориентированных алгоритмов [22]. Пожалуй, на первом месте по популярности находится уже упомянутый метод молекулярной динамики, который частично реализован на GPU. Тем не менее, можно констатировать, что популярность классических CPU-реализаций алгоритмов в естественно-научной среде существенно выше спроса на GPU-версии. На практике получается, что издержки на обучение, установку и обновление GPU-приложений часто оказываются выше, чем положительный эффект от их использования, что связано со сложной организацией задач компьютерной биологии. В этом контексте фундаментальный и практический интерес представляет дальнейший поиск таких этапов в решении задач компьютерной биологии, реализация которых с использованием GPU была бы эффективной и открывала новые возможности для анализа возрастающих объемов биологических данных.
Работа выполнена при поддержке грантов РФФИ №17-07-00751 и РНФ №15-14-00069 с использованием оборудования Центра коллективного пользования сверхвысокопроизводительными вычислительными ресурсами МГУ имени М.В. Ломоносова.
Список литературы Гибридные вычислительные кластеры для изучения структуры, функции и регуляции белков
- Суплатов Д.А., Швядас В.К. Изучение функциональных и аллостерических сайтов в суперсемействах белков//Acta Naturae. 2015. Т. 7, № 4. С. 39-52.
- Zuckerkandl E., Pauling L. Evolutionary Divergence and Convergence in Proteins//Evolving Genes and Proteins. 1965. Vol. 97. P. 97-166.
- Fourment M., Gillings M.R. A Comparison of Common Programming Languages Used in Bioinformatics//BMC bioinformatics. 2008. Vol. 9, No. 1. P. 82. 2105-9-82 DOI: 10.1186/1471-
- Kourist R., Jochens H., Bartsch S., Kuipers R., Padhi S.K., Gall M., Dominique B., Henk-Jan J., Bornscheuer U.T. The α/β-hydrolase Fold 3DM Database (ABHDB) as a Tool for Protein Engineering//ChemBioChem. 2010. Vol. 11, No. 12. P. 1635-1643 DOI: 10.1002/cbic.201000213
- Программное обеспечение BioProdict. URL: https://www.bio-prodict.nl/(дата обращения: 28.09.2017).
- Vouzis P.D., Sahinidis N.V. GPU-BLAST: Using Graphics Processors to Accelerate Protein Sequence Alignment//Bioinformatics. 2011. Vol. 27, No. 2. P. 182-188 DOI: 10.1093/bioinformatics/btq644
- Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z., Miller W., Lipman D.J. Gapped BLAST and PSI-BLAST: a New Generation of Protein Database Search Programs//Nucleic Acids Research. 1997. Vol. 25, No. 17. P. 3389-3402 DOI: 10.1093/nar/25.17.3389
- Suplatov D., Shalaeva D., Kirilin E., Arzhanik V., Švedas V. Bioinformatic Analysis of Protein Families for Identification of Variable Amino Acid Residues Responsible for Functional Diversity//Journal of Biomolecular Structure and Dynamics. 2014. Vol. 32, No. 1. P. 75-87 DOI: 10.1080/07391102.2012.750249
- Suplatov D., Kirilin E., Takhaveev V., Švedas V. Zebra: Web-server for Bioinformatic Analysis of Diverse Protein Families//Journal of Biomolecular Structure and Dynamics. 2014. Vol. 32, No. 11. P. 1752-1758 DOI: 10.1080/07391102.2013.834514
- Suplatov D., Kirilin E., Arbatsky M., Takhaveev V., Švedas V. PocketZebra: a Web-server for Automated Selection and Classification of Subfamily-specific Binding Sites by Bioinformatic Analysis of Diverse Protein Families//Nucleic Acids Research. 2014. Vol. 42, No. W1. P. W344-W349 DOI: 10.1093/nar/gku448
- Suplatov D., Kirilin E., Švedas V. Bioinformatic Analysis of Protein Families to Select Function-related Variable Positions "Understanding Enzymes: Function, Design, Engineering and Analysis" (Allan Svendsen) Pan Stanford Publishing 2016. P. 351-385.
- Suplatov D., Popova N., Zhumatiy S., Voevodin V., Švedas V. Parallel Workflow Manager for Non-parallel Bioinformatic Applications to Solve Large-scale Biological Problems on a Supercomputer//Journal of Bioinformatics and Computational Biology. 2016. Vol. 14, No. 2. P. 1641008 DOI: 10.1142/S0219720016410080
- Le Grand S., Götz A.W., Walker R.C. SPFP: Speed Without Compromise -A Mixed Precision Model for GPU Accelerated Molecular Dynamics Simulations//Computer Physics Communications. 2013. Vol. 184, No. 2. P. 374-380 DOI: 10.1016/j.cpc.2012.09.022
- Salomon-Ferrer R., Goetz A.W., Poole D., Le Grand S., Walker R.C. Routine Microsecond Molecular Dynamics Simulations with AMBER on GPUs. 2. Explicit Solvent Particle Mesh Ewald//Journal of Chemical Theory and Computation. 2013. Vol. 9, No. 9. P. 3878-3888 DOI: 10.1021/ct400314y
- Goetz A.W., Williamson M.J., Xu D., Poole D., Le Grand S., Walker R.C. Routine Microsecond Molecular Dynamics Simulations with AMBER on GPUs. 1. Generalized Born//Journal of Chemical Theory and Computation. 2012. Vol. 8, No. 5. P. 1542-1555 DOI: 10.1021/ct200909j
- Bezsudnova E.Y., Stekhanova T.N., Suplatov D.A., Mardanov A.V., Ravin N.V., Popov V.O. Experimental and Computational Studies on the Unusual Substrate Specificity of Branched-chain Amino Acid Aminotransferase from Thermoproteus uzoniensis//Archives of Biochemistry and Biophysics. 2016. Vol. 607. P. 27-36 DOI: 10.1016/j.abb.2016.08.009
- Suplatov D.A., Voevodin V.V., Svedas V.K. Robust Enzyme Design: Bioinformatic Tools for Improved Protein Stability//Biotechnology Journal. 2015. Vol. 10, No. 3. P. 344-355 DOI: 10.1002/biot.201400150
- Shcherbakova T., Panin N., Suplatov D., Shapovalova I, Švedas V. The βD484N Mutant of Penicillin Acylase from Escherichia coli is More Resistant to Inactivation by Substrates and Can Effectively Perform Peptide Synthesis in Aqueous Medium//Journal of Molecular Catalysis B: Enzymatic. 2015. Vol. 112. P. 66-68 DOI: 10.1016/j.molcatb.2014.11.015
- Suplatov D., Panin N., Kirilin E., Shcherbakova T., Kudryavtsev P., Švedas V. Computational Design of a pH Stable Enzyme: Understanding Molecular Mechanism of Penicillin Acylase's Adaptation to Alkaline Conditions//PLoS ONE. 2014. Vol. 9, No. 6. P. e100643 DOI: 10.1371/journal.pone.0100643
- Suplatov D., Besenmatter W., Švedas V., Svendsen A. Bioinformatic Analysis of Alpha/Beta-Hydrolase Fold Enzymes Reveals Subfamily-Specific Positions Responsible for Discrimination of Amidase and Lipase Activities//Protein Engineering, Design and Selection. 2012. Vol. 25, No. 11. P. 689-697 DOI: 10.1093/protein/gzs068
- Воеводин Вл.В., Жуматий С.А., Соболев С.И., Антонов А.С., Брызгалов П.А., Никитенко Д.А., Стефанов К.С., Воеводин Вад.В. Практика суперкомпьютера «Ломоносов»//Открытые системы. 2012. Т. 7. С. 36-39.
- Nobile M.S., Cazzaniga P., Tangherloni A., Besozzi D. Graphics Processing Units in Bioinformatics, Computational Biology and Systems Biology//Briefings in Bioinformatics. 2016. P. 870-885 DOI: 10.1093/bib/bbw058


