Идентификация нового генотипа ВЛКРС
Автор: Шаева А.Ю., Гараева З.Р., Вафин Р.Р., Хазипов Н.З., Алимов А.М.
Статья в выпуске: 3 т.211, 2012 года.
Бесплатный доступ
На основании филогенетического анализа секвенированных последовательностей локуса env-гена провируса BLV охарактеризован новый генотип ВЛКРС, факт существования которого также подтвержден хорватскими исследователями. Усовершенствована схема ПЦР-ПДРФ-генотипирования BLV в соответствии с филогенетической классификацией данного возбудителя.
Влкрс, генотип, идентификация, пцр, пдрф, филогенетический анализ
Короткий адрес: https://sciup.org/14287578
IDR: 14287578 | УДК: 619:57.065:578.828
Текст научной статьи Идентификация нового генотипа ВЛКРС
Введение. Лейкоз крупного рогатого скота – инфекционная болезнь опухолевой природы, этиологическим агентом которой является одноименный вирус (ВЛКРС, англ. Bovine leukemia virus, BLV), относящийся к роду Deltaretrovirus семейства Retroviridae [2, 3, 4, 5, 6. 7].
Современный подход оценки генотипического разнообразия ВЛКРС на основе филогенетического анализа локуса env -гена возбудителя является более информативным инструментом идентификации генотипов [6, 7], нежели предложенные ранее стратегии типизации, основанные на интерпретации генерируемых env -ПЦР-ПДРФ-профилей [3, 4, 5].
Дальнейшее же развитие таксономической классификации BLV и совершенствование способов генотипической идентификации напрямую связано с получением новых знаний о генетическом многообразии возбудителя по мере выявления оригинальных изолятов ВЛКРС [1, 2].
Материалы и методы. При постановке ПЦР с выделенными образцами провирусной ДНК BLV использовалась модификация «nested» ПЦР с применением внешних (env5032+env5608) и внутренних (env5099+env5521) праймеров, инициирующих на заключительном этапе реакции амплификацию фрагмента env -гена ВЛКРС длиной 444 п.н. [1, 3].
Секвенирование продуктов амплификации локуса env-гена выявленных изолятов провируса ВЛКРС «N10», «N28», «N72» и «N174» выполнено на приборе ABI-300 в лабораториях НПО «СибЭнзим» с использованием праймеров «env5099» и «env5521» в качестве сиквенсных олигонуклеотидов.
Секвенированные последовательности локуса env -гена изолятов BLV «N10» (GenBank A/N: HM102355), «N28» (GenBank A/N: HM102356), «N72» (GenBank A/N: JF683619) и «N174» (GenBank A/N: JF713455) были выравнены по длине 400 нуклеотидов c соответствующими опубликованными в GenBank нуклеотидными последовательностями известных представителей BLV, используя программы BLAST и MEGA-4 с последующим филогенетическим анализом.
Эндонуклеазы рестрикции, подобранные для ПЦР-ПДРФ-генотипирования ВЛКРС в соответствии с филогенетической классификацией: PvuII , SspI , HphI , HaeIII , BstYI .
ПЦР-ПДРФ-моделирование: NEBcutter v.2.0.
ПЦР-ПДРФ-продукты разгоняли в 2,5% агарозном геле (20 В/см, 40 мин) и визуализировали в УФ-трансиллюминаторе (λ=310 нм).
Результаты исследования и их обсуждение. Согласно результатам филогенетического анализа выравненных нуклеотидных последовательностей локуса env -гена типовых представителей известных генотипов ВЛКРС было установлено, что изолят «N28», выявленный в Спасском районе Республики Татарстан, принадлежал к кластеру 7-го генотипа, а изоляты «N10» и «N72», выявленные соответственно в Мензелинском и Дрожжановском районах Республики Татарстан, относились к 4-му генотипу BLV.
Еще один изолят «N174», выявленный также в Дрожжановском районе Республики Татарстан, характеризовался признаком нового, ранее не обоснованного генотипа ВЛКРС, обозначенного нами «8-й генотип», ввиду формирования на выстраиваемой дендрограмме автономного генотипического кластера, в который также входили, на момент обоснования нами нового генотипа BLV [1], близкородственные изоляты, выявленные в Украине [«4-1» (GenBank A/N: HM563766); «3-41» (GenBank A/N: HM563767]) и Хорватии [«M1/ELG_Cro/08» (GenBank A/N: GU724606)], соответственно.
Необходимо отметить, что чуть позже выхода нашей предыдущей статьи [1] с обоснованием 8-го генотипа BLV, в другой научной публикации [2] уже хорватских исследователей был отражен результат сравнительного филогенетического анализа выявленных в Хорватии изолятов ВЛКРС, также подтверждающий факт наличия нового генотипа в таксономической классификации BLV.
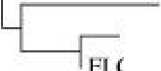
В настоящей же работе нами представлена дендрограмма типовых представителей известных генотипов ВЛКРС с включением в филогенетическое дерево всех депонированных в GenBank NCBI (по состоянию на май 2012 г.) нуклеотидных последовательностей env -гена изолятов нового 8-го генотипа BLV (рис. 1).
Полученные результаты наглядно демонстрируют присутствие автономного 8-го генотипического кластера (рис. 1).
Дополнительно, в рамках совершенствования схемы ПЦР-ПДРФ-генотипирования, согласующейся с филогенетической классификацией ВЛКРС, в процессе системного анализа рестрикционных картирований локуса env -гена известных представителей BLV, нами подобраны новые условия идентификации по 5 эндонуклеазам рестрикции ( PvuII , SspI , HphI , HaeIII и BstYI ). Обобщенная информация с интерпретацией полученных env -ПЦР-ПДРФ-профилей BLV представлена в табл. 1.
--- LO (UMI 02355)
. "I---—— 3(1187872)
BG (EF06563 8)
---------- N72 (JF683619)
4-2 ЙМ5637В1)
— CROC (EF065639)
ERAS-2 (EPO65636)
-----CRLjC-1 (EF065655)
— I 51 {AY185360}
50(DQ0 5 9421)
--------PL-1238 (PJ8O8582)
--------------12 (S83530)
3O(DQO59417)
--------- ^28 {HM] 023 5 6) ---- 1 (AY515280)
-------14 (AY515274)
I--- USCA- 1 (EF065647)
_ । USCA-2 (Ef-065648) ---------JPFU (EFO65 650) _
--АЫ453 (EJ808 577) ------PL-4960 (FJ808 590)
AL-164 (FJ806574}
-
- ARGSF 8 (AE 485773)
— N 174 (JF7I3455]
. 3-4 3 {HM 5 63 767)
_ 4-1 {HM563 766)
Е1О£гаМПЮ9 (JN99OO73)
MI /ELG Cro/08 (GU724606)
EU3(^EM/0 8(JN9900 69}
ELG Gratis AM (JN990070)
ELG CratiRA/to (J N 990071)
ELG Cto/VRAM {JN99O072)
^ Сга.'ЫО/W (JN990074)
KurdEtan {EU266062)

- VdM {М35239)
— Cow 52 7 (AF007764)
------23 (L'87873)
AL-63 {FJ8O857I)
______I AL-2106 {H808 578)
■■ UnK’OOU (Ш9$$
I-------------------------------------1 0005
Рис. 1/ Дендрограмма типовых представителей известных генотипов BLV ( env -ген) [MEGA-4: алгоритм NJ, 400 nt, 40 seq.]
Табл. 1. Усовершенствованная схема ПЦР-ПДРФ-генотипирования BLV в соответствии с филогенетической классификацией
|
Г |
Изолят |
GenBank A/N |
ПЦР-продукт (п.н.) |
ПДРФ-фрагменты (п.н.) |
К |
N |
||||
|
PvuII |
SspI |
HphI |
HaeIII |
BstYI |
||||||
|
1-й |
AL-63 |
FJ808571 |
444 |
444 |
399/45 |
224/220 |
198/94/87/32/27/6 |
198/128/118 |
1 |
50 |
|
1-й |
Cow 527 |
AF007764 |
444 |
444 |
399/45 |
224/220 |
285/94/32/27/6 |
198/128/118 |
2 |
8 |
|
1-й |
23 |
U87873 |
444 |
444 |
399/45 |
224/220 |
312/94/32/6 |
198/128/118 |
3 |
1 |
|
1-й |
AL-2106 |
FJ808578 |
444 |
444 |
399/45 |
224/220 |
198/94/87/32/27/6 |
246/198 |
4 |
39 |
|
1-й |
UruC06II |
FM955558 |
444 |
444 |
399/45 |
224/220 |
285/94/32/27/6 |
246/198 |
5 |
1 |
|
1-й |
VdM |
M35239 |
444 |
444 |
399/45 |
224/181/39 |
198/94/87/32/27/6 |
316/128 |
6 |
1 |
|
1-й |
Kurdistan |
EU266062 |
444 |
444 |
399/45 |
220/196/28 |
198/119/94/27/6 |
198/128/118 |
7 |
1 |
|
2-й |
AL-164 |
FJ808574 |
444 |
280/164 |
399/45 |
224/220 |
198/94/87/32/27/6 |
198/128/118 |
8 |
34 |
|
2-й |
PL-4960 |
FJ808590 |
444 |
280/164 |
399/45 |
224/220 |
198/87/49/45/32/27/6 |
198/128/118 |
9 |
1 |
|
2-й |
ARGSF8 |
AF485773 |
444 |
280/164 |
399/45 |
444 |
198/94/87/32/27/6 |
198/128/118 |
10 |
1 |
|
2-й |
AL-1453 |
FJ808577 |
444 |
280/164 |
444 |
224/220 |
198/94/87/32/27/6 |
198/128/118 |
11 |
1 |
|
3-й |
USCA-1 |
EF065647 |
444 |
444 |
399/45 |
444 |
285/94/32/21/6/6 |
198/128/96/22 |
12 |
1 |
|
3-й |
USCA-2 |
EF065648 |
444 |
444 |
399/45 |
444 |
285/94/32/27/6 |
198/128/96/22 |
13 |
2 |
|
3-й |
JPFU |
EF065650 |
444 |
444 |
399/45 |
444 |
285/94/32/27/6 |
198/128/118 |
14 |
1 |
|
4-й |
N10 |
HM102355 |
444 |
280/164 |
399/45 |
224/220 |
198/94/87/32/27/6 |
444 |
15 |
25 |
|
4-й |
3 |
U87872 |
444 |
444 |
399/45 |
224/220 |
198/94/87/32/27/6 |
444 |
16 |
1 |
|
5-й |
CRAS-2 |
EF065636 |
444 |
280/164 |
399/45 |
224/181/39 |
198/94/87/32/27/6 |
316/128 |
17 |
7 |
|
5-й |
CRGC |
EF065639 |
444 |
280/164 |
399/45 |
224/181/39 |
285/94/32/27/6 |
316/128 |
18 |
1 |
|
5-й |
CRLC-1 |
EF065655 |
444 |
280/164 |
444 |
224/181/39 |
198/94/87/32/27/6 |
316/128 |
19 |
2 |
|
6-й |
PL-1238 |
FJ808582 |
444 |
444 |
399/45 |
224/220 |
285/94/32/27/6 |
316/128 |
20 |
3 |
|
6-й |
151 |
AY185360 |
444 |
444 |
399/45 |
224/220 |
198/94/87/32/27/6 |
316/128 |
21 |
7 |
|
7-й |
N28 |
HM102356 |
444 |
444 |
444 |
224/137/83 |
198/94/87/32/27/6 |
294/128/22 |
22 |
1 |
|
7-й |
1 |
AY515280 |
444 |
444 |
444 |
224/137/83 |
198/94/87/32/27/6 |
316/128 |
23 |
16 |
|
7-й |
I2 |
S83530 |
444 |
444 |
444 |
224/220 |
285/94/32/27/6 |
316/128 |
24 |
1 |
|
7-й |
14 |
AY515274 |
444 |
444 |
444 |
145/137/83/79 |
198/94/87/32/27/6 |
316/128 |
25 |
1 |
|
7-й |
30 |
DQ059417 |
444 |
444 |
444 |
444 |
198/87/49/45/32/27/6 |
316/128 |
26 |
1 |
|
8-й |
N174 |
JF713455 |
444 |
444 |
399/45 |
224/220 |
225/94/87/32/6 |
316/128 |
27 |
1 |
|
8-й |
3-43 |
HM563767 |
444 |
444 |
399/45 |
224/220 |
225/94/87/32/6 |
198/128/118 |
28 |
7 |
|
8-й |
ELG_Cro/VRA/09 |
JN990072 |
444 |
444 |
444 |
224/220 |
225/94/87/32/6 |
198/128/118 |
29 |
2 |
Обозначения: Г – генотип ; К – комбинация ; N – количество проанализированных изолятов BLV с соответствующей комбинацией ПЦР-ПДРФ-профилей, чьи нуклеотидные последовательности локуса env -гена депонированы в базу данных GenBank (по состоянию на май 2012 г).
Так, 1-ый генотип BLV характеризуется наличием 7 комбинаций env -ПЦР-ПДРФ-профилей (К1-7), 2-ой генотип – четырёх комбинаций (К8-11), 3-ий генотип – наличием трёх комбинаций (К12-14); 4-ый генотип – двух комбинаций (К15-16); 5-ый генотип – трех комбинаций (К17-19); 6-ой генотип – двух комбинаций (К20-21), 7-ой генотип – пяти комбинаций (К22-26) и 8 генотип ВЛКРС – наличием трех комбинации (К27-29) env -ПЦР-ПДРФ-профилей BLV (табл. 1).
Следует подчеркнуть, что целый ряд комбинаций env -ПЦР-ПДРФ-профилей на данный момент представлен единственными нуклеотидными последовательностями локуса env -гена изолятов ВЛКРС, депонированными в GenBank NCBI, в числе которых и выявленные нами изоляты N28 (22 комбинация, 7-ой генотип) и N174 (27 комбинация, 8-ой генотип), соответственно (табл. 1).
Примечательно, что для идентификации 4-го генотипа ВЛКРС достаточно даже одной эндонуклеазы рестрикции – BstYI . Охарактеризованный в нашей работе новый 8-ой генотип ВЛКРС также может быть идентифицирован с использованием одной единственной рестриктазы – HaeIII . С применением двух ферментов могут быть определены представители 2-го [ PvuII и BstYI ], 3-го [ HphI и HaeIII ] и 5-го [ PvuII и BstYI ] генотипов BLV (табл. 1).
В целях же исчерпывающей идентификации генотипов ВЛКРС целесообразно руководствоваться полной картиной env -ПЦР-ПДРФ-профилей с сопоставлением полученных данных с результатами секвенирования ПЦР-продуктов и последующим филогенетическим анализом представителей BLV по локусу env -гена.
ЛИТЕРАТУРА. 1. Шаева, А.Ю. Генотипическая идентификация изолятов ВЛКРС, выявленных в хозяйствах Республики Татарстан / А.Ю. Шаева, Р.Р. Вафин, Н.З. Хазипов, Б.В. Камалов, А.М. Алимов, М.Ш. Тагиров // Ученые записки КГАВМ. – Казань, 2011. – Т. 208. – С. 330-337. 2. Balic, D. Identification of a new genotype of bovine leukemia virus / D. Balic, I. Lojkic, M. Periskic, T. Bedekovic, A. Jungic, N. Lemo, B. Roic, Z. Cac, L. Barbic, J. Madic // Arch. Virol. – 2012 – Apr 10 [Epub ahead of print]. 3. Beier, D. Identification of different BLV provirus isolates by PCR, RFLPA and DNA sequencing / D. Beier, P. Blankenstein, O. Marquardt, J. Kuzmak // Berl Munch Tierarztl Wochenschr – 2001. – V. 114. – № 7-8. – P. 252-256. 4. Fechner, H. Provirus variants of the bovine leukemia virus and their relation to the serological status of naturally infected cattle / H. Fechner, P. Blankenstein, A.C. Looman, J. Elwert, L. Geue, C. Albrecht, A. Kurg, D. Beier, O. Marquardt, D. Ebner // Virology –1997. – V. 237. – № 2. – P. 261-269. 5. Licursi, M. Genetic heterogeneity among bovine leukemia virus genotypes and its relation to humoral responses in hosts / M. Licursi, Y. Inoshima, D. Wu, T. Yokoyama, E.T. González, H. Sentsui // Virus Res. – 2002. – V. 86. – № 1-2. – P. 101-110. 6. Moratorio, G. Phylogenetic analysis of bovine leukemia viruses isolated in
South America reveals diversification in seven distinct genotypes / G. Moratorio, G. Obal, A. Dubra, A. Correa, S. Bianchi, A. Buschiazzo, J. Cristina, O. Pritsch // Arch. Virol. – 2010. – V. 155. – № 4. – P. 481-489. 7. Rodriguez, S.M. Bovine leukemia virus can be classified into seven genotypes: evidence for the existence of two novel clades / S.M. Rodriguez, M.D. Golemba, R.H. Campos, K. Trono, L.R. Jones //J Gen Virol. – 2009. – V. 90. – № 11. – P. 2788-2797.
ИДЕНТИФИКАЦИЯ НОВОГО ГЕНОТИПА ВЛКРС
Шаева А.Ю., Гараева З.Р., Вафин Р.Р., Хазипов Н.З., Алимов А.М. Резюме
На основании филогенетического анализа секвенированных последовательностей локуса env -гена провируса BLV охарактеризован новый генотип ВЛКРС, факт существования которого также подтвержден хорватскими исследователями. Усовершенствована схема ПЦР-ПДРФ-генотипирования BLV в соответствии с филогенетической классификацией данного возбудителя.
IDENTIFICATION OF A NEW BLV GENOTYPE
Shaeva A.Y., Garaeva Z.R., Vafin R.R., Khazipov N.Z., Alimov A.M. Summary
Based on phylogenetic analysis of the BLV env -gene sequences was characterized a new genotype of bovine leukemia virus, the existence of which is also confirmed by Croatian researchers. A scheme of PCR-RFLP-genotyping of BLV in accordance with the phylogenetic classification of this pathogen was improved.


