Иммунологически значимые гликопротеины p54 и CD2v вируса африканской чумы свиней: биоинформатический анализ генетических вариаций и гетерогенности
Автор: Мима К.А., Бурмакина Г.С., Титов И.А., Малоголовкин А.С.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Вирусы - возбудители особо опасных инфекций: молекулярная генетика, иммуногенность, культивирование
Статья в выпуске: 6 т.50, 2015 года.
Бесплатный доступ
Вирус африканской чумы свиней (АЧС) - уникальный представитель возбудителей арбовирусных инфекций, который до сих пор остается единственным представителем семейства Asfarviridae. Он служит возбудителем одной из самых опасных болезней представителей семейства Suidae и, кроме того, способен инфицировать мягких клещей рода Ornithodoros. Генетическая и фенотипическая гетерогенность вируса АЧС остается одной из основных причин отсутствия вакцин против этого опасного трансграничного заболевания. Нами изучена структура и функции наиболее вариабельных гликопротеинов вируса АЧС p54 и CD2v с использованием биоинформатического анализа и рекомбинантных конструкций, экспрессируемых в экстрахромосомном состоянии в культурах клеток млекопитающих COS-I (клетки африканской зеленой мартышки) и HEK-293 (клетки почки эмбриона человека). Согласно результатам проведенного биоинформатического анализа, основанного на расчете индекса разнообразия аминокислотных последовательностей в пропорциях по методу Симпсона, вариабельная область (N-концевой участок) СD2v сильно гликозилирована (содержит 28-30 сайтов N- и О-гликозилирования) и находится на наружной поверхности клеточной мембраны. Эта область также содержит иммуноглобулиновый домен (1-225-я аминокислоты), что обусловливает взаимодействие СD2v с антителами. Выявленные различия в посттрансляционных модификациях и наличие генетических вариаций по белку СD2v могут объяснять разнообразие в проявлении феномена гемадсорбции у изолятов вируса АЧС. Напротив, белок p54 имеет вариабельную часть, локализованную с внутренней стороны клеточной мембраны, и гликозилированный экстраклеточный участок, расположенный с ее внешней стороны. Высокий уровень различий в нуклеотидных последовательностях гена p54 ( E183L ) у разных изолятов вируса АЧС может быть результатом мутационной изменчивости в ходе эволюции. Выявленными особенностями посттрансляционных модификаций и генетическими вариациями по белку СD2v, по всей видимости, объясняется разнообразие антигенных свойств изолятов вируса АЧС. Таким образом, в представленной работе впервые проанализирована посттрансляционная модификация наиболее вариабельных гликопротеинов вируса АЧС - p54 и СD2v. Посредством транзиентной экспрессии в клеточных линиях млекопитающих получены рекомбинантные химерные продукты p54-EGFP и CD2v-HA, что позволило выявить различную локализацию вирусных белков в трансфицированных клетках. В частности, при исследовании культуры клеток COS-I, трансфицированных плазмидой р54-pEGFP-N1, была установлена цитоплазматическая локализация флуоресцентно меченного рекомбинантного белка p54-EGFP. В культуре клеток НЕК-293 белок CD2v обнаруживался исключительно в составе клеточной мембраны. По данным иммуноблоттинга, молекулярная масса CD2v равнялась примерно 90 кДа (при расчетной 65 кДа). Следовательно, в этом поверхностном гликопротеине вириона АЧС на углеводный компонент приходится около 30 % массы. Кроме того, результаты иммуноблоттинга указывали на наличие двух вариантов белка CD2v с молекулярной массой 25 кДа и 90 кДа, то есть предположительно его различных гликозилированных форм, что согласуется с сообщениями в специальной литературе. Итак, выполненный биоинформатический анализ и результаты исследований в культурах клеток COS-I и HEK-293 показали, что именно СD2v может считаться наиболее вероятным белком-кандидатом, определяющим взаимодействие вируса АЧС с вирусоспецифическими антителами.
Африканская чума свиней, гликопротеины, вариабельность, гликозилирование, транзиентная экспрессия, иммунотипы
Короткий адрес: https://sciup.org/142134845
IDR: 142134845 | УДК: 636.4:619:616.98:578:577.2.08:51-76 | DOI: 10.15389/agrobiology.2015.6.785rus
Текст научной статьи Иммунологически значимые гликопротеины p54 и CD2v вируса африканской чумы свиней: биоинформатический анализ генетических вариаций и гетерогенности
Вирус африканской чумы свиней (АЧС) до настоящего времени остается единственным известным представителем семейства Asfarviridae (1-3). Он служит возбудителем одной из самых опасных болезней представителей семейства Suidae и, кроме того, способен инфицировать мяг-
Исследование выполнено при финансовой поддержке РФФИ в рамках научного проекта «Мол-а-вед» (15-34-20995).
ких клещей рода Ornithodoros (4-6). Геном вируса представлен двуцепочечной ДНК, размер которой в зависимости от штамма варьирует от 170 до 190 тыс. п.н. (7, 8). Анализ полной нуклеотидной последовательности авирулентного изолята BA71V выявил наличие не менее 151 гена (9). Для некоторых белков, закодированных в геноме, определена функция и роль в патогенезе (10-16), однако значительное их число до сих пор полностью не охарактеризованы.
Вирусная частица по морфологии очень похожа на иридовирусы (17, 18), имеет диаметр около 200 нм и состоит из нескольких концентрических доменов (19, 20). Около 35 % массы вириона составляет основной капсидный белок p72. На структурные белки p150, p37, p34 и p14 (их предшественник — полипротеин p220) приходится до 25 % массы частицы (21, 22). Из всех известных белков вируса АЧС гликопротеины р54 и CD2v, расположенные на поверхности вирусной частицы, наиболее вариабельны. Белок p54 (кодируется геном E183L ) близко к N-концу содержит потенциальную область трансмембранного домена. Этот белок принимает участие в ранних стадиях развития вирусной инфекции и отвечает за присоединение вируса АЧС к клетке-мишени. Молекулярная масса полипеп-тидной части белка р54 у разных вирусных изолятов варьирует от 24 до 28 кДа (23). Белок CD2v обусловливает гемадсорбирующие свойства вируса и кодируется геном EP402R . Для репликации вируса в культурах клеток CD2v несуществен (24, 25). У природных изолятов, не способных к гемад-сорбции, обнаружены изменения в последовательности гена EP402R , соответствующей сигнальному участку белка, которые приводят к сдвигу рамки считывания. CD2v — трансмембранный белок с N-концевым сигнальным пептидом и одним трансмембранным доменом, который имеет сходство с доменом внеклеточного белка CD2 клетки-хозяина (24, 26, 27).
Несмотря на функциональные различия, оба гликопротеина (p54 и СD2v), по данным ряда авторов (24, 28), обусловливают фенотипические характеристики вируса АЧС, проявляя свойства серотипоспецифических антигенов. Определение серотипа изолятов чрезвычайно важно для изучения биологии вируса АЧС и создания средств специфической профилактики этого заболевания.
В представленной работе нами впервые проанализирована посттрансляционная модификация наиболее вариабельных гликопротеинов вируса АЧС — p54 и СD2v. Посредством транзиентной экспрессии в клеточных линиях млекопитающих получены рекомбинантные химерные белки p54-EGFP и CD2v-HA и показана их клеточная локализация.
Цель настоящей работы заключалась в анализе структуры и роли наиболее вариабельных гликопротеинов вируса АЧС p54 и CD2v на основе методов биоинформатического анализа, функциональной геномики, молекулярного клонирования и экспрессии рекомбинантных конструкций.
Методика . Нуклеотидные последовательности генов p54 и CD2v вируса африканской чумы свиней (АЧС) были получены из международной базы данных GenBank (the National Center for Biotechnology Information — NCBI): NC_001659.2, GQ410768.1, GQ410771.1, GQ410767.1, KC610537.1, KC610538.1, KJ671542.1, KC610535.1, KC610532.1, KF303309.1, KF303306.1, KC990883.1, EU620682.1, FR682468.1, FN557520.1, AM712240.1, AF481876.1, DQ026270.1, DQ026267.1, KM262845.1, KM262844.1, DQ026268.1.
Анализ вариабельных областей генов, доменов и структуры белков р54 и СD2v проводили при помощи сервиса Для определения степени N- и О-гликозилирования использовали сервис , для анализа вариабельности аминокислот-786
ных последовательностей — сервис (29, 30).
При дизайне праймеров нуклеотидную последовательность ДНК штамма Georgia2007/wb вируса АЧС (FR682468.1) использовали в качестве референтной. Праймеры, фланкирующие полноразмерные копии вирусных генов р54 и СD2v, конструировали с помощью программ SerialCloner v. 2.6.1 (F. Perez, 2004), Oligo v. 6.71 (P. Rychlik, 2005). В олигонуклеотид-ные праймеры были встроены специфические гексамерные последовательности сайтов рестрикции эндонуклеаз SacI, EcoRI для р54 и BglII и EcoRI для CD2v.
Нуклеотидные последовательности генов p54 и СD2v синтезировали в полимеразной цепной реакции (ПЦР) с использованием One Taq Hot start master mix («New England Biolabs», США) на приборе Maxygene thermal cycler («Axygen Scientific Inc.», США) согласно рекомендациям производителя. Программа включала 30 с начальной денатурации при 94 °С и 40 циклов ПЦР (денатурация — 30 с при 94 °С, отжиг праймеров — 30 с при 55 °С, элонгация — 2 мин при 72 °С). ДНК-мишень выделяли из штамма Став-рополь-08/01 вируса АЧС (получен из коллекции микроорганизмов Всероссийского НИИ ветеринарной вирусологии и микробиологии) при помощи набора QIAamp DNA Mini Kit («Qiagen N.V.», Германия) согласно инструкции производителя. Ампликоны разделяли электрофоретически в 1,5 % агарозном геле, содержащем 0,001 % бромистого этидия, при силе тока 50 мА. Для очистки ПЦР-продуктов от агарозного геля использовали набор QIAquick Gel Extraction Kit («Qiagen N.V.», Германия).
На этапе промежуточного клонирования ПЦР-продукты встраивали в вектор pGEM-T-easy с маркером устойчивости к ампициллину («Pro-mega», США) и сайтами расщепления специфическими эндонуклеазами, используя ДНК-лигазу фага T4 («NEB», США). Трансформацию клеток Escherichia coli Dh5 полученными конструкциями осуществляли методом электропорации на приборе Gene Pulser Xcell («Bio-Rad», США). Из отобранных устойчивых к ампициллину трансформантов выделяли плазмидную ДНК набором QIAGEN Plasmid Mini Kit («Qiagen N.V.», Германия). Наличие специфической вставки в рекомбинантных плазмидах подтверждали в ПЦР с применением геноспецифических праймеров, проверяя ее идентичность секвенированием сайтов рестрикции на генетическом анализаторе Applied Biosystems 3130xl («Applied Biosystems», США). Наличие специфических сайтов рестрикции в созданных рекомбинантных плазмидах подтвердили также с использованием эндонуклеаз SacI, EcoRI при клонировании последовательностей гена р54 и BglII и EcoRI — в случае гена CD2v.
Для контроля функциональной целостности клонированных целевых участков вирусного генома и получения генно-инженерных конструкций, обеспечивающих экспрессию белков p54 и СD2v в клетках млекопитающих, кодирующие последовательности генов E183L (p54) и EP402L (СD2v) из рекомбинантных плазмид pGEM-T-easy, где они находились под контролем промотора бактериофага Т7, последовательно реклониро-вали по указанным выше сайтам рестрикции в акцепторные векторы с цитомегаловирусным промотором. В случае p54 таким вектором была плазмида pEGFP-N1 («Clontech», США) с геном устойчивости к канами-цину и eGFP (enhanced green fluorescence protein), кодирующим маркерный флуоресцентный белок. Для СD2v выбрали вектор pCMV-HA-C («Clontech», США) c геном устойчивости к ампициллину, содержащий короткую (29 нуклеотидов) последовательность, которая кодирует иммуногенный участок гемагглютинина (НА) вируса гриппа. ДНК полученных 787
плазмидных конструкций очищали на анионообменных колонках с использованием набора QIAGEN Plasmid Maxi Kit («Qiagen N.V.», Германия).
Трансфекцию культур клеток африканской зеленой мартышки (COS-I) и почки эмбриона человека (НЕК-293) очищенными рекомбинантными плазмидами (2,5 мкг на 1 лунку) проводили в 6-луночных планшетах с применением липофектамина («Invitrogen», США) согласно инструкции производителя (31).
Через 24, 48, 72 и 96 ч после трансфекции проводили скрининг клеток на наличие репортерной флуоресценции под инвертированным флуоресцентным микроскопом Olympus MIT-2 (Япония), клетки собирали при помощи скребка и лизировали в TBS буфере (0,1 % Твин 20) с добавлением ингибитора протеаз Protease Inhibitor Cocktail Set V («Calbiochem», США). Для подтверждения наличия рекомбинантных белков использовали метод иммуноблоттинга (32) с моноклональными антителами GFP(FL) («Santa Crus Biotechnology», США) к репортерному белку GFP и c моноклональными антителами HA-probe (F-7) («Santa Crus Biotechnology», США) к маркерному НА-участку и вторичными антивидовыми моноклональными антителами, меченными пероксидазой хрена. Субстраты для детекции — соответственно набор Clarity Western ECL Substrat («Bio-Rad», США) и DAB («Thermo Scientific», США).
Результаты . Для анализа вариабельных областей генов был использован метод Симпсона, основанный на расчете индекса разнообразия аминокислотных последовательностей в пропорциях. Индекс описывает вероятность того, что две последовательности с заменами, выбранные случайным образом, будут схожи с остальными аминокислотными последовательностями. Значения этого индекса находятся в диапазоне от 0 до 1 и тем больше, чем больше разнообразие образцов.
А

Рис. 1. Результаты анализа профилей вариабельности капсидных гликопротеинов p54 (А) и СD2v (Б) у различных штаммов вируса африканской чумы (АЧС) свиней по методу Симпсона: ТД — трансмембранный домен. Вариабельными считаются области, для которых полученные значения превышают пороговое (соответствующая горизонтальная линия). Аминокислотные последовательности получали посредством трансляции нуклеотидных последовательностей генов, депонированных в базе данных GenBank (всего проанализированы 22 изо-лята вируса АЧС).
Аминокислотные последовательности рассчитывали на основании нуклеотидных последовательностей генов, депонированных в базе данных GenBank. Анализ аминокислотных последовательностей белков р54 ( E183L ) у различных изолятов ( n = 22) показал, что наиболее вариабелен его С-концевой участок (аминокислоты в позициях с 62-й по 185-ю), локализованный с внутренней стороны клеточной мембраны (рис. 1, А). Указанная область состоит из тандемных повторов четырех аминокислот (Ile-Ile-Ile-Ile, Аla-Аla-Аla-Аla). Ближе к N-концу находится участок белка, проходящий через мембрану клетки, — трансмембранный домен (с 25-й по 45-ю аминокислоту). Белок CD2v ( EP402L ) имеет вариабельную N-концевую часть, расположенную с наружной стороны клеточной мембраны (с 1-й по 225-ю аминокислоту) (рис. 1, Б), и закреплен в мембране при помощи трансмембранного домена. Согласно профилям гетерогенности, CD2v обладал большей вариабельностью по сравнению c p54.
Анализ аминокислотных последовательностей на наличие сайтов N- и О-гликозилирования показал, что для белка р54 характерно преобладание О-гликозилирования и большинство потенциальных сайтов (18 сайтов) гликозилирования располагается на С-конце молекулы. У белка CD2v более выраженным было N-гликозилирование, представленное преимущественно на N-концевом участке (28-30 сайтов) (рис. 2).
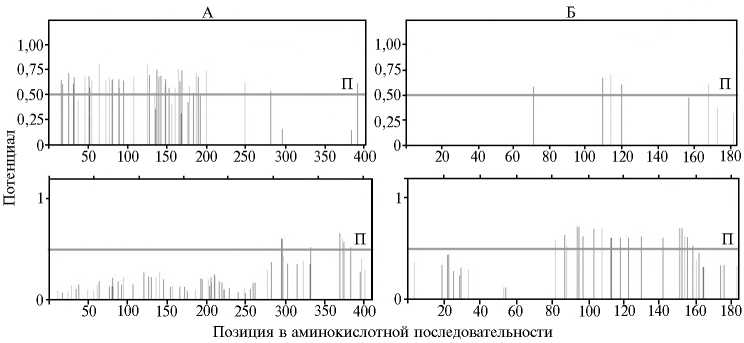
Рис. 2. Анализ распределения потенциальных сайтов N- (верхний ряд) и О-гликозили-рования (нижний ряд) в капсидных гликопротеинах CD2v (А) и р54 (Б) вируса африканской чумы свиней. Пересечение с горизонтальной пороговой линией (П) означает, что в соответствующих аминокислотных позициях гликозилирование наиболее вероятно. N- и О-гли-козилирование в белках прогнозировали на основании анализа аминокислотных последовательностей и наличия определенных сочетаний аминокислот, потенциально соответствующих сайтам гликозилирования. Для CD2v было выявлено 28-30 сайтов N-гликозилирования и 5-7 сайтов О-гликозилирования, для р54 — 5 сайтов N-гликозилирования и 18 сайтов О-гликозилирования.
Следует отметить, что ген EP402L (CD2v), ответственный за гемад-сорбцию, был идентифицирован и у негемадсорбирующих штаммов (14, 24), однако открытая рамка считывания у них нарушена. Мы показали, что у гемадсорбирующих и не способных к гемадсорбции (Л-50, ЛЦПП, Kонго-49, Мфуати-79, Мозамбик-78, Франция-32, РРА, PSA-1/NH, Lee, Уганда, ТС7, Родезия-79) штаммов вируса АЧС число потенциальных сайтов гликозилирования CD2v не различалось. Нам удалось установить, что у всех негемадсорбирующих штаммов вируса АЧС в последовательности аминокислот белка CD2v отсутствует сигнальный пептид и сайт разрезания сигнальной пептидазой. Поэтому CD2v не подвергается гликозилированию даже при наличии соответствующих потенциальных сайтов.
Таким образом, результаты биоинформатического анализа позволили сделать вывод, что вариабельная область (N-концевой участок) молекулы СD2v сильно гликозилирована и находится на наружной поверхности клеточной мембраны. Эта область также содержит иммуноглобулиновый домен, чем обусловлено взаимодействие СD2v с антителами. Выявленные различия в посттрансляционных модификациях и генетические вариации по белку СD2v могут объяснять разнообразие в антигенных свойствах изолятов вируса АЧС. Напротив, белок p54 имеет вариабельную область, локализованную с внутренней стороны клеточной мембраны, и гликозилированную, находящуюся вне клетки. Значительное несходство в нуклеотидных последовательностях гена p54 ( E183L ) у разных изолятов вируса АЧС может быть результатом мутационной изменчивости в ходе его эволюции.
Продуцентами рекомбинантных белков р54 и СD2v вируса АЧС служили культивируемые клетки COS-I и HEK-293, которые несли конструкции, полученные на основе плазмидных векторов pEGFP-N1 и pCMV-HA-C с цитомегаловирусными (CMV) промоторами и способные экспрессироваться в клетках млекопитающих во внехромосомном состоянии. Открытая рамка считывания для белка p54 была соединена с репортерным геном eGFP в составе акцепторного вектора. Слияние таргет-ного белка с EGFP позволяет детектировать клеточную локализацию химерного белка методом флуоресцентной микроскопии. Во избежание структурных изменений в случае белка CD2v была выбрана стратегия слияния с маркерным HA-участком на С-конце молекулы. HA-последовательность на С-концевом участке рекомбинантного белка не влияет на иммунологические свойства и дает возможность детектировать таргентный белок в нативной конформации, что особенно важно при наличии значительной части углеводного компонента в составе гликопротеина.
А Б
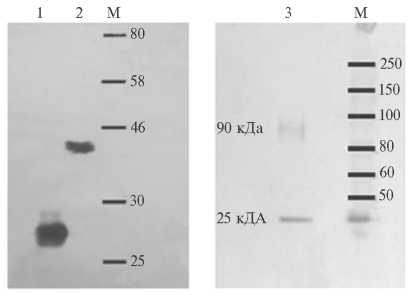
Рис. 3. Обнаружение рекомбинантных капсидных белков р54-EGFP (А) и СD2v-HA (Б) вируса африканской чумы свиней в лизатах культур клеток методом иммуноблоттинга: 1 — EGFP (зеленый флуоресцентный белок), 2 — р54-EGFP, 3 — СD2v-HA; М — маркеры молекулярной массы для р54-EGFP (Unstained Protein Ladder, Broad Range 10-250 kDa; «NEB», США) и СD2v-HA (Prestained Protein Marker, Broad Range 7-175 kDa; «NEB», США). Использованы культуры клеток линии COS-I HEK-293, трансфицированных соответственно рекомбинантными плазмидами р54-pEGFP-N1 (А) и pCMV-CD2v (Б) с применением липофектамина. Моноклональные антитела конъюгированы с пероксидазой хрена;
субстраты для детекции — соответственно Clarity Western ECL Substrat («Bio-Rad», США) и DAB («Thermo Scientific», США). Полосы 25кДа и 90 кДа (Б) — соответственно негликози-лированная и гликозилированная формы белка СD2V-HA.
Репортерную флуоресценцию р54-EGFP наблюдали в цитоплазме трансфицированных клеток СOS-1 на протяжении 96 ч. Наиболее сильный сигнал отмечали между 24 и 48 ч после трансфекции, далее (в период с 72 до 96 ч) он снижался. Экспрессию рекомбинантного СD2v детектировали, наоборот, только в мембране клеток. Рекомбинантные белки p54 и CD2v также обнаруживались в лизате трансфицированных клеток методом иммуноблоттинга с применением моноклональных антител соответственно к зеленому флуоресцентному белку GFP и иммуногенному участку гемагг-790
лютинина (НА) вируса гриппа (рис. 3). Рекомбинантный белок CD2v присутствовал в трансфицированных клетках в двух изоформах (см. рис 3): высокомолекулярная (90 кДа) соответствовала зрелому гликопротеину CD2v, в то время как низкомолекулярная (25 кДа) представляла собой не полностью гликозилированную молекулу.
Итак, гликопротеины р54 и CD2v вируса африканской чумы свиней (АЧС) обладают выраженными структурными и функциональными особенностями, однако их роль в формировании иммунотипов этого патогена до сих пор не выяснена. Из всех структурных белков вируса АЧС р54 и CD2v наиболее вариабельны и представляют интерес с точки зрения изучения его генетической и фенотипической изменчивости в отношении способности индуцировать гемадсорбцию. У белка CD2v преобладает N-гликозилирование внеклеточной части (N-конец), в то время как для р54 характерно О-гликозилирование С-концевой области. Вариабельная часть белка СD2v также находится на N-конце и содержит иммуноглобулиновый домен, что может свидетельствовать о взаимосвязи между гликозилированием и характером взаимодействия с вирусоспецифическими антителами. В то же время в нашем исследовании установлено, что отсутствие феномена гемадсорбции у некоторых штаммов вируса АЧС связано с изменением рамки считывания гена EP402L , кодирующего CD2v, и не зависит от степени его гликозилирования. При исследовании культуры клеток COS-I, трансфицированных плазмидой р54-pEGFP-N1, была установлена цитоплазматическая локализация флуоресцентно меченного рекомбинантного белка p54-EGFP. Белок CD2v обнаруживался исключительно в составе мембраны клеток. По данным иммуноблоттинга, молекулярная масса CD2v cоставила около 90 кДа (при расчетной 65 кДа). Следовательно, в этом поверхностном гликопротеине вириона АЧС на углеводный компонент приходится около 30 % массы. Кроме того, результаты иммуноблоттинга указывают на наличие двух вариантов белка CD2v с молекулярной массой 25 кДа и 90 кДа, то есть предположительно его различных гликозилированных форм, что согласуется с сообщениями в специальной литературе. На основании биоинформатического анализа и исследования культур трансфицированных клеток COS-I и HEK-293 именно СD2v можно считать наиболее вероятным белком-кандидатом, определяющим взаимодействие с вирусоспецифическими антителами и формирование иммунотипов животных при АЧС.
Список литературы Иммунологически значимые гликопротеины p54 и CD2v вируса африканской чумы свиней: биоинформатический анализ генетических вариаций и гетерогенности
- Dixon L.K., Rock D.L., Vinuela E. African swine fever-like viruses. Virus taxonomy: classification and nomenclature of viruses. Arch. Virol., 1995, 69(10): 92-94.
- Michaud V., Randriamparany T., Albina E. Comprehensive phylogenetic reconstructions of African swine fever virus: proposal for a new classification and molecular dating of the virus. PLoS ONE, 2013, 8(7): 1-14 ( ) DOI: 10.1371/journal.pone.0069662
- Hubalek Z., Rudolf I., Nowotny N. Arboviruses pathogenic for domestic and wild animals. Adv. Virus Res., 2014, 89: 201-275 ( ) DOI: 10.1016/B978-0-12-800172-1.00005-7
- Porterfield J.S. The basis of arbovirus classification. Med. Biol., 1975, 53(5): 400-405.
- Burrage T.G. African swine fever virus infection in Ornithodoros ticks. Virus Res., 2013, 173(1): 131-139 ( ) DOI: 10.1016/j.virusres.2012.10.010
- Boinas F., Ribeiro R., Madeira S., Palma M., de Carvalho I.L., Nuncio S., Wilson A.J. The medical and veterinary role of Ornithodoros erraticus complex ticks (Acari: Ixodida) on the Iberian Peninsula. Journal of Vector Ecology, 2014, 39(2): 238-248 ( ) DOI: 10.1111/jvec.12098
- Wilkinson P.J. African swine fever virus. In: Virus infections of porcines/M. Pensaert (ed.). Elsevier Science Publishers, Amsterdam, The Netherlands, 1989: 17-35.
- Chapman D.A., Tcherepanov V., Upton C., Dixon L.K. Comparison of the genome sequences of non-pathogenic and pathogenic African swine fever virus. J. Gen. Virol., 2008, 89(2): 397-408 ( ) DOI: 10.1099/vir.0.83343-0
- Nix R.J., Gallardo C., Hutchings G., Blanco E., Dixon L.K. Molecular epidemiology of African swine fever virus studied by analysis of four variable genome regions. Arch. Virol., 2006, 151(12): 2475-2494 ( ) DOI: 10.1007/s00705-006-0794-z
- Yanez R.J., Rodriguez J.M., Nogal M.L., Yuste L., Enriquez C., Rodriguez J.F., Vinuela E. Analysis of the complete nucleotide sequence of African swine fever virus. Virology, 1995, 208(1): 249-278.
- Villiers E.P., Gallardo C., Arias M., Silva M., Upton C., Martin R., Bishop R.P. Phylogenomic analysis of 11 complete African swine fever virus genome sequences. Virology, 2010, 400(1): 128-136 ( ) DOI: 10.1016/j.virol.2010.01.019
- Chacon M.R., Almazan F., Nogal M.L., Vinuela E., Rodriguez J.F. The African swine fever. Virology, 1995, 214(2): 670-674.
- Martinez P.L., Simon M.C., Lopez-Otín C., Vinuela E. Characterization of the African swine fever virus protein p14.5: a DNA binding protein. Virology, 1997, 229(1): 201-211.
- Dixon L.K., Chapman D.A., Netherton C.L., Upton C. African swine fever virus replication and genomics. Virus Res., 2013, 173(1): 3-14 ( ) DOI: 10.1016/j.virusres.2012.10.020
- Simon M.C., Freije J.M., Andres G., Lopez-Otín C., Vinuela E. Mapping and sequence of the gene encoding protein p17, a major African swine fever virus structural protein. Virology, 1995, 206(2): 1140-1144.
- Simon M.C., Andres G., Almazan F., Vinuela E. Proteolytic processing in African swine fever virus: evidence for a new structural polyprotein, pp62. J. Virol., 1997, 71(8): 5799-5804.
- Carrascosa J.L., Carazo J.M., Carrascosa A.L., Garcia N., Santisteban A., Vinuela E. General morphology and capsid fine structure of African swine fever virus particles. Virology, 1984, 132(1): 160-172.
- Andres G., Simon M.C., Vinuela E. Assembly of African swine fever virus: role of polyprotein pp220. J. Virol., 1966, 71(3): 2331-2341.
- Breese S.S. Jr., DeBoer C.J. Electron microscope observation of African swine fever virus in tissue culture cells. Virology, 1966, 28(3): 420-428.
- Karalova E.M., Voskanian G.E., Sarkisian Kh.V., Abroian L.O., Avetisian A.S., Akopian L.A., Semerdzhian Z.B., Zakarian O.S., Arzumanian G.A., Karalian Z.A. Pathology of lymphoid tissue cells infected by African swine fever virus in vitro. Voprosy virusologii, 2011, 56(1): 33-37.
- Salas M.L., Andres G. African swine fever virus morphogenesis. Virus Res., 2013, 173(1): 29-41 ( ) DOI: 10.1016/j.virusres.2012.09.016
- Suarez C., Salas M.L., Rodriguez J.M. African swine fever virus polyprotein pp62 is essential for viral core development. J. Virol., 2010, 84(1): 176-187 ( ) DOI: 10.1128/JVI.01858-09
- Gomez-Puertas P., Rodriguez F., Oviedo J.M., Brun A., Alonso C., Escribano J.M. The African swine fever virus proteins p54 and p30 are involved in two distinct steps of virus attachment and both contribute to the antibody-mediated protective immune response. Virology, 1998, 243(2): 461-471.
- Середа А.Д., Балышев В.М. Антигенное разнообразие вируса африканской чумы свиней. Вопросы вирусологии, 2011, 4: 38-42.
- Borca M.V., Carrillo C., Zsak L., Laegreid W.W., Kutish G.F., Neilan J.G., Burrage T.G., Rock D.L. Deletion of a CD2-like gene, 8-DR, from African swine fever virus affects viral infection in domestic swine. J. Virol., 1998, 72(4): 2881-2889.
- Quintero J.C., Wesley R.D., Whyard T.C., Gregg D., Mebus C.A. In vitro and in vivo association of African swine fever virus with swine erythrocytes. Am. J. Vet. Res., 1986, 47(5): 1125-1131.
- Rodriguez J.M., Yanez R.J., Almazan F., Vinuela E., Rodriguez J.F. African swine fever virus encodes a Cd2 homolog responsible for the adhesion of erythrocytes to infected cells. J. Virol., 1993, 67(9): 5312-5320.
- Казакова А.С. Конструирование продуцентов рекомбинантных белков Р72, Р30 и Р54 вируса африканской чумы свиней. Канд. дис. Покров, 2013.
- Garcia-Boronat M., Diez-Rivero C.M., Reinherz E.L., Reche P.A. PVS: a web server for protein sequence variability analysis tuned to facilitate conserved epitope discovery. Nucl. Acids Res., 2008, 36: 35-41 ( ) DOI: 10.1093/nar/gkn211
- Díez-Rivero C.M., Reche P. Discovery of conserved epitopes through sequence variability analyses. Bioinformatics for Immunomics, 2010, 3: 95-101 ( ) DOI: 10.1007/978-1-4419-0540-6_8
- Maurisse R., De Semir D., Emamekhoo H., Bedayat B., Abdolmohammadi A., Parsi H., Gruenert D.C. Comparative transfection of DNA into primary and transformed mammalian cells from different lineages. BioMed Central Biotechnol., 2010, 10(9): 2-9 ( ) DOI: 10.1186/1472-6750-10-9
- Laemmli U.K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature, 1970, 227(5259): 680-685.


