Исследование микросателлитных локусов в породах индеек российской селекции
Автор: Фисинин В.И., Селионова М.И., Шинкаренко Л.А., Щербакова Н.Г., Кононова Л.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Птицеводство: наука и технологии
Статья в выпуске: 4 т.52, 2017 года.
Бесплатный доступ
В настоящее время мясо индеек ( Meleagris gallopavo ) занимает второе место в мире в общем объеме производства мяса птицы. По оценке аналитического агентства Global Reach Consulting (GRC), за последние 10 лет этот сегмент вырос более чем в 8 раз. Положительная динамика наблюдается и в России: в 2015 году прирост производства мяса индеек составил 34,9 %, в основном благодаря достижениям современной генетики и использованию высокопроизводительных технологий. В мировом птицеводстве жесткая конкуренция на рынке генетического материала определяет важность контроля его происхождения и генетической консолидированности. Анализ микросателлитных локусов относят к числу наиболее информативных методов изучения генетического биоразнообразия у животных и птицы. Изучение микросателлитов у пород и коммерческих линий индеек, разводимых в США, Турции, Венгрии и других странах, позволило установить генетические профили пород, их дифференциацию, сходство и различия, однако до настоящего времени подобные исследования на российских породах индеек не проводились. В представляемой работе мы впервые оценили полиморфизм микросателлитных локусов и генетическую дифференциацию у пород индеек российской селекции. Исследования выполняли в условиях Северо-Кавказской зональной опытной станции по птицеводству на породах, разводимых в Селекционно-генетическом центре Северо-Кавказской зональной опытной станции по птицеводству (Ставропольский край). MLVA (multiple locus variable, number tandem repeats analysis) генотипирование проводили по 12 VNTR-локусам (MNT9-MNT20). Пробы крови отбирали у индеек семи пород: белой широкогрудой ( n = 12), бронзовой северокавказской ( n = 12), белой северокавказской ( n = 9), серебристой северокавказской ( n = 15), московской белой ( n = 12), черной тихорецкой ( n = 10), узбекской палевой ( n = 8). ДНК выделяли с применением гуанидинтиоционата. Качество образцов ДНК оценивали спектрофотометрически. Детекцию продуктов ПЦР-амплификации осуществляли по наличию специфических полос на электрофореграмме в агарозном геле. Для количественной оценки дискриминирующей способности метода генотипирования использовали индекс Хантера-Гастона (Hunter Gaston Discriminatory Index, HGDI). Кластерный анализ осуществляли методом невзвешенного попарного среднего (UPGMA), дендрограмму строили с помощью компьютерной программы START 2. Из 12 исследованных VNTR-локусов 3 оказались мономорфными (MNT11, MNT15, MNT17), в 6 было выявлено по 2 аллеля (MNT9, MNT10, MNT12, MNT14, MNT19 и MNT20). В локусе MNT16 обнаружили 3 и лишь в двух VNTR-локусах (MNT13 и MNT18) - по 4 аллеля. Из 26 выявленных аллелей 21 ранее не был описан и не аннотирован в специализированных международных базах данных, что указывает на генетическое своеобразие исследованных российских пород индеек. Филогенетический анализ с построением дендрограммы генетических дистанций позволил выделить два неравнозначных кластера - I и II. Первый кластер был образован частью генотипов белой широкогрудой и всеми генотипами узбекской палевой породы. Второй сформировали две крупные группы - C и D. Группа C объединяла генотипы пород бронзовая северокавказская, белая северокавказская и белая широкогрудая; группа D - генотипы пород черная тихорецкая, серебристая северокавказская и московская белая. Особенности генетической дифференциации пород индеек российской селекции обусловлены историей их происхождения и участием генофонда одних пород при создании других. Впервые выявленные аллельные варианты в локусах позволяют провести исследования по генетической дифференциации пород индеек российской и зарубежной селекции.
Породы индеек, микросателлиты, vntr-локусы, филогенетический анализ, генетическое разнообразие
Короткий адрес: https://sciup.org/142214163
IDR: 142214163 | УДК: 636.592:575.174:577.21 | DOI: 10.15389/agrobiology.2017.4.739rus
Текст научной статьи Исследование микросателлитных локусов в породах индеек российской селекции
В настоящее время мясо индеек (Meleagris gallopavo) занимает второе место в мире в общем объеме производстве мяса птицы. По оценке аналитического агентства Global Reach Consulting (GRC, Россия), за последние 10 лет этот сегмент вырос более чем в 8 раз. Положительная динамика наблюдается и в России: прирост производства мяса индеек в 2015 году составил 34,9 %, в основном благодаря достижениям современной генетики и использованию высокопроизводительных технологий. Повы- шенный интерес к этому направлению мирового птицеводства служит причиной жесткой конкуренции на рынке генетического материала, что, в свою очередь, определяет значимость генетического контроля его происхождения и консолидированности.
Важным и информативным методом изучения генетического биоразнообразия у различных видов животных и птицы служит анализ мик-росателлитных локусов (1-3). Известно, что микросателлиты — SSR (simple sequence repeat) или STR (short tandem repeat) — представляют собой тандемные повторы длиной от 2 до 4, иногда до 6 пар нуклеотидов в некодирующих областях генома. Приоритетность их использования в ДНК-диагностике обусловлена относительно равномерным распределением на хромосомах и широким разнообразием. Полиморфизм микроса-теллитных повторов настолько высок, что позволяет различать хромосомы при семейном анализе, прослеживая их передачу в поколениях, а также при идентификации принадлежности биологического образца к виду, породе, популяции (4).
Поскольку данные о генетической структуре популяций исключительно важны для рационального использования генетических ресурсов сельскохозяйственных животных и птицы, Продовольственная и сельскохозяйственная организация ООН — ФАО (The Food and Agricultural Organization of the United Nations — FAO) осуществила глобальный проект по оценке генетического разнообразия домашних животных (Global Project for the Measurement of Domestic Animal Genetic Diversity, MoDAD) (5, 6). С помощью 30 микросателлитных локусов было выполнено генотипирование от 6 до 50 пород одного вида. Примером успешной апробации проекта стали результаты по изучению генетического разнообразия микросател-литных маркеров более чем у 50 популяций кур (7, 8). Несмотря на то, что значительное число микросателлитных локусов у индеек определены и картированы (9-11), до настоящего времени не предложена унифицированная микросателлитная панель для M. gallopavo (12).
В ряде публикаций, посвященных проблеме генетического разнообразия у индеек, продемонстрирована возможность использовать для его оценки микросателлитную панель, рекомендованную ФАО для кур. Так, при описании генетического профиля пород Nero d’Italia, Brianzolo и Colli Euganei, разводимых в Турции, из 31 куриного маркера 22 оказались информативными. По ним для трех пород было выявлено 63 общих аллеля. При этом 10 маркеров исследовали с помощью мультиплексных панелей (Multiplex Master Mix 1 — ADL0268, ADL0278, LEI0094, MCW0216, MCW0248; Master Mix 2 — MCW0034, MCW0069, MCW0081, MCW0222, MCW0295), а 12 локусов (ADL0112, LEI0192, LEI0234, MCW0014, MCW0016, MCW0037, MCW0067, MCW0098, MCW0103, MCW0111, MCW0165, MCW0183) изучали по отдельности в одиночных ПЦР. По 9 маркерам (LEI0166, MCW0020, MCW0078, MCW0080, MCW0104, MCW0123, MCW0248, MCW0284 и MCW0330) ПЦР-продукты не обнаружены (13).
K. Reed с соавт. (14) выполнили наиболее масштабное исследование информативности куриных микросателлитных локусов для оценки генетических особенностей индейки. Они использовали 520 микросател-литных маркеров курицы и получили продукты амплификации с ДНК индейки только в 280 случаях (54 %). Для дальнейшего анализа отобрали 57 локусов, в 20 из которых был установлен невысокий аллельный полиморфизм — в среднем 1,4 аллеля на локус. Авторы пришли к выводу, что около 20 % микросателлитных маркеров курицы информативны при изучении генетического биоразнообразия в популяциях индеек (14). По-740
строены генетические профили двух пород индеек (BIG6 и BIG10) с использованием восьми микросателлитных маркеров кур ( Gallus gallus ) — MCW0111, MCW0067, LEI0104, MCW0123, MCW0081, MCW0069, MCW0104 и MCW0183. Установлен полиморфизм семи из восьми исследованных локусов, что позволило авторам сделать заключение об информативности микросателлитных локусов G. gallus для характеристики генофонда M. gallopavo (3).
Изучение вариабельных тандемных повторов микросателлитов (multiple locus variable, number tandem repeats analysis, MLVA) выполнено на породах индеек в нескольких странах. Так, E.K. Latch с соавт. исследовали популяции дикой восточной ( M. gallopavo silvestris ) и домашней индейки ( M. gallopavo ), разводимых в Турции, по 7 микросателлитным локусам. Число аллелей на локус варьировало от 5 до 15, при этом средняя гетерозиготность была высокой почти для всех локусов. Домашняя индейка характеризовалась значительно меньшим числом аллелей на локус при низкой общей гетерозиготностью по сравнению с восточными дикими индейками (15). K. Reed с соавт. (16) открыли у индеек восемь новых микросателлитных локусов.
D. Kamara с соавт. исследовали генетические взаимоотношения между коммерческими и некоммерческими породами индейки (Narra-gansett, Bourbon Red, Blue Slate, Spanish Black и Royal Palm) из генофонд-ной коллекции фермы колледжа Вирджиния, используя 10 локусов микросателлитов (RHT0009, RHT0011, RHT0024, RHT0095, RHT0131, RHT0216, RHT0294, TUM16, TUM20, ADL0023). Филогенетический анализ выявил, что породы Narra-gansett, Bourbon Red и Blue Slate имели большее генетическое сходство с коммерческими породами, чем Spanish Black и Royal Palm (17). Аналогичные результаты для этих пород были получены при использовании трех маркерных систем: случайно амплифицированных фрагментов ДНК (random amplified polymorphic DNA, RAPD), микросателлитов и SNP (single nucleotide polymorphism) (18).
В совместной работе ученые из США и Турции, выявляя микроса-телитные локусы, удобные и информативные для изучения генома индейки, на основании библиотеки данных выполнили дизайн праймеров для 164 участков геномной ДНК, содержащих микросателлиты. Авторы делают вывод, что 154 обнаруженных генетических маркера вполне приемлемы для проведения анализа, однако для разработки информативной панели требуются дальнейшие исследования (19).
S. Kusza с соавт. (20), изучая популяции индеек венгерской бронзовой и белой широкогрудой пород с использованием 15 микросателлит-ных локусов, установили, что первая популяция высокополиморфна (среднее число аллелей на локус 3,20). Полученные данные и генетический анализ по M. Nei позволили четко дифференцировать изученные популяции.
Для наиболее полного описания генетического разнообразия пород и линий индеек, разводимых в Турции, было проведено секвенирование их генома (21). Установлено, что все современные турецкие коммерческие линии имеют общее происхождение и выведены из диких популяций. При этом дикие индейки характеризуются более высокой гетерозиготностью по сравнению с коммерческими линиями. Авторы пришли к заключению, что у индеек геном гораздо менее разнообразен, чем у других видов сельскохозяйственных животных и птицы.
До настоящего времени изучение генетического профиля российских пород индеек, в том числе по локусам микросателлитов, не проводи-741
лось, что определило актуальность нашего исследования.
Нами впервые выполнено MLVA (multiple locus variable, number tandem repeats analysis) генотипирование пород индеек российской селекции по 12 микросателлитным (MNT9-MNT20) VNTR-локусам (Variable Number of Tandem Repeats). Установлено генетическое своеобразие исследованных пород: из 24 выявленных аллелей 18 ранее не были описаны и не аннотированы в специализированных международных базах данных. Филогенетический анализ с построением дендрограммы генетических дистанций выявил объединение в одном кластере части генотипов белой широкогрудой и всех генотипов узбекской палевой пород, в другом — генотипов бронзовой северокавказской, белой северокавказской, белой широкогрудой, черной тихорецкой, серебристой северокавказской и московской белой пород. Подученные данные свидетельствуют, что особенности генетической дифференциации пород индеек российской селекции обусловлены историей их происхождения и участием генофонда одних пород в создании других.
Цель настоящей работы заключалась в изучении полиморфизма и генетической дифференциации по микросателлитным локусам у пород индеек российской селекции.
Методика. Исследование выполняли на породах, разводимых в Селекционно-генетическом центре Северо-Кавказской зональной опытной станции по птицеводству (Ставропольский край). Для определения генетического разнообразия по 12 VNTR-локусам были отобраны пробы крови у индеек семи пород: белой широкогрудой ( n = 12), бронзовой северокавказской ( n = 12), белой северокавказской ( n = 9), серебристой северокавказской ( n = 15), московской белой ( n = 12), черной тихорецкой ( n = 10), узбекской палевой ( n = 8).
ДНК выделяли с применением гуанидинтиоционата в соответствии с протоколом к коммерческому набору АмплиПрайм ДНК-сорб-АМ («Ин-терЛабСервис», Россия). Чистоту образцов ДНК оценивали спектрофотометрически (A 260/280 ).
Для выполнения MLVA генотипирования были выбраны 12 VNTR-локусов (MNT9-MNT20) (22). ПЦР проводили в конечном объеме смеси 20 мкл, содержащей реагенты из расчета на одну реакцию: 1 мкл forvard primer (F) и 1 мкл reverse primer (R) («ФКУЗ СтавНИПЧИ Роспотребнадзора», Россия), 2 мкл раствора dNTPs, 4 мкл РНК-элюента, 10 мкл раствора ПЦР-смесь-2red («ИнтерЛабСервис», Россия) и 2 мкл пробы ДНК из образца крови. Реакцию осуществляли в следующем режиме: 15 мин при 95 ° С (начальная денатурация); 30 с при 95 ° С, 30 с при 58 ° С (для локусов MNT10, MNT11, MNT20 температура составляла 56 ° С), 30 с при 72 ° С (35 циклов); 5 мин при 72 ° С (завершающая элонгация).
Продукты ПЦР-амплификации детектировали по наличию специфических полос при электрофоретическом разделении в 1,5 % агарозном геле. Точный размер аллелей выявленных маркеров по всем VNTR-локусам для каждой исследуемой пробы определяли с помощью автоматизированной станции микрокапиллярного электрофореза Experion System («Bio-Rad Laboratories», США).
Для оценки дискриминирующей способности протокола генотипирования использовали индекс Хантера-Гастона (Hunter Gaston Discriminatory Index — HGDI) (23). Кластерный анализ осуществляли методом невзвешенного попарного среднего (UPGMA), для построения дендрограмм применяли программу START 2 (24).
Результаты. Селекционно-генетический центр Северо-Кавказской зональной опытной станции по птицеводству (Ставропольский край) — единственное в России предприятии, осуществляющее создание новых пород и кроссов и обеспечивающее сохранение генофонда индеек.
Последовательности праймеров, использованных для амплификации 12 MLVA-локусов, приведены в таблице 1.
1. Праймеры, использованные для амплификации 12 VNTR-локусов у пород индеек российской селекции (Северо-Кавказская зональная опытная станция по птицеводству, Ставропольский край)
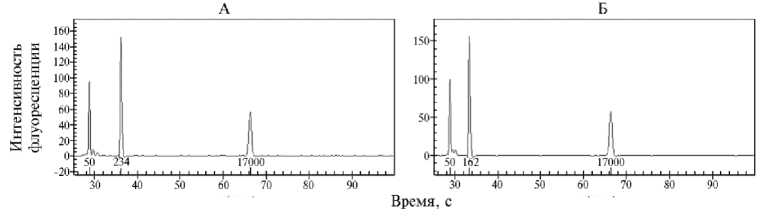
Рис. 1. Примеры определения размера аллелей VNTR-локусов, выявленных у пород индеек российской селекции, по результатам микрокапиллярного электрофореза продуктов ПЦР: А — ранее не описанный аллель 234 п.н. (локус MNT16; проба ¹ 140, черная тихорецкая порода), Б — ранее не описанный аллель 162 п.н. (локус MNT18; проба ¹ 107, московская белая порода); 50 и 1700 — пики, соответствующие стандартам 50 п.н. и 17000 п.н. (Experion System, «Bio-Rad Laboratories», США).
Проведенное исследование позволило выявить некоторые особенности вариабельных VNTR-локусов у изученных пород индеек российской селекции (табл. 2).
2. Вариабельность VNTR-локусов в исследуемой выборке у пород индеек российской селекции (Северо-Кавказская зональная опытная станция по птицеводству, Ставропольский край)
Из 12 исследованных локусов мономорфными оказались MNT11, MNT15 и MNT17. В 6 локусах (MNT9, MNT10, MNT12, MNT14, MNT19, MNT 20) было выявлено по два аллеля, в локусе MNT16 обнаружили 3 аллеля и лишь в двух локусах (MNT13 и MNT18) детектировали по 4 аллеля. Следовательно, среднее число аллелей на один локус равнялось 2. Полученные данные в определенной степени подтверждают выводы других ученых о том, что у индеек геном характеризуется меньшим полиморфиз-
|
VNTR-локус |
Последовательность праймера (5 ′→ 3 ′ ) |
|
|
прямой (F) |
обратный (R) |
|
|
MNT9 |
TGGGAGTGGAAAGGTGAAAG |
TTCTCCTCAGCTCAGCAACC |
|
MNT10 |
TTCCCAGTGCACTACCTGAAC |
TGAACAGTGATTCCACTGAAGC |
|
MNT11 |
TTTCTGACACAGGTACAAGGAAAC |
GCCCTCGAGTATTAGCCACTC |
|
MNT12 |
AGGTGTTTTTGGGCAGTCTC |
TGCAAGCACCATCTGCTAAG |
|
MNT13 |
TTAGGGGATGCTGAACTGTG |
GCGTAATTGGTGCTTTCTCC |
|
MNT14 |
AAACAGAACAACCTCAAGGACAG |
GAATTGGGTTTGCATTTGAG |
|
MNT15 |
TTGTTGCTGTTGTTTTTGTGG |
TTTCTGTGCCTAAGCTTAATGTG |
|
MNT16 |
TGTTTGCCTGCAATAAGCTG |
GCACCCTCCCACTGACTG |
|
MNT17 |
GGAGCACCCAGCTCAAAG |
GAGTAATACCAAGGAAAAGTGTGC |
|
MNT18 |
GCAGGCACAGAGAGCTACG |
CCAATGTTGAAGCAGGTGAG |
|
MNT19 |
GCAGGAGGCTCTGAGCTATG |
TTATACGGAAGGCGGTTGAG |
|
MNT20 |
TAACTGTCTGCCAGGTGGTG |
GATCTCGGGTGGTGATTGC |
мом и более консервативен по микросателлитным локусам, чем у других видаов сельскохозяйственных животных (21, 25).
145-51*11 117-ST-11 116-ST-11 115-ST-11 114-ST-11 108-ST-11 146-ST-11
(168,78,90,145,235,177.188,234,181,161,250,192)
(168,78,90,145,235,177,188,234,181,161,250,192)
(168.78.90,145,235.177.188.234,181,161,250,192)
(168,78,90,145,235,177.188.234,181,161,250,192)
(168,78,90,145,235,177,188.234,181,161,250,192)
(168,78.90,145,235.177.188,234,181,161,250,192)
(168,78,90,145,235,177,188,234,181,161,250,192)
107 - ST-10 (168,78,90,145,235,177,188,234,181,162,250,192) 106 - ST-10 (168,78.90,145,235,177,188,234,181.162,250,192) 120 - ST-10 (168,78,90,145,235,177,188,234,181,162,250,192) 119- ST-10 (168,78,90,145,235,177,188.234,181,162,250,192) 118- ST-10 (168,78.90,145,235.177.188,234.181.162,250,192) 104 - ST-9 (168,78,90,145,235,177,188,226,181,162,250,192) 103- ST-9 (168,78,90,145,235,177.188,226,181,162,250,192) 102 - ST-9 (168,78,90,145,235,177,188,226,181,162,250,192) 101 - ST-9 (168,78,90,145,235,177,188,226,181,162,250,192) 100 - ST-9 (168,78,90,145,235,177,188,226,181.162,250,192) 99 - 57-9(168,78,90,145,235,177,188,226,181,162,250,192) 98 - 57-9(168,78,90,145,235,177,188,226,181,162,250,192) 97 - ST-9 (168.78,90,145,235,177,188,226.181,162,250,192) 96 - ST-9 (168,78,90,145,235,177,188,226,181,162,250,192) 95 - ST-9 (168.78,90,145,235,177,188,226,181,162,250,192) 94 - 57-9(168,78,90,145,235,177,188,226.181,162,250,192) 93 - 57-9(168.78,90,145,235,177,188,226.181,162,250,192) 92 - 57-9(168,78,90,145,235,177,188,226,181,162,250,192) 91 - 57-9(168,78,90.145,235,177,188,226,181,162,250,192) 105 - 57-9 (168,78,90,145,235,177,188,226,181,162,250,192) 137 - 57-12 (164,78.90,145,235.177,188.219.181,161,224,195) 136 - S7-12 (164,78,90,145,235,177,188,219,181,161,224,195) 138 - 57-12 (164,78,90,145,235,177,188,219,181,161,224,195) 140 - S7-14 (164,78.90,145,235.177.188.234.181.161,224,195) 150 - S7-13 (168,78,90,145,235,177,188,219,181,161,224,195) 142 - 57-13 (168,78,90,145,235,177,188,219,181,161,224,195) 141 - S7-13 (168,78.90,145,235.177.188,219.181,161,224,195) 139 - S7-13 (168,78,90,145,235,177,188,219,181,161,224,195) 53 - 57-3 (168.67,90,121.185,177,188,234,181,162,224,195) 52 - 57-3(168,67,90.121.185,177,188,234.181,162,224,195) 54 - 57-3 (168,67,90,121,185,177,188,234,181,162,224,195) 51 - 57-4 (168,67,90,121,235,177,188,234,181,162,224,192) 56 - 57-6(168.67,90.145.183.177,188,234,181,162,224,192) 55 - 57-6(168.67,90.145.183.177,188,234.181.162,224,192) 50 - 57-6 (168.67,90,145,183.177,188,234,181.162,224,192) 49 - 57-6(168,67,90,145,183.177,188,234,181,162,224,192) 76 - 57-8 (168,67,90,145.235.181.188,226.181.162,224,195) 67 - 57-8 (168.67,90.145.235,181,188,226,181,162,224,195) 66 - 57-8(168,67,90,145,235,181,188,226,181,162,224,195) 65 * 57*8 (168.67,90,145,235,181,188,226.181.162,224,195) 64 - 57-8 (168.67,90,145.235.181,188,226,181,162.224,195) 63 - 57-8(168.67,90.145.235.181,188,226.181,162,224,195) 62 - S7-8 (168,67,90,145,235,181,188,226,181,162,224,195) 61 - 57-8 (168.67,90,145,235.181,188,226.181.162,224,195) 90 - 57-8(168.67,90.145,235,181,188,226.181,162,224,195) 89 - 37-8 (168,67,90,145,235,181,188,226,181,162,224,195) 88 - 57-8 (168,67,90,145,235,181,188,226,181,162,224,195) 77 - 57-8 (168.67,90.145.235,181.188,226.181.162,224,195) 85 - 57-7 (168,67,90,145,185,181,188,226,181,162,224,195) 84 - 57-7 (168.67,90,145.185,181,188,226,181,162,224,195) 83 - 57-7(168,67,90,145,185,181,188,226,181,162,224,195) 75 - 57-7 (168,67,90,145,185,181,188,226,181.162,224,195) 68 - 57-1 (164,67,90,121,187,181,188,234,181,159,224,195) 29 - 57-1 (164,67,90,121,187,181,188,234,181,159,224,195) 28 - 57-1 (164,67,90,121,187,181,188,234,181,159,224,195) 27 - 57-1 (164,67,90,121,187,181,188,234,181,159,224,195) 158 * 57-1 (164,67,90,121,187,181,188,234,181,159,224,195) 157 - S7-1 (164.67,90,121,187,181.188,234,181,159,224,195) 156 - S7-1 (164,67,90,121,187,181,188,234,181,159,224,195) 155 - S7-1 (164,67,90,121,187,181,188,234,181,159,224,195) 154 - 57-1 (164,67.90,121.187,181.188,234,181.159,224,195) 153 - S7-1 (164,67,90,121,187,181,188,234,181,159,224,195) 152 - S7-1 (164,67,90,121,187,181,188,234,181,159,224,195) 151 -57-1 (164,67,90,121,187,181,186,234,181,159,224,195) 111 -57-1 (164,67,90,121,187,181,188,234,181,159,224,195) 109 - S7-1 (164,67.90,121,187.181.188.234,181.159.224.195) 78 - 57-1 (164,67,90,121,187,181,188,234,181,159,224,195) 72 * 57-1 (164.67,90.121.187,181.188,234.181.159,224,195) 71 -S7-1 (164.67,90.121.187.181,188,234.181,159.224,195) 69 - 57-1 (164,67,90,121.187,181,188,234,181,159,224,195) 30 - S7-2 (164.67,90.121.187,181.188,226.181,158,224,195)
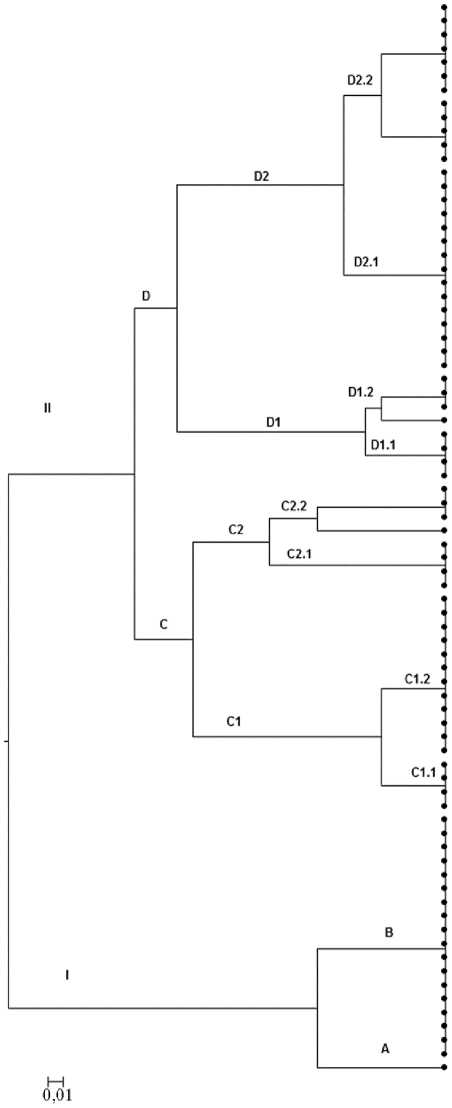
Рис. 2. Дендрограмма генетических дистанций между породами индеек российской селекции по результатам анализа 12 вариабельных VNTR-локусов методом MLVA (multiple locus variable, number tandem repeats analysis): I и II — кластеры; А-В — группы, C1, C2, D1, D2 — подкластеры, C1.1, C1.2, C2.1, C2.2, D1.1, D1.2, D2.1, D2.2 — ветви подкластеров (описание см. в тексте статьи) .
Номера проб по породам: белая широкогрудая — 27-30, 49-56 ( n = 12); бронзовая северокавказская — 61-69, 75-77 ( n = 12); белая северокавказская — 71, 72, 78, 83-85, 88 744
90 ( n = 9); серебристая северокавказская — 91-105 ( n = 15); московская белая — 106-111, 114-120 ( n = 12); черная тихорецкая — 137-142, 145, 146, 150 ( n = 10); узбекская палевая — 151-158 ( n = 8); St 1-14 — 14 генотипов, выявленные у исследованных пород индеек по 12 локусам микросателлитов (в скобках указаны номера аллелей, соответствующие генотипам St 114) (Северо-Кавказская зональная опытная станция по птицеводству, Ставропольский край).
В то же время полученные нами данные позволяют сделать заключение о генетическом своеобразии изученных российских пород индеек. Так, из 26 обнаруженных аллелей VNTR-локусов 21 не был ранее описан у других пород и не аннотирован в специализированных международных базах данных. Выявленные варианты аллелей позволили разделить особей исследованных пород на 14 типов по 12 вариабельным VNTR-локусам, на основе чего была построена дендрограмма генетических дистанций, отображающая степень филогенетического родства (рис. 2).
На дендрограмме выделились два неравнозначных кластера — I и II. Первый кластер образовала часть генотипов белой широкогрудой породы (ветвь А) и все генотипы породы узбекская палевая (ветвь В). Объединение этих пород в один кластер представляется вполне логичным, поскольку узбекская палевая выводилась посредством скрещивания местных узбекских бронзовых индеек с белыми широкогрудыми.
Второй кластер, в свою очередь, был сформирован двумя крупными группами C и D. В группу C вошли генотипы пород бронзовая северокавказская, белая северокавказская и белая широкогрудая. Близкое расположение пород бронзовая северокавказская и белая северокавказская в подкластере С1 (соответственно ветви С1.1 и С1.2) объясняется использованием генофонда одной породы при создании другой. Так, первая отечественная порода индеек — бронзовая северокавказская была выведена в 1950-1960-е годы посредством длительного использования при размножении местных индеек генофонда бронзовой и бронзовой широкогрудой пород. Вторая отечественная порода — белая северокавказская создавалась в 1970-1980-е годы при скрещивании бронзовых северокавказских индеек с самцами белой широкогрудой породы английского происхождения. Генотипы породы белая широкогрудая образовали собственный подкластер С2, ветви С2.1 и С2.2 располагались на очень близком расстоянии друг от друга, что подтверждает принадлежность к одной породе и свидетельствует о ее высокой генетической консолидированности.
Известно, что белая широкогрудая порода индеек — одна из старейших в мире. В Россию она была завезена из Великобритании в начале 1960-х годов. В 1980 году Северо-Кавказская зональная опытная станция по птицеводству получила из Голландии четыре линии кросса Hidon — А, В, С, D. На базе двух линий (В и D) были получены родительские формы, чистопородное разведение которых поддерживается до настоящего времени. Группа D (см. рис. 2) также состояла из двух подгрупп: подгруппа D1 была образована исключительно генотипами породы черная тихорецкая, которые разделились на ветви D1.1 и D1.2. В подгруппе D2 ветвь D2.1 оказалась образована только генотипами серебристой северокавказской породы, в состав ветви D 2.2 вошли генотипы породы московская белая.
Определенная генетическая обособленность этих пород объясняется историей их создания. Так, современная серебристая северокавказская порода, утвержденная в 2008 году, создавалась на основе генофонда палевой узбекской и белой широкогрудой пород. Для повышения показателей воспроизводства и улучшения мясных форм было проведено повторное вводное скрещивание помесей с самцами материнского типа белой широкогрудой породы (линия О4). В последующем совершенствование популя- ции осуществляли посредством разведения в себе. Порода черная тихорецкая, ранее известная как краснодарская черная, создавалась в 19501960 годы в Краснодарском крае в процессе длительной массовой селекции местных индеек с черным оперением. Белая московская порода создана в тот же период посредством сложного высокопроизводительного скрещивания белых местных индеек с белыми голландскими и белтсвилл-скими индейками. В последующих поколениях помесных самок улучшали скрещиванием с белой белтсвиллской породой и далее желательные генотипы разводили в себе.
Полученные результаты позволяют сделать заключение о том, что индейки одной породы, как правило, имеют одинаковый MLVA12-тип и относятся к одной подгруппе на дендрограмме.
Расчеты показали, что дискриминирующая способность выполненного нами анализа по 12 вариабельным VNTR-локусам (метод MLVA12) была удовлетворительной. Согласно общепринятой практике, метод ти-пирования считается приемлемым, если имеет индекс Хантера-Гастона ≥ 0,9. В нашем исследовании величина этого индекса (HGDI = 0,9) соответствовала допустимым значениям.
Таким образом, в результате анализа филогенетического дерева, построенного на основании генотипирования индеек отечественных пород на Северо-Кавказской зональной опытной станции по птицеводству по 12 вариабельным VNTR-локусам, сформированы группы (MLVA12-ти-пы) с разной степенью дискриминации. Индейки одной породы обычно имеют одинаковый MLVA12-тип и группируются вместе на дендрограмме. У изученных пород среднее число аллелей на один локус равнялось 2, что согласуется с преставлениями о том, что у индеек геном более консервативен по микросателлитным локусам. Дискриминирующая способность примененного метода MLVA12 была удовлетворительной (HGDI = 0,9). Аллели исследованных локусов, выявленные нами впервые (всего 21 аллельный вариант), позволяют продолжить изучение генетической дифференциации в породах индеек российской и зарубежной селекции. Для углубленного анализа генетических особенностей пород индеек целесообразно использовать комплексный подход с применением дополнительных методов генетического анализа, таких как SNP, MLST (multilocus sequence typing) и секвенирование по Сэнгеру.
Список литературы Исследование микросателлитных локусов в породах индеек российской селекции
- Putman A.I., Carbone I. Challenges in analysis and interpretation of microsatellite data for population genetic studies. Ecology Evolution, 2014, 4(22): 4399-4428 ( ) DOI: 10.1002/ece3.1305
- Новгородова И.П., Волкова В.В., Гладырь Е.А., Селионова М.И., Растоваров Е.И., Фисинин В.И., Зиновьева Н.А. Изучение информативности микросателлитов кур G. gallus для характеристики аллелофонда индеек M. gallopavo. Достижения науки и техники АПК, 2011, 10: 66-67.
- Фисинин В.И., Гладырь Е.А., Волкова В.В., Севастьянова А.А., Зиновьева Н.А. Анализ генетической структуры пород домашних кур с использованием микросателлитных маркеров. Проблемы биологии продуктивных животных, 2011, 1: 68-72.
- Jarne P., Lagoda P.J.L. Microsatellites, from molecules to populations and back. Trends in Ecology & Evolution, 1996, 11(10): 424-429 ( ) DOI: 10.1016/0169-5347(96)10049-5
- Weigend S., Romanov M.N. The world watch list for domestic animal diversity in the context of conservation and utilisation of poultry biodiversity. World's Poultry Science Journal, 2002, 58(4): 411-430 ( ) DOI: 10.1079/WPS20020031
- Groeneveld L.F., Lenstra J.A., Eding H., Toro M.A., Scherf B., Pilling D., Negrini R., Finlay E.K., Jianlin H., Groeneveld E., Weigend S. Genetic diversity in farm animals: a review. Animal Genetics, 2010, 41(1): 6-31 ( ) DOI: 10.1111/j.1365-2052.2010.02038.x
- Soller M., Weigend S., Romanov M.N., Dekkers J.C.M., Lamont S.J. Strategies to assess structural variation in the chicken genome and its associations with biodiversity and biological performance. Poultry Science, 2006, 85(12): 2061-2078 ( ) DOI: 10.1093/ps/85.12.2061
- Wilkinson S., Wiener P., Teverson D., Haley C.S., Hocking P.M. Characterization of the genetic diversity, structure and admixture of British chicken breeds. Animal Genetics, 2012, 43(5): 552-563 ( ) DOI: 10.1111/j.1365-2052.2011.02296.x
- Reed K.M., Roberts M.C., Murtaugh J., Beattie C.W., Alexander L.J. Eight new dinucleotide microsatellite loci in turkey (Meleagris gallopavo). Animal Genetics, 2000, 31(2): 140 ( ) DOI: 10.1046/j.1365-2052.2000.00571.x
- Burt D.W., Morrice D.R., Sewalem A., Smith J., Paton I.R., Smith E.J., Bentley J., Hocking P.M. Preliminary linkage map of the turkey (Meleagris gallopavo) based on microsatellite markers. Animal Genetics, 2003, 34(6): 399-409 ( ) DOI: 10.1046/j.1365-2052.2003.01033.x
- Chaves L.D., Knutson T.P., Krueth S.B., Reed K.M. Using the chicken genome sequence in the development and mapping of genetic markers in the turkey (Meleagris gallopavo). Animal Genetics, 2006, 37(2): 130-138 ( ) DOI: 10.1111/j.1365-2052.2005.01396.x
- Gholizadeh M., Mianji G.R. Use of microsatellite markers in poultry research. International Journal of Poultry Science, 2007, 6(2): 145-153 ( ) DOI: 10.3923/ijps.2007.145.153
- Colombo E., Strillacci G., Cozzi M.C., Madeddu M., Mangiagalli M.G., Mosca F. Feasibility study on the FAO chicken microsatellite panel to assess genetic variability in the turkey (Meleagris gallopavo). Italian Journal of Animal Science, 2014, 13(4): 887-890 ( ) DOI: 10.4081/ijas.2014.3334
- Reed K., Mendoza K.M., Beattie C.W. Comparative analysis of microsatellite loci in chicken and turkey. Animal Genetics, 2000, 43: 796-802 ( ) DOI: 10.1139/g00-045
- Latch E.K., Smith E.J., Rhodes O.E. Isolation and characterization of microsatellite loci in wild and domestic turkeys (Meleagris gallopavo). Molecular Ecology Notes, 2002, 2: 176-178 ( ) DOI: 10.1046/j.1471-8286.2002.00183.x
- Reed K., Roberts M.C., Murtaugh J., Beattie C.W., Alexander L.J. Eight new dinucleotide loci in turkey (Melagris gallopavo). Animal Genetics, 2000, 31: 140-157 ( ) DOI: 10.1046/j.1365-2052.2000.00571.x
- Kamara D., Gyenai K.B., Geng T., Hammade H. Microsatellite marker-based genetic analysis of relatedness between commercial and heritage turkeys (Meleagris gallopavo). Poultry Science, 2007, 86(1): 46-49 ( ) DOI: 10.1093/ps/86.1.46
- Smith E.J., Geng T., Long E., Pierson F.W., Sponenberg D.P., Larson C., Gogal R. Molecular analysis of the relatedness of five domesticated turkey strains. Biochemical Genetics, 2005, 43(1-2): 35-47 ( ) DOI: 10.1007/s10528-005-1065-5
- Knutson T.P., Chaves L.D., Hall M.K., Reed K.M. One hundred fifty-four genetic markers for the turkey (Meleagris gallopavo). Genome, 2004, 47(6):1015-28 ( ) DOI: 10.1139/g04-076
- Kusza S., Mihók S., Czeglédi L., Jávor A., Árnyasi M. Testing the breeding strategy of Hungarian Bronze turkey strains for maintaining genetic diversity with microsatellites. Archiv Tierzucht, 2011, 54(4): 419-429.
- Aslam M., Bastiaansen J., Elferink M., Megens J. Crooijmans R., Blomberg L., Fleischer R., Van Tassell C., Sonstegard T., Schroeder S., Groenen M., Long J. Whole genome SNP discovery and analysis of genetic diversity in Turkey (Meleagris gallopavo). BMC Genomics, 2012, 13: 391-404 ( ) DOI: 10.1186/1471-2164-13-391
- Reed K., Chaves L.D., Rowe J.A. Twelve new turkey microsatellite loci. Poultry Science, 2002, 81: 1789-1791 ( ) DOI: 10.1093/ps/81.12.1789
- Hunter P.R., Gaston M.A. Numerical index of the discriminatory ability of typing systems: an application of Simpson's index of diversity. Journal of Clinical Microbiology, 1988, 26(11): 2465-2466 (PubMed ID: 3069867).
- START 2. Режим доступа: http://pubmlst.org/software/analysis/start. Без даты.
- Guan X., Silva P., Gyenai K.B., Xu J., Geng T., Tu Z., Samuels D.C., Smith E.J. The mitochondrial genome sequence and molecular phylogeny of the turkey, Meleagris gallopavo. Animal Genetics, 2009, 40(2): 134-141 ( ) DOI: 10.1111/j.1365-2052.2008.01810.x


