ISSR-PCR маркеры и мобильные генетические элементы в геноме домашней лошади Equus caballus
Автор: Бардуков Н.В., Феофилов А.В., Глазко Т.Т., Глазко В.И.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Динамика популяций и изменчивость генома у лошадей
Статья в выпуске: 4 т.49, 2014 года.
Бесплатный доступ
Закономерности и причины различий в полиморфизме отдельных микросателлитов, как и видоспецифичность такой изменчивости до сих пор остаются недостаточно исследованными. Тест-системы, разработанные Международной ассоциацией по генетике животных (International Society of Animal Genetics) для генетической паспортизации сельскохозяйственных животных, оценки межпородных взаимосвязей, консолидированности пород, включают небольшое число маркеров. Поэтому для животных сельскохозяйственных видов более простым и эффективным методом полилокусного генотипирования служит использование фрагментов микросателлитных локусов в качестве ПЦР-праймеров. Нами предложен метод выявления в секвенированных последовательностях позиционирования инвертированных повторов участков микросателлитов. Были исследованы алтайская порода ( n = 96), рысистые породы ( n = 48), карачаевская порода ( n = 34) домашней лошади Equus caballus. Проанализированы нуклеотидные последовательности для фрагментов (ожидаемая длина/фактиче-ская длина) разной локализации: 1250-1350/1326 п.н. (17-я хромосома), 1250-1350/1302 п.н. (2-я хромосома), 1250-350/1296 п.н. (Х-хромосома), 1250-1350/1287 п.н. (20-я хромосома), 980/986 п.н. (23-я хромосома), 800-900/900 п.н. (24-я хромосома), 800-900/876 п.н. (Х-хро-мосома), 800-900/871 п.н. (18-я хромосома), 800-900/857 п.н. (3-я хромосома), 800-900/832 п.н. (6-я хромосома), 800-900/823 п.н. (19-я хромосома), 740/739 п.н. (16-я хромосома), 580/585 п.н. (1-я хромосома), 550/637 п.н. (Х-хромосома), 550/572 п.н. (10-я хромосома), 500/494 п.н. (1-я хромосома), 490/489 п.н. (28-я хромосома), 380/378 п.н. (11-я хромосома), 380/377 п.н. (21-я хромосома), 360 (370)/364 п.н. (5-я хромосома), 310/309 п.н. (5-я хромосома). Рассчитан индекс PIC (polymorphic information content). Выявленные в геноме домашней лошади фрагменты ДНК (99 участков), фланкированные инвертированным повтором (GA) 9C, содержали участки гомологии к мобильным генетическим элементам. При их сопоставлении с продуктами амплификации, полученными в полимеразной цепной реакции у представителей ряда пород лошадей с использованием в качестве праймера последовательности (GA) 9C, выделены близкие по размеру консервативные и полиморфные фрагменты ДНК. Оказалось, что фрагменты ДНК, полиморфные у разных животных, в большинстве случаев несут последовательности, гомологичные фрагментам ретротранспозонов (в частности, ERV III), тогда как консервативные гомологичны участкам ДНК транспозонов. В исследованных фрагментах геномной ДНК лошади, фланкированных инвертированным повтором (GA) 9C, наблюдается увеличенная плотность нуклеотидных последовательностей, потенциально предрасположенных к формированию G 4 квадруплексов, что, по-видимому, может быть ассоциировано с повышенной активностью рекомбинаций в этих участках. Полученные данные позволяют предполагать связь между полиморфизмом фрагментов ДНК, фланкированных инвертированными повторами микросателлитов, и мобильностью ретротранспозонов.
Анонимные днк-маркеры, днк-транс-позоны, ретротранспозоны, неканонические структуры днк
Короткий адрес: https://sciup.org/142133524
IDR: 142133524 | УДК: 636.1/.2:599.723:
Текст научной статьи ISSR-PCR маркеры и мобильные генетические элементы в геноме домашней лошади Equus caballus
Генотипирование по микросателлитным локусам получило широкое распространение в популяционной генетике, особенно при решении таких актуальных задач, как контроль генетической структуры сельскохозяйственных видов, выявление сорто- и породоспецифичных особенностей их генофондов (1). Международной ассоциацией по генетике животных (International Society of Animal Genetics — ISAG) разработаны тест-системы для разных сельскохозяйственных видов, рекомендованные при генетической паспортизации животных, оценке межпородных взаимосвязей и консолидированности пород . В то же время закономерности и причины различий в полиморфизме отдельных микросателлитов, видоспецифичность такой изменчивости до сих пор остаются недостаточно исследованными. Кроме того, предложенные тест-системы включают не более двух десятков маркеров. В этой связи именно для животных сельскохозяйственных видов более простым и эффективным методом полилокусного генотипирования признается использование фрагментов микросателлитных локусов в качестве ПЦР-праймеров, позволяющее за одну-две постановки реакции оценивать полиморфизм нескольких десятков фрагментов ДНК, фланкированных инвертированным повтором такого праймера (inter-simple sequence repeat PCR — ISSR-PCR маркеры) (2).
Следует отметить, что применение в качестве одного праймера в ПЦР консенсусного фрагмента высокоповторенных последовательных ДНК успешно используется для геномного сканирования и выявления полиморфизма различных геномных районов у человека (3). Обнаружена достаточно высокая частота инвертированных повторов таких последовательностей на небольших расстояниях, что позволяет в одной ПЦР с одним праймером выявлять полиморфные участки ДНК (4). В геноме домашней лошади около 36 % нуклеотидов входят в диспергированные повторы; микросателлиты занимают около 2 % генома (5). Тем не менее, с использованием ISSR-PCR маркеров можно получать полилокусные спектры фрагментов ДНК, полиморфизм которых достаточно надежно отличает одну породу лошадей от другой (6). Ранее в наших исследованиях число выявленных в ПЦР фрагментов ДНК, фланкированных инвертированным повтором (ФИП) микросателлита, и их полиморфизм зависели от нуклеотидной последовательности праймера (6). В полученных спектрах продуктов амплификации выделялись участки, консервативные и высокополиморфные у всех исследованных лошадей.
Для того чтобы оценить возможные причины описанных различий по консервативности и полиморфизму, мы сопоставили данные по экспериментально наблюдаемому полиморфизму локусов и первичной структуре участков близкой длины, фланкированных инвертированным повтором микросателлита, который использовался в качестве ПЦР-прай-мера, в секвенированных последовательностях генома лошади из GenBank .
Методика . Исследования выполнялись на геномной ДНК следующих пород домашней лошади Equus caballus : алтайская (48 образцов из хозяйства «Энчи» и 48 образцов из СПК «Чингиз», Алтайский край); группа лошадей рысистых пород, содержащихся в конезаводе «Поворот В.П.» (Московская обл.) (7 образцов от американских рысаков, 6 — от русских рысаков, 4 — от орловских рысаков, 31 — от помесных русских рысаков, улучшенных американскими рысаками); карачаевская порода (34 образца).
ДНК выделяли из образцов периферической крови с помощью набора ДНК-Экстран 1 («Синтол», Россия). Для исследования генетической структуры и полиморфизма внутри вышеуказанных пород лошадей применили полимеразную цепную реакцию (ПЦР) ISSR-PCR (inter-simple sequence repeat PCR), в которой в качестве праймера использовали нуклеотидную последовательность (GA) 9 C. ПЦР выполняли в амплификаторе Терцик («ДНК-технология», Россия) в следующем режиме: первичная денатурация (94 °С, 2 мин); денатурация (94 °С, 30 с), отжиг (55 °С, 30 с), элонгация (72 °С, 2 мин) — 35 циклов; финальная элонгация (72 °С, 10 мин). Для проведения ПЦР использовали наборы реагентов PCR Core («Биоком», Россия) и ПЦР-РВ (ПЦР в реальном времени) («Синтол», Россия). Продукты амплификации, окрашенные в растворе бромистого этидия, разделяли в 1,5 % агарозном геле с последующей визуализацией на трансиллюминаторе УВТ-1 («Биоком», Россия). Длины полученных фрагментов оценивали с помощью маркера молекулярных масс M 27 (100 bp + 1,5 Kb + 3 Kb, 12 фрагментов от 100 до 3000 п.н., «СибЭнзим», Россия).
Индекс PIC (polymorphic information content) вычисляли по форму- ле: PIC =_2f (1 - f), где f — частота одного из двух аллелей, рассчитанная как f = /R, где R — частота вариантов, у которых отсутствовал фрагмент
ДНК соответствующей длины (гомозиготы по «рецессивному» аллелю).
Результаты . Экспериментальные спектры ДНК-фрагментов
ФИП (GA)9C у исследованных пород лошадей. Напомним, что ISSR-маркеры характеризуются доминантным типом проявления. В спектрах ампликонов, которые мы получили с праймером (GA)9C в ISSR-PCR (табл. 1) на геномной ДНК лошадей разных пород, суммарно присутствовали 20 фрагментов (до 20 000 п.н.), при этом 11 из них ока- зались полиморфными хотя бы у одной из исследованных пород. При расчете индекса полиморфного информационного содержания локуса (PIC) каждый фрагмент ДНК рассматривали как отдельный локус. Наибольший полиморфизм отмечали у лошадей алтайской породы, наименьший — у карачаевской. Полиморфными оказались фрагменты ДНК 1700, 1600, 1580, 1100, 980, 880, 840, 740, 550, 500 и 360 п.н.; консервативными — 1880, 1400, 1300, 1200, 950, 900, 580, 490, з90 п.н. При этом полиморфизм каждого локуса в значительной степени варьировал в зависимости от породной принадлежности животных.
Расчетные спек
1. Значения ицдекса полиморфного информационного содержания локуса (PIC) у исследованных пород домашней лошади Equus cabal-lus по результатам ISSR-PCR
|
Длина локуса, п.н. |
PIC по породам |
|||
|
рысистые | алтайская [карачаевская| |
по всем |
|||
|
1880 |
0 |
0 |
0 |
0 |
|
1700 |
0 |
0 |
0 |
0,43 |
|
1600 |
0,49 |
0 |
0 |
0,38 |
|
1580 |
0,27 |
0,19 |
0 |
0,11 |
|
1400 |
0 |
0 |
0 |
0 |
|
1300 |
0 |
0 |
0 |
0 |
|
1200 |
0 |
0 |
0 |
0 |
|
1100 |
0,43 |
0,50 |
0 |
0,42 |
|
980 |
0,41 |
0,48 |
0 |
0,49 |
|
950 |
0 |
0 |
0 |
0 |
|
900 |
0 |
0 |
0 |
0 |
|
880 |
0 |
0,43 |
0 |
0,29 |
|
840 |
0,43 |
0,35 |
0 |
0,29 |
|
7401 |
- |
0,44 |
- |
- |
|
580 |
0 |
0 |
0 |
0 |
|
550 |
0 |
0,35 |
0,28 |
0,29 |
|
500 |
0,43 |
0,35 |
0,47 |
0,44 |
|
490 |
0 |
0 |
0 |
0 |
|
390 |
0 |
0 |
0 |
0 |
|
3601 (370) |
0,47 |
0,40 |
- |
- |
|
PIC ср. |
0,14 |
0,15 |
0,04 |
0,17 |
|
Примечание. Прочерки означают, |
что у карачаевской ло- |
|||
|
шади не учитывали фрагменты меньшей длины, чем 390 п.н.; 1 |
||||
|
(в верхнем индексе) — |
опубликованные данные (6). В |
качестве |
||
|
праймера использована последовательность (GA^C. |
||||
тры ДНК-фрагментов ФИП (GA)9C в секвенированных последовательностях генома домашней лошади (Equus caballus, GenBank). Для выявления внутренней структуры полученных в результате ISSR-PCR полиморфных и мономорфных фрагментов ДНК, фланкированных инвертированным повтором (GA^C, были разработаны алгоритмы на языке Python 2.7.3 :
import re f=open("chromosome1 .txt", "r") for line in f.readlines():
f.close(), где — файл из GenBank для хромосомы с удаленными в текстовом редакторе пробелами и переносами строк (в анализ включали последовательно все аутосомы и Х-хромосому лошади); AGAGA[A...]{0,4}GAGAGAC — прямая последовательность для праймера (GA)9C, где точками указывались нуклеотиды, которые могли быть неспаренными; [л...]{0,4} — символ, которым задается шаг длиной не более 4 нуклеотидов после жестко детерминированных 7 нуклеотидов с 3'-конца праймера; GTCTCTC[A...]{0,4}TCTCT — инвертированная последовательность праймера в той же цепи ДНК; {50,2000} — указание нук- леотидного промежутка между сайтами отжига праймера. Алгоритм поиска возможных амплифицируемых последовательностей ДНК предполагал, что не менее 7 нуклеотидов с 3'-конца праймера должны отжигаться точно и не менее 12 нуклеотидов — иметь комплементарность к праймеру (GA^C. Такое условие основано на экспериментальных исследованиях (7, 8) об источниках ошибок при отжиге праймеров в ПЦР.
Длина последовательности между предполагаемыми сайтами отжига праймеров по условиям поиска составляла не менее 50 и не более 2000 нуклеотидов. До начала работы с сиквенсами хромосом все строки необходимо было объединить в одну, что выполнялось при помощи текстового редактора или программы BioEdit, представленной в свободном доступе .
В качестве внутреннего контроля с использованием тех же подходов выделяли и рассматривали фрагменты секвенированных последовательностей ДНК, на флангах которых локализованы прямые повторы последовательности (GA^C.
Сравнение спектров экспериментальных и расчетных фрагментов ДНК ФИП (GA) g C в геноме домашней лошади. Электрофоретические спектры продуктов амплификации участков ДНК (ампликонов) были представлены зонами с различной интенсивностью окрашивания, что допускает наличие в них фрагментов ДНК с близкими длинами. Условно мы обозначили зоны как мажорные (наиболее крупные и интенсивно окрашенные) и минорные. Для того чтобы уточнить возможность присутствия в мажорных зонах нескольких фрагментов ДНК, были выполнены исследования, в которых на одной и той же геномной ДНК проводили ПЦР при разных температурах отжига. В спектрах ампликонов, полученных с праймером (GA) g C (рис., А), выделились две крупных зоны в размерном диапазоне примерно 800-900 п.н. и 12501350 п.н., которые при повышении специфичности ПЦР за счет увеличения температуры отжига до 62 °С превращались в кластеры, состоящие из групп ампликонов, и смещались вверх (см. рис., А).
А Б
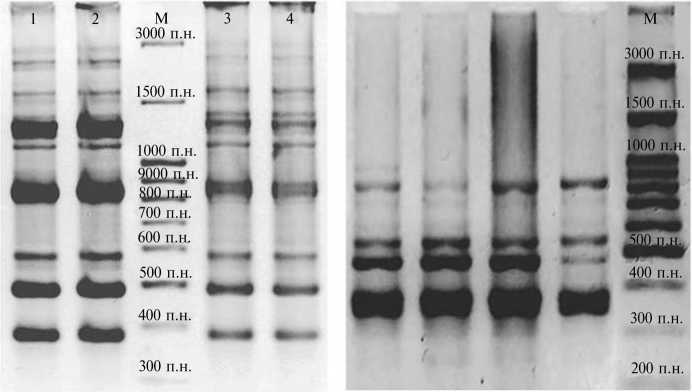
Пример спектра ампликонов ISSR-PCR с праймером (GA) 9 C для генома домашней лошади Equus caballus при разных температурах отжига (А) и использовании лиофилизированной полимеразы (Б, температура отжига 55 ° С) : 1, 2 — 55 ° С; 3, 4 — 62 ° С; М — маркер молекулярных масс M 27 («СибЭнзим», Россия); а, б — соответственно кластеры 1250-1350 п.н. и 800-900 п.н., в — бэнд ~ 590 п.н., г — вероятный гетерогенный бэнд ~ 559 п.н., д, е — мажорные бэнды соответственно ~ 490 п.н. и ~ 380 п.н.
Сопоставив данные о последовательностях ДНК, найденных в сек-венированных хромосомах лошади с использованием описанных выше алгоритмов поиска, с результатами, полученными при электрофоретическом разделении продуктов амплификации, мы выявили соответствие экспериментальных и расчетных длин для многих участков.
Так, для кластера 1250-1350 п.н. фрагментами ДНК с высококомплементарными предполагаемыми сайтами отжига праймеров были последовательности 1326 п.н. (локализована в 17-й хромосоме, имеет комплементарные сайты отжига праймеров по всей длине последних), 1302 п.н. (2-я хромосома), 1296 п.н. (Х-хромосома), 1287 п.н. (20-я хромосома). Разделение зон на кластеры при увеличении температуры отжига праймера соответствовало повышению точности отжига в найденных фрагментах ДНК (см. рис., табл. 2).
2. Фланкированные инвертированным повтором (GA^C нуклеотидные последовательности в секвенированном геноме домашней лошади Equus cabaUus , соответствующие размеру ампликонов в ISSR-PCR, и их предрасположенность к формированию G 4 квадруплексов
Длина фрагмента ДНК 1250-1350/1326 п.н. (17-я хромосома, PIC « - ») GGAAGAGAGAGAGAGAGACAGACCGTGGTCATTTGATCCCTGAATGTAGGTTTGCTGAG ACCAGCACTCAGTCTGACTCCTCAGTTATGTGAGCCAGCATATTACTTTTCTTCATAAA GAGGTTTGAGTGGGTTTCTGACACTGGCACTGAGAGAGTCCTGAGTAACTCAAATATGG AAATAAATGGAAATTATACCAAATGAAGACATATGTAAAAAGGAGAACTTCCTCTGATG TGTGCAACAGTGAAAAAGCTCATCAAATCTGAAGCAGCTCCGAGACAGCTGAAGTCGAC AGATCCTAGGGTCTTTACCATAATCATACTAATTTTGTATATGTGATGCTTTAAAAATA TCACACATATTCTTTACAAGTATAATTTTTTAATTTATTTTTTCTTCTCCCTTTTAAAG AAGGAGATTCTAGAATACAAATGGAGCTCAAGGAATATATCATCCTTTCAGCATGCCTT AAAGTTCTGTATCAAACAATGAATTATTGTATGTGAAAGGGGTTTATGTTTCTGAAAAT TATTATAATAGGGATTATTTAAAATATGATTTTGAAGCATTAACATATTTACTTATTTG CACCCTTTTATATATTAGTTTTTATATTTAAAAGAAATGTAGCCACATTTCTTCAGTAA TATTGAACAGTTGAAAAGTTTACCATCATCCTGGACTTTTCCTCATTATTGGGAAGTAG AGGAATTTTACTGTAATATATTTCAATAATGTTTATCTAGTGTTAGCTTTTGAGGTTCA AAGTTTTAACCCCAGAGAAGATCTTTTTTATGTACATGGTACTTGAAAATGCAATCATG ACTGAAATGAGTTAATCTAAAACTGTGAAACAAGAATTAATCACTTATTTGTCAAGATC AAGACATGATGTGTCAATTTATTTAGCACACAGGAGGGCCATCCCTATACATACTAAAA CATATTTGGAGGATTGTTAGAAATCAACTATATTTGATGGAAACATTTCTGCAAAATTA GCTAAATATAATACTGAATAGTTTGAGCTTCTATTTGCTTTTAAAATAGTAAATCTCTT TTGGTTGACCACAGATATCAATTATAATTATCAGTTACTGTGTAATAGGTTGATGCTCT TTATTGCTTTTTATGCATGTAAGTTCACTTGTCTCAATTTTAAATGATTTCAGACTGGC GTGTTTTTGAAGTGTTTACTTTCATAGTTTCTATCTCCTTCTTTACTGTTGAAATCCCA GCTGTCACTTCTATTTCTGTCAATATGCTAGCCTTCAGCAATCTCTCAATTCCTTCTTG TTCTTCTTGGTCTCTCTCTCTCTTTCTC
Длина фрагмента ДНК 1250-1350/1302 п.н. (2-я хромо сома, PIC « - ») gaaagagagagagagagacaggttctttgtgcatttcgcatcctgttag T gg Gtgt ggG| I GG TGCAT GG T TTATGTTAGTTACATGGTTATGAGGGTGACAACCTGTGTCCCTGTCTCA TACCGTCTGGGGATATCATGTGCCCGTCTCACGATCTGGGCGAAGTGACAGGTTATATG CTTAGCTCATCCTGTTAGGAGGGTGGTCACTGACTTGTGTGCTTGTCTCTGGCTGTTGG GTATGTGTGAGGTGTCACCATGTGCCCGTCTGATGTTGTGAGGAGTGACATGTGTGATT atctcgtgctgttg Gg a gg ttgtcagtaactt gg g gG tt gtctctcaccctaaagagac aacatgttg cccagt gg tgtg gg gctgc gg ttggCa GG gccttcccacgcagaccacag ggccccaaa Gg gct GG ccaga gg aagctct gG gggccctgagtacag Gg acagatgt ggI I gct gg agcga GG gtctcagtccctcatgacCtaccttctggcccaggtgccaacccttg AGACTGTTCCCCAGGAGGAAGGACCCCGTGTATCTTCAC ACAGTGT GG AAAGG GG CCCCl | t gg ctctg gG gca GG tgtgact gggg a gg T agagcacca Gg cg gg CtTcacat gg gg gG CTGTGTCCCCGCAGGCCTGGGGGGCCCTATTCCCTGCCTCTTCTGCCCCTGAAACTGTT aaactaattaaggctgtggagccgcaggattaatcg Gg gcagctc gg cgcgggcc gg ga I gctca gG tagtcgattagtgtgccgcggaggatattggatttctgaaccgcaagtcagc actattcgggcctgttatctctccga Gg cttga gg gtca gg aggc ggI gagcctccgggc GCACTGGCCGCCCCCGCCCGGCCAGCTCTGGAGCGTCCACTCCCCCTGCCCACACACCG CCAGCCTGTCTGCCTGGCATCCCTACCCACTCCCCCCTGCCCTCCTGGCCCCACCGCTG CCCACACGGCACCCTCACCCAGCCCTTCTCCCTGGCTCACCCTGGCCCTACAGTCATGT TCTCTCTCCTTCTTGTTCCCTCCTGGCATGACTCCCACTCTGTGCCTCCCTGACACCTG CTCCTCAGCTTCTCACCAGCCTCTCTCCACTTTGGTCTCCATCTCCCTGTCACTGTCTC TGTCCCCCAGTCTCTGTTAGCCTCTCCTATCACCACTCTCTGTTTTTGTGTTTGTGTTT GTGAGTGCCTGTCTCTTACAGTTTCTGTCTGTCTCCATCTCTCTGTCTCTCTCCCTCTG TCTG
Длина фрагмента ДНК 1250-1350/1296 п.н. (Х-хромосома, PIC « - ») GGATGAGAGAGAGAGAGACATGAATGACAGAGTAAGAGAGACAGATGATGAATAAGTAG ATGACAGAGAGAGATAGATAATAGCTTATTTACTAATTTATTTAGTCATTTACTGTGGC AGACTCCATACTAGATGCTGAGGGCGCAGAGGTGACTAAGATAAGGGTCCCTGTTTTCA acaagcttgttacatagtaggataaatcagctgtctacctccta | gg agcagtagt gg ac| AGACTCA GG CT GG A TTCCATCCCAGCTAGGAATGCCCCTCACCCCAGCGGAAACACAGA aaatcccagcagtgca a gg ttctaga gg gctaa gg cctaaaaa gG tag tcctgaagaga acggcgttgctgggaaaaacctgg Gg tagaat gg taaca gg cca gG cacagacaa Gg agI
Продолжение таблицы 2 G tgtaaga gg aagaaca ggI ggaagctgcttgtcaaaggtcaggaaccagagccacaggt cagcagcagtggctaagattta T gg gaa gg aaagcca gg tga gG cccccacttctaacc , acattattctgttatggatttcagagggcccagcag Gg acgcTgaa gg catgagaaagcI | CTT GGGGI gTGTGTAACTGCTCCAGAGTCTCACACACAAAGCCATGATGTTAGAAATATC TCCAGCTGCTGTTACCCGGTAGTCTTCAACAACTATTTATAAAACCTCTCGCAGAAGTA CTATGTACTACTACGCACTGTGCCATGTGTATCTACTATATACTATGCTTCTAGGCACT GAAGATACAGTACAGAACAGGGTATGTCTCTTATTTTGGGCCCTTCTTCTACATCCAGG ATTCCTGAAGTTCTCCTGAGCCCAGGGTTTAAAATGTGAGCAGTCAGTGTTGATACCAC CTTCTCCCCAACTGTCCAATCCTTCACTGAGTTCTGCTGAGTTTTCTTTTATGATATGT CTCTGGCTTTGCCTCTCTCTCTCCATTCTCAGTGAAACTATTTCACATAATTGCAGTTC TCCTCCACAGAATCCATCACCAACACTTCCTATTCTAGTCCAAGCCCTTAACATTCTAT GTCCAAAATACTGCATCAGTATGCCAGCCAGTGTCCCCTTGTTCCACCTCCAGTCACTG CTTTTAGACCATATCATGTCAAACACCAATTGAATTGTGGCACTCTCCTATTCAAGAAT ATTCACTGGCTCCCTATTTCTTACCCTGTCCTGCATCAAATGTAAGCTGGATTAGACCT TCAAAGCCCTCTATAAGCTATCTTTATCCGCCAGCCCAGTCTCTCACATCTCTGCTC
Длина фрагмента ДНК 1250-1350/1287 п.н. (20-я хромосома, PIC « - »») GGAGAGAGGAGAGAGAGACACAGAGAGAGAAAGACTGAGAATATTGCAGGTATTCCTAG CTGCCTAA ATTTTCTGCTCAAAAATATA GG AGAACCCGCAGCCC GG T GG AA GG AGCCTA tttaagtcc Gg a gg aa gg ttgaactttaaCagaaat gG cagtcaggccagtcgaacggg TTCCTGAAACATCCAAAAAGGTTTTCTGTTGCCCCTGGCTTTGCCTGGAAAGGATAAGT TTAGTGTACTTGAAGATTGATTGCATGTATTGTAAATTGAGGTAAAAATCAGACTGTAT gagcactaaagag Gg aaa gg c gg cacac gg A tgaaaaaccccggctcgcctctgtcctt TAATAAATAAGCATAATCTCCGATATTCAGTGAAAGTCCGCAGACGTCACTGTAAACAA CCGGGCCCACCGGGTTACTTTTCTCTCTCCTCCAAATGATGAGGGAACGCCTTTTTATT TTGTTCTTAATCTGCACACAAAGCACCTAGGGCGATCGACTCTTCCCATTAGGAAAGTC CCTCTCCAGGAACAAGGACACCTACGTTCTCTACCAGCCCCCAGCGATGCTCTCCCTCC CTGCCTTGTCCCGGGGAGCAGGGGCTGCTTTACGCGTGTCCCCGGCTCAGTTAATTGAA GTTCTGCCTCTTTCCTGCTAAACTTTTTCCCCAGTCTGCCTTTGCCAGCCCCCTGGTCT GATTACATCTAGTAATTCTCCACTGAATGAACGTCATTTACAATAGTAGGGGAGCTCTG CGCTCGCTGCTGCTTCACGCTCCATTTGTCCTCCATTCTCCCTTTAAAGTACTCGTGTA atatt aatagtcatgaatatcaattattatga ggcggtgta Gg caagc gg aggga gg aA I gg gG cgcctgagcagtctgagcccagcttc Gg gt gg aaGa gg actgcgtct gg A gcccc GAAGAACGGCAGAAGAAAAAAGGCCAAAAAGACAGCAAGAGACGGAGAAACAGCTCTGT ctgtgtgtgacaggcatcgccaatcttgttt Ggg a ggg g ggg g ggG gtaaatttttttt TTTGATGGCTTGTCCTGGAAACACCTCAAGCTCGGCTCCTGTGTCCAGCTCGTTGCTCA GCTGGACTCCTTGATTGTTTTTTTTTAATCATTGTTTGATTTTGAGCAGTAACCAGGCT TTTTTTTCCAGATGTTAGTCCACACCTATTCATCCATGGTGAGTTTGGATTCACGTTGT ACCAGAATTGCATGCTCCCTCCTCTCACTGTCTCTCTGTCTCTCTGTC
Длина фрагмента ДНК 980/986 п.н. (23-я хромосома, PIC = 0,49) TGGGGAGAGAGAGAGAGACAGTGGCTTCTTTCATCTGATGGGTATCTGCTCTAATAGCA GTATCCAGTGAATACCTAGCTTCGATGGTTCGTTTCACCTGCCTCTCTCTGAGTGGGTA AAAGGGTATCACAGAAAAGAAGTATACATACCTTATATATCTCATAAGTTTGGTTATAG CACAGAATGTAAATGCTATCATGAAACTCACCCAGCTTGTATTTGGTGTATTAGAAAAC TATCTCTCCAGAGGAGAAAAACAAACAAAAACATTCTGCGCTGAAATATGGTAGAAGAC acttggataagcaaaggtaaaggctttctttaccctgggacttc Tcagagt gg actt ggI I GG CTAAAACCCA GG| TCTCTTGCTTCAGAATAATTGCCTTTCCCCCCCTCACAATGCTGC TCTGTTTAGTTCATAGGGCCCAAGGCCCTCAGTTTGAGGCCCAAACCAGCGGCTTTGAG AAATTATGCCCCCTCATCCTGATCATCACAAATTCAAGGTTGCTGACGA GG GAAGAA GG Agaagcca gggg A acaagaatagagt I gg aaataca gggg ttaa ggI gtatttcctctgga GTCAGAAGGTACTGAATGTTTATCCCCATCTGACACTAGCTTTGTGAACTTGGATCAAG AAATTTGTTAAGGTTCAGCTTCCTCATCTTTAATACAGAGTTGTTTAAAGAACAAAATA TGAGAGTTCTTTTTCAATGTTACTGTTACTATTTTTTATTAATTTTTGTCGAGTCTAAC AACCCATCCCTTTTTCAATGGGGTTATCAGTTTCATCTCCAATTATGAGTAGCAGCTCT TTCATTGGTTTCCTTAAAATGGTATCAAGGAAAAAGAAATATGCCTTAGAAGAAATTGA GAGGCTTTATATCTAAATGCACTGTATTAAAACAGTCTCAAAGTAATTAACGTTATTTT AATTAAAGTATGCAAATCTCTCTGTCTCTCTGTCTCTAACAC
Длина фрагмента ДНК 800-900/900 п.н. (24-я хромосома, PIC = 0) TAGAAAGAGAGAGAGAGACCACCAGGGAATTGTGAGGAAAAGAAAAATGAGGGAAATGT CTCGATTTAATAGGAATTTGGGAGTGTTGAGAGCATTTTAAGAAAAATGCTTGCTTTTT CACTAGATGAGTCATTTTGTTCAAAGGGAGAAGAAACTTACTACTTTCTCTGAAGTGTA ATAGAACTGCAGACCTAGCTCCACAACTTTTTTCTCTTTTACATAATCTTGTCCTTATT CAAGAGCTAGTGAGTGAGGGTTTTCCCATGAGAACTGGTCATGTACAGAATGTTAATGC AGAGCTGGGTCAGGCTACGGTGTTCAACATATACAAGGTGAGAAAGCTCTGCCTCTGGT TAGATTAAGGTATGTGGATTGTCCCGTGCATTCTGTCCATGTGTCTGGGAACCTCATGT AATCATGAGAACTAAGGAATCCACCGATTCCTTAGACACC A| gG TTTCTAA GG GCCTA GG| | CATTGTGTCT GG| TTTTGTTTTTATCTTCCCAAACATGGGTTTTTTCCAGCTTCACTTAA TATGTCTAGCAGATAAAGCTATTTCAGTTAGGCACAGTTCCACTGCTCTTAGAAACATT GAGGATGAATTTTCCTTGTTTGTACCTGCTTCTTGCCAGAGACTAAAAACTGATAATTT GGCTGCTGGCAAGATTCCACTGCAACAGCTAGTAGAGTGTTCTGGAACGATGCCAACAG CAGACAGATTGCATTAAATACTTAAACTACTATTTTAGATCAGTTCTGCAGTT | GGGG AC| | CTGAAATCAGC GG T GG A .TCCACTTTCTACTTTCCAGCTTACTTTGCTCTGGGTTTTTTA AATGTCTTCTGTGATTTCCTCCAAATGGTTATCTCAAAGTGGCTTCTCTCTCTCTGTCT CTCTCTCTAACATGC
Длина фрагмента ДНК 800-900/876 п.н. (Х-хромосома, PIC « - ») TTTATAGAGAGAGAGAGACAGAAAAATTCTGTAACTGTGTAACTGTTCTTATATTTAAT CCATGTGACACATTTAATATTGATTTTCTCTGTCCTAGGATCTATGTGATGACTTATTT GAAATAGCCAAGTGGATTTTAGGGTTATCACTAAAAACACACCATTGATTGGAGCAATG CTGTCCAATTGAACTTTCCCAATGAGGGGAATGTTATATATCTGTGCTGGAGTAGCTAC TAGCCAGATAGAGTTTTTGAGCAAATGACATGAAGCTAGTGTAGCCGAGGAACTGAATT
Продолжение таблицы 2 TTTAATTTTATTTAATTTTACTTAGCTTATATTTAAATAAATGCATTTGGCTAGTGGTC ACTCTGTTGGGCAGAGCAGCTCTGTCCAAAGATAAATTTTGAGAAAAAAATTGTTAAAG AAGCCTAGTGTTTGTCCTTATTTTTAAATAAACTATCCTCTAAGCTCTCAGATTCATCA CCTTCCATTAATTAAACAGAATACTATGAAATTCTAAAGGTTCTCAGAAGTGACCATTT gatttgtgagacaag Ggg taaagt gg aaaaca gg tgattg gg a gtctggattctcccctg CTTTCCCATCTCTCTACCTTCAACAAAACAGCATATTTGTATGTGTTTAGTAAATTAAT TTTTCACTTAACTCTTTACTCAAATAAAGATTTTATTCCACTTAAAATCTTTTAGAAAC CACTACCTTGAACAACAGATTTTTATCTATATTCAGGAGCAACATGCAATATCTACCCA GTATGTGCCTGTGCATATTTATAGGAAGGGAAAGTCACTATCCATGTAGAATGATATGA GAGGCATAAGAAGGTCTCTCCACCTCCAAGTGTCTCTCTCTCTCACTCAC
Длина фрагмента ДНК 800-900/871 п.н. (18-я хромосома, PIC « - ») AAGACAGAGAGAGAGAGACA GAGAGACAGAGAGAGAGAGAGA GAGGGAGAGAGACTGAC ttaaaagaggcagcctgaact Gg atctt gggg aagcagctc ggJ aatatattattgtact tgtttttttgtaatactaagccaagccaatagatcaa A gg cagcacaaa gg gaaaa ggcI a GG Ataacgtaacgaacagtttctgactaaaattcctctatcactcccccatatggctc GACACCTCCACCACAACAAGCAAAAGACACACACACACACACACACACACACGCACACA CACGCACAAAACCTAGAGAGAGACAGAGAGACACAGAGAGAGAAGCATCACTTGTCTCT AATCAAAATTCTCTCGTCTTTGTGTTTTCCTCCCTCCAGCACCCCACACTGCGCCCCCA GCCGCCCCCTCCCCGCGCCGGGCAGCTCCG G GG CA GG CC GG G Gti CGCGCGCGATTGTCT tctctggaagaaagttgctccaagtgggcccgggcgctccccgcgagccgagggct Gg G I cttgc gg gaggcc gg aggccg gG ctggcgccgcgtccccgc ggccagctcgggagcgtc aacgcggccccgcgccg Gg tgatcg gg ctcagagcga gggg A acgagaagtttcctttc CCAGTCTGAGCTGGCGCTAGTTCGCTTGTTTGTTTTGGGGGGTTGGTTTTTCTTTTTAA ACTCAGTATCCTCCGTCAGCTCCCCCTCGGCACCCTCTCTTGTCCCCAGCTCTCTCCCC TCCCCCAGGCGCCTTCTGCGCTCTTGCCAATCACTTTTCTCTTTTATTCCCACGATTTT gtttggg Gg taataccta gg g gg tc G gtctctctttctctctctc
Длина фрагмента ДНК 800-900/857 п.н. (3-я хромосома, PIC « - ») CTGTTAAGAGAGGAGAGACAATTTGATGTGCAGCTTCTGACTGTCTCAGGCTACTGATA AGTACAGTTGATGGCAGCATTAGACTCTGTTTTAGTTATCTATTGCCCAACACTTTAAA AAATGATCTTTTTCCTCATGATTCTGTG l GG TTGACTA GG CTGAGTT GG GT GGl TTTTTCG GCCCCTTATGGTGTCAGCTAGGGTTACTCAGTTGGTTGCATTCAGTTAGCATCTTGGTT GGCTAGAAGTTCCAACAAAGCTTCACACAAATGCCTCACCCTTTGCTGGTTCTCCACGT atcctttttacgtggctagcttg Gg cctcctttcagtat gg t gg cttca g"G ctattcaa ACTTCTTACCCTGCCACTGGCTTCAAGGGGGAAAACAGCAGCTGCTAAGCCTCTTAAAG CCTAGAACTGCATCTGGCACAGTGTCACTTCCACCTAAATTATGAGAACAACCCAAATT CAAGGGAAGGGAGGTAGACGCCACTTCTCTATGAGGAGAGTGGCTTGCTGATAGAGTGA GGGAAAGAATTGATGGCAGCCATCTTTGGAGAATCACCACCACAGCCTCATTACTCACT TTACATTAGCACTATTTAATTTTATGTTAGATTTTCTTATGTATCTTCAGCTCCCTTTA TAAGAACAATCTTACATTCACTTACCAGTTCCAGCCAGTGTTTTCTGGTCCTTTAGGAA AAGTGGTCCTAATTATTCTTAATAGTTGGTACTATATAGTTAGGTCTCACTTATGTGCT CAGTACTGAGTTTAGTTCTTTATGTATATGAGCAAGTCCTTACAATAACCCTGTGAGGG AGATATTATTATGTCTCTCTCTCTATTTTTT
Длина фрагмента ДНК 800-900/832 п.н. (6-я хромосома, PIC « - ») GGTGAAGAGGAAGAGAGACAGTGGGCTGAGATCCTACGCCTGAAACTTATCTGGACAGA AAGGATGCCTTTAATCAGGCCTGACTAACTGTCTGCACCCCCACATACACACCTTCACA caaattctttgga Gg gcaagg gg ggaaga gg tgtaaa gG ccaacgcagacggcagtttc ATCCTAGGATAAGAAGAGCCATCACAACATTCAAGAAATGTTAGCATATTTTCTTTTCC TGGAAGTGAGGACCAGGGCACCAAAACAGTGACAGGCACATTGTGCCTCGAACTGCTGG AGGAGGCAGTGCATTTTAGACAAGATGTTTAAGCTCCCTGCTTCCATGTTTTCATCTGC AAATCAGCAGTTACGACAGTAACCACGCCATAGGGTTATGATGAATTTTAATGGGATAA TCCAAGTCAAGCACTTAGCACGTGCCTGGCACAAAGGTGACGCTCAATAAAAGTGAGCT CTTGTCATTCTTTGGAGCACCCCGTACATTCTGTCTACCCCCTGCTCACCCTGACCCAA GGCGTCTGGTTTAATTTAGTGTCCAAAACCAGCCTAGGCAGATGACCTGGAAGCCTCAA agtctgaacacctggggcttac a gggg tgact gg a gG accacccggatctcctcacctg aggatccatctttgtga gcgcccacccagctagccT gggg ttctgtccg gg tcctcaag| T gG gagacagctatgc T gg agtga gg ctgaccct gG acctg gg G ttaggattttcagga TTGGAACCCTAACTCTGCCATTTACCAACTTGGTTACCTTGAGCGAGTCTCTCTCTCTC TAGCAT
Длина фрагмента ДНК 800-900/823 п.н. (19-я хромосома, PIC « - ») tagagagagagagagagacagatg Ggg gga ggg Tgt ggg gga ggG cgagattcattgat tgattgcaaggaattgtaatgcaattgt gagagctgtctgggcatgtcccaaatca Gg C | catca gg gag gg cagttt gg A aactct T gg tca gg agcagatgca gg agtccaca gg A a ATTTCTTCTTCCTCAGGGAAACCTCAGTTCTGTTCTGAAGGCCTTCAGCTGAATGGTTC AGGCTCACTGAGATTATCCAAGATAGTCTCTTATACAGCCAACTGATCCCATCACTGAA ATACCTTCATAGCAACACCTGGAGCACAACAACCTAGAGTGCTGTTCGATTGCATAATA GAGTACCATAGCTTAGCCAAGATGACACAAAACTAACCATCACAATAAGTACAAGGGAA ggggcaagacgctactaca gtattcctT gg acta gg tgaat gg g gG caa taatattggg tgtattgttcggtagtggcc | gg ctgact gg tccctgttt gg gaggc gg a gtgtgcacaa GGTTCGCATGCTGTTGTGTCACAGCCCAGGACCCTTGGTGTACTTTTTTCTATCCCAGC CCTAATATAACCTTCTGTCTTCCAGTGTATAGCATGAAGCTCTAGCTTCATTCTTCCAA TTCAATCCCGTGTTCTCTGAAGAAAAAATGCCCAAAGCAATTTTCACCCCATCACACAC TAGTAATCATTTTATTTTTCTGATTATGTTTCTTTCTCTCTTTCAGCCCTTTGGTAAAT TACGCAGGGACCCTTTATTCTCATTCTCTCTTTTTCTGTCTCTCTGTGTCTCTATG
Длина фрагмента ДНК 740 1 /739 п.н. (16-я хромосома, PIC = 0,29) GACAGAGACAGGGAGAGACAGACAGACAGACAGAGACAGAGAGAGAGAGAGAGAAAGTG AGCCAGACGAAGCTGTATCCCTTTTCATGAGCTAGCATCAGAAAATAAGTCACAAAGAC TAAGACTAGCCCTTATTCAATAGTGAAGGCTTAGATGCGACCATTTGATGAGAATGTCA AGTAATTTTCAGACATGTTTTAAAACCATCACAGTTGTATATGTTCTTCCAGGATGTTG ATCAACTCCATTTCTTTAACATCTCCTCTCATATCTAGCCTCTCCTCCCCAGCCTATAG TTTATTCCTCAGTACGTGTAATGGTTCATATTATGTACCAACTTGACTAGGTTACAAGA
Продолжение таблицы 2 TGCTCATATATTTGGTTAAACAGTATTTCTGGGTGCATCCCTGAGAGTGTCTCCAGAAG attagcatttaagtctgactgagtaaagaagatgtctccctcgtcccccagtt Gggg tg| I gg cattatccgatctatga gG gcctcaacagaacaaaa A gg t gg a ggg a gG gagaattt GCTTTCTCTGCTTAAATGCTTGAACTGGGACATTGGCTTTTTTCCTACCAGCTCTCCTG GTTCTCAGCCTTCAGGCTCAGCTAGAATGACACCGCTAGCTTTTATGCGTCTCCAGCTT GTAGATGGCAGATTGTGGGACTTCTCAGCCTCCATAATTGCATGAGCCAATTCCATATA ACCTCTGTCTCTGTCTCTCTGTCTCTGTCTG
Длина фрагмента ДНК 580/585 п.н. (1-я хромосома, PIC = 0) CGCTGAGAGAGAGAGAGACCAAATGCCCACAGACGGCTCATCCTAACTCGACATGGAGG aaaagtcctactgagtgctaaaagt aattcccagagtatggaattctgaatgac Gg gct| I ga gg taatgatg gg caccttca gG aggctggcacccccatctcaattataccaacctca ATCTGCTTATACCTGTCAGTTTGTAAAAGAACTCTCCTCTTCCTGTCTTAGCCAGGCTG ctcataagagttccaacaaacaaccaataattaatagaa Gg tt gg tccta gg tt gG tct TAACCAAGCCATGAAGGATGTGTGTGTAGATTTTATACTCCAGCAGAGGGCAGTACCAC ATAAAAATTACTATTAAGGAAAAACTGAGGTGCAGCCTCTTTTGTCAATTACCCTGAAT TTTCTGTATTATTGAATAATTATTATTTACTGTCACAAAAACGGGTAACTTGTTCATAC GGTTATCTTGAGTATCAATGGACAATCCTGGCAATCAATTTAAGCCCTAACAATTTTAA ATCTTACAATTTTACATAATACTTGCTTTACGTGAGTCTCTCTCCTCTCACATG
Длина фрагмента ДНК 550/637 п.н. (Х-хромосома, PIC = 0,29) CAAAACGAGAGAGAGAGACCCAGACTTCTCATAGAACCCGAATTAGGACCGATACACTC CTTACTGCCAGAATGTAAGAGGAATTTCCCCTATAAGTCCCGTGAGTCAGAACTCAGGA aaataatga | gggg ctttccat gg ctctcctga gg A aaagagaaatatctatttacttga AGAACATGGAATGTGTTTCATACCACTTTGTCTTTCTCTAGGACTGAGGTCTGTTGGCT TTTTAAAATATAGCTACCCAAGGAGTGGGATCTCTCTCTTTCTCTCTCTCCATGTTTTT CTCTTTCTCCATCTCTCTCCATCTCTCTCCATCTATCTCTCCATCTCTCTCCGTCTCTC TCTCCATCTTTTTCTCTTTCTCCATCTCTCCATCTCTCTCCATCTGTCTCTCCATCTCT CTCCATCTCTCTCCATCTGTCTCTCCATCTCTCTCCATCTCTCTCCATCTCTGTCTATC TCTCTCCATCTTTTTCTCTTTCTCCATCTCTCTGTCCATCTCTGTCCATCTCTCTCCGT CTGTCTCTCTATCTCTCTCCATCTCTGTCCATCTCTCTCCATCTCTCTCTCCATCTATC CTCCATTTCTCTCCATCTCTCTCTCCATGTCTCTCCATCTCTCTCTC
Длина фрагмента ДНК 550/572 п.н. (10-я хромосома, PIC = 0,29) AAGAAAGAGGGAGAGAGACAGAGAAATACTCTGCTCTCTCCAGCACTCCAGTCTTCTGC cagtgcctcccattggccagacctacc T gg aa gg ctgag gg caacagactct gG gagct gtagttcccggaaataccgagaacagtaggaggtg Ggg gtagga ggg atacaaaa gggG GG caaaaagcatctggcattgattctaaaactaacaggaaaaactaggctgtgaaaagt CAAAAGCTCTTAGGAAAATGTCACTTTGGAGATGAGCTCACATCTCTTATGAAGTTTAG CACAGCCAGTTTTACCTCTGGTGTTGGAACAGAAAATTCTCCTTTTGATTGGGCAATAG ATGACATCTTTGGCTTTGCTCTGGGGTCATCTTGTACGAACAATATGAATATTTCAATG CTAAAATCATTCAGCTCAGTGACATGCTCATAACTTGAGAAGTTGCCAGAAACTGAGAA GAAGAACCAGGGTAAAAGCAGTTTCACACCTCCCATTTCGTCCTTACTCAGAAATCTCA GTCTCTCTCTGTTTCTATCTCTGTCTCTCTCTCTTTCTCTT
Длина фрагмента ДНК 500/494 п.н. (1-я хромосома, PIC = 0,44) CACAGAGGAGGAGAGAGACTGTTCCCCAGAACAGAACTGGGTGCAGAACCAGAAGAAAG TGAGACTCCTGCTGGGGCCAAAAACGTCAATGAGTTCTGATGTAGTGGACAACGACACC ACTGACAGAGCGGTTGTAAGGAGAAAAGGGACCAAACAGTAAAGCACTGGGCACAGTGC CCTGTCCCTTCTCAGGGGTCAGCAGCGTTAGCTGTGCTAACCATCGCTTCTCTCAATCC tcagcaactctgtgaagcatctcccccattccccagatgggcgaccgg Gg ctcgagct G I g tgatgg gg ttgcctga gG ctgcctaagtgccaggcaagtctctgatccagcccgtccg CAGGGCCGGCTACATACACTGCAAAGCCTTGTGCAAACGAAAGCAAGGAAGCCCTTGTT CAAAAAGCAGGAAAAATACCATTAAGAGAGCTAAAATATAAAGCTTTTTCCTTTCTTCC AGTGTCTCTCTCTCTCTCGCCA
Длина фрагмента ДНК 490/489 п.н. (28-я хромосома, PIC = 0) GAGAGAAAGAGAGAGAGACCACAATGAGATATAAAATACAGGACATAGTTTAAATTACT GACAAAGTGGCAAACTGGTGTGTGTGTCTTGTGGTACCCTTCGGGAAGCTGTCCTGTCT TTTTTTTTTCATATCACCTGATACCTCCAGCTCCACTCGGCTCAAGGTTTGTCAGGTCA AGAGCTCAGAAGAGCAGCCGCACACAGCGCTCTCCACAATCTCTCGCCTGTGCTCGCCC AGATAGACCTCAGGCCCTGGCCCTTTCATGTCCTCTCCCAGCTCTCAGCCTGGCTCCCC cctcagaaagccagagtcccagccgaatcagaggagctggtgttagagtccccgaaga G G tcacctc gg ccgtcct gg g gG cacagctgtcatgggtttcagagcagcaggtaaaatg gactgcaagttgagagcagacccaaca Gggg a gg agtcagaca gG cgcccacacaccgt CTCTCTCTCTCACACAC
Длина ф р а г м е н т а ДНК 380/378 п.н. (11-я хромосома, PIC = 0) acacacagaggagagagacgtag GG agaagagaa GG ccgtgtgaagac GG a GG cagaga TTGGAATGATGCAGCCACAAGCCAAGGAGCATCTGGAACCATCCGGAAGCTACAAGAGG CAAGCAAGAATTCTCCCCTACAGCCTTTGGAGGGAACGTGGCTCTGCCAACATCTTGAT TTCAGACTTCTGGCCGCCAGAACTGTGAGAGAATGAATTTCTGTTGTTTATAGCCACCA AGTTTGTAGTCATTTGTTATGGCAGCCTGAGCAAAGTAACACAGGAAATTACTGTGATA ATGTAGCAAAACGTTTAAAAGCCTGTTTCCCCAAGCACGTCTCTCCTTCTCTGTCTCGC TGCGTGTCTCTCTCTGTCTCTCTC
Длина ф р а г мента ДНК 380/377 п.н. (2 1-я хромосома, PIC = 0) tgaggagagggagagagac a gg ggaacact gg ttggt GG a gcagtcataacacacacat TTATCAATTGTTAGCCATCTTATATGGTTGAGGTTCATCGCCCCTCAAAACAATTACAA TAGTGCCATCAAAGATCACTGACACAGATCACCACTACAAATATAATAATAATTAAAAA TTTGAAATTTTGTGAAAATTACCAAAATGTGACACATAAAAGTGACCAAATGCTGCTGG GAAAATGACTCCACTAGACTTTCTTATTGCAGGGTTGCCACAAACCTTCAATTTGTAAA AATGCAATATCTGCAAAGTGCAATAAAGTGGGCTGCGAAAAAATGAGTTAGGCTTCTCT CTCAGTCTCTCTATCTCTCTCTA
Длина фрагмента ДНК 360 (370) 1 /364 п.н. (5-я хромосома, PIC = 0,42 1 ) GAAAAAGAGGAAGAGAGACCGGAGCTCTTTCTTTCTCTCTTTCGATCATGTGAGGATAC
Продолжение таблицы 2
AGCAAGACTGTCTGGGAACCAGGAAGAAGGCCCTTACTGAGAACCTAATTGGCAGGCAC
CTTGCTCTTGGACTTCCCCATCTCTTATTCTGTTGTTTAAGCCACTCAGTCTATGGTAT
TTGTTATAGCAGCCCATCTTAGTCATAAGACATACATATATATACACATATACGTATAT
GTGAGTCTGTGTGTGTGTATATATATATGTATTTTTTTCCATTTCCTAAATTCATGCAC TTTCAAAAAATGCCTTCTCTTCCTTTTTCACATTCTGTAGAAATAGTACAGTCTCTCTT CTCTTCCCCT
Длина фрагмента ДНК 310/309 п.н. (5-я хромосома, PIC « - »)
TTCACAGAGAGAGAGAGACAGAGACAGAGATGAGAGAGAGAGGACAGGAAATGCAAAGA GATAACTCTAAATAGAGAGGCAAAAGGGAAGAAAATAAGGAACATATAGATAGAATAAT
ATTATCATTCCAGAGACTCTCAGTGAATTTCTGTAAAGTCCATAGGGACAGGGCCCCTT
ATCGCCATGTTTCAGGGATTTGATTAAGGATGCTCAGCAGGCACTTGACAGAGTGAGCT
TGTTCAGTTCTAATCCTTCGAGGAACCATTACTCATTCTGCAACATTGAGCCATGTCTC
TCTTTCCCTCTTCT
Примечание. Представлены результаты анализа сиквенсов из Gen-Bank для последовательностей размером до 2000 п.н. на соответствие полученным собственным экспериментальным данным по ISSR-PCR амплификации фрагментов ДНК с праймером (GA)9C (фактическая дли-на/расчетная длина). PIC — индекс полиморфного информационного содержания локуса, PIC «-» означает, что в эксперименте невозможно оценить полиморфизм фрагмента в связи с отсутствием четкого совпадения экспериментальных и расчетных длин фрагментов, 1 (в верхнем индексе) — опубликованные данные (6). Светло-серым выделены сайты отжига праймера, серым — точки ошибки отжига; последовательности, предрасположенные к формированию G4 квадруплексов, отграничены рамками.
3. Представленность диспергированных повторов различных классов в секвенированных участках генома у домашней лошади Equus caballus , фланкированных инвертированным повтором (GA^C
|
Класс |
| Число | От о< |
бщего числа повторов, % |
|
ERV/ERV3 |
47 |
15,93 |
|
DNA/hAT |
32 |
10,85 |
|
NonLTR/SINE/SINE2 |
30 |
10,17 |
|
NonLTR/L1 |
29 |
9,83 |
|
LTR/Gypsy |
28 |
9,49 |
|
NonLTR/CR1 |
17 |
5,76 |
|
DNA |
15 |
5,08 |
|
DNA/EnSpm/CACTA |
13 |
4,41 |
|
DNA/Mariner |
10 |
3,39 |
|
LTR/Copia |
10 |
3,39 |
|
ERV/ERV1 |
9 |
3,05 |
|
ERV/ERV2 |
7 |
2,37 |
|
DNA/Ginger2 |
5 |
1,69 |
|
DNA/Kolobok |
5 |
1,69 |
|
LTR/BEL |
4 |
1,36 |
|
DNA/Dada |
3 |
1,02 |
|
DNA/Harbinger |
3 |
1,02 |
|
Interspersed Repeat |
3 |
1,02 |
|
DNA/Helitron |
2 |
0,68 |
|
DNA/MuDR |
2 |
0,68 |
|
DNA/Polinton |
2 |
0,68 |
|
LTR |
2 |
0,68 |
|
NonLTR/L2 |
2 |
0,68 |
|
NonLTR/Tad1 |
2 |
0,68 |
|
Simple/Sat |
2 |
0,68 |
|
DNA/Novosib |
1 |
0,34 |
|
DNA/piggyBac |
1 |
0,34 |
|
DNA/Transib |
1 |
0,34 |
|
DNA/Zator |
1 |
0,34 |
|
LTR/DIRS |
1 |
0,34 |
|
NonLTR/CRE |
1 |
0,34 |
|
NonLTR/I |
1 |
0,34 |
|
NonLTR/Rex1 |
1 |
0,34 |
|
NonLTR/RTE |
1 |
0,34 |
|
NonLTR/RTEX |
1 |
0,34 |
|
NonLTR/Tx1 |
1 |
0,34 |
Для кластера 800900 п.н. также были найдены потенциально амплифи-цируемые участки ДНК из разных хромосом: 900 п.н. (24-я хромосома), 876 п.н. (X-хромосома), 871 п.н. (18-я хромосома), 857 п.н. (3-я хромосома), 832 п.н. (6-я хромосома), 829 п.н. (19-я хромосома). Наибольшую точность отжига имел фрагмент длиной 871 п.н. из 18-й хромосомы (см. табл. 2). Отметим, что при повышении температуры отжига примерно такому размеру фрагмента соответствовала зона, которая четче визуализировалась в виде кластера. Далее на электрофореграмме располагался фрагмент ~ 580 п.н. Согласно расчетам, он мог соответствовать участку длиной 585 п.н., локализованному в 1-й хромосоме (см. табл. 2).
Особый интерес представляла зона, соответствующая 550 п.н. Исходя из найденных нуклеотидных последовательностей, она, ве роятно, гетерогенна и включает участки из 10-й хромосомы (полная длина фрагмента 572 п.н.) и X-хромосомы (полная длина 637 п.н.). В каждом из них находились три сайта отжига праймера — по одному прямому и по два обратных. Таким образом, существует возможность амплификации как полных фрагментов (соответственно 572 и 637 п.н.), так и их коротких вариан- тов (550 и 552 п.н.) (см. табл. 2). Не исключено, что по указанной причине этот участок относился к числу локусов, которые воспроизводились наиболее стабильно, хотя он и не был одним из самых яркоокрашенных.
Мажорный локус длиной около 490 п.н., по-видимому, включал фрагмент ДНК из 28-й хромосомы, имеющий длину 489 п.н. и высокую комплементарность флангов к праймеру (GA^C. Мажорная зона ~ 380 п.н. могла состоять из нескольких фрагментов: 377 п.н. (21-я хромосома), у которого последовательности флангов наиболее полно комплементарны праймеру (GA) 9 C, и 378 п.н. (11-я хромосома).
Таким образом, в хромосомах домашней лошади было найдено 99 фрагментов ДНК, которые потенциально могут быть амплифицированы в ПЦР с праймером (GA) 9 C (фрагменты с наиболее точными сайтами отжига на флангах — см. табл. 2).
Анализ нуклеотидных последовательностей в расчетных спектрах фрагментов ДНК ФИП (GA)9C. Мы проанализировали 99 участков геномной ДНК лошади, фланкированных инвертированным повтором (GA)9C и потенциально способных к формированию продуктов амплификации в ПЦР с этим микросателлитом в качестве праймера, на наличие фрагментов мобильных генетических элементов (МГЭ) разных классов с помощью базы данных (табл. 3). Оказалось, что наибольшее распространение имеют участки гомологии с эндогенными ретровирусами III класса, которые составляли приблизительно 15,93 % от общего числа всех выявленных МГЭ. На долю гомологии к эндогенным ретровирусам I и II классов приходилось соответственно 3,05 и 2,37 %. Значительно распространенными были участки гомологии к DNA/hAT (10,85 %), NonLTR/SINE/SINE2 (10,17 %), NonLTR/L1 (9,83 %) и LTR/Gypsy (9,49 %).
Оценивая специфичность связи участков гомологии во фрагментах ДНК, фланкированных инвертированным повтором (GA) g C, именно с этими МГЭ, мы выполнили поиск участков ДНК с прямыми повторами на флангах. Оказалось, что таких участков в секвенированном геноме лошади существенно больше, чем фланкированных инвертированным повтором (GA) 9 C (99 фрагментов).
4. Частота мобильных генетических элементов (ЧМГЭ) и доля участков гомологии к ним (ДУГ) во фрагментах ДНК, фланкированных инвертированными и прямыми повторами микросателлита (GA^C, по сравнению с долей нуклеотидов МГЭ в секвенированном геноме (ДМГЭГ) у домашней лошади Equus caballus
|
Группа МГЭ |
ДМГЭГ, % |
Инвертированные повторы |
Прямые повторы |
||
|
ЧМГЭ, % | |
ДУГ, % |
ЧМГЭ, % | |
ДУГ, % |
||
|
LINE L1 |
16,25 |
10,31 |
4,83 |
11,56 |
9,29 |
|
LINE CR1 |
0,26 |
5,84 |
4,70 |
6,80 |
2,85 |
|
SINE |
2,85 |
9,97 |
4,45 |
13,61 |
5,63 |
|
LTR/Gypsy |
- |
9,62 |
5,22 |
16,33 |
5,42 |
|
LTR ERV, III класс |
1,82 |
15,81 |
9,55 |
7,48 |
4,17 |
|
LTR ERV, I класс |
1,53 |
3,09 |
0,96 |
8,16 |
4,34 |
|
LTR ERV, II класс |
0,07 |
2,06 |
0,64 |
1,36 |
0,56 |
|
DNA/hAT |
1,55 1 |
11,34 |
5,94 |
3,40 |
0,90 |
Примечание. Прочерк означает отсутствие гомологии в секвенированном геноме крупного рогатого скота; 1 (в верхнем индексе) — приведены данные по геному человека (P. Arensburger с соавт. Genetics, 2011, 188: 45-57).
В секвенированной ДНК лошади спектр участков с прямыми повторами (GA^C, имеющих гомологию к различным типам МГЭ, существенно отличался от представленного выше (см. табл. 3) по частоте областей гомологии к эндогенным ретровирусам III класса и DNA/hAT (табл. 4).
Интересно отметить, что по нашим данным доля нуклеотидов, приходящихся на области гомологии к эндогенным ретровирусам и особенно к ДНК транспозонов, выше полученной для секвенированного генома домашней лошади (5) (см. табл. 4). Таким образом, можно ожидать, что инвертированные повторы микросателлита (GA) 9 C наиболее тесно ассоциированы с участками гомологии к эндогенным ретровирусам III класса, LINE_L1 и DNA/hAT, поскольку в этих участках их доля выше, чем в се-квенированном геноме крупного рогатого скота.
-
5. Предполагаемые рекомбинационные характеристики мобильных генетических элементов (МГЭ) в секвенированных последовательностях генома домашней лошади Equus caballus , соответствующих полиморфным и консервативным фрагментам ДНК в ISSR-PCR
Длина фрагмента
PIC
Число, размер, локализация участка, совпадающего по длине
Потенциальные участки рекомбинаций с МГЭ/повторы/класс
Частота образования квадруплексов (не пе-рекрывающиеся/пере-крывающиеся)
980 п.н.
0,49
1 (986 п.н.,
Полиморфные локусы DNA transposon/hAT/MER103C
3 (G-Score = 11, 13, 14)/
7401 п.н.
0,291
23-я хромосома)
1 (739 п.н,
Non-LTR Retrotransposon/L1/L1-8 GM
14 (G-Score = 8, 9, 11, 13, 13, 7, 7, 7, 6, 7, 14, 14, 10, 11) 2 (G-Score = 4, 20)/1
550 п.н.
500 п.н.
0,29
0,44
16-я хромосома)
2 (637 п.н., Х-хромосома;
572 п.н., 10-я хромосома)
1 (494 п.н.,
Endogenous Retrovirus/ERV3/MLT1G2
Endogenous Retrovirus/ERV3/MLT1F1
Endogenous Retrovirus/ERV3/MLT2B2
637 п.н.:
LTR Retrotransposon/Gypsy/Gypsy-53_PM-LTR
Non-LTR Retrotransposon/L1/L1M6_5end
572 п.н.:
DNA transposon/Mariner/Tc1/MamRep137
Endogenous Retrovirus/ERV3/LTR33A
Endogenous Retrovirus/ERV3/LTR33
DNA transposon/hAT/MER45C
2 (G-Score = 3, 4, 2, 3, 4, 4, 5, 5, 20, 20, 19, 20)
-
1 (G-Score = 12)/
-
2 (G-Score = 11, 12)
2 (G-Score = 12, 34)/
438 (G-Score = 4815 — суммарный)
1 (G-Score = 19)/
900 п.н.
0
1-я хромосома)
1 (900 п.н.,
Консервативные локусы
DNA transposon/hAT/Homo5
46 (G-Score = 563 — суммарный)
2 (G-Score = 16, 8)/
580 п.н.
0
24-я хромосома) 1 (585 п.н.,
DNA transposon/Dada/Dada-1_CSa) 0
3 (G-Score = 16, 15, 8) 2 (G-Score = 18, 18)/
490 п.н.
0
1-я хромосома)
1 (489 п.н.,
0
14 (G-Score = 17, 18, 16, 17, 13, 14, 17, 11, 18, 12, 4, 13, 14, 18)
2 (G-Score = 15, 12)/
380 п.н. 0
Примечани
28-я хромосома) 10 (G-Score = 14, 15, 13, 8,
10, 9, 7, 9, 8, 12)
2 (377 п.н., 377 п.н.:
21-я хромосома; DNA transposon/Tigger1B_Crp 1 (G-Score = 17)/
378 п.н., 33 (G-Score = 410 — сум-
11-я хромосома) марный)
378 п.н.:
Endogenous Retrovirus/ERV3/MLT1C1 1 (G-Score = 10)/
2 (G-Score = 10, 10)
е. PIC — индекс полиморфного информационного содержания локуса, G-Score — баллы
вероятности формирования G 4 (рассчитаны по программе QGRS, цифрами указано число потенциальных
G4). В качестве
праймера использована последовательность (GA) 9 C; 1 (в верхнем индексе) — данные,
опубликованные ранее (5).
-
Мы детально проанализировали по четыре предполагаемые последовательности для полиморфных и консервативных локусов, исходя из данных о секвенированных участках генома с наиболее высокой точностью отжига праймера, близких по длине соответствующим фрагментам амплификации (см. табл. 2, 5). В результате в четырех полиморфных локусах выявили 11 участков мобильных элементов, из которых к ретротранспозонам (мобильные элементы I класса) относятся 8, то есть 73 % от числа областей гомологии к разным МГЭ. Почти половина из этих 11 уча- стков (45 %) приходилась на долю эндогенного ретровируса III класса ERV3, тогда как на транспозоны II класса (ДНК-транспозоны) — всего 23 %. В отношении консервативных фрагментов ДНК были обнаружены только четыре участка гомологии с МГЭ, причем три из них — с ДНК-транспозонами (МГЭ II класса). То есть в геноме домашней лошади в полиморфных ISSR-PCR маркерах, выявляемых с (GA)gC в качестве праймера, по сравнению с консервативными с относительно повышенной вероятностью могут присутствовать участки гомологии к ERV III класса. Напомним, что эндогенные ретровирусы млекопитающих подразделяются на три класса в зависимости от их происхождения от экзогенных ретровирусов, которое оценивается на основании гомологии последовательностей. Для ERV I класса предполагается тесная связь c ретровирусами Gammaretrovirus и Epsilonretrovirus, для ERV II класса — с Alpharetrovirus , Betaretrovirus, Deltaretrovirus и Lentivirus, для ERV III класса — с Spumavirus (9).
На 1000 п.н. в расчетных участках ДНК, фланкированных инвертированным повтором (GA) 9 C и соответствующих по длине полиморфным фрагментам ДНК при использовании этого микросателлита в качестве праймера в ПЦР, приходилось 1,5 участка различных МГЭ. Только в двух фрагментах из 99 исследованных отсутствовали области гомологии к МГЭ. Иными словами, плотность локализации участков гомологии к МГЭ во фрагментах, фланкированных инвертированным повтором (GA^C, существенно выше прогнозируемой на основании распространенности таких участков в геноме лошади (5). Полученные данные свидетельствуют о том, что большинство ISSR-PCR маркеров представлены фрагментами ДНК, сформированными при рекомбинациях между различными МГЭ. Это соответствует результатам секвенирования фрагментов ДНК лошади и крупного рогатого скота, фланкированных инвертированным повтором последовательности (GA) 9 C, которые мы получили ранее (10, 11).
Возможные причины неполного совпадения экспериментальных и расчетных спектров для фрагментов ДНК ФИП (GA)9C. Следует отметить, что в силу различных причин полученные экспериментальные спектры фрагментов геномной ДНК не всегда полностью соответствуют теоретически ожидаемым. Например, существует вероятность того, что секвенированные области не включали анализируемый участок либо сам сиквенс оказался неполным (известно, что теломерные и центромерные повторы иногда секвенируются частично) и/или содержал ошибки. Локус может быть связан с Y-хромосомой лошади, сек-венированная последовательность которой не представлена в базе данных . Дополнительными факторами, вероятно, служат структурные особенности района геномной ДНК, в которой локализован фрагмент с инвертированным повтором микросателлита на флангах.
Наши расчетные данные поиска последовательностей ДНК тоже не всегда совпадали с результатами ПЦР-амплификации. В частности, возникали затруднения при выявлении фрагментов с длиной менее 350 п.н. на электрофореграммах, тогда как в расчетных спектрах их находили.
Кроме того, что одна часть данных по полилокусному генотипированию домашней лошади была получена нами при использовании лиофилизированных ПЦР-наборов PCR Core («Биоком», Россия), другая — с помощью набора ПЦР-РВ («Синтол», Россия), состоящего из жидких реагентов. Генотипируя одних и тех же животных с применением в ПЦР одного и того же праймера, мы регистрировали неодинаковые спектры, ха- рактерные для каждого из наборов (см. рис.). Причина различий заключалась в лиофилизированной Taq-полимеразе, при применении которой предпочтительно воспроизводилась более «легкая» часть спектра и происходило не только общее снижение числа локусов, но и превращение неко торых минорных фрагментов ДНК в мажорные. При добавлении в реакцию жидкой полимеразы фирмы «Синтол» или «СибЭнзим» (данные не представлены) спектр полностью восстанавливался. Очень важным представляется то обстоятельство, что точность отжига праймера, по-види-мому, не играет в этом процессе определяющей роли. Подтверждением тому служит полное отсутствие в спектрах с лиофилизированной Taq-по- лимеразой фрагмента 877 п.н. с самыми точными сайтами отжига среди всех найденных последовательностей, при этом некоторые локусы, имею щие даже большую длину, воспроизводились стабильно (см. рис., Б).
В спектре ампликонов, полученных с лиофилизированной Taq-полимеразой, обнаруживался минорный компонент длиной примерно
310 п.н., для которого также было найдено соответствие в сиквенсах хро мосом (см. табл. 2). При использовании набора ПЦР-РВ выявить этот фрагмент не удалось. В то же время с помощью набора ПЦР-РВ при повышении температуры отжига из кластера 800-900 п.н. выделялся локус размером 880 п.н., что соответствует расчетному фрагменту ДНК длиной 877 п.н., имеющему на флангах последовательности с высокой компле-ментарностью к праймеру (GA)9C.
Неканонические структуры ДНК в расчетных и экспериментальных спектрах фрагментов ДНК ФИП (GA)9C. Как от- мечалось, причиной неполного совпадения расчетных и экспериментальных спектров ISSR-PCR маркеров могут быть особенности внутренней структуры анализируемых фрагментов ДНК, в частности предрасположенность к формированию неканонических вторичных структур. Само их присутствие на флангах инвертированного повтора микросателлита свидетельствует о вероятности образования шпилек, что может способствовать повышению геномной нестабильности в таких участках (4). В этой связи мы проанализировали возможности для формирования шпилек (длиной не менее 8 п.н.) и G4 квадруп
6. Характеристика потенциальных шпилечных структур во фланкированных инвертированным повтором (GA)9C последовательностях секвенированного генома у домашней лошади Equus caballus
|
Длина локуса, п.н. |
Длина фрагмента, п.н. |
Хромосома |
Число шпилек внутри локуса |
Доля G-C пар в возможных шпильках, % |
|
900 |
900 |
24-я |
7 |
25,0-2,5 |
|
800-900 |
876 |
Х |
9 |
0-50,0 |
|
800-900 |
871 |
18-я |
4 |
25,0-100 |
|
800-900 |
857 |
3-я |
1 |
62,5 |
|
800-900 |
832 |
6-я |
4 |
50,0-62,5 |
|
800-900 |
823 |
19-я |
2 |
37,5 |
|
550 |
637 (552) |
Х |
5 |
22,2-50,0 |
|
550 |
572 (550) |
10-я |
1 |
37,5 |
|
500 |
494 |
1-я |
1 |
37,5 |
|
490 |
489 |
28-я |
4 |
37,5-75,0 |
|
380 |
378 |
11-я |
1 |
50,0 |
|
380 |
377 |
21-я |
0 |
- |
|
360 (370) 364 |
5-я |
0 |
- |
|
|
310 |
309 |
5-я |
0 |
- |
|
Примечание. Представлены данные для локусов длиной до |
||||
|
2000 п.н |
., соответствующих по размеру фрагментам ДНК, ам- |
|||
|
плифицированным в ISSR-PCR с последовательностью (GA) 9 C в |
||||
|
качестве праймера. |
Прочерки означают отсутствие таких после- |
|||
|
довательностей. Минимальное число спаренных |
оснований для |
|||
|
формирования шпилек — 8. |
||||
лексов (структурные элементы с повышенной термоустойчивостью, образующие четырехцепочечные структуры, в которых гуанины формируют плоскость благодаря поперечным связям) внутри ампликонов (12). Рас смотрены связи между плотностью локализации таких структур в расчетных спектрах и полиморфизмом фрагментов ДНК соответствующей длины в экспериментальных спектрах у лошадей (см. табл. 5, 6).
Оказалось, что в последовательности длиной 877 п.н., локализованной в Х-хромосоме, выявлялось 9 достаточно крупных шпилек, что больше, чем во всех остальных участках (для анализа использовали олигокалькулятор . В последовательности длиной 871 п.н. из 18-й хромосомы обнаружились 4 потенциальные шпильки. Важно подчеркнуть, что в экспериментальных спектрах участки близкой длины выявлялись только при использовании набора ПЦР-РВ. В то же время для наиболее воспроизводимых в разных условиях ПЦР фрагментов ДНК была характерна пониженная частота таких последовательностей. Например, фрагмент длиной 380 п.н. имел всего одну шпильку, причем в наиболее близком по длине фрагменте длиной 377 п.н. (21-я хромосома) потенциальные шпильки отсутствовали. Не имела потенциальных шпилек и нуклеотидная последовательность минорного локуса длиной 310 п.н. (309 п.н., 5-я хромосома), амплифицуруемого в эксперименте при использовании лиофилизированной Taq-полимеразы. Полученные данные позволяют предполагать, что предрасположенность к формированию вторичных структур может влиять на воспроизводимость фрагментов ДНК в разных условиях ПЦР.
Известно, что G4 квадруплексы тесно связаны с возникновением двуцепочечных разрывов ДНК (DSBs) (13). Имеются данные, свидетельствующие о выраженных ассоциациях между наличием G4 квадруплексов и двуцепочечными разрывами ДНК (DSBs) в митотических и мейотических клетках (14). В этой связи мы сопоставили частоту потенциальных G4 квадруплексов в расчетных спектрах секвенированных нуклеотидных последовательностей, близких по длине к консервативным и полиморфным фрагментам ДНК из экспериментальных спектров ISSR-PCR маркеров . При этом выраженных различий мы не обнаружили (см. табл. 5, 6). В обеих группах G4 квадруплексы встречались примерно с равной частотой (около 3,4 потенциального G4 квадруплекса на 1000 п.н.). Тем не менее, один из расчетных участков, по длине наиболее близкий к высокополиморфному в экспериментальных спектрах фрагменту ДНК длиной 550 п.н., проявлял выраженную предрасположенность к формированию таких структур (см. табл. 5). Более того, в геноме человека, например, аналогичные последовательности в среднем встречаются с частотой примерно 0,125 потенциального G4 квадруплекса на 1000 п.н. (15). В полученных нами расчетных последовательностях ампликонов ISSR-PCR спектра по праймеру (GA^C эта цифра примерно в 27 раза больше, что может свидетельствовать о повышенной частоте локализации G4 квадруплексов в участках генома, расположенных между инвертированными повторами микросателлитов.
Итак, у домашней лошади инвертированные повторы микросател-литных локусов, фланкирующие участки ДНК длиной до 2000 п.н. (ISSR-PCR маркеры), преимущественно сформировались в результате сложных рекомбинационных событий между мобильными генетическими элементами, принадлежащими к разным классам транспозонов. Полиморфные ампликоны ДНК, фланкированные инвертированным повтором (GA)9C, которые по длине близки к обнаруженным в секвенированных геномных последовательностях, могут содержать больше участков гомологии с ретротранспозонами (в частности, с последовательностями эндогенного ретровируса III класса — ERV III), чем консервативные фрагменты. Наличие в ISSR-PCR маркере продуктов рекомбинаций с ретротранспозонами (в отличие от ДНК-транспозонов), по всей видимости, ассоциировано с по- вишенным полиморфизмом этого геномного участка. Образование вторичных структур внутри последовательности ДНК, фланкированной инвертированным повтором микросателлита, способно повлиять на воспроизводимость полилокусного генотипирования по ISSR-PCR маркерам. В участках геномной ДНК длиной до 2000 п.н., фланкированных инвертированным повтором (GA)9C, обнаруживается повышенная плотность нуклеотидных последовательностей, потенциально предрасположенных к формированию G4 квадруплексов, что, вероятно, может быть обусловлено повышенной активностью рекомбинаций в этих участках.


