Изучение аллелофонда и степени генетической интрогрессии домашней и дикой популяций северного оленя (Rangifer tarandus L., 1758) с использованием микросателлитов
Автор: Харзинова В.Р., Доцев А.В., Крамаренко А.С., Лайшев К.А., Романенко Т.М., Соловьева А.Д., Денискова Т.Е., Костюнина О.В., Брем Г., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Геномное сканирование, биоразнообразие
Статья в выпуске: 6 т.51, 2016 года.
Бесплатный доступ
Совместное существование домашней и дикой форм северного оленя ( Rangifer tarandus L., 1758) - важная особенность вида. Обе формы обитают в условиях, которые остаются практически неизменными очень продолжительное время. Установлено, что между домашней и дикой популяциями происходит обмен генами, приводящий к смешению их генофонда. Наряду с эволюционными факторами (дрейф генов, мутации, естественный отбор) на изменение генофонда популяции влияет миграционный процесс. В настоящей работе на основе анализа микросателлитов дана характеристика биоразнообразия двух самых многочисленных популяций северного оленя: домашних оленей ненецкой породы и дикой популяции, обитающей на территории Ненецкого (НАО) и Таймырского (ТАО) автономных округов, а также оценена степень интрогрессии этих популяций. Материалом для исследований служили пробы ткани 178 северных оленей. Биоматериал от животных ненецкой породы домашних оленей (DOM, n = 115, 4 субпопуляции) был взят на сельхозпредприятиях НАО и в оленеводческих бригадах на территории ТАО. Материал от оленей дикой таймырской популяции (WLD, n = 63, 5 субпопуляций) собирали в разных регионах ТАО. Геномную ДНК выделяли с использованием колонок фирмы Nexttec («Nexttec Biotechnologie GmbH», Германия). Полиморфизм 9 STR-локусов (NVHRT21, NVHRT24, NVHRT76, RT1, RT6, RT7, RT9, RT27, RT30) определяли по ранее разработанной методике на ДНК-анализаторе ABI3130xl («Applied Biosystems», США). Для оценки аллелофонда каждой популяции рассчитывали среднее число аллелей (Na) и эффективное число аллелей (Ne) на локус, аллельное разнообразие, вычисленное с применением процедуры рарификации (Ar), число приватных аллелей на локус (PrAr), наблюдаемую (Ho) и ожидаемую (He) гетерозиготность, коэффициент инбридинга (FIS). Степень генетической дифференциации популяций оценивали на основании попарных значений FST и генетических дистанций по M. Nei. На основе частот аллелей микросателлитов рассчитывали показатели миграции генов между популяциями. Распределение общей генетической изменчивости между популяциями и в их пределах изучали методом AMOVA (анализ молекулярной дисперсии). Олени дикой популяции характеризовались бóльшим генетическим разнообразием по сравнению с домашними: среднее число аллелей на локус - 10,00±0,78 против 8,44±0,80, наблюдаемая гетерозиготность - 0,633±0,060 против 0,589±0,049. Показано формирование двух независимых кластеров, соответствующих дикой и домашней популяциям, с высокими значениями членства в собственных кластерах: QWLD = 0,940±0,013 и QDOM = 0,938±0,010. При этом были выявлены несколько особей (4,4-4,8 %), имеющих смешанное генетическое происхождение. Степень взаимной интрогрессии популяций составляла около 6 %. Кластерный анализ генетической структуры отдельно дикой и домашней популяций не выявил четкой кластеризации, что указывало на однородность генетической структуры внутри изучаемых популяций. Разложение общей генетической изменчивости с использованием AMOVA показало, что большая часть разнообразия приходилась на изменчивость внутри популяций (95,40 %, p ST и DN составили соответственно 0,046 против 0,023 и 0,353 против 0,151). Полученные нами данные будут использованы для разработки программ селекционно-племенной работы с ненецкой породой домашних северных оленей, а также организации мероприятий по охране и рациональному использованию биологических ресурсов диких северных оленей.
Аллелофонд, интрогрессия, популяции, северный олень, микросателлиты
Короткий адрес: https://sciup.org/142213984
IDR: 142213984 | УДК: 636.294:575.174 | DOI: 10.15389/agrobiology.2016.6.811rus
Текст научной статьи Изучение аллелофонда и степени генетической интрогрессии домашней и дикой популяций северного оленя (Rangifer tarandus L., 1758) с использованием микросателлитов
Оленеводство — одна из древнейших отраслей животноводства, занимающая ведущее место в сельском и промысловом хозяйстве Крайнего Севера. Это единственная отрасль сельского хозяйства, с которой прямо
Исследования выполнены при поддержке Российского научного фонда, проект ¹ 16-16-10068.
или косвенно связаны представители 18 коренных малочисленных народов, насчитывающих свыше 130 тыс. человек, преимущественно сельских жителей (1). Благодаря северным оленям в тундровой и лесотундровой зонах продуктивно используются миллионы гектаров пастбищ, недоступные для других видов животных (2). По данным проекта Арктического Совета «Устойчивое оленеводство» (3), в России находится примерно 2/3 мирового поголовья домашних северных оленей, которые пасутся в тундре, лесотундре, тайге и горных областях на территории более 3 млн км2.
Северный олень ( Rangifer tarandus L., 1758) — единственный представитель рода Rangifer C. H. Smith, 1827. За исторически длительный период методами народной селекции была выведена многочисленная группа домашних северных оленей с четко выраженными морфо-биологическими и хозяйственно полезными признаками, устойчиво передающимися потомству. В 1985 году утверждены и занесены в государственный реестр четыре породы северного оленя: ненецкая, чукотская, эвенкийская и эвенская. Численность домашних оленей в России за последнее десятилетие сократилась в 2 раза и по состоянию на 1 января 2012 года оценивалась в 1583 тыс. голов, что составило лишь 70,0 % от соответствующего показателя в 1990 году (4).
Ненецкая аборигенная порода северных оленей — самая большая в России и мире по численности (950 тыс. голов) и используемой пастбищной территории (110 млн га) (5). Олени этой породы средней величины, с достаточно хорошо выраженными рабочими качествами (6). Они отлично приспособлены к местным природно-климатическим условиям (7). Олени ненецкой породы широко распространены на территории Ненецкого, Ямало-Ненецкого, Ханты-Мансийского и Таймырского АО, Мурманской и Архангельской областей и Республики Коми. По данным племенного учета на 1 января 2015 года, на территории Ненецкого автономного округа (НАО) в 23 хозяйствах содержится около 170 тыс. оленей (8). Несмотря на наличие нескольких эколого-географических типов, олени ненецкой аборигенной породы наиболее однотипны и консолидированы. Более крупные особи встречаются только на арктических островах (Колгуев, Вайгач и др.), что связывают с благоприятными условиями питания, а не с генетическими особенностями (9).
За указанный период снизилась численность и диких северных оленей (10-12). В настоящее время она составляет 1,4 млн гол., при этом более 70 % поголовья сосредоточено на севере Средней Сибири. Самая крупная популяция — таймырская, представляющая исключительную ценность как ключевой компонент биоразнообразия Таймыра. Дикий олень во многом определяет условия возобновления растительности, и именно через этот вид осуществляется воздействие на среду обитания млекопитающих и птиц региона (13-14). Таймырская популяция северных оленей — географический и экологический феномен, не имеющий аналогов по численности, длительности миграций (до 1500 км) и широте освоения зональных типов растительности (от северной тайги до полярных пустынь) (15).
Совместное существование домашней и дикой форм — важная особенность вида. Домашний северный олень не сильно отличается от своего дикого предка. Обе формы обитают в почти одинаковых условиях, которые остаются практически неизменными очень продолжительное время. В оленеводстве не используются искусственные корма, не разработаны зоотехнические приемы кормления и искусственного содержания. Домашние северные олени живут на полувольном выпасе, питаются теми же кормами, что и дикие сородичи, однако используют кормовые ресурсы более 812
полно, поскольку не совершают длительных миграций (3). Некоторые авторы, обсуждая внутривидовой статус домашнего северного оленя, считают, что в одних и тех же географических районах он образует единый генофонд с дикой формой (16).
Для оленеводческой отрасли сосуществование домашней и дикой форм северного оленя — серьезная проблема (11), среди причин которой отмечают увод домашних оленей дикими, стравливание пастбищ и взаимная пастбищная конкуренция, сохранение очагов инфекций, а также перенос болезней. Наиболее серьезны первые две, в результате действия которых полностью исчезло домашнее оленеводство в центральной части Таймырского полуострова, значительно сократилось поголовье домашних оленей на Восточном Таймыре и в ряде других районов (17). При этом Е.Е. Syroechkovskii (18) и L.M. Baskin (19) считают, что сокращение дикой популяции северного оленя вызвано интенсивным развитием домашнего оленеводства и его обширным вытеснением дикой популяции.
Установлено, что между домашней и дикой популяциями происходит обмен генами, приводящий к смешению их генофонда. Наряду с эволюционными факторами (дрейф генов, мутации, естественный отбор) на изменение генофонда популяции влияет миграционный процесс. Дивергенция между популяциями может быть спровоцирована генетическим дрейфом по причине полной или частичной изоляции, а также гетерогенной селекции среди субпопуляций. Миграции особей, как бы незначительны они ни были, препятствуют дивергенции, вызывая снижение генетического разнообразия между популяциями, но в то же время повышая этот показатель внутри субпопуляций (20).
Основные виды сельскохозяйственных животных (крупный рогатый скот, овцы, свиньи, козы, лошади, куры, собаки) были одомашнены несколько тысячелетий назад (21-22), их генетическая изменчивость сохраняется внутри и между породами, и они в значительной степени отличаются от своих диких предшественников (23-24). Несмотря на то, что северный олень был одомашнен 5-10 тыс. лет назад (25-26), между дикой и домашней популяцией наблюдается обмен генами (27).
Оценку генетического разнообразия и мониторинг эволюционных процессов можно адекватно осуществлять посредством молекулярно-генетических методов, обеспечивающих объективность суждения о генетической структуре популяций и позволяющих определять степень интрогрессии между ними (28). Один из методов, широко применяемых для оценки генетической структуры популяций северного оленя разных подвидов, — анализ микросателлитов, известных также под названием STR (short tandem repeats, короткие тандемные повторы) (29-33). Наиболее часто микросателлиты используются в популяционных и экологических исследованиях для изучения переноса генов, эффективного размера популяций, миграционных процессов, внутривидового распределения генетической изменчивости и степени дифференциации популяций (34-37).
В представленном исследовании на примере популяций, обитающих на севере Ненецкого и Таймырского автономных округов, впервые показана возможность выявления особей северного оленя, принадлежащих к дикой, домашней или смешанным формам, с использованием высокополиморфных ДНК-маркеров.
Целью настоящей работы было изучение аллелофонда домашней и дикой популяций северного оленя, обитающих на севере Ненецкого и Таймырского автономных округов, и оценка степени генетической интрогрессии между ними с использованием микросателлитов.
Методика. Исходным материалом для исследований служили пробы ткани 178 северных оленей ( Rangifer tarandus L., 1758), включая домашних оленей ненецкой породы (DOM, n = 115) и животных дикой таймырской популяции (WLD, n = 63). Животные ненецкой породы были отобраны на двух предприятиях Ненецкого автономного округа (НАО) — СРО «Илебц» (ILB, n = 17) и СПК «Индига» (IND, n = 64), а также в двух оленеводческих бригадах, расположенных в Таймырском автономном округе (ТАО) — бригаде ¹ 4 в районе р. Дудинки (DUD, n = 26) и ¹ 11 в районе реки Пелятки (PEL, n = 9). Образцы от диких особей были собраны при экспедиционных исследованиях в различных географических регионах ТАО: о. Портнягино (POR, и = 7; 74 ° 8'48.12"N, 107 ° 9'54"E), д. Белогорка (BEL, и = 13; 72 ° 9'33.13"N, 91 ° 18'92"E), пос. Конгудояр (KON, и = 19; 72 ° 7'62.05"N, 91 ° 36'09"E), пос. Усть-Авам (UAV, и = 20; 71 ° 11'40.71"N, 92 ° 82'07"E) и пос. Волочанка (VOL, и = 4; 70 ° 97'60.83"N, 94 ° 54'13"E). Материал для исследований собирали в течение 2016 года. Построение карты мест отбора проб выполняли в пакете R-ggmap (38) c визуализацией в пакете ggplot2 (39).
Геномную ДНК выделяли с использованием колонок фирмы Nexttec («Nexttec Biotechnologie GmbH», Германия) согласно рекомендациям фирмы-изготовителя. Полиморфизм 9 STR-локусов (NVHRT21, NVHRT24, NVHRT76, RT1, RT6, RT7, RT9, RT27, RT30) определяли с помощью ранее разработанной методики (33) на ДНК-анализаторе ABI3130xl («Applied Biosystems», США). Размеры аллелей, определенные программой GeneMapper 4.0 («Applied Biosystems», США), были конвертированы в числовые выражения, на основании которых сформировали матрицу генотипов в формате MS Excel.
Для оценки аллелофонда каждой популяции определяли среднее число аллелей (Na) и эффективное число аллелей (Ne) на локус (40), аллельное разнообразие, рассчитанное с применением процедуры рарифика-ции (Ar), число приватных аллелей на локус (PrAr) (41), наблюдаемую (Ho) и ожидаемую (He) гетерозиготность, коэффициент инбридинга (FIS) (42). Степень генетической дифференциации популяций оценивали на основании попарных значений FST (43) и генетических дистанций по M. Nei (44). Для вычислений использовали программное обеспечение GenAIEx 6.5.1 (45), HP-Rare 1.1 (46) и Genetix 4.05 (47).
Степень миграции генов между популяциями рассчитывали на основе частот аллелей микросателлитов, используя divMigrate модель графических сетей (48), реализованную в пакете R diveRsity (49). Эта модель позволяет сравнивать популяции на основе разных типов оценок генетической дифференциации: попарных значений FST (45), GST (50), DN (46) и DJost (51). Анализ главных компонент (Principal Component Analysis, PCA) выполняли с помощью R пакета аdegenet (52) с визуализацией в R пакете gglot2 (39).
Распределение общей генетической изменчивости между популяциями и в их пределах изучали методом анализа молекулярной дисперсии (AMOVA) с использованием программного обеспечения Arlequin suite 3.5.2.2 (53). Генетическую структуру популяций оценивали кластеризацией в программе STRUCTURE 2.3.4 (54), с использованием смешанной модели (число предполагаемых кластеров, К — от 1 до 6; длина burn-in периода — 100 000; модель Марковских цепей Монте-Карло — 100 000). Для каждого значения К выполнялось по 10 итераций. Приложение STRUCTURE HARVESTER (55) использовали для определения оптимального числа кластеров (ΔК) для выборки, оцененного по методу G. Evanno с со-814
авт. (56). Для каждой из выборок определяли среднее значение коэффициента членства Q в i-м кластере для общего числа кластеров K.
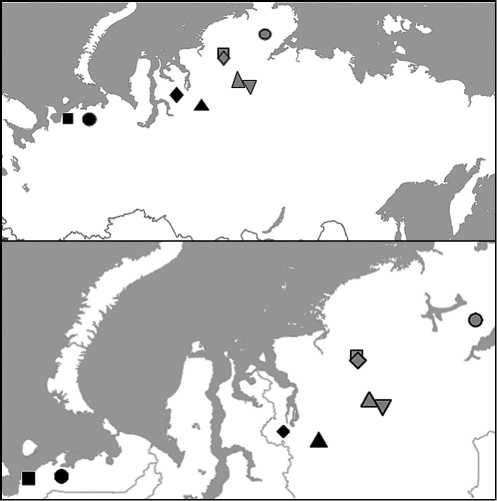
Рис. 1 . Места отбора проб у животных из дикой и домашней популяций северного оленя ( Rangifer tarandus L., 1758): • — ILB, ■ — IND, ♦ — PEL, ▲ — DUD, • — POR, ■ — BEL, ♦ — KON, ▲ — UAV, ▼ — VOL. Описание субпопуляций см. в разделе «Методика».
Результаты. Места отбора образцов для исследования показаны на рисунке 1.
Анализ показателей Na, Ne, Ar, PrAr, Ho и He выявил тенденцию более высокого генетического разнообразия в дикой популяции по сравнению с домашней. Дефицит гетерозигот отмечался в обеих популяциях (табл. 1), однако в субпопуляции VOL был выявлен их незначительный избыток. Аналогичная закономерность описана K.H. Mager (57) при изучении генетической структуры дикой популяции северного оленя карибу на Аляске и домашней популяции северного оленя полуост- рова Сьюард (западная Аляска): Ar = 12,28-13,15, He = 0,86 — в дикой популяции; Ar = 10,06, He = 0,75 — в домашней популяции.
1. Генетическое разнообразие исследованной дикой (WLD) и домашней (DOM) популяций северного оленя ( Rangifer tarandus L., 1758) по локусам микросателлитов
|
Популяция, субпопуляция |
n |
Na |
Ne |
Ar |
PrAr |
H o |
H e |
F IS |
|
POR |
7 |
6,00±0,62 |
4,47±0,61 |
4,59±0,39 |
0,32±0,14 |
0,730±0,065 |
0,738±0,036 |
0,011 |
|
BEL |
13 |
7,44±0,56 |
5,29±0,52 |
4,76±0,24 |
0,36±0,11 |
0,633±0,072 |
0,790±0,029 |
0,199 |
|
KON |
19 |
8,00±0,60 |
5,17±0,46 |
4,66±0,23 |
0,23±0,08 |
0,620±0,068 |
0,787±0,029 |
0,213 |
|
UAV |
20 |
7,89±0,72 |
4,74±0,69 |
4,42±0,31 |
0,29±0,10 |
0,617±0,080 |
0,741±0,044 |
0,167 |
|
VOL |
4 |
3,67±0,50 |
2,80±0,45 |
3,67±0,50 |
0,20±0,10 |
0,611±0,074 |
0,576±0,056 |
- 0,060 |
|
WLD |
63 |
10,00±0,78 |
5,44±0,63 |
4,60±0,26 |
2,29±0,29 |
0,633±0,060 |
0,786±0,035 |
0,195 |
|
ILB |
16 |
5,56±0,65 |
3,39±0,50 |
3,60±0,35 |
0,10±0,03 |
0,563±0,052 |
0,643±0,056 |
0,125 |
|
IND |
64 |
7,22±0,66 |
3,97±0,43 |
3,88±0,21 |
0,11±0,05 |
0,603±0,045 |
0,723±0,030 |
0,167 |
|
DUD |
26 |
6,44±0,56 |
3,67±0,53 |
3,74±0,27 |
0,15±0,04 |
0,556±0,067 |
0,684±0,042 |
0,188 |
|
PEL |
9 |
5,89±0,63 |
4,24±0,56 |
4,24±0,37 |
0,21±0,07 |
0,642±0,076 |
0,722±0,042 |
0,111 |
|
DOM |
115 |
8,44±0,80 |
4,12±0,56 |
3,92±0,26 |
1,61±0,19 |
0,589±0,049 |
0,719±0,037 |
0,180 |
П р и м е ч а н и е. Na — среднее число аллелей на локус, Ne — среднее эффективное число аллелей на локус, Ar — аллельное разнообразие, PrAr — число приватных аллелей, Ho — наблюдаемая гетерогзигот-ность, He — ожидаемая гетерозиготность, FIS — коэффициент инбридинга. Описание субпопуляций см. в разделе «Методика».
Для проведения кластерного анализа было определено наиболее вероятное число кластеров (К), то есть объективно обособленных генетических групп в изучаемой выборке, согласно алгоритму, основанному на значениях величины ΔК (56). Оптимальным числом кластеров для выборки оказалось K = 2 (ΔK = 87,23). Структурный анализ при K = 2 (рис. 2) показал высокую степень генетической обособленности как дикой, так и домашней популяций северного оленя, о чем свидетельствуют средние зна- чения коэффициента подобия Q в каждой популяции (QWLD = 0,940±0,013 и QDOM = 0,938±0,010). Вместе с тем в дикой популяции выявили три (QWLD = 0,441, QWLD = 0,508, QWLD = 0,631), а в домашней — пять особей (QDOM = 0,310, QDOM = 0,505, QDOM = 0,621, QDOM = 0,368, QDOM = 0,684) (соответственно 4,4 и 4,8 %), имеющих смешанное генетическое происхождение. Наличие таких смешанных (помесных) особей северного оленя, несущих в генотипе долю аллелей другой популяции, свидетельствует о периодическом обмене генами между ними. Кластерный анализ, проведенный отдельно по домашней популяции (К = 1-5), показал однородность генетической структуры всех выборок. Наши результаты подтверждают исследования, проведенные по основным морфо-биологическим показателям, показателям роста и развития и полиморфным белковым системам крови, которые дали основание считать породу единой по происхождению и генотипу (58).
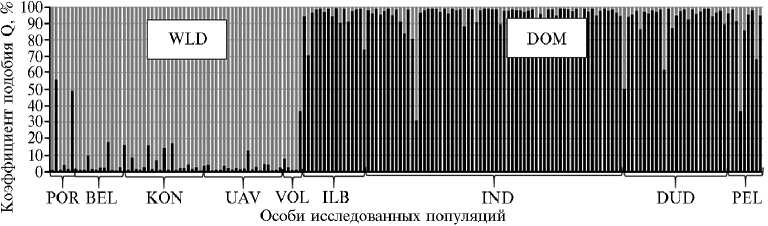
Рис. 2. Результаты кластерного анализа дикой (WLD) и домашней (DOM) популяций северного оленя ( Rangifer tarandus L., 1758) на основе девяти STR-локусов с использованием программы STRUCTURE 2.3.4 (54) для числа кластеров К = 2 . Q — величина вклада каждого из кластеров в генотип особи (вероятность того, что особь принадлежит к тому или иному кластеру). Домашняя популяция отмечена на рисунке черным, дикая популяция — серым цветом. Описание субпопуляций см. в разделе «Методика».
Несмотря на то, что дикая таймырская популяция северного оленя, представлена несколькими изолированными группировками (10), по мнению Н.В. Малыгиной (59), ее следует рассматривать как единую целую экологическую популяцию. Возможно, именно поэтому все выборки дикой таймырской популяции, изученные нами, характеризовались общей генетической структурой без четкой кластеризации между ними (анализ для дикой популяции был проведен при К = 1-6).
Оценка молекулярной вариации (AMOVA) показал, что 95,4 % генетической изменчивости северного оленя приходилось на изменчивость внутри выборок домашней и дикой популяций и 4,6 % — на изменчивость между популяциями (p < 0,001).
Генетическая изменчивость в природных популяциях, как правило, свидетельствует о структуре, возникающей вследствие различных процессов (географическая изоляция, эффект родоначальника, миграции и адмик-сия). Один из методов, который широко используется для выявления такой структуры популяций, — PCA-анализ, определяющий две-три основные оси в изменчивости данных и проецирующий исследуемые объекты на эти оси (60). Такой анализ на основе частот аллелей исследованных популяций позволяет одновременно охарактеризовать размах вариабельности, выявить основные аллели, вносящие вклад в дифференциацию групп особей, проследить главные паттерны популяционно-генетической дифференциации и обнаружить их связь с экологическими и географическими факторами (61). Результаты проведенного нами PCA-анализа представлены на рисунке 3.
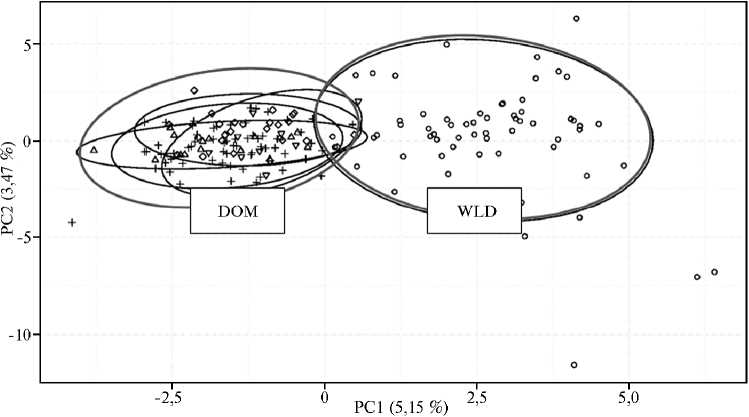
Рис. 3. Расположение особей северного оленя ( Rangifer tarandus L., 1758) из дикой (WLD) и домашней (DOM) популяций в пространстве первых двух главных компонент: DOM — домашняя популяция, А — ILB, + — IND, О — DUD, V — PEL. Описание субпопуляций см. в разделе «Методика».
Первая главная компонента отражала 5,15 % наблюдаемой изменчивости популяций и четко разделяла домашнюю и дикие популяции северного оленя. Наличие незначительного перекрывания массивов, соответствующих домашней и дикой популяциям, можно рассматривать как указание на наличие неконтролируемой миграции между ними. По мнению А.В. Давыдова (62), именно в период миграций стада диких северных оленей уводят часть домашних особей, а дикие самцы, зашедшие во время гона в домашние стада, спариваются с самками и оставляют свое потомство (до 3 % от числа новорожденных). Внутри популяций олени из разных субпопуляций демонстрировали сходство в отношении частоты общих аллелей, представляя собой общий генетический пул.
2. Генетические дистанции между изученными субпопуляциями дикой и домашней популяций северного оленя ( Rangifer tarandus L., 1758)
|
Субпопуляция |
POR |
BEL |
KON |
UAV |
VOL |
ILB |
IND |
DUD |
PEL |
|
POR |
0,194 |
0,187 |
0,241 |
0,353 |
0,436 |
0,387 |
0,378 |
0,393 |
|
|
BEL |
0,003 |
0,122 |
0,191 |
0,324 |
0,402 |
0,369 |
0,339 |
0,368 |
|
|
KON |
0,004 |
0,003 |
0,119 |
0,230 |
0,397 |
0,349 |
0,328 |
0,320 |
|
|
UAV |
0,024 |
0,021 |
0,009 |
0,118 |
0,450 |
0,425 |
0,326 |
0,348 |
|
|
VOL |
0,046 |
0,040 |
0,025 |
0,002 |
0,562 |
0,521 |
0,400 |
0,387 |
|
|
ILB |
0,097* |
0,093* |
0,096* |
0,116* |
0,150* |
0,072 |
0,094 |
0,151 |
|
|
IND |
0,072* |
0,074* |
0,075* |
0,099* |
0,120* |
0,008* |
0,077 |
0,101 |
|
|
DUD |
0,076* |
0,073* |
0,075* |
0,081* |
0,097* |
0,020* |
0,015* |
0,113 |
|
|
PEL |
0,047* |
0,047* |
0,045* |
0,060* |
0,066* |
0,023 |
0,018 |
0,010 |
П р и м е ч а н и е. Несмещенные дистанции по M. Nei (DN) показаны над диагональю; значения FST при парном сравнении — под диагональю. Описание субпопуляций см. в разделе «Методика».
* Различия достоверны по FST (p < 0,05).
Расчеты значений FST при парном сравнении и генетических дистанций по M. Nei (DN) (44) (табл. 2) в целом показали относительно более высокую степень генетической дифференциации субпопуляций внутри дикой популяции по сравнению с домашней: максимальные значения FST и DN составили соответственно 0,046 против 0,023 и 0,353 против 0,151. Следует отметить, что внутри дикой популяции выделялись генетически более близкие субпопуляции, чем внутри домашней.
На рисунке 4 представлена оценка степени миграции генов между популяциями северного оленя. Исследуемые выборки особей отображаются узлами в сетях. Каждый узел гипотетически соединен с каждым другим узлом с помощью двух линий, представляющих два взаимных компонента потока генов между любой из пар популяций. Длина и толщина каждой представленной линии изменяются в соответствии с интенсивностью генного потока. Такие характеристики достаточно информативны, поскольку отображают именно те популяции, между которыми обмен генами происходит с высокой интенсивностью локально, но с низкой интенсивностью вне популяций. Паттерны генетической структуры популяции оказываются представлены в виде единого кластера в сетевом пространстве (63).
Между домашней и дикой популяциями интрогрессия аллелей происходила в одностороннем порядке — от дикой к домашней (см. рис. 4). Максимальная степень миграции внутри домашней популяции была выявлена между субпопуляциями ILB-IND и DUD-IND, при этом IND выступала в качестве реципиента потока генов от ILB и DUD. Для диких оленей наибольшее сходство отмечалось между субпопуляциями BEL-KON-UAV, максимальная степень миграции — между UAV и BEL.
По мнению L. Su-ndqvis с соавт. (63), этот подход отображает графиче-
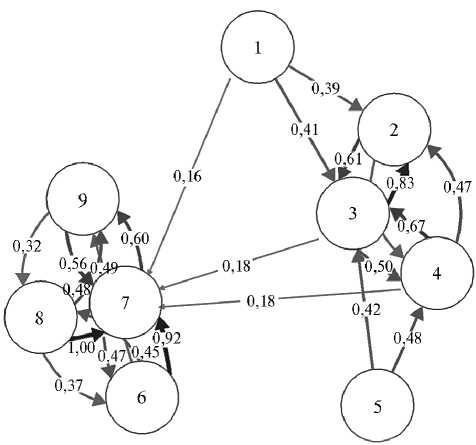
Рис. 4. Относительная направленная миграция генов между популяциями северного оленя ( Rangifer tarandus L., 1758), визуализированная с использованием модели divMigrate (48) : слева — домашняя популяция (DOM), справа — дикая популяция (WLD); 1 — POR, 2 — BEL, 3 — KON, 4 — UAV, 5 — VOL, 6 — ILB, 7 — IND, 8 — DUD, 9 — PEL. Описание субпопуляции см. в разделе «Методика».
определили интрогрессивную гибридизацию
ски интегрированные сетевые паттерны потока генов между популяциями и определяет достоверную разницу в направлении генного потока между парами исследуемых выборок.
Изучение генетического разнообразия и процессов интрогрессивной гибридизации в популяциях северного оленя с помощью генетических маркеров всегда вызывало повышенный интерес ученых. Вопрос о взаимоотношениях между диким и домашним видами северного оленя был затронут еще в 1989 году (64), а также в ранних исследовательских работах (65). В. Jepsen с соавт. (66) между популяциями диких карибу и домашней популяции северного оленя, обитающих на территории города Нуук (Гренландия). М.А. Cronin с соавт. (67) выявили общие аллели по нескольким генам, присутствующие как в дикой, так и в домашней популяциях северного оленя на Аляске, предположив незначительную генетическую интрогрессию в обоих направлениях. Вероятно, гибридные животные менее приспособлены к жизни в дикой природе, а метизация в неволе сравнительно невелика (64, 68). При изучении генетического разнообразия мигрирующих популяций карибу на северном склоне Аляски и их потенциальной гибридизации с домашними оленями K.Н. Mager с соавт. (69) обнаружили несколько особей, имеющих сме- шанное генетическое происхождение (8 % — в популяциях карибу и 14 % — среди домашних оленей).
Ожидается, что расширение набора STR-локусов и увеличение размера исследуемой выборки обеих популяций с включением особей из других точек ареала позволит точнее определить степень интрогрессии этих форм у рассматриваемого вида. Более целостное представление о взаимодействии дикой и домашней популяций на генетическом уровне может дать использование нового поколения молекулярных маркеров, основанных на анализе однонуклеотидных полиморфизмов (SNP, single nucleotide polymorphism) (70). Понимание генетической изменчивости в популяциях северного оленя в масштабах всего ареала раскроет закономерности эволюции этого вида. Кроме того, постоянный мониторинг генетического разнообразия и численности необходим для сохранения дикой популяции северного оленя.
Таким образом, нами на основе полиморфизма микросателлитов охарактеризован аллелофонд выборок двух самых многочисленных популяций северного оленя — домашних оленей ненецкой породы и дикой популяции северного оленя, обитающей на территории Ненецкого и Таймырского автономных округов. Несмотря на то, что кластерный анализ показал высокую степень генетической обособленности обеих форм, были обнаружены несколько особей, имеющих смешанное генетическое происхождение. При сравнении внутрипопуляционных показателей выяснилось, что у диких оленей генетическое разнообразие выше, чем у домашних. Информация о генетическом разнообразии и популяционной структуре ненецкой породы необходима для организации мероприятий по охране и рациональному использованию ее биологических ресурсов.
Список литературы Изучение аллелофонда и степени генетической интрогрессии домашней и дикой популяций северного оленя (Rangifer tarandus L., 1758) с использованием микросателлитов
- Мухачев А.Д. Путешествие в мир оленеводов. Новосибирск, 2001.
- Бороздин Э.К., Забродин В.А., Востряков П.Н. Северное оленеводство/Под ред. В.А. Забродина. М., 1979.
- Йернслеттен Й.-Л., Клоков К. Оленеводство в России. Глава из отчета по проекту Арктического cовета «Устойчивое оленеводство» (Университет Тромсё, Санкт-Петербургский университет; Арктический совет, 2000-2002). Илин, 2005, 1(42). Режим доступа: http://ilin-yakutsk.narod.ru/2005-1/18.htm. Без даты.
- Логинов В.Г. Оленеводство как базовая отрасль традиционного сектора АПК Севера. Аграрный вестник Урала, 2014, 11(129): 74-77.
- Южаков А.А. Ненецкая аборигенная порода северных оленей. Автореф. докт. дис. Новосибирск, 2004.
- Югай В.К. Экстерьерные особенности северных оленей в условиях Ямала. Аграрный вестник Урала, 2009, 10(64): 48-51.
- Stammler F. Reindeer nomads meet the market: culture, property and globalisation at the end of the land. Lit-Verlag Muenster, 2005 (ISBN 3-8258-8046-x).
- Романенко Т.М., Филиппова Г.И. Генетический полиморфизм в популяции домашнего северного оленя Канинско-тиманской тундры Ненецкого АО. Символ науки, 2015, 11: 44-52.
- Эрнст Л.К., Дмитриев Н.Г., Паронян И.А. Генетические ресурсы сельскохозяйственных животных в России и сопредельных странах. СПб, 1994.
- Колпащиков Л.А. Таймырская популяция дикого северного оленя (биологические основы управления и устойчивого использования ресурсов). Автореф. докт. дис. Норильск, 2000.
- Сыроечковский Е.Е., Рогачева Э.В., Савченко А.П., Соколов Г.А., Баранов А.А., Емельянов В.И. Красная книга Красноярского края. Редкие и находящиеся под угрозой исчезновения виды животных. Красноярск, 2000.
- Мухачев А.Д., Колпащиков Л.A., Лайшев К.А. Мясная продуктивность северных оленей. Новосибирск, 2001.
- Тихомиров Б.А. Взаимосвязи животного мира и растительного покрова тундры. М.-Л., 1959.
- Тишков A.A. Взаимодействие животных фитофагов с растительностью тундры. Журнал общей биологии, 1977, 38(1): 15-23.
- Колпащиков Л.А., Михайлов В.В. К проблеме изучения пространственно-временной динамики таймырской популяции диких северных оленей. Современные проблемы природопользования, охотоведения и звероводства, 2012, 1: 108-109.
- Семенов-Тян-Шанский О.И. Северный олень. М., 1977.
- Клоков К.Б. Современное состояние циркумполярного оленеводства. Мат. III Межд. школы по юридической антропологии «Олень всегда прав». СПб-Пушкин, 2003: 53-74.
- Syroechkovskii E.E. Wild reindeer. Amerind Publishing, New Delhi, India, 1995.
- Baskin L.M. Number of wild and domestic reindeer in Russia in the late 20th century. Rangifer, 2005, 25(1): 51-57 ( ) DOI: 10.7557/2.25.1.337
- Papa R. Gene flow and introgression between domesticated crops and their wild relatives. Proc. Int. Workshop «The role of biotechnology for the characterization and conservation of crop, forestry, animal and fishery genetic resources». Turin, Italy, 2005: 71-76.
- MacHugh D.E., Bradley D.G. Livestock genetic origins: goats buck the trend. PNAS USA, 2001, 98: 5382-5384 ( ) DOI: 10.1073/pnas.111163198
- Bruford M.W., Bradley D.G., Luikart G. DNA markers reveal the complexity of livestock domestication. Nat. Rev. Genet., 2003, 4: 900-910 ( ) DOI: 10.1038/nrg1203
- MacHugh D.E., Shriver M.D., Loftus R.T., Cunningham P., Bradley D.G. Microsatellite DNA variation and the evolution, domestication and phylogeography of taurine and zebu cattle (Bos taurus and Bos indicus). Genetics, 1997, 146: 1071-1086.
- Luikart G., Gielly L., Excoffier L., Vigne J.-D., Bouvet J., Taberlet P. Multiple maternal origins and weak phylogeographic structure in domestic goats. PNAS USA, 2001, 98: 5927-5932 ( ) DOI: 10.1073/pnas.091591198
- Baskin L.M. Reindeer husbandry in the Soviet Union. Wildlife production systems: economic utilization of wild ungulates/R.J. Hudson, K.R. Drew, L.M. Baskin (eds.). Cambridge University Press, Cambridge, UK, 1989: 197-206.
- Staalannd H., Eikelmann I.M.H. Status of the reindeer industry in Fennoscandia. Wildlife production: conservation and sustainable development/L.A. Renecker, R.J. Hudson (eds.). AFES Misc. University of Alaska, Fairbanks, 1991, 91(6): 77-88.
- Давыдов А.В., Холодова М.В., Мещерский И.Г., Груздев А.Р., Сипко Т.П., Кол Н.В., Царев С.А., Железнов-Чукотский Н.К., Мирутенко B.C., Губарь Ю.П., Линьков А.Б., Рожков Ю.И. Дифференциация диких и домашних форм северного оленя (Rangifer tarandus L.) по результатам анализа мтДНК. Сельскохозяйственная биология, 2007, 6: 48-53.
- Хохлов А.М. Теоретическое обоснование процесса доместикации, селекционно-генетический и технологический мониторинг в свиноводстве. Автореф. докт. дис. Харьков, 2006.
- Ball M.C., Finnegan L., Manseau M., Wilson P. Integrating multiple analytical approaches to spatially delineate and characterize genetic population structure: an application to boreal caribou (Rangifer tarandus caribou) in central Canada. Conserv. Genet., 2010, 11: 2131-2143 ( ) DOI: 10.1007/s10592-010-0099-3
- Zittlau K.A. Population genetic analyses of North American caribou (Rangifer tarandus). PhD Thesis. University of Alberta, Canada, 2004.
- Cronin M.A., MacNeil M.D., Patton J.C. Variation in mitochondrial DNA and microsatellite DNA in caribou (Rangifer tarandus) in North America. J. Mammal., 2005, 86: 495-505 (doi: 10.1644/1545-1542(2005)862.0.CO;2).
- Tyler S.K., Mcfarlane K.A., Pamela Groves P., Mooers A.Ø., Shapiro B. Modern and ancient DNA reveal recent partial replacement of caribou in the southwest Yukon. Mol. Ecol., 2010, 19: 1312-1323 ( ) DOI: 10.1111/j.1365-294X.2010.04565.x
- Kharzinova V.R., Gladyr' E.A., Fedorov V.I., Romanenko T.M., Shimit L.D., Layshev K.A., Kalashnikova L.A., Zinovieva N.A. Development of multiplex microsatellite panel to assess the parentage verification in and differentiation degree of reindeer populations (Rangifer tarandus). Agricultural Biology, 2015, 50(6): 756-765 ( ) DOI: 10.15389/agrobiology.2015.6.756eng
- Pfeiffer A., Olivieri A.M., Morgante M. Identification and characterization of microsatellites in Norway spruce (Picea abies K.). Genome, 1997, 40: 411-419.
- Scotti I., Magni F., Paglia G.P., Morgante M. Trinucleotide microsatellite in Norway spruce (Picea abies): their features and the development of molecular markers. Theor. Appl. Genet., 2002, 106: 40-50 ( ) DOI: 10.1007/s00122-002-0986-1
- Fluch S., Burg A., Kopecky D., Homolka A., Spiess N., Vendramin G.G. Characterization of variable EST SSR markers for Norway spruce (Picea abies L.). BMC Res. Not., 2011, 4: 401-412 ( ) DOI: 10.1186/1756-0500-4-401
- Cremer E., Liepelt S., Sebastiani F., Buonamici A., Michalczyk I.M., Ziegenhagen B., Vendramin G.G. Identification and characterization of nuclear microsatellite loci in Abies alba Mill. Mol. Ecol. Notes, 2006, 6: 374-376 ( ) DOI: 10.1111/j.1471-8286.2005.01238.x
- Kahle D., Wickham H. ggmap: Spatial visualization with ggplot2. The R Journal, 2013, 5(1): 144-161.
- Wickham H. ggplot2: Elegant graphics for data analysis. Springer-Verlag, NY, 2009.
- Brown A.H.D., Weir B.S. Measuring genetic variability in plant populations. In: Isozymes in plant genetics and breeding, Part A/S.D. Tanksley, T.J. Orton (eds.). Elsevier Science Publ., Amsterdam, 1983.
- Kalinowski S.T. Counting alleles with rarefaction: Private alleles and hierarchical sampling designs. Conserv. Genet., 2004, 5: 539-543 ( ) DOI: 10.1023/B:COGE.0000041021.91777.1a
- Hartl D.L., Clark A.G. Principles of population genetics. Sunderland, Massachusetts, 1997.
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38: 1358-1370.
- Nei M. Genetic distance between populations. Am. Nat., 1972, 106(949): 283-292 ( ) DOI: 10.1086/282771
- Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research -an update. Bioinformatics, 2012, 28: 2537-2539 ( ) DOI: 10.1093/bioinformatics/bts460
- Kalinowski S.T. HP RARE 1.0: a computer program for performing rarefaction on measures of allelic richness. Mol. Ecol. Notes, 2005, 5: 187-189 ( ) DOI: 10.1111/j.1471-8286.2004.00845.x
- Belkhir K., Borsa P., Chikhi L., Raufaste N., Bonhomme F. GENETIX 4.05, logiciel sous Windows TM pour la génétique des populations. Laboratoire Génome, Populations, Interactions, CNRS UMR 5171, Université de Montpellier II, Montpellier (France), 1996-2004.
- Sundqvist L., Zackrisson M.Z., Kleinhans D. Directional genetic differentiation and asymmetric migration. Режим доступа: https://arxiv.org/abs/1304.0118 . Без даты.
- Keenan K., McGinnity P., Cross T.F., Crozier W.W., Prodohl P.A. diveRsity: An R package for the estimation of population genetics parameters and their associated errors. Methods in Ecology and Evolution, 2013, 4: 782-788 ( ) DOI: 10.1111/2041-210X.12067
- Meirmans P.G., Hedrick P.W. Assessing population structure: FST and related measures. Mol. Ecol. Resour., 2011, 11: 5-18 ( ) DOI: 10.1111/j.1755-0998.2010.02927.x
- Jost L. GST and its relatives do not measure differentiation. Mol. Ecol., 2008, 17: 4015-4026 ( ) DOI: 10.1111/j.1365-294X.2008.03887.x
- Jombart T. adegenet: a R package for the multivariate analysis of genetic markers. Bioinformatics, 2008, 24: 1403-1405 ( ) DOI: 10.1093/bioinformatics/btn129
- Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Mol. Ecol. Resour., 2010, 10: 564-567 ( ) DOI: 10.1111/j.1755-0998.2010.02847.x
- Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155: 945-959.
- Earl D.A., von Holdt B.M. Structure Harvester: A website and program for visualizing Structure output and implementing the Evanno method. Conserv. Genet. Resour., 2012, 4: 359-361 ( ) DOI: 10.1007/s12686-011-9548-7
- Evanno G., Regnaut S., Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: A simulation study. Mol. Ecol., 2005, 14: 2611-2620 ( ) DOI: 10.1111/j.1365-294X.2005.02553.x
- Mager K.H. Population structure and hybridization of Alaskan caribou and reindeer: integrating genetics and local knowledge. Dissertation of the degree doctor of philosophy. Fairbanks, Alaska, 2012.
- Южаков А.А. Ненецкая аборигенная порода северных оленей. Автореф. докт. дис. Новосибирск, 2004.
- Малыгина Н.В. Дикий северный олень (Rangifer tarandus L.) Восточного Таймыра. Автореф. канд. дис. М., 2000.
- McVean G. A genealogical interpretation of principal components analysis. PLoS Genet., 2009, 5(10): e1000686 ( ) DOI: 10.1371/journal.pgen.1000686
- Юрченко А.А. Генетическая структура популяций сахалинского тайменя Parahucho perryi brevoort и вопросы природоохранной генетики вида. Автореф. канд. дис. М., 2015.
- Давыдов А.В. Морфологическая и генетическая дифференциация популяций северного оленя Евразии. Автореф. канд. дис. М., 1997.
- Sundqvist L., Keenan K., Zackrisson M., Prodöhl P., Kleinhans D. Directional genetic differentiation and asymmetric migration. Ecol. Evol., 2016, 6(11): 3461-3475 ( ) DOI: 10.1002/ece3.2096
- Burns J.J. Report of the scientific workshop on caribou in the NPRA. Barrow, Alaska, 1989.
- Klein D.R. Conflicts between domestic reindeer and their wild counterparts: A review of Eurasian and North American experience. Arctic, 1980, 33: 739-756.
- Jepsen B., Siegismund H., Fredholm M. Population genetics of the native caribou (Rangifer tarandus groenlandicus) and the semi-domestic reindeer (Rangifer tarandus tarandus) in Southwestern Greenland: Evidence of introgression. Conserv. Genet., 2002, 3: 401-409 ( ) DOI: 10.1023/A:1020523303815
- Cronin M.A., Renecker L.A., Pierson B.J., Patton J.C. Genetic variation in domestic reindeer and wild caribou in Alaska. Anim. Genet., 1995, 26: 427-434 ( ) DOI: 10.1111/j.1365-2052.1995.tb02695.x
- Cronin M.A., Patton J.C., Balmysheva N., MacNeil M.D. Genetic variation in caribou and reindeer (Rangifer tarandus). Anim. Genet., 2003, 34: 33-41 ( ) DOI: 10.1046/j.1365-2052.2003.00927.x
- Mager K.H., Colson K.E., Hundertmark K.J. High genetic connectivity and introgression from domestic reindeer characterize northern Alaska caribou herds. Conserv. Genet., 2013, 14: 1111-1123 ( ) DOI: 10.1007/s10592-013-0499-2
- Kharzinova V.R., Sermyagin A.A., Gladyr E.A., Okhlopkov I.M., Brem G., Zinovieva N.A. A study of applicability of SNP chips developed for Bovine and Ovine species to whole-genome analysis of reindeer Rangifer tarandus. J. Heredity, 2015, 106(6): 758-761 ( ) DOI: 10.1093/jhered/esv081


