Изучение генетического разнообразия домашних и диких популяций северного оленя (Rangifer tarandus L., 1758) с использованием маркеров ядерного и митохондриального геномов
Автор: Кошкина О.А., Соловьева А.Д., Денискова Т.Е., Харзинова В.Р., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика, геномика, генетическая инженерия
Статья в выпуске: 6 т.57, 2022 года.
Бесплатный доступ
Енных воздействий он может оказаться под угрозой исчезновения, поэтому изучение и сохранение генетического разнообразия северного оленя остается актуальной задачей. В представленной работе мы впервые дали характеристику генетического разнообразия северных оленей, обитающих на территории Российской Федерации, выявили филогенетические связи и дали оценку степени дифференциации исследованных животных с использованием комплексного молекулярно-генетического подхода, который заключался в анализе ядерного и митохондриального геномов. Нашей целью была оценка генетического разнообразия, генетической структуры и филогенетических взаимоотношений домашних и диких популяций северного оленя, разводимых на территории Российской Федерации, на основе полных последовательностей гена CytB митохондриальной ДНК и полиморфизма локусов микросателлитов. Исследования проводили в 2022 году. Материалом служили срезы с рогов северного оленя. Выборка включала диких северных оленей тундровой популяции (WLD), домашних оленей ненецкой (NEN), чукотской (CHU), эвенской (EVN) пород, а также красноярской (EVK_KRA) и якутской (EVK_YAK) популяций эвенкийской породы. Для исследования мтДНК были отобраны 123 неродственные особи. Микросателлитный анализ проводили у 213 особей домашних пород и 119 представителей дикой популяции. Полные последовательности гена CytB определяли с использованием NGS (next generation sequencing) технологии (секвенатор miSeq, «Illumina, Inc.», США). Полиморфизм 9 STR (short tandem repeat) локусов (NVHRT21, NVHRT24, NVHRT76, RT1, RT6, RT7, RT9, RT27, RT30) определяли с помощью фрагментного анализа (генетический анализатор ABI3130xl, «Applied Biosystems», США). Для оценки генетического разнообразия каждой группы северных оленей рассчитывали показатели митохондриальной (число полиморфных сайтов S, среднее число нуклеотидных различий K, число гаплотипов H, гаплотипическое разнообразие HD, нуклеотидное разнообразие p) и микросателлитной (аллельное разнообразие, вычисленное с применением процедуры рарификации AR, наблюдаемая HO и несмещенная ожидаемая uHE гетерозиготность, несмещенный коэффициент инбридинга FIS) изменчивости. Степень генетической дифференциации групп оценивали на основании попарных значений FST и JostD . Статистическую обработку данных выполняли с использованием программ MEGA 7.0.26, PopART 1.7, PartitionFinder 2, Arlequin 3.5.2.2, MrBayes 3.2.7, FigTree 1.4.3, DnaSP 6.12.01, SplitsTree 4.14.5, STRUCTURE 2.3.4 и R пакетов diveRsity, pophelper, аdegenet и ggplot2. Анализ последовательностей гена CytB мтДНК показал, что все популяции характеризовались высоким гаплотипическим HD = 0,519 (CHU)-0,997 (WLD) и нуклеотидным разнообразием p = 0,00238 (CHU)-0,00626 (WLD). По мтДНК обособленной генетической структуры исследуемых популяций северного оленя мы не выявили. При анализе микросателлитной изменчивости значения аллельного разнообразия находились в пределах от 6,188 у CHU до 8,76 у WLD. Во всех шести популяциях наблюдаемая гетерозиготность варьировала от 0,566 (CHU) до 0,687 (EVK_YAK) и 0,693 (WLD). Все группы северного оленя характеризовались дефицитом гетерозигот, на что указывали положительные значения индекса фиксации FIS = 0,11 (EVK_YAK)-0,262 (EVK_KRA). Анализ структуры генетической сети показал дифференциацию чукотской породы от остальных, о чем свидетельствуют наивысшие показатели индекса FST и критерия JostD - от 0,203 и 0,488 у EVK_KRA до 0,212 и 0,564 у EVN. Как по ядерным, так и по митохондриальным маркерам популяция диких оленей отличалась от одомашненных популяций более высоким генетическим разнообразием. Можно предположить, что так или иначе селекционная работа с домашними породами северного оленя привела к созданию уникальных массивов животных, отличающихся от исходных диких сородичей. Тем не менее как домашняя, так и дикая популяции характеризовались высокой генетической изменчивостью.
Rangifer tarandus, северный олень, генетическое разнообразие, филогенетическая оценка, митохондриальная днк, микросателлитные локусы
Короткий адрес: https://sciup.org/142237379
IDR: 142237379 | УДК: 636.294:636.082:577.21 | DOI: 10.15389/agrobiology.2022.6.1101rus
Текст научной статьи Изучение генетического разнообразия домашних и диких популяций северного оленя (Rangifer tarandus L., 1758) с использованием маркеров ядерного и митохондриального геномов
Северный олень ( Rangifer tarandus L., 1758) — это биологический ресурс, имеющий важное значение с экологической точки зрения (влияние на растительный покров) и в качестве источника средств к существованию для многих коренных народов Заполярного Севера. Северный олень, вероятно, был необходим для миграции и колонизации человеком Евразийской Арктики и Субарктики после последнего ледникового периода. В последнее время северного оленя также используют как объект для формирования заповедных территорий (1, 2). В отличие от большинства других видов домашнего скота, у которых дикие формы вымерли (например, крупный рогатый скот, лошади), находятся под угрозой исчезновения (ослы, ламы, альпаки) или географически ограничены (овцы, козы), дикие популяции северных оленей по-прежнему широко распространены по Северной Евразии и Северной Америке (карибу). Почти 50 % из примерно 3 000 000 оленей в Старом Свете — дикие животные, а во многих районах дикие и домашние стада тесно сосуществуют (2, 3). Это дает уникальную возможность проанализировать взаимодействие между домашними и дикими популяциями.
Северный олень в основном распространен в арктическом регионе Северного полушария, включая Норвегию, Финляндию, Швецию, Россию, Гренландию, Соединенные Штаты, Монголию, Китай и Канаду. Найденные окаменелости свидетельствуют о том, что в плейстоцене Rangifer обитал к югу от ледникового покрова как в Евразии, так и в Северной Америке, а также в Берингии, охватывающей Беринг-Ленд-Бридж, Аляску и значительную часть Сибири.
Популяции современных северных оленей на основании морфологических и исторических данных классифицируют на три экологические группы: лесные (оседлая форма), тундровые (мигрирующая форма) и высокоарктические островные формы (4). Эти экологические группы включают девять подвидов, из которых к настоящему времени сохранились семь (5). Домашние северные олени в Российской Федерации подразделяются на четыре официально утвержденные породы: ненецкую, эвенскую, эвенкийскую и чукотскую (6).
Северный олень, как и другие голарктические виды, может оказаться под угрозой исчезновения из-за изменения климата и антропогенных воздействий, поэтому четкая идентификация состояния генетического разнообразия и филогенетической структуры на уровне вида важна для сохранения генетического разнообразия (5), которое позволяет видам адаптироваться к изменениям окружающей среды и развивать локальные адаптации (7). Вопросу изучения генетического разнообразия популяций северного оленя посвящено множество работ, основанных на использовании ядерных и митохондриальных маркеров.
J.-C. Zhai с соавт. охарактеризовали генетическое разнообразие восьми популяций северного оленя, обитающих в горах Большого Хингана, с использованием 11 микросателлитных локусов (13). Авторы выявили дефицит гетерозигот во всех популяциях и низкую степень генетической дифференциации. Т.Е. Денискова с соавт. (14) с использованием 14 микроса-теллитных локусов оценили генетическое разнообразие 15 популяций ненецкой породы. Позднее, в 2020 году, V.R. Kharzinova с соавт. (15) изучили структуру популяций 528 домашних северных оленей четырех пород, разводимых на территории Российской Федерации. В том же году Ю. Столпов-ский с соавт. (16) исследовали 397 особей домашних и диких северных оле- ней, разводимых в различных климатических зонах России. Анализ микро-сателлитных локусов показал, что 70 % аллельного разнообразия принадлежит диким популяциям северного оленя.
В 2018 году V.R. Kharzinova с соавт. (17) впервые провели генотипирование северных оленей четырех пород, обитающих на территории России, с использованием BovineHD BeadChip. Была дана полная характеристика генетического разнообразия этих пород, а также их экотипов, обитающих в четырех федеральных округах Крайнего Севера России. Низкие значения генетического разнообразия имели чукотская порода и якутский внутрипо-родный экотип харгин, в связи с чем авторы рекомендовали обратить особое внимание на сохранение и увеличение генетической изменчивости в этих группах (17).
В качестве маркеров митохондриального генома широкое распространение при изучении популяций северного оленя получили два высоковариабельных участка мтДНК — ген CytB и контрольный регион (D-петля). В 2018 году C.D. Wilkerson с соавт. (19) на основе анализа последовательностей D-петли и гена CytB мтДНК выявили 4 гаплогруппы (А, В, С и D) и 32 гаплотипа у лесного карибу на острове Ньюфаундленд. Островные карибу характеризовались достаточно высоким генетическим разнообразием (Hd = 0,894 и π = 0,00216), за исключением оленей с полуострова Авалон, у которых выявили лишь 3 гаплотипа с относительно низкой степенью гап-лотипического (H D = 0,569) и нуклеотидного ( π = 0,00052) разнообразия. Филогенетический анализ позволил авторам проследить направление послеледникового повторного заселения острова северными оленями (19).
В настоящее время набирает популярность комплексный подход, который заключается в использовании нескольких типов молекулярно-генетических маркеров, что позволяет осуществить более точный анализ и получить полную информацию о генетическом разнообразии животных. В 2012 году F. Barbanera с соавт. (11) провели успешное расследование браконьерства кипрского муфлона ( Ovis orientalis ophion ), используя в качестве молекулярно-генетических маркеров 12 микросателлитных локусов совместно с митохондриальным геном CytB. Позднее, в 2021 году, было раскрыто еще одно преступление браконьерства в Кабардино-Балкарской Республике. A. Rodionov с соавт. (12), применив комплексный анализ на основе 14 микросателлитов и SNP генотипирования (ДНК-чип), доказали факт браконьерства кавказского тура ( Capra caucasica ).
M.A. Cronin с соавт. (20) дали количественную оценку генетического разнообразия 11 стад карибу, обитающих в Северной Америке, с использованием 18 микросателлитных локусов и последовательностей гена CytB . Такой комплексный анализ подтвердил внутривидовую классификацию северного оленя на три экотипа: обитающие в тундре на бесплодной земле, горная форма и лесная форма. Позднее те же авторы охарактеризовали генетическое разнообразие домашних северных оленей, обитающих на территории Аляски, Сибири и Скандинавии, в сравнении с дикими карибу с использованием 18 микросателлитных локусов и последовательностей митохондриального гена CytB. Авторы выявили различия в частотах гаплотипов и микросателлитных локусов у домашних северных оленей и диких карибу. Дикие олени характеризовались большим генетическим разнообразием по обоим маркерам (21).
В 2018 году коллектив авторов из Китая (22) исследовал генетическое разнообразие единственной популяции Аолугуйского северного оленя с использованием 10 микросателлитных локусов совместно с геном CytB.
Было показано наличие различной степени инбридинга в популяции. Полиморфизм мтДНК определил относительно низкое генетическое разнообразие (H D = 0,468±0,091, π = 0,0017±0,001), было выделено всего пять уникальных гаплотипов. Полученные данные авторы предлагают использовать для формирования стратегий по сохранению вида и восстановлению популяции (22).
В то время как за рубежом проводятся работы по изучению генетического разнообразия северных оленей с использованием комбинации нескольких маркеров, аналогичных попыток охарактеризовать российские популяции еще не предпринималось. В представленной работе мы впервые дали характеристику генетического разнообразия северных оленей, обитающих на территории Российской Федерации, выявили филогенетические связи и дали оценку степени дифференциации исследованных животных с использованием комплексного молекулярно-генетического подхода, который заключался в анализе ядерного и митохондриального геномов.
Нашей целью была оценка генетического разнообразия, генетической структуры и филогенетических взаимоотношений домашних и диких популяций северного оленя, разводимых на территории Российской Федерации, на основе полных последовательностей гена CytB митохондриальной ДНК и полиморфизма локусов микросателлитов.
Методика. Исследования проводили в 2022 году. Материалом служили срезы с рогов северного оленя. Выборка включала диких северных оленей тундровой популяции (WLD), домашних оленей ненецкой (NEN) (Республика Коми), чукотской (CHU) (Иультинский р-н, Чукотский АО), эвенской (EVN) (Нерюнгринский р-н, Республика Саха) пород, а также красноярской (EVK_KRA) (Красноярский край) и якутской (EVK_YAK) (Алданский р-н, Республика Саха ) популяций эвенкийской породы. Для исследования мтДНК были отобраны 123 неродственные особи. Микроса-теллитный анализ проводили у 213 особей домашних пород и 119 представителей диких популяций.
ДНК выделяли с использованием набора ДНК-Экстран-2 (ООО «Синтол», Россия) по стандартному протоколу производителя. Далее проводили контроль качества и количества полученных образцов ДНК. Концентрацию ДНК в растворе измеряли на флуориметре Qubit 4.0 («Invitrogen/Life Technologies», США), соотношение поглощения OD 260/280 — на спектрофотометре NanоDropTM 8000 («Thermo Fisher Scientific, Inc.», США). В результате отобрали образцы с концентрацией от 15 до 50 нг/мкл и соотношением OD 260/280 от 1,8 и выше.
Для анализа полных нуклеотидных последовательностей использовали технологию секвенирования следующего поколения (next generation sequencing, NGS). Секвенирование образцов проводили в несколько этапов. На первом этапе (пробоподготовка) полные митохондриальные геномы северных оленей получали при амплификации шести перекрывающихся фрагментов (область перекрывания 120-780 п.н.) длиной от 2000 до 4500 п.н. с использованием следующих пар праймеров: F1 5´-TCCTCCCTTCTAGAC-TTAATCTGACT-3´, R1 5´-CTCCTCCCACGACTAGTTGCAC-3´; F2 5´-ACTCCAACCTATTGCAGATGCCAT-3´, R2 5´-AAGGTTATTTCGACTGC-ATGTGCGGTTAC-3´; F3 5´-CTAACACTCAGATTAATTAGAGGACA-3´, R3 5´-GTACTCCGCGGTTCATATTAATGAGAGG-3´; F4 5´-TGCTTGAG-CAGGCATAGAAGGGAC-3´, R4 5´- TGGTGTGTCATTATGACTTGTTGT-GCA-3´; F5 5´-GGAGGAATTACACTGGGATTAATAAG-3´, R5 5´-AATAC-CCTCTACTGCTATTGGCTTGA-3´; F6 5´-GGAACCGTAAAATTGATACA-
ACTCCAA-3´, R6 5´-GGGATTGCAAGCTTATATAGTTATGG-3´. Амплификацию проводили по собственной методике на термоциклере Applied Biosystems SimpliAmp («Thermo Fisher Scientific, Inc.», США) в следующем режиме: 2 мин при 96 ° С (начальная денатурация); 30 с при 96 ° С, 30 с при 60 ° С, 3 мин при 72 ° С (40 циклов), 10 мин при 72 ° С (заключительная элонгация). На втором этапе библиотеки готовили к секвенированию с использованием набора NEBNext Ultra II DNA Library Prep Kit for Illumina («New England Biolabs», США) по стандартному протоколу производителя. Образцы секвенировали методом парных концевых прочтений по 300 п.н. на приборе MiSeq («Illumina, Inc.», США). Завершающим этапом была обработка полученных данных.
Из полных последовательностей мтДНК оленей после их выравнивания, выполненного с помощью алгоритма MUSCLE (23) в программе MEGA 7.0.26 (24), были восстановлены последовательности гена CytB. Аутгруппой служила последовательность гена CytB северного оленя из базы данных NCBI , GenBank accession number NC_007703.1). Построение медианной сети осуществляли в программе PopART 1.7 (25). Наилучшие модели эволюции определяли в программе PartitionFinder 2 (26) с использованием скорректированного критерия информации Акайке (AICc) (27). Оптимальными оказались эволюционные модели HKY+I, HKY+I, GTR+I соответственно для первого, второго и третьего нуклеотида в кодоне. Анализ FST проводили в программе Arlequin 3.5.2.2 (28). Байесовское филогенетическое дерево строили с помощью программного обеспечения MrBayes 3.2.7 (29) с последующей визуализацией в FigTree 1.4.3 (30). Поиск по методу Монте-Карло с цепями Маркова осуществляли с использованием четырех цепей с 10 000 000 шагов, деревья отбирались каждые 500 поколений (первые 25 % отобранных деревьев были отбракованы с помощью алгоритма burn-in).
Параметры генетического разнообразия — число полиморфных сайтов (S), среднее число нуклеотидных различий (K), число гаплотипов (H), гаплотипическое разнообразие (Hd), нуклеотидное разнообразие ( п ), ошибки средних арифметических (±SEM) рассчитывали в программе DnaSP 6.12.01. Гипотезу о расширении популяции проверяли посредством расчета статистики нейтральности Fu’s Fu и теста Tajima’s D в DnaSP 6.12.01 (31).
Анализ полиморфизма 9 микросателлитов (NVHRT21, NVHRT24, NVHRT76, RT1, RT6, RT7, RT9, RT27, RT30) осуществляли согласно ранее описанной методике (32). Полученные фрагменты ДНК визуализировали посредством фрагментного анализа с помощью программного обеспечения Gene Mapper v. 4 («Applied Biosystems», США). Анализ популяционно-генетических параметров, включая рарифицированное аллельное разнообразие (Ar), наблюдаемую (H O ) и несмещенную ожидаемую (uH E ) гетерозиготность, а также несмещенный коэффициент инбридинга (F IS ) c доверительным интервалом 95 % (CI), проводили с использованием R пакета diveRsity с последующей визуализацией в пакете pophelper (33). Степень генетической дифференциации оценивали на основании матриц попарных значений Fst (34) и JostD (35). Для построения филогенетических деревьев по алгоритму «сети соседей» (Neighbor-Net) применяли программное обеспечение SplitsTree 4.14.5 (36) и R пакет diveRsity с последующей визуализацией в пакете pophelper.
Генетическую структуру исследуемых групп северного оленя оценивали с помощью анализа главных компонент (Principal Component Analysis, PCA) в R пакете аdegenet (37) и с визуализацией в R пакете ggplot2 (38), а также посредством кластеризации в программе STRUCTURE 2.3.4 (39) с использованием смешанной модели (число предполагаемых кластеров К — от 1 до 10, длина burn-in периода — 100 000, модель Марковских цепей Монте-Карло — 100 000). Для каждого значения К выполнялось по 10 итераций. Приложение STRUCTURE HARVESTER (40) использовали для определения оптимального числа кластеров (ΔК) по методу, предложенному G. Evanno с соавт. (41). Исходные файлы формировали в программной среде R 3.5.0 (R Core Team) (42).
Результаты. Использование технологии секвенирования следующего поколения (next generation sequencing, NGS) позволило получить полные последовательности митохондриального гена CytB у исследуемых популяций северного оленя.
Анализ нуклеотидных последовательностей митохондриального гена CytB. В полученных нами последовательностях митохондриального гена CytB длиной 1140 п.н. от 123 особей были идентифицированы 40 гаплотипов. Все животные характеризовались высоким генетическим разнообразием (H D = 0,918±0,014; π = 0,00448±0,00023) (табл. 1). В популяциях дикого северного оленя наблюдалось наивысшее гаплотипи-ческое (H D = 0,997±0,013) и нуклеотидное ( π = 0,00626±0,00041) разнообразие в сравнении с домашними (H D = 0,865±0,021; π = 0,00364±0,00022).
1. Индексы генетического разнообразия в популяциях домашних и диких северных оленей ( Rangifer tarandus L., 1758), рассчитанные на основе анализа нуклеотидных последовательностей митохондриального гена CytB (2022 год)
|
Популяции |
1 n 1 |
S |
1 K 1 |
H 1 |
H d ±SEM |
1 π ±SEM |
1 Tajima’s D |
Fu’s Fu |
|
CHU |
22 |
17 |
2,710 |
5 |
0,519±0,114 |
0,00238±0,00066 |
- 1,53176 ns |
1,780 ns |
|
EVK_KRA |
12 |
8 |
3,333 |
5 |
0,788±0,090 |
0,00292±0,00029 |
1,03140 ns |
0,159 ns |
|
EVK_YAK |
14 |
15 |
4,396 |
7 |
0,846±0,074 |
0,00386±0,00055 |
- 0,27767 ns |
0,159 ns |
|
EVN |
21 |
12 |
3,200 |
10 |
0,848±0,059 |
0,00281±0,00029 |
- 0,14393 ns |
- 2,304 ns |
|
NEN |
21 |
17 |
4,038 |
6 |
0,663±0,105 |
0,00354±0,00067 |
- 0,53756 ns |
2,014 ns |
|
Все популяции домашних оленей |
90 |
35 |
4,153 |
21 |
0,865±0,021 |
0,00364±0,00022 |
- 1,24481 ns |
- 4,365 ns |
|
WLD |
33 |
48 |
7,129 |
24 |
0,997±0,013 |
0,00626±0,00041 |
- 1,45464 ns |
- 10,970 |
|
Всего |
123 |
61 |
5,098 |
40 |
0,918±0,014 |
0,00448±0,00023 |
- 1,73284 ns |
- 19,784 |
Примечани е. n — число образцов, S — число полиморфных сайтов, K — среднее число нуклеотидных различий, H — число гаплотипов, H D — гаплотипическое разнообразие, π — нуклеотидное разнообразие, ns — 0,10 > P > 0,05. CHU — чукотская порода, EVK_KRA — эвенкийская красноярская, EVK_YAK — эвенкийская якутская, EVN — эвенская, NEN — ненецкая, WLD — дикие олени.
В популяциях домашних северных оленей наибольшее гаплотипиче-ское разнообразие наблюдалось у эвенской породы (H D = 0,848±0,059). Самое высокое нуклеотидное разнообразие и наибольшее среднее число нуклеотидных различий были обнаружены в эвенкийской якутской популяции ( π = 0,00386±0,00055, K = 4,396). Чукотская порода характеризовалась наименьшим генетическим разнообразием по всем показателям (Hd = 0,519±0,114, π = 0,00238±0,00066, K = 2,710).
Полученные значения критериев Tajima’s D (-1,24481 ns) и Fu’s Fu (-4,365 ns) свидетельствовали о тенденции к полной идентичности между домашними популяциями. Согласно этим значениям, существует ограниченное различие в числе полиморфных сайтов и среднем числе парных различий нуклеотидов между исследуемыми популяциями. То есть изученные домашние породы северного оленя находятся в генетическом равновесии, что указывает на состояние аллелей и генотипов в генофонде их популяций, обеспечивающих адаптацию к изменениям окружающей среды, вызванных прежде всего антропогенными факторами. Напротив, у диких оленей мы выявили высокий отрицательный показатель Fu’s Fu (-10,970), что свидетельствует о потоке чужеродных генов вследствие пространственной экспансии, в то время как низкое значение D (-1,45464 ns) указывало на стабильное состояние популяции.
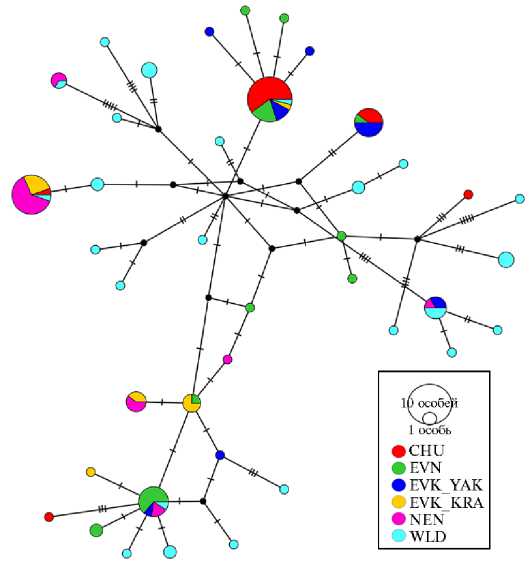
Рис. 1. Медианная сеть, характеризующая связи гаплотипов, идентифицированных у домашних и диких северных оленей ( Rangifer tarandus L., 1758) на основе анализа нуклеотидных последовательностей митохондриального гена CytB : CHU — чукотская порода ( n = 22), EVK_KRA — эвенкийская красноярская ( n = 12), EVK_YAK — эвенкийская якутская ( n = 14), EVN — эвенская ( n = 21), NEN — ненецкая ( n = 21), WLD — дикие олени ( n = 33). Диаметр круга соответствует числу особей, принадлежащих к соответствующему гаплотипу. Число поперечных линий указывает на число нуклеотидных замен. Черные круги в точках ветвления сети обозначают гипотетические гаплотипы (2022 год).
Среди северных оленей как диких, так одомашненных популяций, обитающих на территории Российской Федерации, четкой дифференциации по материнскому маркеру мтДНК мы не выявили.
Из всех 40 гаплотипов общими были только 8, остальные 32 гаплотипа встречались лишь у одного представителя из исследуемой выборки оленей (рис. 1). В основном такое разнообразие гаплотипов достигалось за счет популяций диких животных: из 24 гаплотипов, идентифицированных у диких оленей, лишь 5 оказались общими с популяциями домашних. Представители всех 5 популяций домашних оленей имели по одному общему гаплотипу с популяциями диких северных оленей.
В дополнение к общим гаплотипам с дикими оленями популяции домашних оленей образовали 3 общих гаплотипа: красноярская популяция эвенкийской породы имела по одному общему гаплотипу с представителями ненецкой и эвенской пород, а также один общий гаплотип присутствовал у представителей чукотской, эвенской и якутской популяции эвенкийской породы.
Анализ байесовского филогенетического дерева (рис. 2) выявил четкое расхождение исследуемых групп оленей на два основных кластера. Один кластер включал 17 гаплотипов, которые встречались у CHU, EVN и EVK_YAK. Отдельные представители этих пород также имели гаплотипы, входящие в состав второго кластера. И лишь домашним оленям EVK_KRA принадлежали гаплотипы, характерные только для второго кластера. Дикие олени несли гаплотипы обоих кластеров. Ненецкая порода была наиболее отдалена от особей CHU, что подтверждалось наибольшими значениями показателя критерия FST = 0,32645 (табл. 2, рис. 3).
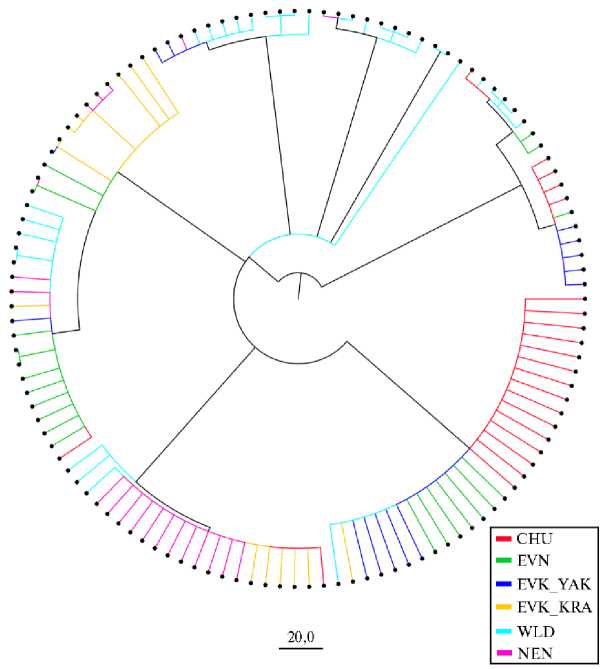
Рис. 2. Байесовское филогенетическое дерево, отражающее генетические связи представителей домашних и диких северных оленей ( Rangifer tarandus L., 1758) на основе анализа нуклеотидных последовательностей митохондриального гена CytB : CHU — чукотская порода ( n = 22), EVK_KRA — эвенкийская красноярская ( n = 12), EVK_YAK — эвенкийская якутская ( n = 14), EVN — эвенская ( n = 21), NEN — ненецкая ( n = 21), WLD — дикие олени ( n = 33) (2022 год).
-
2. Попарные генетические дистанции F ST , вычисленные на основе анализа нуклеотидных последовательностей митохондриального гена CytB , в популяциях домашних и диких северных оленей ( Rangifer tarandus L., 1758) (2022 год)
|
Популяция |
CHU EVN EVK_YAK EVK_KRA NEN WLD |
|
CHU EVN EVK_YAK EVK_KRA NEN WLD |
0 0,16899 0 0,03967 0,13174 0 0,29011 0,08439 0,21856 0 0,32645 0,21418 0,27135 0,00824 0 0,12380 0,08769 0,05715 0,09139 0,12807 0 |
Примечани е. CHU — чукотская порода, EVK_KRA — эвенкийская красноярская, EVK_YAK — эвенкийская якутская, EVN — эвенская, NEN — ненецкая, WLD — дикие олени.
Самые близкие генетические дистанции мы определили между EVK_KRA и представителями NEN, у которых значение индекса фиксации было равно 0,00824.
Четкой породной кластеризации исследуемых групп домашних оленей мы не выявили. Большинство представителей пород несли схожие гаплотипы мтДНК, но некоторые особи имели совершенно отдаленные митохондриальные генотипы. Это можно объяснить случайным спариванием до- машних оленей с дикими особями.
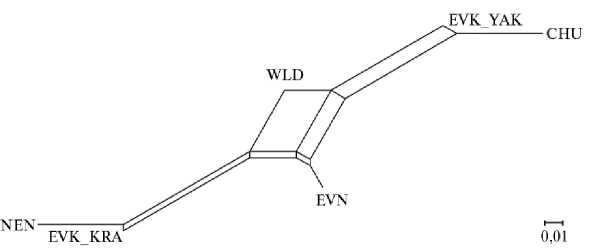
Рис. 3. Генетические взаимосвязи популяций домашних и диких северных оленей ( Rangifer tarandus L., 1758), представленные в виде графика Neighbor Net на основе матрицы значений генетических дистанций F ST при анализе нуклеотидных последовательностей митохондриального гена CytB : CHU — чукотская порода, EVK_KRA — эвенкийская красноярская, EVK_YAK — эвенкийская якутская, EVN — эвенская, NEN — ненецкая, WLD — дикие олени (2022 год).
Микросателлитный анализ. В работе был проведен анализ 332 особей домашнего и дикого северного оленя, обитающего на территории Российской Федерации, с помощью девяти микросателлитных локусов.
Анализ генетического разнообразия (табл. 3) показал, что популяция диких северных оленей характеризовалась относительно высокими значениями аллельного разнообразия (A R = 8,76) по сравнению с популяциями домашних оленей. Этот показатель варьировал от 6,188 в CHU до 7,036 в NEN. Аналогично наивысшие показатели наблюдаемой и несмещенной ожидаемой гетерозиготности (H O = 0,693; uH E = 0,841) были у представителей диких популяций в сравнении с домашними. Наименьшими значениями этих показателей характеризовалась чукотская порода (H O = 0,566; u He = 0,681). EVK_YAK среди домашних северных оленей выделялась наивысшим генетическим разнообразием (H O = 0,687; uH E = 0,775). Во всех популяциях наблюдался дефицит гетерозигот, о чем свидетельствовали положительные величины коэффициента инбридинга uH E , которые колебались от 0,11 у EVK_YAK до 0,262 у EVK_KRA. Значения доверительного интервала коэффициента инбридинга у CHU были близки к нулю ( - 0,049; 0,345), что свидетельствует о состоянии генетического равновесия в этой популяции оленей.
3. Показатели генетической изменчивости в популяциях домашних и диких северных оленей ( Rangifer tarandus L., 1758), рассчитанные на основе полиморфизма 9 микросателлитных локусов (2022 год)
A r — рарифицированное аллельное разнообразие. В скобках — размах изменчивости u F is при доверительном интервале 95 %. CHU — чукотская порода, EVK_KRA — эвенкийская красноярская, EVK_YAK — эвенкийская якутская, EVN — эвенская, NEN — ненецкая, WLD — дикие олени.
Генетические дистанции между исследуемыми популяциями оленей были оценены попарно на основе значений критериев FST и JostD (табл. 4, рис. 4). Анализ структуры генетической сети позволил выявить два условных кластера (см. рис. 4). Первый был представлен популяциями EVN и EVK_YAK, что говорит об их генетическом родстве. Это подтверждается наименьшими значениями показателей FST и JostD между ними (соответственно 0,045 и 0,089) (см. табл. 4). Второй кластер образовали популяции EVK_KRA, NEN, CHU и WLD. В свою очередь, популяция CHU была наиболее отдаленной от остальных, что объясняется географической удаленностью ее ареала.
-
4. Попарные генетические дистанции F ST и JostD, рассчитанные на основе полиморфизма 9 микросателлитных локусов для популяций домашних и диких северных оленей ( Rangifer tarandus L., 1758) (2022 год)
Популяция
EVN
EVK_YAK
EVK_KRA
CHU
NEN
WLD
EVN
0
0,089
0,219
0,538
0,198
0,194
EVK_YAK
0,045
0
0,171
0,564
0,130
0,180
EVK_KRA
0,085
0,068
0
0,488
0,141
0,150
CHU
0,212
0,207
0,203
0
0,364
0,408
NEN
0,099
0,068
0,066
0,175
0
0,178
WLD
0,055
0,051
0,039
0,147
0,053
0
Примечание. кийская якутская,
CHU — чукотская порода, EVK_KRA — эвенкийская красноярская, EVK EVN — эвенская, NEN — ненецкая, WLD — дикие олени.
YAK — эвен-
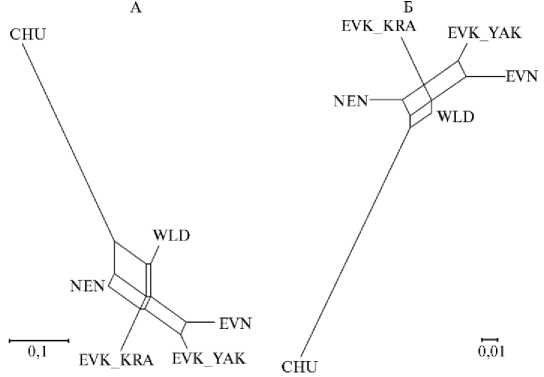
Рис. 4. Генетические взаимосвязи популяций домашних и диких северных оленей ( Rangifer tarandus L., 1758), представленные в виде графика Neighbor Net на основе матрицы значений генетических дистанций JostD (A) и F ST (Б) при анализе полиморфизма по 9 микросателлитным локусам: CHU — чукотская порода, EVK_KRA — эвенкийская красноярская, EVK_YAK — эвенкийская якутская, EVN — эвенская, NEN — ненецкая, WLD — дикие олени (2022 год).
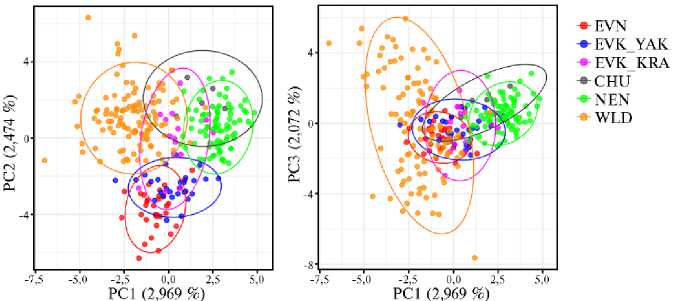
Рис. 5. Результаты анализа главных компонент (PCA), выполненного для популяций домашних и диких северных оленей ( Rangifer tarandus L., 1758) на основе полиморфизма 9 микросателлитных локусов: CHU — чукотская порода ( n = 22), EVK_KRA — эвенкийская красноярская ( n = 12), EVK_YAK — эвенкийская якутская ( n = 14), EVN — эвенская ( n = 21), NEN — ненецкая ( n = 21), WLD — дикие олени ( n = 33) (2022 год).
Для определения популяционной структуры изученных групп оленей был использован анализ главных компонент (PCA) (рис. 5) и кластерный анализ (рис. 6).
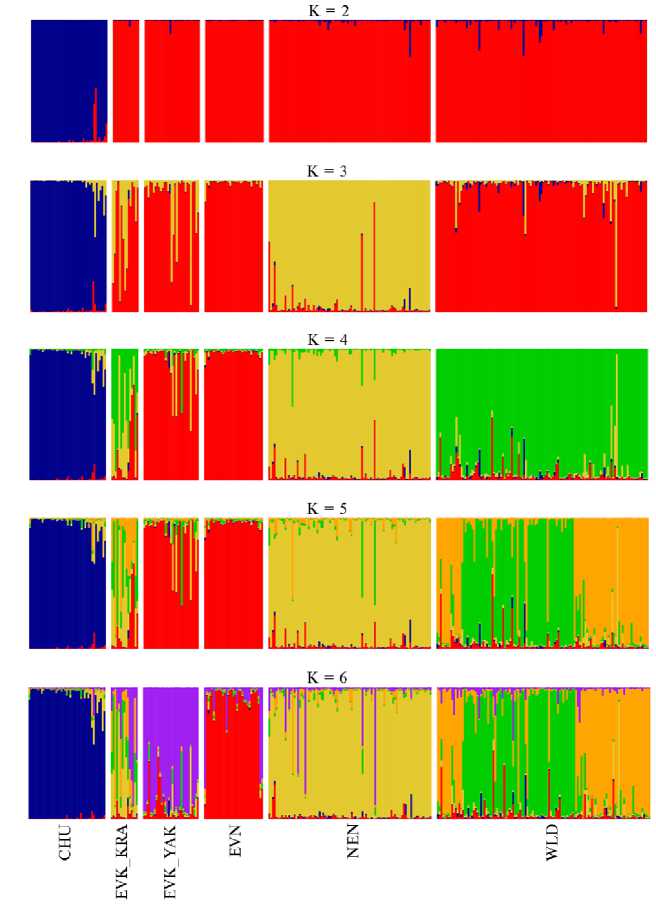
Рис. 6. Результаты кластерного анализа популяций домашних и диких северных оленей ( Rangifer tarandus L., 1758) на основе полиморфизма 9 микросателлитных локусов, проведенного с использованием программы STRUCTURE 2.3.4 для разного числа кластеров (К = 2, К = 3, К = 4, К = 5, К = 6) : CHU — чукотская порода ( n = 22), EVK_KRA — эвенкийская красноярская ( n = 12), EVK_YAK — эвенкийская якутская ( n = 14), EVN — эвенская ( n = 21), NEN — ненецкая ( n = 21), WLD — дикие олени ( n = 21) (2022 год).
Результаты РСА продемонстрировали генетическую дифференциацию между породами и объединили оленей в кластеры, соответствующие аналогичным на филогенетическом дереве (см. рис. 4). Все исследуемые популяции северного оленя показали конвергентный характер генетического состава, образовав пересекающиеся кластеры. Вклад в общую генетическую изменчивость, приходящийся на первую, вторую и третью главные компоненты, составил соответственно 2,969; 2,474 и 2,072 %. Генетические дифференциации по первым двум и по первой и третьей главным компонентам были схожи между собой. PC1 отделила CHU и NEN от диких популяций. Особи EVK_YAK вместе с EVN отделились от остальных животных по PC2. Все исследуемые животные были отнесены к оси PC3.
Несмотря на то, что алгоритм, основанный на величине ΔК, выявил оптимальное число кластеров для всей анализируемой выборки северного оленя, равное 2 (K = 2, ΔK = 136,79), использование К = 4, К = 5 и К = 6 при кластерном анализе также оказалось результативным.
При К = 2 были выявлены два основных генетических пула: первый состоял из трех пород — эвенской, эвенкийской и ненецкой, а также представителей дикой популяции, второй был образован лишь особями чукотской породы. При К = 3 мы обнаружили четкое отделение NEN от остальных популяций. При К = 4 происходило отделение EVK_KRA, а также диких особей. При К = 5 наблюдалось разделение диких оленей на две основные группы с элементами генетических примесей домашних пород. Также при К = 5 происходило разделение популяции WLD, что предположительно может быть связано с большими территориями обитания выбранных диких особей. EVN отделялась от EVK_YAK лишь при К = 6. Стоит отметить, что EVK_KRA демонстрировала полное смешение с представителями всех популяций, кроме CHU.
Для коренных народов Заполярного Севера России северный олень играет важную биологическую роль, поскольку служит источником пищи, одежды и крова, а также средством передвижения (1). В мире изучение генетического разнообразия северного оленя проводится с использованием комбинации нескольких маркеров (20-22). Однако российские популяции северного оленя на сегодняшний день охарактеризованы лишь по одному типу маркеров. В проделанной нами работе для исследования домашних и диких северных оленей впервые был использован комплексный подход на основе анализа полиморфизма CytB мтДНК и микросателлитных локусов. Гаплотипическое и нуклеотидное разнообразие в российских популяциях северных оленей (H D = 0,519-0,997; π = 0,002-0,006) было сопоставимо со значениями, полученными в предыдущих исследованиях российских и норвежских оленей (H D = 0,570-0,978; π = 0,002-0,019) (1), популяций Аолугуя из Китая (H D = 0,468; π = 0,0017) (22), а также северных оленей Канады (Hd = 0,890; π = 0,0022) (1), что свидетельствует об адекватности нашего подхода к расчетам показателей.
Полученные значения наблюдаемой гетерозиготности (uHE = 0,6810,841) были близки к данным, полученным в других работах на российских популяциях северных оленей, — HE = 0,670 (14), HE = 0,62-0,73 (16), He = 0,699 (15), He = 0,6491-0,7608 (13) и в популяциях оленей, обитающих на территории Китая, — HE = 0,650 (22). Аллельное разнообразие AR= 6,1888,760 также было сопоставимо с результатами других авторов — Ar = 5,7307,070 (14), Ar = 3,700-7,400 (21). Анализ структуры генетической сети показал дифференциацию чукотской популяции оленей от остальных, что было продемонстрировано в работе V.R. Kharzinova с соавт. (17), которые изучали генетическое разнообразие северного оленя с использованием анализа одиночных нуклеотидных последовательностей. В исследованиях других авторов, как и в настоящей работе, популяции диких оленей характеризовались более высоким генетическим разнообразием в сравнении с домашними. Выявленная закономерность обусловлена, скорее всего, двумя факторами: селекционной работой с домашними породами северного оленя и миграциями дикой популяции, обеспечивающими более интенсивный обмен гене- тическим материалом.
Таким образом, проведенный нами молекулярно-генетический анализ четырех пород домашнего северного оленя, а также тундровой популяции дикого северного оленя с использованием комбинации нескольких маркеров показал, что все выборки животных характеризовались высоким генетическим разнообразием. При этом по параметрам генетического разнообразия выборка тундровых диких северных оленей превосходила домашнюю популяцию, представленную особями ненецкой, чукотской, эвенской пород, а также красноярской и якутской популяций эвенкийской породы. В результате проведенного филогенетического анализа нуклеотидных последовательностей митохондриального гена CytB не было выявлено обособленной генетической структуры среди популяций северного оленя. Однако в исследуемой выборке наблюдалось четкое расхождение групп оленей на два основных кластера, что свидетельствует об общем происхождении животных по материнской линии внутри одного кластера. Все статистические подходы, примененные при анализе генетической структуры исследуемой выборки северных оленей с использованием микросателлитов (анализ главных компонент, филогенетический и кластерный анализ), выявили четкую генетическую дифференциацию домашних и диких северных оленей. Полученные результаты важны как для повышения эффективности селекционно-племенной работы в оленеводстве, так и в качестве основ для рекомендаций по природопользованию и охране дикого северного оленя — важнейшего промыслового ресурса, традиционного для коренных народов Крайнего Севера.
Список литературы Изучение генетического разнообразия домашних и диких популяций северного оленя (Rangifer tarandus L., 1758) с использованием маркеров ядерного и митохондриального геномов
- Kvie K.S., Heggenes J., Anderson D.G., Kholodova M.V., Sipko T., Mizin I., Røed K.H. Colo-nizing the high arctic: mitochondrial DNA reveals common origin of Eurasian archipelagic reindeer (Rangifer tarandus). PLoS ONE, 2016, 11(11): e0165237 (doi: 10.1371/journal.pone.0165237).
- Røed K.H., Flagstad Ø., Nieminen M., Holand Ø., Dwyer M.J., Røv N., Vilà C. Genetic analyses reveal independent domestication origins of Eurasian reindeer. Proc. R. Soc. B., 2008, 275(1645): 1849-1855 (doi: 10.1098/rspb.2008.0332).
- Baskin L.M. Number of wild and domestic reindeer in Russia in the late 20th century. Rangifer; 2005, 25(1): 51-57 (doi: 10.7557/2.25.1.337).
- Banfield A.W.F. A revision of the reindeer and caribou genus Rangifer. Ottawa, National Museums of Canada, 1961.
- Kvie K.S., Heggenes J., Røed K.H. Merging and comparing three mitochondrial markers for phylogenetic studies of Eurasian reindeer (Rangifer tarandus). Ecology and Evolution, 2016, 6(13): 4347-4358 (doi: 10.1002/ece3.2199).
- Dmitriev N.G., Ernst L.K. Animal genetic resources of the USSR. Rome, FAO, 1989.
- Conner J.K., Hartl D.L. A primer of ecological genetics. Sinauer Associates, Inc., Sunderland, MA, 2004.
- Abdelmanova A.S., Dotsev A.V., Sermyagin A.A., Brem G.G., Zinovieva N.A. Assessment of the genetic resources of Russian local cattle breeds by genome-wide SNP analysis. Journal of Animal Science, 2021, 99(S3): 225 (doi: 10.1093/jas/skab235.410).
- Денискова Т.Е., Селионова М.И., Гладырь Е.А., Доцев А.В., Бобрышова Г.Т., Костю-нина О.В., Брем Г., Зиновьева Н.А. Изменчивость микросателлитов в породах овец, разводимых в России. Сельскохозяйственная биология, 2016, 51(6): 801-810 (doi: 10.15389/agrobiology.2016.6.801rus).
- Кошкина О.А., Денискова Т.Е., Доцев А.В., Kunz E., Upadhyay M., Krebs S., Соловь-ева А.Д., Medugorac I., Зиновьева Н.А. Оценка материнской изменчивости российских локальных пород овец на основе анализа полиморфизма гена цитохрома B. Сельскохозяй-ственная биология, 2021, 56(6): 1134-1147 (doi: 10.15389/agrobiology.2021.6.1134rus).
- Barbanera F., Guerrini M., Beccani C., Forcina G., Anayiotos P., Panayides P. Conservation of endemic and threatened wildlife: molecular forensic DNA against poaching of the Cypriot mouf-lon (Ovis orientalis ophion, Bovidae). Forensic Science International: Genetics, 2012, 6(5): 671-675 (doi: 10.1016/j.fsigen.2011.12.001).
- Rodionov A., Deniskova T., Dotsev A., Volkova V., Petrov S., Kharzinova V., Koshkina O., Abdelmanova A., Solovieva A., Shakhin A., Bardukov N., Zinovieva N. Combination of multiple microsatellite analysis and genome-wide SNP genotyping helps to solve wildlife crime: a case study of poaching of a Caucasian tur (Capra caucasica) in Russian Mountain National Park. Animals, 2021, 11(12): 3416 (doi: 10.3390/ani11123416).
- Zhai J.-C., Liu W.-S., Yin Y.-J., Xia Y.-L., Li H.-P. Analysis on genetic diversity of reindeer (Rangifer tarandus) in the Greater Khingan Mountains using microsatellite markers. Zoological Studies, 2017, 56: e11 (doi: 10.6620/ZS.2017.56-11).
- Денискова Т.Е., Харзинова В.Р., Доцев А.В., Соловьева А.Д., Романенко Т.М., Южа-ков, А.А., Лайшев К.А., Брем Г., Зиновьева Н.А. Генетическая характеристика региональ-ных популяций ненецкой породы северного оленя (Rangifer tarandus). Сельскохозяйствен-ная биология, 2018, 53(6): 1152-1161 (doi: 10.15389/agrobiology.2018.6.1152rus).
- Kharzinova V.R., Dotsev A.V., Solovieva A.D., Fedorov V.I., Shimit L.D.O., Romanenko T.M., Senchik A.V., Sergeeva O.K., Goncharov V.V., Laishev K.A., Yuzhakov A.A., Brem G.G., Zi-novieva N.A. Genetic variability of Russian domestic reindeer populations (Rangifer tarandus) by mi-crosatellites. Journal of Animal Science, 2020, 98(4): 237-238 (doi: 10.1093/jas/skaa278.435).
- Столповский Ю., Бабаян О., Kashtanov S., Piskunov A., Сёмина М., Kholodova M., Лайшев К., Южаков А., Романенко Т., Лисичкина М., Дмитриева Т., Етылина О., Прокудин А., Свищёва Г. Генетическая оценка пород северного оленя (Rangifer tarandus) и их дикого предка с помощью новой панели STR-маркеров. Генетика, 2020, 56(12): 1410-1426 (doi: 10.31857/S0016675820120139).
- Kharzinova V.R., Dotsev A.V., Deniskova T.E., Solovieva A.D., Fedorov V.I., Layshev K.A., Romanenko T.M., Okhlopkov I.M., Wimmers K., Reyer H., Brem G., Zinovieva N.A. Genetic diversity and population structure of domestic and wild reindeer (Rangifer tarandus L. 1758): a novel approach using BovineHD BeadChip. PLoS ONE, 2018, 13(11): e0207944 (doi: 10.1371/journal.pone.0207944).
- Gissi C., Iannelli F., Pesole G. Evolution of the mitochondrial genome of Metazoa as exemplified by comparison of congeneric species. Heredity, 2008, 101(4): 301-320 (doi: 10.1038/hdy.2008.62).
- Wilkerson C.D., Mahoney S.P., Carr S.M. Post-glacial recolonization of insular Newfoundland across the Strait of Belle Isle gave rise to an endemic subspecies of woodland caribou, Rangifer tarandus terranovae (Bangs, 1896): evidence from mtDNA haplotypes. Genome, 2018, 61(8): 575-585 (doi: 10.1139/gen-2017-0199).
- Cronin M.A., Macneil M.D., Patton J.C. Variation in mitochondrial DNA and microsatellite DNA in caribou (Rangifer Tarandus) in North America. Journal of Mammalogy, 2005, 86(3): 495-505 (doi: 10.1644/1545-1542(2005)86[495:VIMDAM]2.0.CO;2).
- Cronin M.A., Macneil M.D., Patton J.C. Mitochondrial DNA and microsatellite DNA variation in domestic reindeer (Rangifer tarandus tarandus) and relationships with wild caribou (Rangifer tarandus granti, Rangifer tarandus groenlandicus, and Rangifer tarandus caribou). The Journal of heredity, 2006, 97(5): 525-530 (doi: 10.1093/jhered/esl012).
- Ju Y., Liu H., Rong M., Zhang R., Dong Y., Zhou Y., Xing X. Genetic diversity and population genetic structure of the only population of Aoluguya Reindeer (Rangifer tarandus) in China. Mi-tochondrial DNA Part A, 2019, 30(1): 24-29 (doi: 10.1080/24701394.2018.1448081).
- Edgar R.C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research, 2004, 32(5): 1792-1797 (doi: 10.1093/nar/gkh340).
- Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874 (doi: 10.1093/mol-bev/msw054).
- Leigh J.W., Bryant D. Popart: Full-feature software for haplotype network construction. Methods in Ecology and Evolution, 2015, 6(9): 1110-1116 (doi: 10.1111/2041-210X.12410).
- Lanfear R., Frandsen P.B., Wright A.M., Senfeld T., Calcott B. PartitionFinder 2: New methods for selecting partitioned models of evolution for molecular and morphological phylogenetic anal-yses. Molecular Biology and Evolution, 2017, 34(3): 772-773 (doi: 10.1093/molbev/msw260).
- Akaike H. A new look at the statistical model identification. IEEE Transactions on Automatic Control, 1974, 19(6): 716-723 (doi: 10.1109/TAC.1974.1100705).
- Excoffier L., Lischer H.E. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 2010, 10(3): 564-567 (doi: 10.1111/j.1755-0998.2010.02847.x).
- Ronquist F., Teslenko M., van der Mark P., Ayres D.L., Darling A., Höhna S., Larget B., Liu L., Suchard M.A., Huelsenbeck J.P. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology, 2012, 61(3): 539-542 (doi: 10.1093/sysbio/sys029).
- Molecular evolution, phylogenetics and epidemiology. Режим доступа: http://tree.bio.ed.ac.uk/soft-ware/figtree. Дата обращения: 30.06.2022.
- Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C., Guirao-Rico S., Librado P., Ramos-On-sins S.E., Sánchez-Gracia A. DnaSP 6: DNA sequence polymorphism analysis of large data sets. Molecular Biology and Evolution, 2017, 34(12): 3299-3302 (doi: 10.1093/molbev/msx248).
- Kharzinova V.R., Gladyr’ E.A., Fedorov V.I., Romanenko T.M., Shimit L.D., Layshev K.A., Kalashnikova L.A., Zinovieva N.A. Development of multiplex microsatellite panel to assess the parentage verification in and differentiation degree of reindeer populations (Rangifer tarandus). Agricultural Biology, 2015, 50(6): 756-765 (doi: 10.15389/agrobiology.2015.6.756eng).
- Keenan K., McGinnity P., Cross T.F., Crozier W.W., Prodöhl P.A. diveRsity: An R package for the estimation and exploration of population genetics parameters and their associated errors. Methods in Ecology and Evolution, 2013, 4(8): 782-788 (doi: 10.1111/2041-210X.12067).
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evo-lution, 1984, 38(6): 1358-1370 (doi: 10.1111/j.1558-5646.1984.tb05657.x).
- Jost L. GST and its relatives do not measure differentiation. Molecular Ecology, 2008, 17(18): 4015-4026 (doi: 10.1111/j.1365-294X.2008.03887.x).
- Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution, 2006, 23(2): 254-267 (doi: 10.1093/molbev/msj030).
- Jombart T. adegenet: a R package for the multivariate analysis of genetic markers. Bioinformatics, 2008, 24(11): 1403-1405 (doi: 10.1093/bioinformatics/btn129).
- Wickham H. ggplot2: Elegant graphics for data analysis. Springer-Verlag, NY, 2009.
- Pritchard J.K. Stephens M., Donnelly P. Inference of population structure using multilocus gen-otype data. Genetics, 2000, 155(2): 945-959 (doi: 10.1093/genetics/155.2.945).
- Earl D.A., von Holdt B.M. Structure Harvester: A website and program for visualizing Structure output and implementing the Evanno method. Conservation Genetics Resources, 2012, 4: 359-361 (doi: 10.1007/s12686-011-9548-7).
- Evanno G., Regnaut S., Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: A simulation study. Molecular Ecology, 2005, 14(8): 2611-2620 (doi: 10.1111/j.1365-294X.2005.02553.x).
- R Core Team. R: A language and environment for statistical computing. R Foundation for statistical computing. Vienna, Austria, 2012. Режим доступа: https://www.semanticscholar.org/paper/R%3A-A-language-and-environment-for-statistical-Team/659408b243cec55de8d0a3bc51b81173007aa89b. Без даты.


