Изучение генома свиней (Sus scrofa) с использованием ДНК-маркеров
Автор: Гладырь Е.А., Эрнст Л.К., Костюнина О.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Научная школа академика Л.К. Эрнста: исследования и перспективы (к 80-летию ученого)
Статья в выпуске: 2 т.44, 2009 года.
Бесплатный доступ
Провели экспериментальные исследования генетического полиморфизма пяти популяций двух подвидов кабанов, обитающих на территории Российской Федерации, и пород свиней крупная белая, йоркшир, дюрок и ландрас. Рассмотрены некоторые аспекты изучения генотипа дикого кабана в сравнении с домашними свиньями разных пород на примере анализа микросателлитов и маркерных генов.
Генетический полиморфизм, гены-кандидаты, генетика популяций, микросателлиты, породы свиней, кабаны
Короткий адрес: https://sciup.org/142133235
IDR: 142133235 | УДК: 636.4:575.174.015.3:575.224.22
Текст научной статьи Изучение генома свиней (Sus scrofa) с использованием ДНК-маркеров
Дикие свиньи, или кабаны, благодаря крепкой конституции, высокой устойчивости к болезням, прекрасной приспособленности к условиям существования, долголетию и другим ценным признакам, служат постоянным источником генетических ресурсов при выведении новых пород (2).
Свинья особенно интересна в эволюционных исследованиях не только потому, что существующие породы характеризуются большим фенотипическим различием по морфологии, физиологии и поведению, но также в силу того, что до настоящего времени сохранилось достаточное число особей и подвидов диких предков одомашненных свиней (3). Свинья ( Sus scrofa domesticus ) была одомашнена от Sus scrofa , или дикого кабана, приблизительно 9 тыс. лет назад в различных регионах мира (4-6). Генетические исследования, проведенные группой G. Larson с соавт. (7), показали, что одомашнивание свиньи — одно из ключевых событий «неолитической революции» — впервые было осуществлено на Ближнем Востоке. Согласно генетическому анализу, первые домашние свиньи попали в Европу из Азии, однако вскоре после этого европейцы одомашнили и местного дикого кабана. Впоследствии потомство европейского кабана полностью вытеснило мигрировавшую ближневосточную популяцию. Анализ мтДНК показал, что современная граница ареалов этих двух популяций совпадает с границей между Европой и Азией: в Турции живут азиатские ближневосточные, в Греции, Болгарии и Румынии — уже европейские кабаны.
В 2006 году был создан консорциум по исследованию генома свиньи, а также начат глобальный проект по его секвенированию (SGSC) . В дополнение к получаемой важной информации об эволюционном развитии вида Sus scrofa этот уникальной проект позволяет идентифицировать гены или геномные области локусов количественных признаков (QTLs), что будет способствовать пониманию молекулярных механизмов, управляющих фенотипами, поможет объединить традиционные и биоинформационные методы для решения вопросов повышения устойчивости животных к заболеваниям и оперативного реагирования на изменяющиеся требования рынка (3).
В настоящее время молекулярные методы анализа генотипа очень широко используются в характеристике диких видов животных и практическом животноводстве многих стран. Используя их, можно решать проблемы генетической дифференциации пород и подвидов кабанов, выявлять или уточнять таксономические отношения между различными подвидами диких свиней, а также устанавливать степень родства между дикими и домашними свиньями (2).
Для характеристики генотипа животных используют как микросателлиты, так и селекционно значимые SNPs (single nucleotide polymorphism). Микросателлиты представляют собой короткие (по разным данным, от 100 до 350 п.н.) тандемно расположенные участки ДНК, обладающие высокой степенью полиморфизма (8, 9). Эти маркеры известны под несколькими названиями: микросателлиты, STMS (sequence tagged microsattelite site), STR (short tandem repeat), SSR (simple sequence repeat) (8, 10, 11). Их применяют для определения степени гетерозиготности малочисленных популяций, пород, консолидированности линий и групп, раннего прогнозирования потенциала продуктивности животных. Из-за высокой степени полиморфизма микросателлитные маркеры часто используют при оценке генетических расстояний между различными популяциями домашних свиней и их дикими предками (12-14) . В научной литературе представлены результаты целого ряда исследований, посвященных молекулярно-генетической характеристике популяций кабанов, обитающих в Азии (15, 16) Америке (17), Западной Европе (18-20), Австралии и Папуа Новой Гвинее (21).
Для оценки генетического потенциала свиней непосредственно на уровне генотипа и для характеристики различных пород вида Sus scrofa проводятся эксперименты по выявлению информативных SNPs и разработке систем ДНК-анализа генов, влияющих на проявление селекционно и экономически значимых признаков. Согласно общепринятому определению, SNPs — это однонуклеотидные позиции в геномной ДНК, для которых в популяции имеются различные варианты последовательностей (аллели) с частотой редкого аллеля не менее 1 % (22). M. Ernst с соавт. (23) изучали внутривидовой и межвидовой генетический полиморфизм генов локусов количественных признаков у диких разновидностей Sus scrofa scrofa и Sus scrofa аttila , обитающих на территории Хорватии.
Целью настоящей работы было изучение внутривидового полиморфизма 12 микросателлитных маркеров и 7 генов локусов количественных признаков для сравнительной генетической характеристики кабанов пяти популяций, обитающих на территории Российской Федерации, и домашних свиней четырех основных пород России.
Методика. Молекулярно-генетические исследования выполняли в 2007-2008 годах на биологическом материале кабанов, полученных из охотоведческих хозяйств России, и чистопородных свиньях. В эксперименте были исследованы 38 кабанов пяти популяций: Кировской ( n = 15), Ярославской ( n = 6), Оренбургской ( n = 2) областей, Краснодарского ( n = 4) и Хабаровского краев ( n = 11), а также свиньи пород крупная белая ( n = 60, 6 гнезд), дюрок ( n = 52, 5 гнезд), йоркшир ( n = 66, 5
гнезд) (ЗАО «СВ-Поволжское», ПЗ «Гибридный») и ландрас ( n = 54) (ЗАО ПЗ «им. В.Н. Цветкова»).
У животных исследуемых групп отбирали пробы ткани (ушные выщипы) для выделения ДНК. Использовали колонки NexttecTM (Германия) и набор реагентов DIAtomTM DNA Prep100 (ООО «Биоком», Россия) согласно рекомендациям фирм-изготовителей. Выделение ДНК и постановку полимеразной цепной реакции (ПЦР) проводили, как описано (24).
Выбор микросателлитных локусов осуществляли в соответствии с рекомендациями ISAG (Международное общество генетики животных). Электрофоретическое разделение фрагментов ДНК методом капиллярного электрофореза проводили на приборе ABI 3130xl Genetic Analyzer («Applied Biosystems», США). Для идентификации аллелей исследованных локусов микросателлитов использовали программу Gene Mapper v. 4.0. Данные об аллелях каждого животного суммировали в электронной таблице Microsoft Excel. Полученная матрица генотипов служила основой для статистической обработки результатов.
Все исследуемые кабаны тестировались по 7 генам ( ESR , FSHB , NCOA1 , EPOR , MUC4 , ECR , RYR ), являющимся маркерами плодовитости и устойчивости к заболеваниям. Исследования выполнялись при помощи оригинальных тест-систем ДНК-анализа, разработанных в Центре биотехнологии Всероссийского НИИ животноводства.
Статистическую обработку данных проводили по стандартным методикам (25-27) с использованием программного обеспечения MS Excel, MSA_WIN v. 2.65, GenAlEx 6.0, Phylip.
Результаты. При молекулярно-генетических исследованиях 38 кабанов и 232 домашних свиней по 12 микросателлитным маркерам Sus scrofa (S0155, S0355, S0386, SW24, SW72, SW951, S0101, SW240, SW857, S0228, SW911 и SW936) оказалось, что во всех исследованных популяциях в совокупности диагностировалось 125 аллелей с частой встречаемости от 0,8 (SW240 и SW951) до 100 % (S0355, SW936 и SW951). Последнее, вероятно, объясняется малыми выборками, представленными кабанами Ярославской, Оренбургской областей и Краснодарского края. Максимальное число аллелей обнаружили в локусе SW24 — 17. Следует отметить, что больше половины микросателлитных маркеров, а именно 7 из 12, характеризовались наличием десяти и более аллелей, что указывает на высокий
1. Оценка полиморфизма микросателлитных маркеров в изучаемых группах домашних свиней и кабанов разных пород и популяций
ландрас (табл. 1).
Среднее число аллелей на локус в исследованных популяциях составило соответственно 5,00±0,46; 5,25±0,59 и 5,00±0,58. Вместе с тем анализ этого показателя в среднем по четырем породам домашних свиней выявил несколько более высокий уровень полиморфизма, чем у кабанов, и составил 4,06±0,34 против 3,88±0,44 аллеля на локус.
Микросателлиты, включенные в анализируемые панели, характеризовались 100 % уровнем полиморфизма. Число информативных аллелей с частотой встречаемости более 5 % у кабанов равнялось 3,13, что на 4,86 % ниже, чем у домашних свиней (см. табл. 1). Однако число эффективных аллелей в популяциях кабанов и домашних свиней было одинаково и составило в среднем 2,60. В исследуемых популяциях представителей вида Sus scrofa был выявлен 31 приватный аллель, то есть аллель, который встречался только у одной исследуемой нами популяции свиней. Подчеркнем, что распределение по породам и популяциям было неравномерным: 21 приватный аллель диагностировали у кабанов и только 10 — у домашних свиней. При этом наиболее полиморфными оказались кабаны, представляющие азиатскую популяцию и имеющие 16 приватных аллелей по восьми микросателлитным маркерам. Описанный факт, возможно, еще раз подтверждает гипотезу о различном происхождении европейского и азиатского кабана. Из домашних свиней наибольшим разнообразием характеризовалась популяция ландрасов, в которой выявлено пять приватных аллелей по трем микросателлитным маркерам с частотой встречаемости от 8,30 до 28,00 %. Относительно невысокое число приватных аллелей в культурных породах свиней можно объяснить тем, что мы анализировали близкородственных животных из гнезд-триад (мать—отец—потомки).
Установлено, что территориальная удаленность ареалов обитания проявляется в существенных генетических различиях, вследствие которых кабаны группировались в два кластера (рис. 1, А). К первому относились кабаны, обитающие в европейской части России, ко второму — животные с ареалом обитания в Сибири. Стандартная генетическая дистанция, рассчитанная по M. Nei (DSG), между европейской и азиатской популяциями составила 72,92 %.
А Б
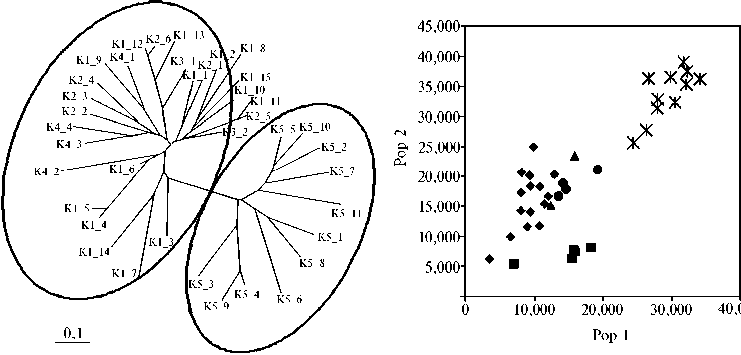
Рис. 1. Филогенетическое древо кабанов России (А) и популяционная принадлежность изучаемых животных (Б) : европейская популяция — Кировская (К1), Ярославская (К2), Оренбургская (К3) области, Краснодарский край (К4) (обозначены соответственно ромбами, квадратами, треугольниками и кружками), азиатская популяция — Хабаровский край (К5) (обозначена звездочками).
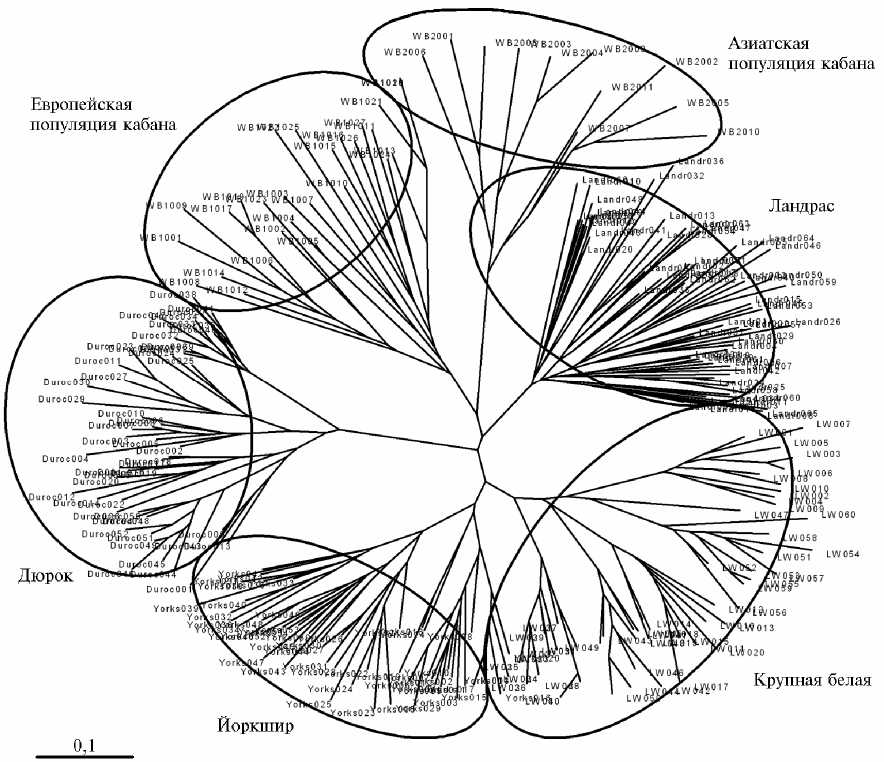
Рис. 2. Индивидуальное филогенетическое древо разных популяций кабанов России и пород домашних свиней (последние представлены гнездами: мать—отец—потомки) .
Результаты сравнительной оценки генетической принадлежности индивидуума к популяции по D. Peatkau (28) на основании анализа микросателлитов (см. рис. 1, Б) полностью подтвердили региональную принадлежность анализируемой выборки кабанов: в 100 % случаев животные принадлежали к собственной популяции.
Различия между региональными группами кабанов внутри европейской популяции находились на уровне 14,52-24,56 %, что согласуется с межпородным разнообразием среди культурных пород свиней.
Наибольшей генетической удаленностью от кабанов характеризовались свиньи пород йоркшир и крупная белая (табл. 2).
2. Генетические дистанции между домашними свиньями и кабанами разных пород и популяций, определенные по M. Nei (29)
|
Порода, популяция |
1 КБ |
1 Д 1 |
Й 1 |
Л |
1 К1 1 |
К2 |
К3 |
К4 |
1 К5 |
|
Крупная белая (КБ) |
1,000 |
||||||||
|
Дюрок (Д) |
0,500 |
1,000 |
|||||||
|
Йоркшир (Й) |
0,710 |
0,433 |
1,000 |
||||||
|
Ландрас (Л) |
0,525 |
0,453 |
0,516 |
1,000 |
|||||
|
К1 (Кировская область) |
0,355 |
0,359 |
0,283 |
0,553 |
1,000 |
||||
|
К2 (Ярославская область) |
0,280 |
0,348 |
0,274 |
0,429 |
0,869 |
1,000 |
|||
|
К3 (Оренбургская область) |
0,303 |
0,356 |
0,280 |
0,485 |
0,833 |
0,847 |
1,000 |
||
|
К4 (Краснодарский край) |
0,365 |
0,360 |
0,312 |
0,416 |
0,765 |
0,812 |
0,716 |
1,000 |
|
|
К5 (Хабаровский край) |
0,186 |
0,171 |
0,254 |
0,258 |
0,280 |
0,217 |
0,193 |
0,165 |
1,000 |
Как известно, при выведении породы ландрас активно использовался генофонд дикого кабана (2, 30). Полученные нами данные молекулярных исследований полностью согласуются с исторической закономерностью формирования этой породы.
Индивидуальное сравнение изучаемых популяций кабанов с наиболее распространенными породами свиней России — крупной белой, ланд-рас, дюрок и йоркшир показало обособленное распределение животных в кластеры, обусловленное их породной и индивидуально-родственной принадлежностью, при этом различные популяции кабанов — азиатская и европейская распределились в два независимых кластера (рис. 2).
Наблюдаемый уровень гетерозиготности (Hо) среди изучаемых пород и популяций (табл. 3) находился в пределах от 45,03 % (дюрок) до 56,94 % (йоркшир) среди культурных пород свиней и от 46,30 % в кировской популяции кабанов до 66,97 и 72,22 % соответственно в азиатской и оренбургской. Это свидетельствует о достаточном генетическом разнообразии особей в исследованных популяциях.
3. Показатели гетерозиготности в исследуемых группах Sus scrofa
|
Порода, популяция |
1 Наблюдаемая, Ho |
Ожидаемая, He |
Индекс фиксации, F |
|
Крупная белая |
0,5542±0,0400 |
0,5804±0,0300 |
0,0440 |
|
Дюрок |
0,4503±0,0600 |
0,5062±0,0600 |
0,1053 |
|
Йоркшир |
0,5694±0,0600 |
0,5722±0,0500 |
- 0,0036 |
|
Ландрас |
0,5467±0,0500 |
0,5856±0,0500 |
0,0656 |
|
К1 (Кировская область) |
0,4630±0,0700 |
0,5959±0,0600 |
0,1980 |
|
К2 (Ярославская область) |
0,5347±0,0800 |
0,5424±0,0700 |
- 0,0068 |
|
К3 (Оренбургская область) |
0,7222±0,1200 |
0,5278±0,0300 |
- 0,3267 |
|
К4 (Краснодарский край) |
0,6136±0,1100 |
0,5824±0,0700 |
- 0,0368 |
|
К5 (Хабаровский край) |
0,6697±0,0800 |
0,6376±0,0400 |
- 0,0253 |
Несмотря на достаточно высокий уровень наблюдаемой гетерозиготности, в ряде пород и популяций выявлен дефицит гетерозигот, что проявляется в положительных значениях индекса фиксации (F).
Использовав разработанную D. Paetkau с соавт. (31) модель расчета ожидаемой частоты встречаемости генотипа в каждом из локусов и принимая во внимание случайный характер спаривания особей внутри популяции, получили логарифмическое значение правдоподобия. Его рассчи- тывали по каждой популяции на основании полученной для нее частоты встречаемости аллелей (рис. 3, А). Анализируемые породы домашних свиней образовали четкий общий кластер, в который вошли и кабаны азиатской популяции (см. рис. 3, Б). Свиньи с общей историей породообразова-ния находятся в непосредственной филогенетической близости друг от дру- га и характеризуются минимальными генетическими дистанциями, например крупная белая и йоркшир (32). Кабаны как родоначальники всех культурных пород свиней занимают центральное положение. Следует отметить, что все животные были отнесены к собственной популяционной группе.
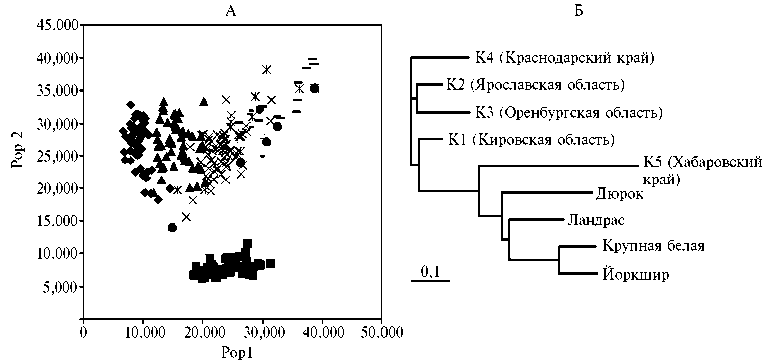
Рис. 3. Популяционная принадлежность исследуемых животных (А) и дендрограмма филогенетического родства кабанов и свиней (Б) : породы свиней крупная белая, дюрок, йоркшир и ланд-рас (обозначены соответственно ромбами, квадратами, треугольниками и косыми крестиками); популяции кабанов К1, К2, К3, К4 и К5 (обозначены соответственно звездочками, кружками, прямыми крестиками, короткими и длинными черточками).
Работы по изучению групп крови и полиморфных систем у дикого кабана и домашней свиньи проводятся уже не одно десятилетие как у нас в стране (2), так и в мире (33). В 2000 году группой E. Miller с соавт. (34) впервые было выполнено исследование европейских кабанов по гену RYR1 , а спустя год N. Okumura с соавт. (35) сообщили об анализе генетических различий между азиатским кабаном и домашними свиньями по мтДНК.
В рамках работ по генотипированию Sus scrofa различных пород и популяций мы изучили 38 кабанов из разных популяций по 7 селекционно значимым генам (табл. 4). Как в европейской, так и в азиатской популяции были диагностированы желательные аллели В генов эстрогенового рецептора и β -субъединицы фолликулостимулирующего гормона, ассоциированные с повышенным многоплодием. Так, М. Rothshild с соавт. (36) в 1996 году установили, что свиноматки с генотипом BB гена ESR имели размер гнезда в первом опоросе на 2,3 поросенка больше, в среднем по трем опросам — на 1,5 поросенка больше по сравнению с животными с генотипом AA (p < 0,001). Считается, что источником аллеля В гена эстрогенового рецептора в популяциях свиней России и мира служили азиатские кабаны и свиньи. Мы установили, что этот желательный аллель встречается как в европейской, так и в азиатской популяции кабанов с высокой частотой (соответственно 27,78 и 36,36 %), а частота встречаемости аллеля В гена FSHB составляет от 75,93 % в европейской до 81,82 % в азиатской популяциях, что существенно выше величины, полученной M. Ernst с соавт. (23) (в их исследовании 109 особей европейского кабана частота аллеля В составляла 56 %).
4. Генотипы и аллельные частоты генов QTL в исследуемых популяциях кабанов
|
Популяция |
Ген |
n |
Частота встречаемости, % |
Описание |
|
|
генотипов |
аллелей |
||||
|
Европейская |
ESR |
27 |
AA AB BB 59,26 25,93 14,81 |
A B 72,22 27,78 |
Ген эстрагенового рецептора: аллель А — нежелательный, |
|
Азиатская |
11 |
AA AB * 27,27 72,73 |
A B 63,64 36,36 |
аллель B — желательный (плодовитость) |
|
|
Европейская |
FSHB |
27 |
AA AB BB 14,81 18,52 66,67 |
A B 24,07 75,93 |
Ген β -субъединицы фолликулостимулирующего гормона: |
|
Азиатская |
11 |
AA AB BB 9,09 18,18 72,73 |
A B 18,18 81,82 |
аллель A — нежелательный, аллель B — желательный (плодовитость) |
|
|
Европейская |
EPOR |
27 |
CC * * 100 |
C T 100 0 |
Ген эритропоэтинового рецептора: аллель С — нежела- |
|
Азиатская |
11 |
CC * * 100 |
C T 100 0 |
тельный, аллель T — желательный (плодовитость) |
|
|
Европейская |
NCOA1 |
27 |
A1A1 A1A2 * 88,89 11,11 |
A1 A2 94,44 5,56 |
Ген коактиватора ядерных рецепторов: аллель A1 — же- |
|
Азиатская |
11 |
A1A1 * * 100 |
A1 A2 100 0 |
лательный (плодовитость), аллель A2 — нежелательный |
|
|
Европейская |
MUC4 |
27 |
CC CG * 92,59 7,41 |
C G 96,30 3,70 |
Ген устойчивости к Escherichia coli K88 (муцин 4, устойчи- |
|
Азиатская |
11 |
CC * * 100 |
C G 100 0 |
вость к диарее): аллель С — желательный, аллель G — нежелательный |
|
|
Европейская |
ECR |
27 |
GG * * 100 |
G A 100 0 |
Ген рецептора Escherichia coli (устойчивость к диарее): |
|
Азиатская |
11 |
GG * * 100 |
G A 100 0 |
аллель A — желательный, аллель G — нежелательный |
|
|
Европейская |
RYR1 |
27 |
NN * * 100 |
N n 100 0 |
Ген рианодинового рецептора (стрессоустойчивость): аллель |
|
Азиатская |
11 |
NN * * 100 |
N n 100 0 |
N — желательный, аллель n — нежелательный |
|
* Отсутствие генотипа в исследуемой популяции животных.
Комплекс NCOA1 взаимодействует с эстрогеновым рецептором, стимулируя его транскрипционную активность и обусловливая, тем самым, последующий физиологический ответ. Проведенный J. Melville с со-авт. (37) в 2002 году анализ помесных свиней пород крупная белая и мэй-шан показал превосходство свиноматок с генотипом А1А1 и гетерозиготным генотипом над животными с генотипом А2А2 по величине приплода (соответственно на 1,82 и 0,90 поросенка за опорос). В наших исследованиях желательный аллель А1 гена NCOA1 был выявлен у подавляющего большинства кабанов обеих популяций. Частота встречаемости этого аллеля варьировала от 94,44 % в европейской популяции кабанов до 100 % у кабанов Хабаровского края. Последний ген из блока плодовитости — ген эритропоэтинового рецептора ( EPOR ) оказался мономорфным по нежелательному аллелю С .
В свиноводстве кишечные расстройства относятся к наиболее распространенным и составляют серьезную проблему, поскольку ежегодно во всем мире хозяйства несут большие потери поголовья от этих болезней. Известно, что возбудителями, ответственными за возникновение диареи у молодняка свиней, являются энтеротоксичные штаммы Escherichia coli (ETEC), чаще всего E. coli с типами фимбрий F18 и F4 (K88) (38). Патология развивается после того, как ETEC попадают в кишечный тракт поросят. Бактерии ETEC прикрепляются к стенкам тонкого кишечника через поверхностные белковые антигены, их число увеличивается и токсины поступают непосредственно в клетки кишечного эпителия. В результате действия токсинов прекращается жидко-абсорбирующая деятельность эпителиальных клеток кишечника, что приводит к диарее (39). В основе генетической устойчивости животных к диарее лежит отсутствие на поверхности клеток кишечника соответствующих рецепторов. В качестве генов-кандидатов, принимающих участие во взаимодействии ETEC и кишечных рецепторов, рассматриваются MUC4 и ECR. В основе полиморфизма гена MUC4 лежит точечная мутация, при этом предполагается, что желательным с точки зрения устойчивости к диарее является аллель С (40). В изученных популяциях кабанов частота встречаемости желательного аллеля была максимальной и составляла от 96,30 % у европейского кабана до 100 % у азиатского. Второй ген из блока устойчивости к ETEC — ген рецептора E. сoli F18 (ECR F18). Установлено тесное сцепление этого гена с геном α-1-фукозилтрансферезы (FUT1). В гене FUT1 выявлен полиморфизм, причиной которого служит точечная мутация А→G в позиции 307. Показано, что поросята, имеющие генотип GG и AG, предположительно восприимчивы к колибактериозу, а животные с генотипом АА — устойчивы. В исследованных популяциях кабанов мы не обнаружили желательный аллель А этого гена.
Ранее сообщалось об идентификации одного гетерозиготного генотипа гена RYR1 в европейской популяции кабанов (41). В наших исследования все особи исследуемых популяций были гомозиготными по устойчивому к стрессу аллелю N .
Таким образом, выполненная работа показала высокое генетическое разнообразие двух подвидов кабанов, обитающих на территории России, которое успешно можно диагностировать как посредством микросателлитов, так и с использованием генов-кандидатов QTL. Подтверждена универсальность системы ДНК-анализа генотипа свиней по 12 микроса-теллитным маркерам, разработанной и предложенной для оценки достоверности происхождения племенных животных вида Sus scrofa . Результаты исследований послужат основой для более глубокого изучения генотипа диких предков домашней свиньи и продолжат цикл экспериментов по анализу генофонда наиболее распространенного вида сельскохозяйственных животных — Sus scrofa .
Л И Т Е Р А Т У Р А
-
1. FAO SoW-AnGR. The State of the World's Animal Genetic Resources. 2006, Rome.
-
2. Г о р е л о в И.Г. Биология сибирского кабана. Новосибирск, 1994.
-
3. C h e n K., B a x t e r T., M u i r W.M. e.a. Genetic resources, genome mapping and evo
lutionary genomics of the pig ( Sus scrofa ). Int. J. Biol. Sci., 2007, 3: 153-165.
-
4. G i u f f r a E., K i j a s J.M., A m a r g e r V. e.a. The origin of the domestic pig: independent domestication and subsequent introgression. Genetics, 2000, 154(4): 1785-1791.
-
5. K i j a s J.M., A n d e r s s o n L. A phylogenetic study of the origin of the domestic pig estimated from the near-complete mtDNA genome. J. Mol. Evol. 2001, 52(3): 302-308.
-
6. L a r s o n G., D o b n e y K., A l b a r e l l a U. Worldwide phylogeography of wild boar reveals multiple centers of pig domestication. Science, 2005, 307: 1618-1621.
-
7. L a r s o n G., A l b a r e l l a U., D o b n e y K. e.a. Ancient DNA, pig domestication, and the spread of the Neolithic into Europe. PNAS USA, 2007, 104(39): 15276-15281.
-
8. T a u t z D. Hypervariability of simple sequences as a general source for polymorphic DNA markers. Nucl. Acids Res., 1989, 17: 6463-6471.
-
9. D e W o o d y J.A., A v i s e J.C. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. J. Fish Biol., 2000, 56: 461-473.
-
10. L i t t M., L u t y J.A. A hypervariable microsattellite revealed by in vitro amplification of a dinucleotide within the cardiac muscle action gene. Am. J. Hum. Genet., 1989, 44(3): 397-401.
-
11. W e b e r J.L., M a y P.E. Abundant class of human DNA polymorphisms which can be typed using the polymerase chain reaction. Am. J. Hum. Genet., 1989, 44(3): 388-396.
-
12. L a v a l G., I a n n u c c e l l i N., L e g a u l t C. e.a. Genetic diversity of eleven European pig breeds. Genet. Sel. Evol., 2000, 32: 187-203.
-
13. R a j e e v K., S i n g h A., V i j h R.K. e.a. Evaluation of the genetic variability of 13 mi-
crosatellite markers in native Indian pigs. J. Genet., 2001, 80: 149-153.
-
14. B e h l R., K a u l R., S h e o r a n N. e.a. Genetic identity of two Indian pig types using microsatellite markers. Anim. Genet., 2002, 33: 158-159.
-
15. L i K., C h e n Y., M o r a n C. e.a. Analysis of diversity and genetic relationships between four Chinese indigenous pig breeds and one Australian commercial breed. Anim. Genet., 2000, 31: 322-325.
-
16. K a u l R., S i n g h A., V i j h R.K. e.a. Evaluation of genetic variability of 13 microsatellite markers in native Indian pigs. J. Genet., 2001, 80: 149-153.
-
17. L e m u s - F l o r e s C., U l l o a - A r v i z u R., R a m o s -K u r i M. e.a. Genetic analysis of Mexican hairless pig population. J. Anim. Sci., 2001, 79: 3021-3026.
-
18. L a v a l G., I a n n u c c e l l i N., L e g a u l t C. e.a. Genetic diversity of eleven European pig breeds. Genet. Sel. Evol., 2000, 32: 187-203.
-
19. M a r t i n e z A.M., D e l g a d o J.V., R o d e r o A. e.a. Genetic structure of the Iberian pig breed using microsatellites. Anim. Genet., 2000, 31: 295-301.
-
20. V e r n e s i C., C r e s t a n e l l o B., P e c c h o i l i E. e.a. The gene tic impact of demographic decline and reintroduction in the wild boar ( Sus scrofa ): a microsatellite analysis. Mol. Ecol., 2003, 12: 585-595.
-
21. S p e n c e r P.B.C., H a m p t o n J., L a p i d g e S.J. e.a. An assessment of the genetic diversity and structure within and among populations of wild pig ( Sus scrofa ) from Australia and Papua New Guinea. J. Genet., 2006, 85(1): 63-66.
-
22. С у л и м о в а Г.Е. ДНК-маркеры в генетических исследованиях: типы маркеров, их свойства и области применения. Электронный журнал Лаборатории сравнительной генетики животных, 2004, 1. ( http://www.lab-cga.ru/articles/Jornal01/Statia1.htm ).
-
23. E r n s t M., K u c i e l J., U r b a n T. Analysis of genetic variation of eight candidate genes in two wild boar subspecies. Czech J. Anim. Sci., 2003, 48(12): 533-539.
-
24. З и н о в ь е в а Н.А., П о п о в А.Н., Э р н с т Л.К. и др. Методические рекомендации по использованию метода полимеразной цепной реакции в животноводстве. Дуб-ровицы, 1998.
-
25. М е р к у р ь е в а Е.К. Биометрия в селекции и генетике сельскохозяйственных животных. М., 1979.
-
26. Ж и в о т о в с к и й Л.А. Популяционная биометрия. М., 1991.
-
27. В е й р Б. Анализ генетических данных. Пер. с англ. Д.В. Зайкина, А.И. Пудовкина, А.Н. Татаренкова. М., 1995.
-
28. P e a t k a u D., S l a d e R., B u r d e n s M. e.a. Genetic assignment methods for the direct, real-time estimation of migration rate: a simulationbased exploration of accuracy and power. Mol. Ecol., 2004, 13: 55-65.
-
29. Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 1978, 89: 583-590.
-
30. B r o o k e s A.J. The essense of SNPs. Gene, 1999, 234(2): 177-186.
-
31. P a e t k a u D., S t r o b e c k C. The molecular basis and evolutionary history of a mi
crosatellite null allele in bears. Mol. Ecol., 1995, 4: 519-520.
-
32. Электронный ресурс DAD-IS (Domestic Animal Diversity Information System). 2006, http://www.fao.org/dad-is/ .
-
33. T i k h o n o v V.N., B o b o v i c h V.E. Immuno- and cytogenetic peculiarities of wild boar subspecies ( Sus scrofa ) from different regions of Eurasia and examination of their genetic purity for conservation of subspecies. In: Proc. the 1st Int. Symp. on physiology and ethology of wild and zoo animals. Berlin, Germany, 1997, Suppl. II: 229-232.
-
34. M u 11 e r E., M o s e r G., B a r t e n s c h l a g e r H. e.a. Trait values of growth, carcass and meat quality in Wild Boar, Meishan and Pietrain pigs as well as their crossbred generations. J. Anim. Breed. Genet., 2000, 117(3), 189-202.
-
35. O k u m u r a N., K u r o s a w a Y., K o b a y a s h i E. e.a. Genetic relationship
amongst the major non-coding regions of mitochondrial DNAs in wild boars and several breeds of domesticated pigs. Anim. Genet., 2001, 32: 139-147.
-
36. R o t h s c h i l d M., J a c o b s o n C., V a s k e D. e.a. The estrogen receptor locus is
associated with a major gene influencing litter size in pigs. PNAS USA, 1996, 93: 201-205.
-
37. M e l v i l l e J.S., G i b b i n A.M., R o b i n s o n J.A. e.a. Meishang positire QTL for
prolificacy traits found at the NCOA1 locus on SSC3. In: Proc. of the 7th World Congress on Genetics Applied to Livestock Production. Montpellier, France, 2002: 19-23.
-
38. P y t h o n P., J o e r g H., N e u e n s c h w a n d e r S. e.a. Submitted (MAR-2003) to the EMBL/GenBank/DDBJ databases. ( http://au.expasy.org/uniprot/Q864U6 ).
-
39. J 0 r g e n s e n C.B., C i r e r a S., A n d e r s o n S.I. e.a. Linkage and comparative mapping of the locus controlling susceptibility towards E. coli F4ab/ac diarrhoea in pigs. Cyto-genet. Genome Res., 2003, 102: 157-162.
-
40. P e n g Q.-L., R e n J., Y a n X.-M. e.a. The g.243A>G mutation in intron 17 of MUC4 is significantly associated with susceptibility/resistance to ETEC F4ab/ac infection in pigs.
-
41. A n d e r s s o n - E k l u n d L., M a r k l u n d L., L u n d s t r 0 m K. e.a. Mapping quantitative trait loci for carcass and meat quality traits in a Wild Boar x Large White intercross. J. Anim. Sci., 1998, 76: 694-700.
-
1ГН У Всероссийский НИИ животноводства Поступила в редакцию
Россельхозакадемии , 1 февраля 2009 года
142132 Московская обл., Подольский р-н, пос. Дубровицы, e-mail: elenagladyr@mail.ru ;
Anim. Genet., 2007, 38(4): 397-400.
-
2 Ро ссийская академия сельскохозяйственных наук,
117218 г. Москва, ГСП-7, ул. Кржижановского, 15, корп. 2
STUDY OF Sus scrofa GENOME WITH THE USE OF DNA-MARKERS
E.A. Gladyr’1, L.K. Ernst2, O.V. Kostyunina1
S u m m a r y
The authors made the experimental investigations of genetic polymorphism in five populations of two subspecies of wild boar, which are inhabit at the territory of Russian Federation, and the pigs of the Large White, the Yorkshire, the Duroc and the Landrace breeds. The some aspects of studying of genotype of wild boar in comparison with different breeds of domestic pigs were considered by the example of analysis of microsatellites and marker genes.

ВНИМАНИЮ ЧИТАТЕЛЕЙ! ВЫШЛА В СВЕТ КНИГА:
Л.К. Эрнст, Н.А. Зиновьева. Биологические проблемы животноводства в XXI веке. М.: РАСХН, 2008, 501 с., с илл.
Монография представляет собой обобщение данных литературы и результатов многолетних собственных исследований авторов в области биологии, биотехнологии, молекулярной биологии, питания и экологии сельскохозяйственных животных.
Рассмотрены новые технологии изучения генома, генодиагностики, биотехнологии и био инженерии сельскохозяйственных животных, основанные на использовании современных аналитических методов, вопросы изучения изменчивости генотипа в процессе эволюции (селекции), а также взаимодействия генотипа и фенотипа в норме и при воздействии различных факторов, в частности интеграции чужеродных генов у трансгенных животных.
Монография состоит из введения, 11 глав, заключения и списка использованной литературы. Включает 35 черно-белых иллюстраций в виде схем и графиков и 8 цветных графиков и фотографий, 148 таблиц.
Авторы монографии: Лев Константинович Эрнст, доктор сельскохозяйственный наук, профессор, академик и вице-президент Российской академии сельскохозяйственных наук (РАСХН), руководитель Центра биотехнологии и молекулярной диагностики Всероссийского НИИ животноводства (ВИЖ), кавалер ордена «За заслуги перед Отечеством» III и IV степеней; Наталия Анатольевна Зиновьева, доктор биологических наук, профессор, член-корреспондент РАСХН, заместитель директора ВИЖ по научной работе, заместитель руководителя Центра биотехнологии и молекулярной диагностики ВИЖ, лауреат Государственной премии Российской Федерации для молодых ученых в области науки и техники.


