Изучение генов, контролирующих реакцию дегалогенирования, бактерий-деструкторов 4-хлорбензоата
Автор: Шумковa Е.С., Лобова И.В., Ананьина Л.Н., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 5, 2007 года.
Бесплатный доступ
Методом полимеразной цепной реакции проведен скрининг бактерий-деструкторов 4-хлорбензойной кислоты на наличие fcb-генов, контролирующих реакцию дегалогенирования 4-хлорбензоата. Установлено, что Arthrobacter sp. H4, H5, Pseudomonas fluorescens H2, Micrococcus sp. G120 и Rhodococcus ruber Р25 содержат fcbA и fcbB гены, кодирующие 4ХБК-СоА-лигазу и 4ХБК-СоА-дегалогеназу, соответственно. Уровень сходства fcb-генов штамма R. ruber Р25 с деструкторами 4ХБК рода Arthrobacter cjcnfdkztn 95-99%.
Короткий адрес: https://sciup.org/147204393
IDR: 147204393 | УДК: 579.25
Текст научной статьи Изучение генов, контролирующих реакцию дегалогенирования, бактерий-деструкторов 4-хлорбензоата
-
a Институт экологии и генетики микроорганизмов, 614081, Пермь, ул. Голева, 13
-
b Пермский государственный университет, 614990, Пермь, ул. Букирева, 15
Методом полимеразной цепной реакции проведен скрининг бактерий-деструкторов 4-хлор-бензойной кислоты на наличие fcb -генов, контролирующих реакцию дегалогенирования 4-хлорбензоата. Установлено, что Arthrobacter sp. H4, H5, Pseudomonas fluorescens H2, Micrococcus sp . G120 и Rhodococcus ruber Р25 содержат fcb A и fcb B гены, кодирующие 4ХБК-СоА-лигазу и 4ХБК-СоА-дегалогеназу, соответственно. Уровень сходства fcb- генов штамма R. ruber Р25 с деструкторами 4ХБК рода Arthrobacter cjcnfdkztn 95–99%.
К наиболее распространенным и экологически значимым загрязнителям окружающей среды относятся галогенированные ароматические соединения, в том числе полихлорированные бифенилы (ПХБ) и хлорбензойные кислоты (ХБК). Данные ксенобиотики являются высокотоксичными веществами, способными вызывать серьезные нарушения нервной, репродуктивной и иммунной систем животных и человека (Кулинский, 1999). Они характеризуются высокой устойчивостью к физическим и химическим воздействиям, способны накапливаться в значительных количествах в объектах окружающей среды. Известно, что основная роль в трансформации ПХБ и ХБК в природной среде принадлежит микроорганизмам (Dua et al., 1991; Hickey et al., 1993; Abraham et al., 2002). Изучение процессов разложения этих соединений микроорганизмами приобретает все большую актуальность и является основой для разработки методов биоремедиации загрязненных почв и водных объектов.
Микробная деструкция ПХБ в большинстве случаев идет до стадии образования хлорбензойной и пентадиеновой кислот. Полное разложение хлорированных бифенилов осуществляется смешанными культурами бактерий, в состав которых входят деструкторы ХБК (Hickey et al., 1993; Fava et al., 1994; Fava et al. 1996; Abraham et al., 2002). О природных бактериях, способных утилизировать моно- и дихлорированные бифенилы, имеются лишь единичные публикации (Arensdorf, Focht, 1995; Kim, Picardal, 2000). Однако, появляются работы по созданию рекомбинантных штаммов, осуществляющих полную деструкцию хлорбифе- нилов (Havel, Reineke, 1991; Hrywna et al., 1999; Dua et al., 2002; Rodrigues et al., 2006). При конструировании таких микроорганизмов используются клонированные гены, контролирующие реакцию дегалогенирования ХБК. Отщепление галогена от молекулы хлорбензойной кислоты проводит к резкому снижению ее биологической устойчивости и делает это соединение доступным в качестве источника питания для большинства микроорганизмов (Карасевич, 1982).
Реакция дехлорирования 4ХБК, в результате которой происходит замещение иона хлора в пара положении на гидроксильную группу воды и образование пара -оксибензойной кислоты, катализируется ферментной системой, включающей 3 компонента: 4ХБК-СоА-лигазу ( ген fcbA), 4ХБК-СоА-дегалогеназу (ген fcbB ) и пара -оксибензоат-СоА-тиоэстеразу (ген fcbC ) (рис. 1). Подобные ферментные системы обнаружены у ряда деструкторов 4ХБК, в том числе у Arthrobacter globiformis КЗТ1 (Tsoi et al., 1991), Arthrobacter sp. SU (Schmitz et al., 1992), Arthrobacter sp. TM1 (Garte-mann, Eichenlaub, 2001), Pseudomonas sp. DJ-12 (Chae et al., 2000).
Из почв, загрязненных отходами производства галогенированных соединений (ОАО «Галоген», Пермь), выделены бактериальные штаммы, способные использовать в качестве единственного источника углерода разнообразные ароматические соединения (бифенил, нафталин, толуол, бензол, фенол), а также моно- и дихлорированные бензойные кислоты. Изолированные биодеструкторы отнесены к родам Arthrobacter, Micrococcus, Pseudo-
monas , Planococcus и Rhodococcus (Рыбкина, 2003;
Рыбкина и др., 2005). Наиболее интересный из изолированных бактерий штамм Rhodococcus ruber P25 является эффективным деструктором ряда ксенобиотиков, в том числе орто- и пара-замещенных моно- и полихлорированных бифенилов, и он обладает ферментативными системами, отвечающими за деградацию 2ХБК и 4ХБК (Рыб- кина, 2003).
OCoA
АТФ, KoA
O OH
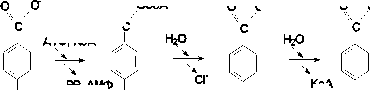
O OСoA
C
Cl 4ХБК fcbC
4ХБК-КоА тиоэстераза
PP, АМФ
Cl
4ХБК-КоА
KoA
OH 4ОБК
OH 4ОБК-КоА fcbB
4ХБК-КоА дегалогеназа
fcbA
4ХБК:КоА лигаза
Рис. 1. Схема гидролитического дегалогенирования 4ХБК штаммом A. globidformis КЗТ1 (Tsoi et al., 1991)
Цель настоящей работы – поиск и изучение fcb -генов, контролирующих реакцию дегалогенирования, бактерий-деструкторов 4ХБК.
Материалы и методы
Рабочая коллекция. В работе использованы бактериальные штаммы, выделенные ранее из образцов почв, отобранных на территории предприятия ОАО «Галоген» (Пермь): Arthrobacter sp. H4, H5, Micrococus sp . G126, G120, Planococcus sp. G110, P. fluorescens H2, P. putida G107, R. ruber P25 (Рыбкина, 2003; Рыбкина и др., 2005), а также биодеструктор 4ХБК A. globiformis КЗТ1 (Tsoi et al., 1991).
Среды и условия культивирования. Для культивирования бактериальных культур использовали минеральную среду К1 следующего состава (г/л): K 2 HPO 4 x 3H 2 O – 4, NaH 2 PO 4 x 2H 2 O – 0,4, (NH 4 ) 2 SO 4 – 0,5, Ca(NO 3 ) 2 – 10, MgSO 4 х 7H 2 O – 0,15, дополненную раствором витаминов (мкг/л): биотин – 0.02, тиамин – 0.5, рибофлавин – 0.5, инозитол – 0.5, рН среды 7.3 (Zaitsev et al., 1991). В качестве ростового субстрата использовали химически чистую 4ХБК (1 г/л) (Fluka, США), которую добавляли в виде водного раствора. Культивирование микроорганизмов проводили в колбах объемом 250 мл в 100 мл среды при 28°С с аэрацией на шейкере (100 об/мин).
Молекулярно-генетические методы. Выделение тотальной ДНК у исследуемых штаммов проводили по стандартной методике (Ausbel et al., 1995). Плазмидную ДНК выделяли модифицированным методом щелочного лизиса (Marko et al., 1982).
Для проведения полимеразной цепной реакции (ПЦР) и определения нуклеотидных последовательностей исследуемых генов были использованы праймеры, сконструированные на основе известных последовательностей fcb-генов штаммов Arthrobac- ter sp. SU и A. globiformis КЗТ1 (GenBank). Праймеры к гену fcbA:
f:5’-AACTGATCCGCCGAGACAACATTC-3’, r:5’-AGGCATTTTTCGAGACGCTTCA-3’;
fcbB : f:5’-GGTCCAGCGAGCGAAATCCAGTC-3’, r:5’-CCCCCGCACACCGCATCAAG-3’.
ПЦР осуществляли на приборе MyCycler (BioRad, США) по стандартной методике (Ausbel et al., 1995). Условия проведения ПЦР: 5 мин. при 95°С, далее 30 циклов: 30 сек. при 94°С, 30 сек. при 60°С, 2 мин. 10 сек. при 72°С.
Для обнаружения ДНК и ПЦР-продуктов применяли электрофорез в горизонтальном агарозном геле (1%) при напряжении 60–90 В в трис-ацетатном буфере (89 мМ Трис, 12.5 мМ Na 2 ЭДТА, 89 мМ ацетата Na, рН 8,0). Агарозные гели окрашивали раствором бромистого этидия (2 мкг/мл) в течение 20 мин. и фотографировали в УФ-свете с помощью системы гель-документирования Gel DocTM XR (BioRad, США). В качестве метчиков использовали ДНК λ / Hind III и молекулярный маркер IX (Sigma, Германия). Размер плазмидной ДНК оценивали сравнивая ее электрофоретическую подвижность с подвижностью плазмидной ДНК известного размера. В качестве маркерной плазмиды использовали pКЗТ1 (110 т.п.н.).
Анализ полиморфизма длин рестрикционных фрагментов (ПДРФ) исследуемых участков fcb -генов осуществляли с помощью эндонуклеаз рестрикции Hha I , Hae III , Eco RI , Eco RV , Bam HI , Pvu II , Mse I (Sigma, Германия). Реакцию проводили при 37°С в течение 1–2 часов в объеме реакционной смеси 20 мкл. Построение карт сайтов рестрикции для участков исследуемых генов осуществляли с помощью программы Vector NTI.
Секвенирование продуктов амплификации проводили по методу Сэнгера с помощью набора CEQ Cycle Sequencing kit на автоматическом секвенаторе CEQ 2000XL DNA Analysis System (Beckman Coulter, США) согласно инструкциям производителя. Полученные нуклеотидные последовательности анализировали с использованием программы Chro-mas 1.45. Нуклеотидные последовательности были выровнены с соответствующими последовательностями других бактерий-деструкторов с помощью программы CLUSTALX 1.83. Построение дендрограмм сходства исследуемых участков ДНК проводили с помощью метода «neighbor-joining», реализованного в пакете программ TREECONW и PHY-LIP. Показатель достоверности порядка ветвления определяли на основании «butstrap»-анализа 100 альтернативных деревьев. Для поиска гомологичных нуклеотидных последовательностей в международных базах данных GenBank/EMBL/DDBJ использовали программу BLAST.
Результаты и обсуждение
Характеристика штаммов-деструкторов
Для настоящей работы были отобраны восемь штаммов, способных использовать в качестве единственного источника углерода и энергии 4-хлорбензоат: Arthrobacter sp. H4, Arthrobacter sp. H5 , Micrococcus sp. G120, Micrococcus sp. G126 , Planococcus sp. G110, P. fluorescens H2, P. putida G107 и R. ruber P25.
Показано, что все бактерии-деструкторы способны к росту на возможных продуктах разложения 4ХБК: пара- оксибензойной и протокатеховой кислотах. Исследуемые штаммы проявляют широкую субстратную специфичность по отношению к различным ароматическим соединениям, способны к росту на феноле, орто -фталате, бензоле и толуоле. Кроме того, изоляты разлагают 2-хлорбензоат (штаммы G120, G126, H2, G107, G110), 3-хлорбензоат и 2,4-дихлорбензоат (штаммы G120, G126, G107, G110) (Рыбкина, 2003; Рыбкина, Гусев, Плотникова, 2005).
Rhodococcus ruber P25 является активным деструктором хлорбифенилов (ХБ): способен утилизировать 2-моноХБ, 4-моноХБ, 2,4-диХБ, 2,2’-диХБ и 4,4’-диХБ, осуществляет 2,3-диоксигенирование ди( орто-пара )хлорированного кольца молекулы 2,4,2’-триХБ и окисляет моно( пара )хлорированное кольцо 2,4,4’-триХБ. Штамм способен к росту на 2ХБК, 4ХБК и 2,4ДХБК, используя данные субстраты в качестве единственного источника углерода и энергии (Рыбкина, 2003).
Известно, что способность к деструкции различных ксенобиотиков, в том числе ХБК, часто определяется генами, расположенными в плазмидах. Исследуемые нами штаммы были проверены на наличие плазмидной ДНК. Плазмиды большого размера были обнаружены в штаммах Arthrobacter sp. Н4 (120 т.п.н.) и P.fluorencens H2 (100 т.п.н.). Ранее было показано, что штамм R. ruber P25 содержит три плазмиды, размеры которых - 110, 90 и 80 т.п.н. Результаты экспериментов по элиминации плазмид позволили предположить, что гены, ответственные за деструкцию 2ХБК и 4ХБК расположены в плазмиде размером 110 т.п.н., гены деструкции бифенила – в плазмиде размером 90 т.п.н., а гены, контролирующие деструкцию нафталина, – в плазмиде размером 80 т.п.н. (Рыбкина, 2003). Таким образом, размер обнаруженных плазмид (100– 120 т.п.н.), предположительно содержащих гены деградации 4ХБК, сопоставим с размером плазмид известных биодеструкторов 4ХБК (Gartemann, Eichen-laub, 2001; Zaitsev et al., 1991). Кроме того, в плазмид-ных ДНК штаммов A.globiformis КЗТ1, Arthrobacter sp. SU были обнаружены fcb -гены, контролирующие первый этап разложения 4ХБК до 4-оксибензоата, дальнейшее разложение которого контролируется хромосомальными генами (Zaitsev et al., 1991; Schmitz et al., 1992).
Скрининг штаммов на наличие fcb-генов методом полимеразной цепной реакции
На матрице ДНК исследуемых штаммов была проведена амплификация нуклеотидных последовательностей, соответствующих участкам fcb- генов, контролирующих первый этап разложения 4ХБК (дегалогенрование) у бактерий. Для выявления генов fcbA и fcbB, кодирующих 4-хлоробензоат-СоА-лигазу и 4-хлоробензоат-СоА-дегалогеназу соответственно, были использованы праймеры, разработанные на основе известных последовательностей fcb- генов штаммов Arthrobacter sp. SU и A. globi-formis КЗТ1 (GenBank).
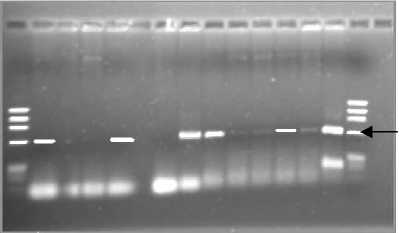
500 п.н.
1 2 3 4 5 6 7 8 9 10 11 12 13 14
Рис. 2. Гель-электрофорез продуктов ПЦР-амп-лификации генов fcb из препаратов ДНК штаммов: H2 ( 2 – fcbA , 8 – fcbB ), G107 ( 3 – fcbA , 9 – fcbB ), G110 ( 4 – fcbA , 10 - fcbB ), G120 ( 5 – fcbA , 11 – fcbB ), G126 ( 6 – fcbA , 12 – fcbB ), КЗТ1 ( 7 – fcbA , 13 – fcbB ), маркер IX ( 1 , 14 )
У пяти из восьми изучаемых штаммов (H4, H5, H2, G120 и Р25) обнаружены ПЦР-продукты длиной около 600 п.н., соответствующие фрагменту гена fcbA. Ампликоны такого же размера, соответствующие участку гена fcbB, получены для всех исследуемых штаммов (рис. 2, 3). Размер гена fcbA у A. globi-formis КЗТ1 (GenBank) составляет 1556 п.н. (позиции 277–1833 секвенированной последовательности ДНК), а гена fcbB – 830 п.н. (позиции 1843–2673 сек-венированной последовательности ДНК). Амплифи-цируемая область гена fcbA , располагающаяся в районе 568–1157 нуклеотидов, соответствует участку около 1/3 длины гена и находится в его центральной части. Амплифицируемый фрагмент гена fcbB располагается в районе 2068–2666 нуклеотидов. Этот участок соответствует большей части гена, за исключением первых 200 п.н. Длина амплифицирован-ных фрагментов fcb -генов с матриц ДНК исследуемых штаммов совпадала с ожидаемой. Полученные данные позволяют предположить наличие у штаммов H4, H5, H2, G120 и Р25 генов, контролирующих дегалогенирование 4ХБК.
Более подробно были исследованы нуклеотидные последовательности фрагментов fcbA и fcbB генов, амплифицированных с матрицы тотальной ДНК R. ruber P25 и Arthrobacter sp. Н4.
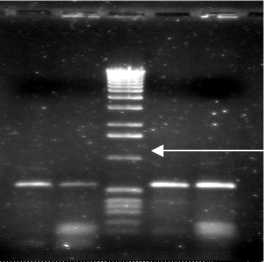
1000 п.н.
1 2 3 4 5
Рис. 3. Гель-электрофорез продуктов ПЦР-амплификации fcb -генов штамма КЗТ1 ( 1, 4 ) и штамма Р25 ( 2,5 ): 1,2 – fcbA , 4,5 – fcbB, 3 – маркер мол. масс 1000 п.н.
галогенирование 4ХБК у штаммов P25 и КЗТ1.
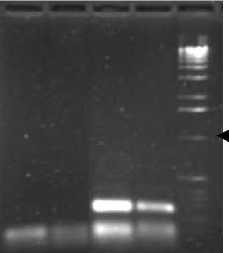
1000 п.н.
1 2 3 4 5
Рис. 5. Электрофореграмма рестрикционных фрагментов, полученных в результате обработки продуктов ПЦР fcbA и fcbB штамма P25 ( 1, 3 ) и штамма КЗТ1 ( 2, 4 ) эндонуклеазой Hha I: 1, 2 – fcbA ; 3, 4 – fcbB ; 5 – маркер мол. масс 1000 п.н.
ПДРФ-анализ fcb -генов штамма R. ruber 25
Для подбора эндонуклеаз рестрикции с помощью программы VectorNTI были построены рестрикционные карты генов fcbA и fcbВ штамма A. glo-biformis КЗТ1. ПЦР-продукты fcb -генов исследуемого штамма P25 и контрольного штамма КЗТ1 обработали следующими рестриктазами: ДНК fcbA - Hha I, Hae III, Eco RV, Bam HI, Mse I, а ДНК fcbB -Hha I, Hae III, Bam HI, Pvu II, Mse I. Картины гидролиза перечисленными выше ферментами ампликонов fcb генов обоих штаммов совпадали (рис. 4, 5) и согласовывались с предполагаемыми результа-
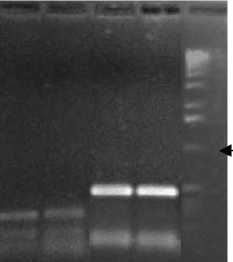
1000 п.н.
1 2 3 4 5
Рис. 4. Электрофореграмма реестрикци-онных фрагментов, полученных в результате обработки продуктов ПЦР fcbA и fcbB штамма P25 ( 1, 3 ) и штамма КЗТ1 ( 2, 4 ) эндонуклеазой Hae III: 1,2 – fcbA , 3,4 – fcbB ; 5 – маркер мол. масс 1000 п.н.
тами построенных рестрикционных карт. Таким образом, полученные результаты говорят о сходстве амплифицированных нуклеотидных последовательностей штаммов, что является дополнительным свидетельством в пользу предположения о сходстве генов/ферментов, осуществляющих де-
Анализ нуклеотидных последовательностей fcb -генов штаммов-деструкторов 4ХБК
На следующем этапе исследования были определены нуклеотидные последовательности ампли-фицированных фрагментов fcbA и fcbB генов штаммов R. ruber P25 и Arthrobacter sp . H4. Для проведения сравнительного анализа полученных нуклеотидных последовательностей с уже имеющимися в международных базах данных Gen-Bank/EMBL/DDBJ последовательностями fcb генов штаммов-деструкторов 4ХБК была использована программа BLAST. Для участка гена fcbA штамма Arthrobacter sp . H4 обнаружено 100% сходство с гомологичными последовательностями штаммов рода Arthrobacter : КЗТ1, FG1, SU, TM1. Уровень гомологии секвенированного участка гена fcbA штамма R. ruber P25 с перечисленными выше штаммами составил 98-99% и 95% – со штаммом Arthrobacter sp . FHP1 (рис. 6).
В результате проведения сравнительного анализа гомологичных нуклеотидных последовательностей, соответствующих участку гена fcbB, обнаружено 100% сходство штамма Arthrobacter sp . Н4 и 98–99% штамма R. ruber P25 со всеми исследованными штаммами рода Arthrobacter (рис. 6).
Уровень гомологии нуклеотидных последовательностей грамотрицательных штаммов ( Pseudomonas sp. DJ-12, Alcaligenes sp. AL3007) с грампо-ложительными штаммами бактерий-деструкторов 4ХБК составил около 60% для обоих исследуемых fcb- генов.
Заключение
Шесть грамположительных и два грамотрица-тельных штамма-деструктора 4-хлорбензоата были
а)
0,1
A. globiformis КЗТ-1 (pCA31 1)
Arthrobacter sp. TM 1 4
Arthrobacter ramosus FG1
Arthrobacter sp. SU (pASU1)
Arthrobacter sp. FHP1
Rhodococcus ruber P25
Pseudomonas sp. DJ-12
Alcaligenes sp. AL3007 (pss70)
б)
0,1
A. globiformis КЗТ-1 (pCA31 1)
Arthrobacter sp. TM 1 4
Arthrobacter ramosus FG1
Arthrobacter sp. SU (pASU1)
Arthrobacter sp. FHP1 [2]
Arthrobacter sp. FHP1 [1]
Rhodococcus ruber P25
Alcaligenes sp. AL3007 (pss70)
Pseudomonas sp. DJ-12
Рис. 6. Древо сходства нуклеотидных последовательностей, гомологичных исследуемым участкам генов fcbА (а) и fcbB (б) R. ruber P25. Масштаб соответствует 10 нуклеотидным заменам на каждые 100 нуклеотидов. “Bootstrap”-анализ проведен на 100 повторностях исследованы на наличие генов, контролирующих начальный этап разложения 4ХБК – гидролитическое дегалогенирование. Скрининг исследуемых бактериальных культур методом полимеразной цепной реакции установил наличие у штаммов Arthrobacter sp. H4, H5, P. fluorescens H2, Microcoсcus sp. G120 и R. ruber P25 генов fcbA и fcbB, кодирующих 4ХБК-СоА-лигазу и 4ХБК-СоА-дегалогеназу соответственно. Данные белки являются компонентами ферментной системы, осуществляющей реакциию дегалогенирования 4ХБК по гидролитическому пути. У штаммов P. fluorescens H2, Arthrobacter sp. Н4 и R. ruber Р25 гены fcb, возможно, локализованы в плазмидах.
ПЦР-анализ ДНК бактериальных культур Micrococcus sp. G126, Planococcus sp. G110, P. putida G107 показал лишь наличие гена fcbB . Можно предположить, что нуклеотидные последовательности fcbA -генов этих деструкторов отличаются от последовательности генов, на основе которых были синтезированы праймеры. Изучение таких генов представляет интерес и будет продолжено.
Гены fcbA и fcbB грамположительных штаммов R. ruber P25 и Arthrobacter sp. H4 были исследованы более детально. ПДРФ-анализ ПЦР-продуктов и сравнительный анализ нуклеотидных последовательностей fcb-генов штамма P25 с гомологичны- ми последовательностями показали их значительное сходство со штаммами рода Arthrobacter.
Таким образом, исследуемые биодеструкторы обладают существенным потенциалом в виде генетических систем деградации пара хлорбензоатов, который в перспективе может быть использован в биотехнологиях по очистке почв и водных объектов, загрязненных галогенсодержащими ксенобиотиками.
Работа поддержана грантом РФФИ-Урал №0404-96042.
Список литературы Изучение генов, контролирующих реакцию дегалогенирования, бактерий-деструкторов 4-хлорбензоата
- Карасевич Ю.Н. Основы селекции микроорганизмов, утилизирующих синтетические органические соединения. М.: Наука, 1982. 144 с.
- Кулинский В.И. Обезвреживание ксенобиотиков.//Соросов. образов. журн. 1999. № 1. C. 8-12.
- Рыбкина Д.О. Исследование аэробных бактерий, разлагающих полихлорированные бифенилы и хлорбензойные кислоты: Дис. …канд. биол. наук. Пермь, 2003. 181 с.
- Рыбкина Д.О., Гусев В.А., Плотникова Е.Г. Почвенные микроорганизмы, разлагающие ароматические углеводороды и карбоновые кислоты.//Вестн. Перм. ун-та. 2005. № 6. Биология. С. 115-122.
- Abraham W.R. et al. Polychlorinated biphenyldegrading microbial communities in soils and sediments.//Curr. Opin. Microbiol. 2002. Vol. 5. P. 246-253.
- Arensdorf J.J., Focht D.D. A meta cleavage pathway for 4-chlorobenzoate, an intermediate in the metabolism of 4-chlorobiphenyl by Pseudomonas cepacia P166.//Appl. Environ. Microbiol. 1995. Vol. 61. P. 443-447.
- Ausbel F.M. et al. Short protocols in molecular biology. Third edition. 1995. 450 p.
- Chae J.C. et al. Genetic structure and functional implication of the fcb gene cluster for hydrolytic dechlorination of 4-chlorobenzoate from Pseudomonas sp. DJ-12.//Gene. 2000. Vol. 258. P. 109-116.
- Dua M. et al. Biotechnology and bioremediation: successes and limitations.//Appl. Microbiol. Biothechnol. 2002. Vol. 59. P. 143-152.
- Fava F. et al. Degradation and dechlorination of lowchlorinated biphenyls by a three-membered bacterial co-culture.//Appl. Microbiol. Biotechnol. 1994. Vol. 41. P. 117 -123.
- Fava F. et al. Aerobic dechlorination of lowchlorinated biphenyls by bactterial biofilms in packed-bed batch bioreactors.//Appl. Microbiol. Biotechnol. 1996. Vol. 45. P. 562-568.
- Gartemann K.H. and Eichenlaub R. Isolation and characterization of IS1409, an insertion element of 4-chlorobenzoate-degrading Arthrobacter sp. strain TM1, and development of a system for transposon mutagenesis//J. Bacteriol. 2001. Vol. 183. P. 3729-3736.
- Havel J., Reineke W. Total degradation of various chlorobiphenyls by cocultures and in vivo constructed hybrid pseudomonads.//FEMS Microbiol. Lett. 1991. Vol. 78. P. 163-170.
- Hickey W.J., Searles D.B., Focht D.D. Enhanced mineralization of polychlorinated biphenyls in soil inoculated with chlorobenzoate-degradin bacteria.//Appl. Environ. Microbiol. 1993. Vol. 59. P. 1194-1200.
- Hrywna Y. et al. Construction and characterization of two recombinant bacteria that grow on ortho-and para-substituted chlorobiphenyls.//Appl. Environ. Microbiol. 1999. Vol. 65. P. 2163-2169.
- Kim S., Picardal F.W. A novel bacterium that utilizes monochlorobiphenyls and 4-chlorobenzoate as a growth substrates.//FEMS Microb. Letters. 2000. V.185. P. 225-229.
- Marko M.A., Chipperfield R., Birnboim H.C. A procedure for the large-scale isolation of highly purified plasmid DNA using alkaline extraction and binding to glass powder.//Analit. Biochem. 1982. Vol. 121. P. 382-388.
- Rodrigues J.L. et al. Degradation of aroclor 1242 dechlorination products in sediments by Burkholderia xenovorans LB400 (ohb) and Rhodococcus sp. strain RHA1 (fcb).//Appl Environ Microbiol. 2006. Vol. 72. P. 2476-2482.
- Schmitz A. et al. Cloning and sequence analysis of genes for dehalogenation of 4-chlorobenzoate from Arthrobacter sp. strain SU.//Appl. Environ. Microbiol. 1992. Vol.58. P. 4068-4071.
- Tsoi T.V. et al. Cloning and expression of the Arthrobacter globiformis KZT-1 fcb gene encoding dehalogenase (4-chlorobenzoatehydroxylase) in E. coli.//FEMS Microbiol. Lett. 1991. Vol. 81. P. 165-170.
- Zaitsev G.M. et al. Genetic control of degradation of chlorinated benzoic acids in Arthrobacter globiformis, Corynebacterium sepedonicum and Pseudomonas cepacia strains.//FEMS Microbiol. Lett. 1991. Vol. 81. P. 171-176.


