Экспрессия поддерживающего белка NEDD9 как негативный прогностический маркер при немелкоклеточном раке легкого (НМРЛ)
Автор: Тихомирова М.В., Бармин В.В., Топчу Ю.А., Мазитова А.М., Ратнер Е.Ю., Сабиров А.Г., Абрамова З.И., Денека А.Я.
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 4 т.21, 2022 года.
Бесплатный доступ
Цель исследования. Оценка прогностической значимости поддерживающего белка Nedd9 у пациентов с немелкоклеточным раком легкого (НМРЛ). Nedd9 (Neural precursor cell expressed, developmentally down-regulated 9) является поддерживающим белком во множестве внутриклеточных белковых каска-дов, таких как pi3K-aKt-mtoR, Ras-eRK, играющих ключевые роли при прогрессировании опухолей. В литературе охарактеризована регуляторная роль Nedd9 во множестве солидных опухолей, в т. ч. и НМРЛ, однако детального анализа с целью оценки значимости Nedd9 как предиктивного маркера про-грессирования немелкоклеточного рака легкого ранее проведено не было. Материал и методы. Про-ведены log-rank анализ (тест логарифмического ранга) между клинико-патологическими параметрами когорты пациентов с НМРЛ (n=16) и ИГХ-экспрессии Nedd9, оцененной с помощью метода количе-ственной оценки h-score, в гистологических образцах пациентов, а также корреляционное исследование между показателями течения заболевания, такими как общая (ОВ) и беспрогрессивная выживаемость (БПВ), и экспрессией Nedd9. Полученные данные были валидированы в более обширной когорте дан-ных РНК-секвенирования tcga (n=566). Результаты. Показана достоверная разница (p function show_abstract() { $('#abstract1').hide(); $('#abstract2').show(); $('#abstract_expand').hide(); }
Немелкоклеточный рак легкого, общая выживаемость, беспрогрессивная выживаемость, поддерживающие белки, nedd9, маркеры-предикторы
Короткий адрес: https://sciup.org/140295758
IDR: 140295758 | УДК: 616.24-006.6:577.112 | DOI: 10.21294/1814-4861-2022-21-4-47-55
Текст научной статьи Экспрессия поддерживающего белка NEDD9 как негативный прогностический маркер при немелкоклеточном раке легкого (НМРЛ)
В настоящее время смертность от немелкоклеточного рака легкого (НМРЛ) занимает лидирующее место среди всех онкологических заболеваний [1]. Несмотря на то, что в последнее время увеличивается количество пациентов, для которых заболевание выявляется на ранних стадиях, большинство больных обращаются за лечением с распространенным НМРЛ, когда прогноз становится неблагоприятным, особенно при наличии рефрактерного к лечению метастатического поражения [2]. Поэтому важную роль для совершенствования методов лечения НМРЛ играют не только разработка новых препаратов и терапевтических подходов, более эффективных на поздних стадиях, но также улучшение адъювантной терапии, а также поиск новых прогностических биомаркеров НМРЛ.
Одним из новых и потенциально значимых биомаркеров является NEDD9. Этот белок является поддерживающим медиатором в множестве внутриклеточных сигнальных каскадов, таких как PI3K-AKT-mTOR, Ras-ERK, и интегринов, играющих ключевые роли в росте и прогрессировании множества опухолей, в т. ч. и НМРЛ [3–6], а также активатором тирозинкиназ семейства SRC, повышенная экспрессия которых ассоциируется с процессами эпителиально-мезенхимального перехода (ЭМП) – одного из ключевых биологических механизмов, лежащих в основе метастазирования НМРЛ [7–10]. В литературе показано, что повышенная экспрессия NEDD9 в опухолевых клетках ассоциируется с увеличением метастатического потенциала за счет приобретения клетками более мезенхимального фенотипа и увеличения миграционной способности клеток [11–14]. Важно отметить данные о том, что изменение уровней экспрессии NEDD9 в солидных опухолях ассоциируется с метастазированием и сниженной выживаемостью пациентов [15, 16], однако этот вопрос в контексте НМРЛ изучен недостаточно досконально: детального анализа корреляции экспрессии NEDD9 с клиническими показателями выживаемости и прогрессированием заболевания проведено не было.
Целью исследования явилась оценка прогностической значимости белка NEDD9 у пациентов с НМРЛ.
Материал и методы
В данном ретроспективном исследовании нами были изучены образцы первичных опухолей от 16 пациентов с НМРЛ, фиксированные в парафине, полученные из базы данных образцов пациентов, проходивших лечение в Республиканском клиническом онкологическом диспансере (РКОД) г. Казань, Республики Татарстан. Исследование одобрено этическим комитетом РКОД и Казанского федерального университета, протокол № 8 от 13.02.18. У каждого пациента на момент проведения забора материала с целью гистологической верификации, а также перед хирургическим лечением было взято письменное согласие на последующую анонимизированную обработку. Все образцы были анонимизированы, аннотированы.
Ткани первичной опухоли легкого были зафиксированы формальдегидом и погружены в парафин. Парафиновый блок, содержащий образцы тканей, нарезали, толщиной 3–5 мкм и фиксировали их на предметном стекле. ИГХ-окрашивание выполняли по следующей методике: срезы с целью депарафинизации трижды погружали в 100 % ксилен на 5 мин. Затем срезы дегидратировались в серии растворов этанола (100, 70, 50 %) при погружении на 3 мин в каждый. Затем проводилась термическая демаскировка эпитопов в 0,01М цитратном буфере (pH 6.0). ИХГ окрашивание производилось с использованием первичных антител к NEDD9 в концентрации 5 мкг/мл (2G9, Abcam, Бостон, США) и набора вторичных антител против мыши HRP/DAB IHC Detection kit–Micro-polymer (Abcam ab236466, Бостон, США). В качестве негативного контроля окрашивания вместо первичного антитела к NEDD9 использовалось мышиное IgG антитело. После ИГХ-окрашивания производилось дополнительное окрашивание гематоксилином. В конце срезы были повторно дегидратированы с помощью погружения в серию растворов этанола (70, 96, 100 %) и ксилена на 3 мин в каждый. Покровные стекла фиксировались с помощью раствора Histomount (HS-103, National Diagnostics, Атланта, США).
Окрашенные препараты сканировались на рабочей станции Leica Aperio CS2 (Leica Biosystems, Сан-Диего, США). Оценку ИГХ-экспрессии NEDD9 проводили с использованием полуколи-чественного метода h-score в программном обеспечении QPath (QuPath: ) [17], при котором уровень экспрессии определяется по формуле
[1× ( % клеток 1+) + 2 × ( % клеток 2+) +
+ 3 × ( % клеток 3+)]
и варьируется от 0 до 300 баллов [18].
Для корреляционного анализа РНК экспрессии NEDD9 и клинико-прогностических показателей пациентов с НМРЛ использовались материалы общедоступных баз данных Атласа геномных данных опухолей (TCGA) пациентов с аденокарциномой легкого, находящиеся на портале (ноябрь 2021). Использовался доступный онлайн набор инструментов визуализации и обработки данных [19].
Статистическая обработка полученных данных проводилась с использованием программного обеспечения GraphPad Prism 8.0 (Сан Диего, США), а также инструментов статистического анализа, доступных на сайте Для сравнения количественного показателя h-score на разных стадиях НМРЛ использовался непараметрический тест Манна–Уитни. Корреляция клинико-патологических показателей и экспрессии NEDD9 была оценена методом log-rank test. Кривые выживаемости были построены с использованием метода Каплана–Мейера для оценки БПВ и ОВ.
Результаты
В исследовании анализировалась когорта из 16 пациентов, из них 69 % мужчин, 31 % женщин (табл. 1). Медиана возраста – 57 лет, средний возраст – 57,3 года, стандартное отклонение – 7,4 года; диапазон от 38 до 68 лет. Четырем (25 %) пациентам был поставлен диагноз НМРЛ II стадии, 6 и 6 пациентам – НМРЛ III и IV стадии (по 37,5 % соответственно). Больным проведено лечение в соответствии с текущими клиническими рекомендациями; ни один из пациентов не участвовал в клинических исследованиях. Химио- и таргетная терапия проводилась в адъювантном режиме. Образцы опухолевой ткани были получены перед началом лечения. Пациентам с IV стадией НМРЛ была выполнена расширенная лобэктомия или пневмонэктомия с паллиативной целью. Данных за ассоциацию между клинико-патологическими характеристиками, такими как возраст, пол, стадия заболевания, и общей (ОВ) или бесспрогрессивной (БПВ) выживаемостью не обнаружено (p>0,05) (табл. 2).
На первом этапе исследования нами была проведена оценка уровней экспрессии белка NEDD9 с использованием метода ИГХ окрашивания и анализа h-score. Обнаружено, что при НМРЛ III стадии отмечается значимое снижение уровня белка NEDD9 по сравнению с НМРЛ II стадии (усредненный h-score 31,5 против 52,1; р=0,04), с дальнейшим повышением при НМРЛ IV стадии (усредненный h-score 31,5 против 76,05; р=0,006) (рис. 1А, Б). Для подтверждения полученных данных в более широкой выборке была произведена оценка экспрессии РНК NEDD9 в базе данных
Таблица 1/Table 1
Клиническая характеристика исследуемой когорты больных НМРЛ
Clinical characteristics of the cohort of NSClC patients
Для оценки прогностической значимости экспрессии NEDD9 нами проведен корреляционный анализ между уровнями экспрессии NEDD9 (как белка, так и РНК) и общей выживаемостью (ОВ), а также беспрогрессивной выживаемостью (БПВ) пациентов с НМРЛ. На первом этапе анализ проводился в когорте образцов РКОД. Значимой корреляции между экспрессией NEDD9 (дихотомическое распределение по усредненному значению h-score=56,14) и ОВ и БПВ у пациентов на стадиях заболевания не обнаружено (ОВ – р=0,31; БПВ – р=0,33; рис. 2А, Б), однако отмечался тренд между низким уровнем экспрессии NEDD9 и снижением показателей ОВ и БПВ.
Поскольку одним из ограничений данного исследования является небольшой размер выборки, для верификации полученных данных на следующем этапе был проведен аналогичный анализ с использованием более обширной когорты TCGA_PanCancer. Обнаружено, что снижение экспрессии РНК NEDD9 (нижний квартиль диапазона экспрессии) в первичных опухолях НМРЛ значимо коррелирует со снижением ОВ (р=0,05; рис. 2 В), но не с БПВ (р=0,45; рис. 2Г), по сравнению с повышенной (верхний квартиль) экспрессией NEDD9.
Обсуждение
Совершенствование методов хирургического лечения и системной терапии не всегда приводит к существенному улучшению отдаленных результатов, но внедрение таргетной терапии в адъювантном режиме позволяет значительно снизить частоту рецидивов и отдаленных метастазов (исследование ADAURA) [20]. Поэтому поиск новых предиктивных биомаркеров, позволяющих оценить риски прогрессирования НМРЛ, является чрезвычайно актуальным [21, 22].
В данном исследовании обнаружено, что при НМРЛ III стадии отмечается значимое снижение экспрессии белка NEDD9. Однако по данным анализа выживаемости, проведенного с использованием расширенной выборки пациентов (566 образцов), именно сниженная экспрессия РНК NEDD9 являлась негативным прогностическим фактором в отношении общей выживаемости пациентов. Поскольку экспрессия белков, участвующих во внутриклеточных сигнальных каскадах, связанных с пролиферативной и миграционной активностью опухолевых клеток, в которых NEDD9 играет важную роль, в значительной степени регулируется посттрансляционно [23], на основании полученных данных сделан вывод о том, что стабильная экспрессия РНК NEDD9 не всегда коррелирует с уровнями белка в клетках. Это позволяет, с одной
Таблица 2/Table 2
log-rank анализ между клинико-патологическими параметрами пациентов НМРЛ с общей и беспрогрессивной выживаемостью
log-rank analysis of clinicopathological parameters of the NSClC patient with overall or progression-free survival
|
Показатель/Parameter |
Общая выживаемость, мес, ДИ 95 %)/ Overall survival, months, 95 % CI |
Log-rank |
p-value |
Беспрогрессивная выживаемость, мес, ДИ 95 %)/ Progression-free survival, months, 95 % CI |
Log-rank |
p-value |
|
|
Возраст, лет/Age (years) |
|||||||
|
< медиана/ median > медиана / median |
7 (43,7 %) 9 (56,3 %) |
16,0 (13,8–18,2) 22,0 (13,2–30,8) |
0,64 |
0,43 |
7,0 (6,0–8,0) 9,0 (8,1–9,9) |
2,13 |
0,14 |
|
Ст. откл./ Standard |
7,4 |
||||||
|
deviation |
|||||||
|
Пол/Sex |
|||||||
|
Муж/ Male Жен/Female |
11 (69 %) 5 (31 %) |
15,0 (6,4–23,6) 18,0 (15,8–20,2) |
0,05 |
0,82 |
7,0 (3,9–10,1) 8,0 (6,3–9,7) |
0,53 |
0,47 |
|
Стадия/ Stage |
|||||||
|
II |
4 (25 %) |
19,0 (13,9–24,1) |
9,0 (7,8–10,2) |
||||
|
III |
6 (37.5 %) |
19,0 (15,3–22,7) |
3,62 |
0,16 |
7,0 (5,2–8,8) |
1,04 |
0,6 |
|
IV |
6 (37,5 %) |
12,0 (3,3–20,7) |
4,0 (2,5–5,5) |
||||
|
Всего/ Total |
16 (100 %) |
||||||
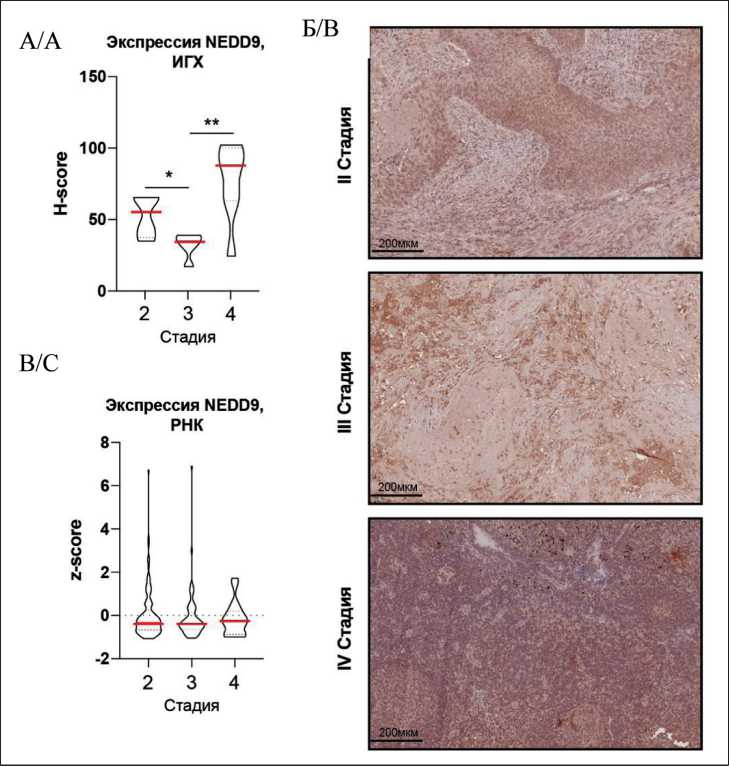
Рис. 1. Разделение уровней экспрессии NEDD9 у больных НМРЛ разных стадий: А) уровни белка NEDD9 в образцах когорты РКОД, установленные с помощью анализа h-score данных ИГХ, а также репрезентативные микрофотографии (Б); В) уровни мРНК NEDD9 (относительные z-score), установленные по данным анализа РНК-секвенирования доступных в базе данных TCGA-PanCan на
На графиках: * – p<0,05;
** – p<0,01. Шкала: 200 мкм Fig. 1. Segregation of NEDD9 expression levels of patients with NSCLC at different stages of the disease: A) NEDD9 protein levels in samples from the RCOD cohort as determined by h-score analysis of IHC data, and representative photomicrographs (B); C) NEDD9 mRNA levels (relative z-score) determined from TCGA-PanCan RNAseq dataset available at All graphs: * – p<0.05; ** – p<0.01.
Scale: 200 µm
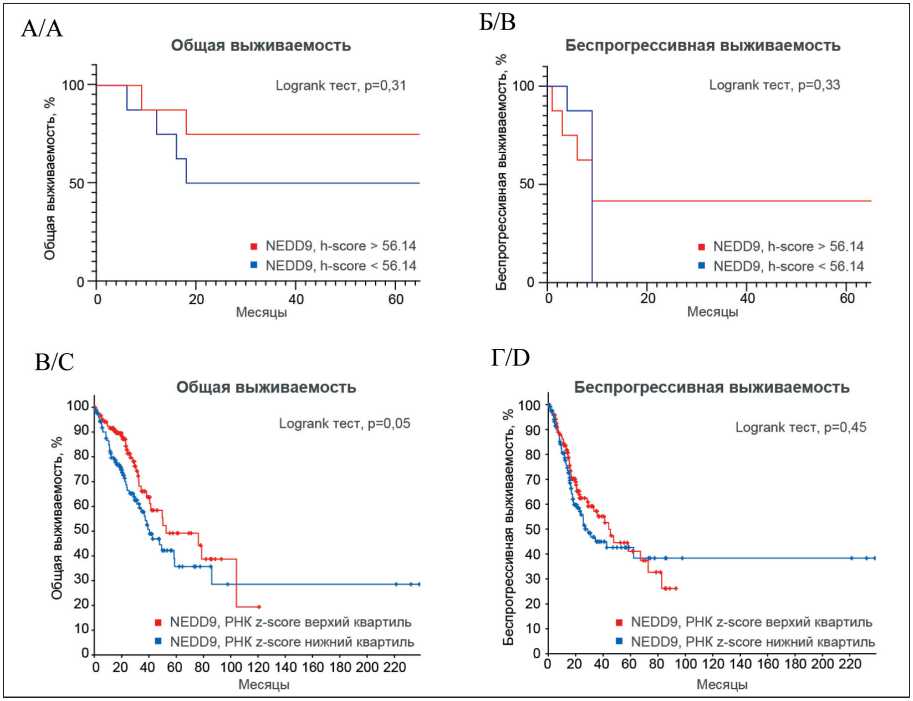
Рис. 2. Корреляция экспрессии NEDD9 в исследуемых когортах НМРЛ с ОВ и БПВ. Кривые общей (ОВ) и бесспрогрессивной (БПВ) выживаемости по методу Каплана–Мейера на основе данных h-score экспрессии белка NEDD9 в образцах когорты РКОД (А, Б) и значений z-score мРНК NEDD9, доступных в базе данных TCGA-PanCan на (В, Г)
Fig. 2. Correlation of NEDD9 expression with OS and PFS in the NSCLC cohorts. Overall survival (OS) and progression-free survival (PFS) curves plotted using the Kaplan–Meier method, based on h-score data of NEDD9 protein expression in RCOD cohort samples (A, B) and NEDD9 mRNA z-score data available in the TCGA-PanCan dataset at (C, D)
стороны, объяснить различия между экспрессией белка (оцененной с помощью ИГХ) и РНК (РНК-секвенирование) NEDD9 на различных стадиях. С другой стороны, эта разница может указывать на то, что на разных этапах прогрессирования НМРЛ в опухолевых клетках активируются биологические механизмы, зависящие поэтапно от уровней РНК и белка NEDD9.
Полученные нами данные, а также анализ литературы позволяют потенциально номинировать NEDD9 в качестве нового прогностического маркера течения заболевания у пациентов с НМРЛ II–III стадий. Одним из достоинств данного исследования является то, что для оценки экспрессии NEDD9 использован метод ИГХ, рутинно применяющийся в клинической практике, в отличие от РНК-секвенирования, которое пока не получило широкого распространения. Достоинствами последнего являются возможность аннотирования профилей экспрессии большой панели генов, обнаружение новых сигнатур экспрессии, специфичных для отдельных групп клеток, однако, учитывая наличие опухолевой гетерогенности, метод ИГХ позволяет провести качественную оценку большей опухолевой поверхности и может служить дополнительным верификационным критерием результатов РНК-секвенирования [24]. Несмотря на ограниченный размер выборки образцов ИГХ, нами был обнаружен тренд к снижению выживаемости пациентов со сниженным уровнем экспрессии белка NEDD9, что, несомненно, указывает на необходимость проведения последующих ИГХ-исследований с использованием расширенных когорт образцов.
Заключение
На основании результатов исследования можно сделать вывод о том, что измерение уровня экспрессии поддерживающего белка NEDD9 у пациентов с НМРЛ может быть использовано в качестве нового прогностического маркера течения заболевания на ранних стадиях. Дальнейшие исследования значимости этого белка с использованием более широкой экспериментальной выборки представляют значительный интерес.
Список литературы Экспрессия поддерживающего белка NEDD9 как негативный прогностический маркер при немелкоклеточном раке легкого (НМРЛ)
- SiegelR.L., MillerK.D., Jemal A. Cancer statistics, 2019. CA Cancer J Clin. 2019; 69(1): 7-34. doi: 10.3322/caac.21551.
- SEER Cancer Statistics Review, 1975-2016. National Cancer Institute. Bethesda, MD.
- Law S.F., Estojak J., WangB., Mysliwiec T., Kruh G., GolemisE.A. Human enhancer of filamentation 1, a novel p130cas-like docking protein, associates with focal adhesion kinase and induces pseudohyphal growth in Saccharomyces cerevisiae. Mol Cell Biol. 1996; 16(7): 3327-37. doi: 10.1128/MCB.16.7.3327.
- Minegishi M., Tachibana K., Sato T., Iwata S., Nojima Y., Mo-rimoto C. Structure and function of Cas-L, a 105-kD Crk-associated substrate-related protein that is involved in beta 1 integrin-mediated signaling in lymphocytes. J Exp Med. 1996; 184(4): 1365-75. doi: 10.1084/ jem.184.4.1365.
- vanSeventerGA, Salmen H.J., LawS.F., O'NeillG.M.,MullenM.M, Franz A.M., Kanner S.B., GolemisE.A., van Seventer J.M. Focal adhesion kinase regulates beta1 integrin-dependent T cell migration through an HEF1 effector pathway. Eur J Immunol. 2001; 31(5): 1417-27. doi: 10.1002/1521-4141(200105)31:5-1417::AID-IMMU1417>3.0.C0;2-C.
- BuiL.C., Tomkiewicz C., Chevallier A., Pierre S., BatsA.S., Mota S., Raingeaud J., Pierre J., Diry M., Transy C., Garlatti M., Barouki R., CoumoulX. Nedd9/Hef1/Cas-L mediates the effects of environmental pollutants on cell migration and plasticity. Oncogene. 2009; 28(41): 3642-51. doi: 10.1038/onc.2009.224.
- Kim L.C., Song L., Haura E.B. Src kinases as therapeutic targets for cancer. Nat Rev Clin Oncol. 2009; 6(10): 587-95. doi: 10.1038/ nrclinonc.2009.129.
- Ombrato L., Malanchi I. The EMT universe: space between cancer cell dissemination and metastasis initiation. Crit Rev Oncog. 2014; 19(5): 349-61. doi: 10.1615/critrevoncog.2014011802.
- Singh A., Settleman J. EMT, cancer stem cells and drug resistance: an emerging axis of evil in the war on cancer. Oncogene. 2010; 29(34): 4741-51. doi: 10.1038/onc.2010.215.
- Smith B.N., BhowmickN.A. Role of EMT in Metastasis and Therapy Resistance. J Clin Med. 2016; 5(2):17. doi: 10.3390/jcm5020017.
- Morimoto K., Tanaka T., Nitta Y., Ohnishi K., Kawashima H., Nakatani T. NEDD9 crucially regulates TGF-ß-triggered epithelial-mes-enchymal transition and cell invasion in prostate cancer cells: involvement in cancer progressiveness. Prostate. 2014; 74(8): 901-10. doi: 10.1002/ pros.22809.
- Feng J., Zhao J., Xie H., Yin Y., Luo G., Zhang J., Feng Y., Li Z. Involvement of NEDD9 in the invasion and migration of gastric cancer. Tumour Biol. 2015; 36(5): 3621-8. doi: 10.1007/s13277-014-2999-1.
- 13.Miao Y., LiA.L., WangL., Fan C.F., ZhangX.P., XuH.T., YangL.H., Liu Y., Wang E.H. Overexpression of NEDD9 is associated with altered expression of E-Cadherin, ß-Catenin and N-Cadherin and predictive of poor prognosis in non-small cell lung cancer. Pathol Oncol Res. 2013; 19(2): 281-6. doi: 10.1007/s12253-012-9580-2.
- Topchu Y.A., Mazitova A.M., Tikhomirova M.V., Abramova Z.I., Deneka A.Y. Nedd9 Regulates Metastasis of Non-Small Cell Lung Cancer through Activation of Epithelial-Mesenchymal Transition and Tumor Cells Migration. Uchenye Zapiski Kazanskogo Universiteta. Seriya Estestvennye Nauki. 2020. 162(1): 123-33.
- DenekaA.Y., KoppM.C., NikonovaA.S., GaponovaA.V., Kisele-vaA.A., HensleyH.H., FliederD.B., SerebriiskiiI.G., GolemisE.A. Nedd9 Restrains Autophagy to Limit Growth of Early Stage Non-Small Cell Lung Cancer. Cancer Res. 2021; 81(13): 3717-26. doi: 10.1158/0008-5472. CAN-20-3626.
- Shagisultanova E., Gaponova A.V., Gabbasov R., Nicolas E., Golemis E.A. Preclinical and clinical studies of the NEDD9 scaffold protein in cancer and other diseases. Gene. 2015; 567(1): 1-11. doi: 10.1016/j. gene.2015.04.086.
- Bankhead P., Loughrey M.B., Fernández J.A., Dombrowski Y., McArt D.G., Dunne P.D., McQuaid S., Gray R.T., Murray L.J., Coleman H.G., James J.A., Salto-TellezM., Hamilton P. W. QuPath: Open source software for digital pathology image analysis. Sci Rep. 2017; 7(1): 16878. doi: 10.1038/s41598-017-17204-5.
- Hirsch F.R., Varella-Garcia M., Bunn P.A. Jr., Di Maria M.V., Veve R., Bremmes R.M., Barón A.E., Zeng C., Franklin W.A. Epidermal growth factor receptor in non-small-cell lung carcinomas: correlation between gene copy number and protein expression and impact on prognosis. J Clin Oncol. 2003; 21(20): 3798-807. doi: 10.1200/JC0.2003.11.069.
- Gao J., Aksoy B.A., Dogrusoz U., Dresdner G., Gross B., Sumer S.O., Sun Y., Jacobsen A., Sinha R., Larsson E., Cerami E., Sander C., Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013; 6(269). doi: 10.1126/scisig-nal.2004088.
- Wu Y.L., John T., Grohe C., Majem M., Goldman J.W., Kim S.W., Kato T., Laktionov K., Vu H.V., Wang Z., Lu S., Lee K.Y., Akewanlop C., Yu C.J., de Marinis F., Bonanno L., Domine M., Shepherd FA., Zeng L., Atasoy A., Herbst R.S., Tsuboi M. Postoperative Chemotherapy Use and Outcomes From ADAURA: Osimertinib as Adjuvant Therapy for Resected EGFR-Mutated NSCLC. J Thorac Oncol. 2022; 17(3): 423-33. doi: 10.1016/j.jtho.2021.10.014.
- Tian Y., Xu J., Chu Q., Duan J., Zhang J., Bai H., Yang Z., Fang W., Cai L., Wan R., Fei K., He J., Gao S., Zhang L., Wang Z., Wang J. A novel tumor mutational burden estimation model as a predictive and prognostic biomarker in NSCLC patients. BMC Med. 2020; 18(1): 232. doi: 10.1186/ s12916-020-01694-8.
- Camidge D.R., Doebele R.C., Kerr K.M. Comparing and contrasting predictive biomarkers for immunotherapy and targeted therapy of NSCLC. Nat Rev Clin Oncol. 2019; 16(6): 341-55. doi: 10.1038/ s41571-019-0173-9.
- KudinovA.E., Karanicolas J., GolemisE.A., Boumber Y. Musashi RNA-Binding Proteins as Cancer Drivers and Novel Therapeutic Targets. Clin Cancer Res. 2017; 23(9): 2143-53. doi: 10.1158/1078-0432.CCR-16-2728.
- ByronSA., VanKeuren-JensenK.R., Engelthaler D.M., CarptenJ.D., Craig D.W. Translating RNA sequencing into clinical diagnostics: opportunities and challenges. Nat Rev Genet. 2016; 17(5): 257-71. doi: 10.1038/nrg.2016.10.


