ЭЛЕКТРОХИМИЧЕСКОЕ ДЕТЕКТИРОВАНИЕ ОДИНОЧНЫХ МОЛЕКУЛ ДНК С ПОМОЩЬЮ ТВЕРДОТЕЛЬНОЙ НАНОПОРЫ
Автор: П. К. Афоничева, Н. В. Ваулин, Д. В. Лебедев, А. С. Букатин, Д. А. Кириленко, А. Н. Аблеев, Ю. Д. Иванов, И. С. Мухин, А. А. Евстрапов,
Журнал: Научное приборостроение @nauchnoe-priborostroenie
Рубрика: Научные статьи, посвященные памяти Л.Н. Галль
Статья в выпуске: 4, 2024 года.
Бесплатный доступ
В работе продемонстрирована возможность электрохимического детектирования одиночных молекул ДНК с помощью тонкой SiN-мембраны с интегрированной наноразмерной порой. Были проведены эксперименты по регистрации ионного тока для буферного раствора без/с добавлением молекул ДНК 10 000, 5000 и 500 пар оснований (п.о.). Для анализа полученных данных разработан алгоритм поиска событий транслокаций одиночных молекул через нанопору. Такой алгоритм позволяет анализировать амплитуду, длительность и частоту обнаруженных событий с целью анализа детектируемых молекул. Для ДНК 5000 и 500 п.о показана нелинейная зависимость продолжительности прохождения молекул от их длины. Продемонстрировано, что при уменьшении длины молекул в 10 раз продолжительность транслокаций снижается на 35%. Продемонстрированная возможность детектирования транслокаций создает основу для создания твердотельного нанопорового сенсора.
Нанопора, твердотельная нанопора, микрофлюидика, ионный транспорт, SiN-мембраны, биосенсоры, ДНК, транслокация ДНК
Короткий адрес: https://sciup.org/142242731
IDR: 142242731 | УДК: 543.08+543.55
Текст научной статьи ЭЛЕКТРОХИМИЧЕСКОЕ ДЕТЕКТИРОВАНИЕ ОДИНОЧНЫХ МОЛЕКУЛ ДНК С ПОМОЩЬЮ ТВЕРДОТЕЛЬНОЙ НАНОПОРЫ
В настоящее время наблюдается повышенный интерес к междисциплинарным исследованиям, направленным на разработку новых устройств с интегрированными микро- и наноструктурами для биомедицинских приложений [1]. Для реализации подобных устройств применяется сочетание методов микро- и нанофлюидики с технологиями микроэлектроники, что позволяет создавать высокочувствительные сенсоры [2], принцип действия которых основан на взаимодействии исследуемого объекта с твердотельными наноразмерными структурами. Одним из наиболее перспективных направлений развития такого подхода является формирование наноразмерных отверстий (нанопор) в тонких, непроницаемых мембранах [3]. Изготовление нанопор с характерными величинами диаметра/длины от 1 до 100 нм открывает широкий спектр возможностей для их применения в биомедицине: концентрации и выделения нуклеиновых кислот (НК) [4, 5], анализа и детектирования одиночных биомолекул [6], секвенирования НК [7, 8].
Наиболее распространенный принцип нанопо-рового детектирования заключается в регистрации ионного тока, протекающего через нанопору под действием внешнего электрического поля [9]. Детектирование происходит в измерительной электрохимической ячейке, представляющей собой независимые (сis и trans) объемы, между которыми фиксируется тонкая мембрана с интегрированной нанопорой. Приложенное электрическое поле формирует направленный транспорт исследуемых молекул из cis-объема в trans-объем через внутренний объем нанопоры. Процесс перемещения молекул через нанопору (событие транслокации) сопровождается скачкообразным изменением (модуляцией) уровня регистрируемого ионного тока. Амплитуда и форма события транслокации зависят от формы и размеров детектируемой молекулы, а также от ее жесткости, заряда и конформационных свойств [10].
Прямыми конкурентами твердотельных нанопор применительно к секвенированию являются биологические нанопоры [11], представляющие собой искусственно синтезированные транспортные белки (α-HL, AeL, ClyA) [12], интегрированные в липидные мембраны. Несмотря на то, что биологические поры активно применяются для секвенирования ДНК [13] и исследования трансмембранного транспорта биологических молекул, они обладают рядом существенных недостатков, такими как ограниченный срок службы и строгие требования к условиям эксплуатации, хранения (температура, концентрация электролита и pH) [9], что значительно сужает возможности их применения. В свою очередь твердотельные нанопоры и наноканалы являются перспективным аналогом, поскольку обладают хорошей механической, тер- мической и химической стабильностью [14, 15], широкими возможностями по модификации их сенсорных и селективных свойств [16], а также хорошим технологическим потенциалом для серийного производства.
Несмотря на ряд преимуществ, твердотельные нанопоры еще не продемонстрировали сенсорные свойства, необходимые для секвенирования ДНК. Основным препятствием этому выступают высокая скорость транслокации молекул в сочетании со сложным механизмом взаимодействия между молекулой ДНК (содержащей как гидрофобные основания, так и гидрофильную основную цепь) и поверхностью поры. Эти эффекты приводят к регистрации коротких сигналов с низком соотношением сигнал/шум, что не позволяет реализовать эффективную идентификацию отдельных нуклеотидов в ДНК. Разработка твердотельных нанопоровых датчиков, способных полностью охарактеризовать детектируемые биомолекулы, является сложной задачей из-за высокой скорости электрофоретической транслокации [17], вызванной сильной локализацией электрического поля в области поры. Сильные поля обусловлены наноразмерами структуры и являются необходимым фактором для формирования уровня тока, превышающего тепловой шум, в то время как для преодоления барьеров, определяемых конформационной энтропией, электростатическим отталкиванием и электроосмотическим потоком, требуется приложить напряжение смещения порядка не менее 20–60 мВ [18].
Типичные скорости транслокации НК через твердотельные нанопоры составляют 1 основание в 10–1000 нс [19]. При таких высоких скоростях короткие фрагменты нуклеиновых кислот (< 100 пар оснований, п.о.) часто невозможно обнаружить. Таким образом, для регистрации сигнала необходима широкая полоса пропускания сигнала (> 1 МГц), что в свою очередь приводит к увеличению уровня тепловых шумов и затрудняет детектирование. Часто для снижения скорости транслокаций применяются дополнительные механизмы, такие как изменение вязкости электролита [20], использование оптического пинцета или плазмонных структур [21].
Помимо оптимизации условий эксперимента и методов детектирования решающее значение имеет обработка полученных сигналов и определение события транслокации. Большинство существующих методов обнаружения событий представляют собой идентификацию скачкообразных изменений уровня ионного тока. Изменения тока вычисляются/ранжируются и отсекаются при достижении определенного порогового значения (триггер). Такие методы поиска событий можно разделить на две категории: классический подход определения пользователем порогового значения с помощью таких программ, как MiniAnalysis [22], Easy Electrophysiology [23] и Clampfit [24], и более продвинутые методы, использующие адаптивные или локальные пороги, исходя из полученных данных, предполагая, что базовый ток стабилен во времени, реализованные в программах OpenNanopore [25], MOSAIC [26], Transalyzer [27], EasyNanopore [28].
Данная работа посвящена исследованию возможности детектирования одиночных молекул ДНК различной длины с помощью наноразмерной SiN-поры, а также статистическому анализу событий транслокаций с целью изучения механизмов прохождения молекул ДНК через внутренний объем нанопоры.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использованы образцы, представляющие собой свободно подвешенную SiN-мембрану толщиной 40 нм с одиночной нанопорой диаметром ~5 нм. На первом этапе синтеза структуры слой SiN толщиной 40 нм формировался на отожженной Si-подложке методом химического осаждения из газовой фазы при пониженном давлении (LP-CVD). Далее с использованием методов плазмохимического и анизотропного травления Si-подложка удалялась, формируя свободно подвешенную SiN-мембрану площадью ~900 мкм2.
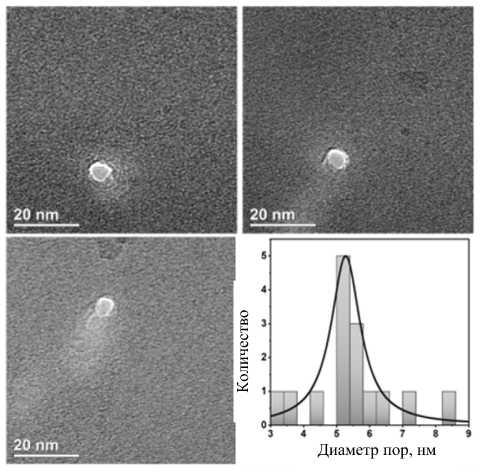
Рис. 1. ПЭМ-изображения трех SiN-нанопор длиной 40 нм и диаметром ~5 нм и распределение диаметров формируемых нанопор по размерам
Формирование нанопор в полученной мембране происходило методом травления сфокусированным электронным пучком с помощью просвечивающего электронного микроскопа (ПЭМ). Данный метод позволяет формировать твердотельные нанопоры необходимых размеров с хорошей воспроизводимостью (рис. 1).
В экспериментах по детектированию молекул ДНК использовалась разработанная нами измерительная ячейка, представляющая собой два независимых cis- и trans-объема, формируемые методом фотополимерной 3D-печати. В ячейку устанавливались Ag/AgCl электроды для проведения измерений по двухэлектродной схеме, а также система капилляров для подачи/отвода буферного раствора электролита. Мембрана с интегрированной нанопорой герметично фиксировалась в ячейке с помощью эластичных прокладок, соединяя cis- и trans-объемы. В процессе заполнения буферным раствором измерительная ячейка проходила процедуру дегазации для предотвращения образования микропузырей воздуха вблизи нанопоры. Методика заполнения ячейки была отработана и подробно описана в предыдущих работах [29].
В качестве буферного электролита использовался раствор 0.1 M KCl и 1X PBS (1:1), pH = 7.4. Измерения ионного тока проводились с помощью источника-измерителя напряжения и тока, позволяющего проводить измерения с частотой 50 кГц. Исследуемыми молекулами в экспериментах выступали двухцепочечные ДНК разной длины: 10000 п.о., 5000 п.о. и 500 п.о (ЗАО "СИНТОЛ").
ЭКСПЕРИМЕНТАЛЬНЫЕ ИССЛЕДОВАНИЯ
На первом этапе была исследована возможность нанопорового детектирования молекул ДНК 10000 п.о. Для этого был проведен эксперимент, направленный на обнаружение транслокаций после добавления молекул ДНК в чистый буферный раствор. Сначала была проанализирована стабильность ионного тока в чистом буферном растворе до добавления ДНК. Измерения тока осуществлялись в потенциостатическом режиме (приложенная разность потенциалов фиксирована во времени). На полученной потенциостатической кривой зависимости тока от времени наблюдался уровень тока 244.9 пА при среднеквадратичном отклонении (СКО) 10.6 пА ( U = 0.1 В), что говорит о высокой чистоте раствора, обеспечивающей отсутствие паразитных событий транслокаций. Далее, в объем ячейки, включающий в себя отрицательно заряженный электрод (cis-объем см. рис. 2), был добавлен буферный раствор, содержащий молекулы ДНК длиной 10 000 п.о. в концентрации ~0.1 нг/мкл. Под действием приложенного элек-
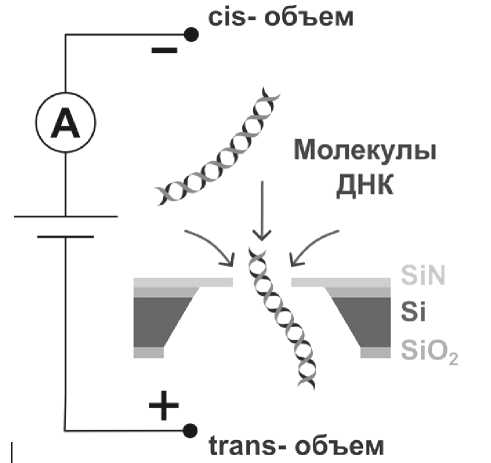
Рис. 2. Схема эксперимента по детектированию одиночных молекул ДНК трического поля отрицательно заряженные молекулы ДНК перемещались через нанопору из cis-в trans-объем (рис. 2). При этом средний уровень ионного тока составил 218.3 пА, при СКО 7.5 пА. После чего были зарегистрированы короткие скачкообразные изменения уровня ионного тока (события транслокаций), что говорит о возникновении транспорта молекул ДНК через внутренний объем нанопоры.
Вторым этапом работы стало исследование зависимости продолжительности событий транслокаций от длины молекул ДНК. Эксперимент был проведен аналогично первому этапу для молекул ДНК длиной 5000 и 500 п.о. в концентрациях 1.25 нг/мкл и 3.3 нг/мкл соответственно. Для увеличения отношения сигнал/шум в данном эксперименте было приложено напряжение 0.3 В. Для обеспечения чистоты эксперимента измерения были проведены на другом образце SiN-нанопоры диаметром ~5 нм.
РЕЗУЛЬТАТЫ
Для идентификации событий транслокаций было необходимо провести анализ результатов нано-порового молекулярного детектирования. С этой целью измеренные зависимости тока от времени-были охарактеризованы в три этапа: построение и вычитание базовой линии, поиск событий транслокаций по пороговому значению тока, фильтра- ция событий и последующий анализ достоверных данных.
Сначала, используя ПО OriginLab, задавалась базовая линия тока методом (фильтром) Савицкого – Голея [30], принцип действия которого заключается в проведении локальной полиномиальной регрессии вокруг каждой точки (значения тока) и создании нового сглаженного значения для этих точек. Метод Савицкого – Голея позволяет сохранить амплитуду и длительность коротких событий транслокации, при этом нивелировать влияние длительных изменений уровня тока, связанных с электрохимическими особенностями измерений ионного тока по двухэлектродной схеме. После сглаживания кривой значение базовой линии вычиталось из значения тока, формируя массив данных, удобный для автоматического поиска событий транслокаций.
На втором шаге, с помощью программы Clampfit (Molecular Devices) производился автоматический поиск событий транслокаций, удовлетворяющих заданным параметрам. Минимально необходимые параметры обнаружения событий включают в себя значения базовой линии и значение триггера (trigger). Поскольку мы исследуем сглаженные данные, значение базовой линии устанавливается, равное 0. Триггер — это пороговое значение тока, задаваемое как It = SD · x, где SD — среднеквадратичное отклонение, x — произвольный коэффициент, значение которого влияет на эффективность детектирования событий транслокации. Слишком маленькое значение коэффициента приводит к детектированию паразитных сигналов, в то время как слишком большое приводит к потере достоверных событий транслокаций. Исходя из этих соображений, нами было выбрано оптимальное значение x = 4.5.
Дополнительные параметры помогают точнее настроить поиск. Значение Re-arm , определяющее окончание события, устанавливалось равным 1 пА: данное значение наиболее близко к значению базовой линии, что необходимо для достоверного определения продолжительности событий. Значение Rejection (отклонение) используется для отсечения событий большой амплитуды, связанных с внешними наводками и/или артефактами измерений. Дополнительно устанавливались значения пре-триггера ( pre-trigger ) и пост-триггера ( post-trigger ), помогающие добавить фиксированные участки до и после события для получения информации о форме обнаруженного сигнала. В нашем случае T pre-trigger = T post-trigger = 2 мс. В результате поиска транслокаций формировался массив данных, содержащий информацию о каждом обнаруженном событии.
Третий шаг обработки полученных данных представляет собой фильтрацию найденных событий. Для этого вручную удалялись кластеры шумовых событий, а также автоматически удалялись артефакты поиска и события, удовлетворяющие соотношению I peak > 2 I antipeak . Такое соотношение необходимо для удаления симметричных событий, являющихся следствием электрических помех.
В ходе анализа событий транслокаций молекул ДНК 10000 п.о. была построена зависимость амплитуды транслокаций от их продолжительности (рис. 3), демонстрирующая возможность одномолекулярного детектирования.
До добавления ДНК, +100 мВ 0.06 событий/с
После добавления ДНК, +100 мВ ☆ 0.25 событий/с
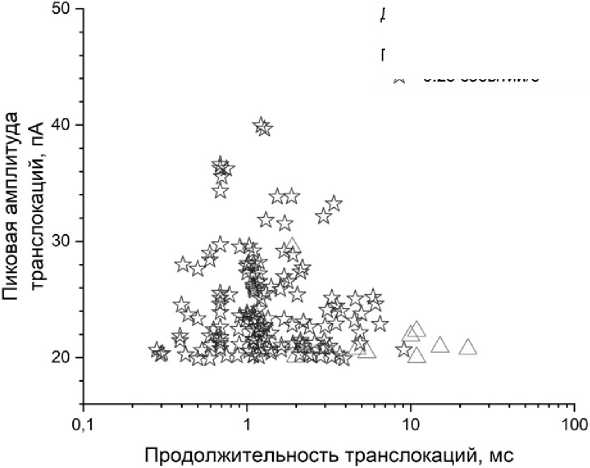
Рис. 3. Зависимость пиковой амплитуды транслокаций от продолжительности транслокаций для молекул ДНК 10000 п.о. при U = 0.1 В
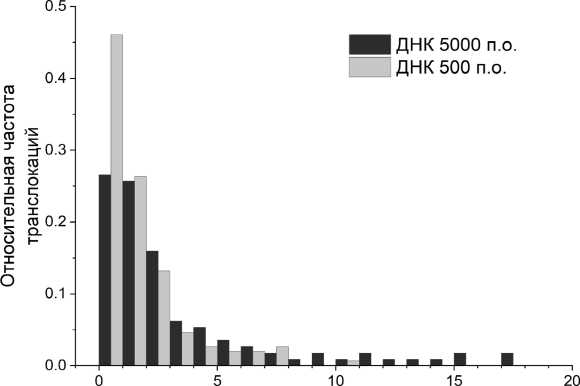
Продолжительность транслокации, мс
Рис. 4. Распределение событий транслокаций по продолжительности для молекул ДНК 5000 и 500 п.о.
Частота событий до/после добавления ДНК изменилась с 0.06 событие/с до 0.25 событие/с. Кроме того, события после добавления ДНК характеризуются более продолжительными транслокациями, что может указывать на прохождение через пору крупных молекулярных соединений. Значительный разброс амплитуды и длительности транслокаций для молекул одной длины может быть связан с различной конформацией (полностью распутанная и частично запутанная) молекул в момент прохождения через внутренний объем поры.
Анализ экспериментальных данных, полученных в ходе эксперимента с молекулами ДНК длинами 5000 и 500 п.о., показал нелинейную зависимость продолжительности транслокаций от длины молекул (рис. 4). Медианное значение времени транслокаций для 5000 и 500 п.о. составило 1.7 мс и 1.09 мс соответственно, что характеризует уменьшение времени транслокаций для более коротких молекул на 35%. Это может быть связано со сложным и не до конца изученным механизмом прохождения молекул ДНК через внутренний объем поры, в котором существенную роль играют процессы взаимодействия молекул со стенками поры. Можно предположить, что взаимодействие молекул ДНК со стенками нанопоры оказывает доминирующее влияние на длительность транслокаций по сравнению с длиной молекул.
ЗАКЛЮЧЕНИЕ
В работе была продемонстрирована возможность электрического детектирования одиночных молекул ДНК в твердотельной нанопоре. С помощью разработанной измерительной ячейки были получены зависимости ионного тока от времени для буфера без/с добавлением ДНК. Для анализа событий транслокаций использован алгоритм, заключающийся в обнаружении событий по пороговому значению. С его помощью были проанализированы события транслокаций для ДНК 10000 п.о., построена зависимость амплитуды транслокаций от их длительности и оценена частота событий. Проведены эксперименты по детектированию молекул ДНК 5000 и 500 п.о., обнаружена нелинейная зависимость длительности событий транслокаций от длины молекулы. Продемонстрировано, что при уменьшении длины молекул в 10 раз продолжительность транслокаций снижается на 35%.
Исследование поддержано Российским научным фондом (проект № 20-74-10117, : формирование нанопор, измерение ионных токов, анализ данных. Н.В. Ваулин и И.С. Мухин благодарят за финансовую поддержку грант Министерства образования и науки (FSRM-2023-0007): синтез образцов с твердотельными SiN-мембранами.
Список литературы ЭЛЕКТРОХИМИЧЕСКОЕ ДЕТЕКТИРОВАНИЕ ОДИНОЧНЫХ МОЛЕКУЛ ДНК С ПОМОЩЬЮ ТВЕРДОТЕЛЬНОЙ НАНОПОРЫ
- 1. Liu H., Zhou Q., Wang W., Fang F., Zhang J. Solid-State Nanopore Array: Manufacturing and Applications // Small. 2023. Vol. 19, iss. 6. Id. 2205680. DOI: 10.1002/smll.202205680
- 2. Kan X., Wu C., Wen L., Jiang, L. Biomimetic nanochannels: From fabrication principles to theoretical insights // Small Methods. 2022. Vol. 6, no. 4. Id. 2101255. DOI: 10.1002/smtd.202101255
- 3. Kang Q., Guo W. Biomimetic smart nanopores and nanochannels // Chemically Modified Nanopores and Nanochannels. 2017. P. 85–102. DOI: 10.1016/B978-0-323-40182-1.00004-X
- 4. Ouyang W., Han J. Universal amplification-free molecular diagnostics by billion-fold hierarchical nanofluidic concentration // Proceedings of the National Academy of Sciences. 2019. Vol. 116, no. 33. P. 16240–16249. DOI: 10.1073/pnas.1904513116
- 5. Zhang X., Dou H., Chen X., Lin M., Dai Y., Xia F. Solidstate nanopore sensors with enhanced sensitivity through nucleic acid amplification // Analytical Chemistry. 2023. Vol. 95, no. 47. P. 17153–17161. DOI: 10.1021/acs.analchem.3c03806
- 6. Shi W., Friedman A.K., Baker L.A. Nanopore sensing // Analytical chemistry. 2017. Vol. 89, no. 1. P. 157–188. DOI: 10.1021/acs.analchem.6b04260
- 7. Sen P., Gupta M. Single nucleotide detection using bilayer MoS2 nanopores with high efficiency // RSC advances.
- 2021. Vol. 11, no. 11. P. 6114–6123. DOI: 10.1039/d0ra10222a
- 8. Yang H., Li Z., Si W., Lin K., Ma J., Li K., Sun L., Sha J., Chen Y. Identification of single nucleotides by a tiny charged solid-state nanopore // The Journal of Physical Chemistry B. 2018. Vol. 122, no. 32. P. 7929–7935. DOI: 10.1021/acs.jpcb.8b06056
- 9. He Y., Tsutsui M., Zhou Y., Miao X.S. Solid-state nanopore systems: from materials to applications // NPG Asia Materials. 2021. Vol. 13, no. 1. Id. 48. DOI: 10.1038/s41427-021-00313-z
- 10. Goto Y., Akahori R., Yanagi I., Takeda K.I. Solid-state nanopores towards single-molecule DNA sequencing // J Hum Genet. 2020. Vol. 65, no 1. P. 69–77. DOI: 10.1038/s10038-019-0655-8
- 11. Deamer D., Akeson M., Branton D. Three decades of nanopore sequencing // Nature biotechnology. 2016. Vol. 34, no. 5. P. 518–524. DOI: 10.1038/nbt.3423
- 12. Lee K., Park K.B., Kim H.J., Yu J.S., Chae H., Kim H.M., Kim K.B. Recent progress in solid state nanopores // Advanced materials. 2018. Vol. 30, no. 42. Id. 1704680. DOI: 10.1002/adma.201704680
- 13. Asandei A., Di Muccio G., Schiopu I., Mereuta L., Dragomir I.S., Chinappi M., Luchian T. Nanopore-Based Protein Sequencing Using Biopores: Current Achievements and Open Challenges // Small Methods. 2020. Vol. 4, iss. 11. Id. 1900595. DOI: 10.1002/smtd.201900595
- 14. Hu R., Tong X., Zhao Q. Four aspects about solid-state nanopores for protein sensing: fabrication, sensitivity, selectivity, and durability // Advanced healthcare materials. 2020. Vol. 9, no. 17. Id. 2000933. DOI: 10.1002/adhm.202000933
- 15. Xue L., Yamazaki H., Ren R., Wanunu M., Ivanov A.P., Edel J.B. Solid-state nanopore sensors // Nature Reviews Materials. 2020. Vol. 5. P. 931–951. DOI: 10.1038/s41578-020-0229-6
- 16. Lebedev D., Malyshev G., Ryzhkov I., Mozharov A., Shugurov K., Sharov V., Panov M., Tumkin I., Afonicheva P., Evstrapov A., Bukatin A., Mukhin, I. Focused ion beam milling based formation of nanochannels in silicon-glass microfluidic chips for the study of ion transport. // Microfluidics and nanofluidics. 2021. Vol. 25. Id. 51. DOI: 10.1007/s10404-021-02450-x
- 17. Dekker C. Solid-state nanopores // Nature nanotechnology. 2007. Vol. 2, no. 4. P. 209–215. DOI: 10.1038/nnano.2007.27
- 18. Fologea D., Uplinger J., Thomas B., McNabb D.S., Li J. Slowing DNA translocation in a solid-state nanopore // Nano letters. 2005. Vol. 5, no. 9. P. 1734–1737. DOI: 10.1021/nl051063o
- 19. Plesa C., Kowalczyk S.W., Zinsmeester R., Grosberg A.Y., Rabin Y., Dekker C. Fast translocation of proteins through solid state nanopores // Nano letters. 2013. Vol. 13, no. 2. P. 658–663. DOI: 10.1021/nl3042678
- 20. Kawaguchi T., Tsutsui M., Murayama S., Leong I.W., Yokota K., Komoto Y., Taniguchi M. Enhanced Nanoparticle Sensing in a Highly Viscous Nanopore // Small Methods. 2024. Vol. 8, iss. 8. Id. 2301523. DOI: 10.1002/smtd.202301523
- 21. Yuan Z., Liu Y., Dai M., Yi X., Wang, C. Controlling DNA translocation through solid-state nanopores // Nanoscale research letters. 2020. Vol. 15. Id. 80. DOI: 10.1186/s11671-020-03308-x
- 22. Mini Analysis Program (RRID: SCR_002184). URL: https://scicrunch.org/resolver/SCR_002184 (дата обращения 28.07.2024).
- 23. Easy Electrophysiology makes patch-clamp analysis simple. URL: https://www.easyelectrophysiology.com/ (дата обращения 28.07.2024).
- 24. Advancing scientific discovery. URL: https://www.moleculardevices.com/ (дата обращения 28.07.2024).
- 25. Raillon C., Granjon P., Graf M., Steinbocka L.J., Radenovic A. Fast and automatic processing of multi-level events in nanopore translocation experiments // Nanoscale. 2012. Vol. 4, no. 16. P. 4916–4924. DOI: 10.1039/C2NR30951C
- 26. Forstater J.H., Briggs K., Robertson J.W.F., Ettedgui J., Marie-Rose O., Vaz C., Kasianowicz J.J., TabardCossa V., Balijepalli A. MOSAIC: A Modular Single Molecule Analysis Interface for Decoding Multi-state Nanopore Data // Analytical chemistry. 2016, Vol. 88, no. 23.
- P. 11900–11907. DOI: 10.1021/acs.analchem.6b03725
- 27. Plesa C., Dekker C. Data analysis methods for solid-state nanopores // Nanotechnology. 2015. Vol. 26, no. 8. Id. 084003. DOI: 10.1088/0957-4484/26/8/084003
- 28. Tu J., Meng H., Wu L., Xi G., Fu J., Lu Z. EasyNanopore: a ready-to-use pocessing software for translocation events in nanopore translocation experiments // Langmuir. 2021. Vol. 37, iss. 33. P. 10177–10182. DOI: 10.1021/acs.langmuir.1c01597
- 29. Lebedev D.V., Vaulin N.V., Afonicheva P.K., Vorobyov A.A., Mozharov A.M., Sharov V.A., Mikhailovskii V.Yu., Kirilenko D.A., Permyakov D.V., Logunov L.S., Ivanov Yu.D., Ryzhkov I.I., Bukatin A.S., Evstrapov A.A., Makarov S.V., Mukhin I.S. Ultra-Low Intensity LightDriven Ionic Conductivity through a Plasmonic Nanopore // ACS Applied Nano Materials. 2024. Vol. 7, no. 14. P. 16172–16181. DOI: 10.1021/acsanm.4c02108
- 30. Savitzky A., Golay M.J.E. Smoothing and differentiation of data by simplified least squares procedures // Analytical chemistry. 1964. Vol. 36, no. 8. P. 1627–1639. DOI: 10.1021/ac60214a047


