Эпизоотология, генетика и эволюция цирковируса свиней типа 2 в Китае
Автор: Юйцзе Ф.
Статья в выпуске: 2 т.258, 2024 года.
Бесплатный доступ
Генотипический подтип цирковируса свиней типа 2 (PCV2) продолжает эволюционировать, осложняя эпизоотическую ситуацию. Для выяснения характера дрейфа генотипов в эволюции штаммов PCV2 в Китае 2529 полностью кодированных генов ORF2 из базы данных NCBI, собранных в Китае с 2004 по 2023 гг., были классифицированы по году выделения. Результаты показали, что за последние 20 лет в Китае в эволюции PCV2 наблюдались два дрейфа генотипов, причем до 2004 года генотипы дрейфовали от PCV2a. В общей сложности 43 штамма были дополнительно исследованы за период 2018-2023 гг. из трех провинциях Северо-Восточного Китая - Ляонин, Хэйлунцзян и Цзилинь, которые прилегают к России. Среди них генотип PCV2a составил 18,6 %, генотип PCV2b - 27,9 %, а генотип PCV2d - 53,4 %. Это указывает на то, что генетическое разнообразие PCV2 увеличилось в Северо-Восточном Китае, а PCV2b и PCV2d стали основными генотипами, распространенными в этот период. Сравнение аминокислот гена ORF2, кодируемого последовательностью вирулентных штаммов, показало, что существует пять основных областей вариаций в белке Сар среди вирулентных штаммов. Эти остатки включали аминокислотные участки 51-91, 121-151, 183-191, 206-217 и 232-234.
Цирковирус свиней типа 2, генотип, генетическое разнообразие, эволюция, сар-белок
Короткий адрес: https://sciup.org/142241650
IDR: 142241650 | УДК: 619:616.98:578.8:636.4(510) | DOI: 10.31588/2413_4201_1883_2_258_237
Текст научной статьи Эпизоотология, генетика и эволюция цирковируса свиней типа 2 в Китае
Цирковирусная болезнь свиней 2-го типа (PCVAD) признана во всем мире как одно из наиболее экономически значимых заболеваний свиноводства. Цирковироз свиней, или синдром послеотъемного мультисистемного истощения (сокр. СПМИ, англ. PMWS) – инфекционная болезнь свиней, главным образом поросят-отъёмышей в возрасте 6-14 недель, основные клинические признаки – отставание в росте и истощение, также наблюдают угнетение, одышку, диарею, цианоз ушей, анемичность и желтушность кожи, дерматит. Иногда отмечают атаксию, парез конечностей, врожденный тремор, внезапную гибель поросят. Высокая плотность содержания поросят в станках ведет к распространению цирковироза, при этом заболеваемость может составлять 5-20 %, в отдельных случаях 50-70 %; летальность может достигать 70-80 %. Инкубационный период длится 3-4 недели. Возбудитель цирковироза обладает иммунодепрессивным действием, т.к. он реплицируется в органах и клетках организма, отвечающих за формирование и выработку иммунитета [1, 2, 3, 4, 5].
Цирковирус типа 2 (сокр. ЦВС-2, англ. PCV2) – это небольшой одноцепочечный вирус с циркулярной ДНК без оболочки. Возбудитель цирковирусной болезни свиней относится к семейству Circoviridae, роду Circovirus, в котором различают ЦВС-1, ЦВС-2, ЦВС-3 и ЦВС-4, генетически отличающиеся друг от друга. На основании филогенетического анализа генома PCV2 и его гена ORF2, вирусы PCV2 можно разделить на шесть генотипов: a, b, c, d, e, f [11, 12]. В последние годы полевые изоляты PCV2 были преимущественно генотипов a, b и d. Исследования генома PCV2 были сосредоточены на этих трех генотипах с меньшей распространенностью генотипов c и e. С момента открытия PCV2 претерпел два генотипических сдвига. В середине 2000-х годов в мире генотип преимущественно распространенного штамма сменился с PCV2a на PCV2b [9]. PCV2a и PCV2b демонстрируют высокую степень сходства как на генетическом, так и на аминокислотном уровне, однако было показано, что PCV2b более патогенен и антигенен, чем PCV2a [6, 13]. Второй сдвиг привел к увеличению распространенности генотипа PCV2d в США, Европе, Китае, Южной Корее и Южной Америке. Генотип PCV2c встречается только в Дании и Бразилии [9, 11, 16]. В последние годы сообщалось, что генотип PCV2d является потенциально доминирующим генотипом в США, Китае, Южной Корее и Таиланде [7, 8, 10, 18, 19]. Интересно, что недавний генотипический сдвиг от PCV2b к PCV2d наблюдался в основном у свиней, вакцинированных PCV2.
На мутацию PCV2d может влиять давление вакцины. Однако, существующие вакцины могут только уменьшить репликацию PCV2d, но не устранить ее. Некоторые исследования показали, что генетические варианты PCV2 могут оказывать влияние на вирулентность, что связано с вакцинацией, патогенезом и диагностикой [11, 20]. В связи с этим необходимо изучить вариабельность последовательностей изолятов PCV2, распространенных в Китае.
В данном исследовании мы проанализировали генетическое разнообразие штаммов PCV2 в Китае на основе 3946 последовательностей китайских штаммов PCV2, собранных в период с 2004 по 2023 гг. Нашей целью было выяснить молекулярную эпизоотологию PCV2 в Китае за 20-летний период и уточнить динамику эволюции в Китае.
Материал и методы исследований. Из базы данных NCBI (GenBank) было загружено 3946 последовательностей китайского штамма PCV2 (доступ получен в апреле 2023 г.). Из 3946 последовательностей штаммов PCV2 были удалены неполностью закодированные последовательности генов ORF1 и ORF2. Так было получено 2529 полных кодирующих последовательности ORF2, включая регион и год выделения образца. Затем последовательности были классифицированы в соответствии с годом выделения. Для всего генома штаммов PCV2 за период с 2004 по 2023 гг. было построено филогенетическое дерево, проведена классификация подтипов и рассчитано соотношение каждого подтипа в каждом году, чтобы в конечном итоге определить характер дрейфа генотипов в ходе эволюции PCV2.
Для уточнения эпизоотологического статуса PCV2 в северо-восточном Китае с января 2018 г. по апрель 2023 г. из базы данных NCBI было загружено в общей сложности 515 последовательностей китайских штаммов PCV2, а также собраны последовательности китайских штаммов PCV2 по результатам исследований ряда авторов. Для построения филогенетического дерева для всего генома штаммов PCV2 с целью классификации подтипов использовали программу MEGA 10.0. MEGAlign использовали для анализа генетической эволюционной связи между проверенными последовательностями и последовательностями всего генома, вариабельности генов ORF1 и ORF2 и гомологии референс-штаммов PCV2 в Китае и за его пределами, перечисленных в GenBank.
Геномные последовательности 10 референтных штаммов PCV2 были загружены из базы данных GenBank (Таблица 1). Филогенетическое дерево на основе целых геномов исследуемых штаммов и 10-и референс-штаммов было построено методом соседних соединений (NJ) с использованием модели Juke-cantor в качестве модели нуклеотидных замен в процессе построения, и 1000 реплик набора данных были использованы для определения надежности филогенетического дерева. Анализ последовательности ORF1, кодирующего белок Rep, и ORF2, кодирующего белок Cap цирковируса свиней, был проведен с помощью биологического программного обеспечения DNA MAN. Также был проведен анализ делеций и мутаций оснований.
Таким образом, для выяснения характера генотипического дрейфа в эволюции штаммов PCV2 в Китае 2529 полностью кодированных генов ORF2 были классифицированы по годам выделения, построено филогенетическое дерево для генов ORF2 в штаммах каждого года, и определен характер генотипического дрейфа путем расчета соотношения основных генотипов (PCV2a, PCV2b и PCV2d) в каждом году.
Метод ПЦР использовали для выявления в различных тканях свиней и изучения PCV2d. Брали 200 мкг ткани, измельчали с раствором PBS (буферный раствор). Затем, согласно протоколу, извлекали геномную ДНК. В соответствии с инструкциями набора TIANamp Genomic DNA kit вирусная ДНК из сыворотки была извлечена в качестве матрицы. Праймеры были сконструированы с использованием специфичных для вируса последовательностей, восходящий праймер представлял собой PCV2d - F959:
5′CCCATGCCCTGAATTTCCATA-3′, нисходящий праймер представлял собой
PCV2d – R1311:
5′TAAACTACTCCTCCCGCCATAC-3′, а фрагмент амплификации составлял 353 п.н. Обычные условия ПЦР: предварительная денатурация при 94 °С в течение 2 мин, 30 циклов при 94 °С в течение 30 с, 55 °С в течение 30 с и 72 °С в течение 30 с, всего 35 циклов и, наконец, удлинение при 72 °С в течение 10 мин. Продукты ПЦР идентифицировали с помощью электрофореза в 1 % агарозном геле.
Результат исследований. СевероВосточный Китай включает три провинции, соседствующие с Россией, с хорошо развитым транспортом, большим количеством свиноводческих предприятий и мелких свиноферм с частым ввозом и вывозом свиней, а также низким уровнем биобезопасности. Все эти факторы создают возможности для распространения, мутации и рекомбинации патогенных микроорганизмов.
Эволюционный анализ PCV2 с использованием биологического программного обеспечения показал, что время появления самого позднего общего предка (TMRCA) приходится на период со второй половины 19 века до начала 20 века [5]. Для типирования PCV2 исследователи предложили различные методы и принципы. Комитет ЕС по цирковирусам свиней определил генетическое расстояние гена ORF2, превышающее 0,035, как принцип типирования PCV2 [11]. По принципу генотипирования PCV2 в настоящее время подразделяется на 6 генотипов – PCV 2а-2f, из которых PCV2а, PCV2b и PCV2d являются основными генотипами; остальные 3 генотипа PCV2с, PCV2e и PCVf выявлены только в некоторых странах и имеют очень низкую распространенность [15].
С момента своего открытия PCV2 претерпел два сдвига генотипа в ходе своей эволюции. Глобальный сдвиг генотипа с PCV2а на PCV 2b произошел примерно в 2003 г. [21], при этом почти 60 % последовательностей, депонированных в GenBank, принадлежат к генотипу PCV2b (таблица 1). В дальнейшем было подтверждено, что PCV2с является вымершей группой вирусов, поскольку с тех пор, как первоначально сообщалось о трех штаммах PCV2с, не было идентифицировано никаких дополнительных последовательностей PCV2с [14]. После 2010 г. распространенность генотипа PCV2b постепенно снижалась, а генотипа PCV2d постепенно увеличивалась, он в настоящее время стал основным генотипом.
Таблица 1 – Филогенетический анализ распространения цирковируса PCV2 в мире за период с 2004 по 2023 гг.
|
№ п/п |
Название вируса |
Географическое происхождение |
Год выявления |
Генетический подтип |
GenBank, № |
Размер генома, п.н. |
|
1 |
LG |
Цзилинь, Китай |
2008 |
PCV2a |
HM038034 |
1768 |
|
2 |
NB0301 |
Чжэцзян, Китай |
2003 |
PCV2b |
AY391729 |
1767 |
|
3 |
GD-TS |
Китай |
2002 |
PCV2b |
AY181945 |
1767 |
|
4 |
BDH |
Хэйлунцзян, Китай |
2008 |
PCV2d |
HM038017 |
1767 |
|
5 |
DK1980 |
Дания |
1998 |
PCV2c |
EU148503 |
1767 |
|
6 |
DK1987 |
Дания |
1998 |
PCV2c |
EU148504 |
1767 |
|
7 |
SD |
Китай |
2009 |
PCV2d |
AY181947 |
1767 |
|
8 |
TJ |
Китай |
2009 |
PCV2d |
AY181946 |
1767 |
|
9 |
pmws |
Канада |
1997 |
PCV2a |
AF027217 |
1768 |
|
10 |
BF |
Китай |
2001 |
PCV2a |
AF381175 |
1768 |
Для выяснения распространенности PCV2 в Северо-Восточном Китае из базы данных NCBI было загружено в общей сложности 515 последовательностей китайских штаммов ЦВС-2 за последние пять лет (2018-2023 гг.). Размер генома этих штаммов составлял 1766-176 8 п.н., среди которых размер ORF1 составлял 945 п.н., а размер ORF2 - 702-708 п.н. Всего было выявлено 43 штамма вируса в трех провинциях Ляонин, Хэйлунцзян и Цзилинь. Подробная информация, включая название штамма, инвентарный номер в GenBank, размер фрагмента, год выделения и провинцию, представлена в таблице 2. Гомология всего генома PCV2 составляет 95,69–100 %, гомология гена ORF1 составляет 96,44–100 %, а гомология гена ORF2 составляет 94,21–100 %. Стоит отметить, что скорость эволюции ORF2 выше, чем скорость эволюции всего генома
PCV2. Капсидный белок, кодируемый ORF2, является основной мишенью иммунной системы, поэтому он, естественно, находится под селективным давлением со стороны иммунной системы с высокой частотой мутаций для этого гена.
Таблица 2 – Информация о штаммах PCV2 в Северо-Восточном Китае с 2018 по 2023 гг.
|
Название штамма |
Место изоляции |
Время изоляции |
Генотип |
GenBank, № |
Размер генома, п.н. |
|
CC361 |
Китай, Цзилинь |
2021 |
PCV2d |
OP279264.1 |
1767 |
|
CC11 |
Китай, Цзилинь |
2021 |
PCV2a |
OP233007.1 |
1768 |
|
CC23 |
Китай, Цзилинь |
2021 |
PCV2b |
OP233009.1 |
1767 |
|
CC33 |
Китай, Цзилинь |
2021 |
PCV2a |
OP233010.1 |
1768 |
|
CC462 |
Китай, Цзилинь |
2021 |
PCV2d |
OP233011.1 |
1767 |
|
CC291 |
Китай, Цзилинь |
2021 |
PCV2d |
OP233012.1 |
1767 |
|
CC443 |
Китай, Цзилинь |
2021 |
PCV2b |
OP233013.1 |
1767 |
|
CC471 |
Китай, Цзилинь |
2021 |
PCV2b |
OP233014.1 |
1767 |
|
CC251 |
Китай, Цзилинь |
2021 |
PCV2a |
ON012606.1 |
1768 |
|
CC346 |
Китай, Цзилинь |
2021 |
PCV2a |
ON012607.1 |
1768 |
|
CC348 |
Китай, Цзилинь |
2021 |
PCV2b |
ON012608.1 |
1767 |
|
SDRS |
Китай, Хэйлунцзян |
2019 |
PCV2d |
OL452024.1 |
1767 |
|
MDJ |
Китай, Хэйлунцзян |
2019 |
PCV2b |
OL452025.1 |
1767 |
|
BY |
Китай, Хэйлунцзян |
2020 |
PCV2b |
OL452026.1 |
1767 |
|
LNHC |
Китай, Хэйлунцзян |
2019 |
PCV2d |
OL452027.1 |
1767 |
|
HLJHH |
Китай, Хэйлунцзян |
2019 |
PCV2d |
OL452028.1 |
1767 |
|
GXA58 |
Китай, Ляонин |
2019 |
PCV2d |
MT302517.1 |
1767 |
|
GXA24 |
Китай, Ляонин |
2019 |
PCV2d |
MT302519.1 |
1767 |
|
GXA14 |
Китай, Ляонин |
2019 |
PCV2d |
MT302521.1 |
1767 |
|
GXA16 |
Китай, Ляонин |
2019 |
PCV2a |
MT302528.1 |
1768 |
|
LJ0313 |
Китай, Хэйлунцзян |
2018 |
PCV2b |
MT104511.1 |
1767 |
|
LJ0715 |
Китай, Хэйлунцзян |
2018 |
PCV2b |
MT104512.1 |
1767 |
|
LJ0719 |
Китай, Хэйлунцзян |
2018 |
PCV2b |
MT104513.1 |
1767 |
|
HD62506 |
Китай, Хэйлунцзян |
2018 |
PCV2b |
MK347349.1 |
1767 |
|
PT625135 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347355.1 |
1767 |
|
XA07174 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347358.1 |
1767 |
|
YB2206 |
Китай, Хэйлунцзян |
2018 |
PCV2b |
MK347362.1 |
1767 |
|
YK1805 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347369.1 |
1767 |
|
JMS07252 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347373.1 |
1767 |
|
XT2807 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347375.1 |
1767 |
|
PF29053 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347379.1 |
1767 |
|
LYD0721 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347383.1 |
1767 |
|
BX3109 |
Китай, Хэйлунцзян |
2018 |
PCV2a |
MK347388.1 |
1768 |
|
ZB2811 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347389.1 |
1767 |
|
DL1802 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347391.1 |
1767 |
|
HC72302 |
Китай, Хэйлунцзян |
2018 |
PCV2b |
MK347393.1 |
1767 |
|
FX3116 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347398.1 |
1767 |
|
LY625123 |
Китай, Хэйлунцзян |
2018 |
PCV2a |
MK347399.1 |
1768 |
|
DP2209 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347401.1 |
1767 |
|
CY29052 |
Китай, Хэйлунцзян |
2018 |
PCV2a |
MK347405.1 |
1768 |
|
LY625121 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347409.1 |
1767 |
|
TL3004 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347410.1 |
1767 |
|
BD2210 |
Китай, Хэйлунцзян |
2018 |
PCV2d |
MK347412.1 |
1767 |
M> 68047401 1 I DP2209 76 68047412 1 | 00 2210
53 MK34 7389 1 I 202811
68047356 1 | PT625135 6*047409 1 I UY625121 06452028 1 |pc»2 HWHH - 06452024 1 |pcv2 SDRS
53 6X34 7369 1 I 3X18 0 5
* AX34 7 3 79 1 | PF29053
MK34 7375 1 I XT2807
AX34 7398 1 | EX 3116
AX34 74 1 0 1 I TL3OO4 6X347373 1 I J64S0 7252 OP279264 1 |pcw2CC361 OP233012 1 tpo2CC291
L MT 30251 7 1 | PCV2/CH^019ДЗХЛ68 . AW 302519 1 | PCV2/CN/2019/GXA24
" wl MT 302521 1 I PCV2CN/2O19X3XA14
06452027 1 lpo2LNHC MK34 7391 1| CX. 1802 04*233011 1 Ipcv2 CC462 68047358 1 | XA0 7174 6804 7 383 1 | UYD07215 | SO PCV2d
АГЛ1 /29 1 | N80301 PCV2b
AY181945 1 IGO TSPCVTb
69047393 1 I ИС72302
06*012606 1|pcv2CC348
• AX34 7362 1 I YB2206 6X34 7349 1 I H0625062 .04*233009 1 lpcv2CC23
■ 04*233013 1 lpo-2CC443
$ 06452025 1 lpc*2 MD J - Ot452026 1 |pcv2 BY
К . MT 104511 1 |pcv2 LJ0313
Ч MT104512 11 U0715
-
- ED 148603 1 I OK198O PC V2c
-
- EM 148504 1 | OK1987 PCV2c й 68047399 1 I 6Y625123
95 . 04*233007 1 Ipcv2 CC11
MK347405 1 I CV29052 HMO38034 1 (LGPCV2*
AF027217 1 I pmw» PCV2a AF381175 1 | BF PCV2*
PCV2d
PCV2b
PCV2C
PCV2a
53,4 %(23/43 )
27,9 % (12/43)
18,6 % (8/43)
-
■ OH012606 1 |por2CC251 MT 30252* 1 |PCV2CWV1»WAie
изоргзя10 1|ро2сс 4^-6804 7388 1 I 0X3109
Рисунок 1
–
Филогенетическое древо всего генома PCV2 в Северо-Восточном Китае с 2018
по 2023 гг. Примечание: PCV2а отмечен красным цветом, PCV2b отмечен синим цветом, PCV2с отмечен зеленым цветом, PCV2d отмечен розовым цветом
Филогенетическое дерево всего генома: 43 штамма принадлежат к подтипам генов PCV2a (8/43), PCV2b (12/43) и PCV2d (23/43), что указывает на то, что PCV2b и PCV2d (35/43) являются основными преобладающими в этот период генотипами. Сравнение последовательностей всего генома за период с 2003 по 2023 гг. показало, что гомология штамма PCV2a и эталонного штамма PCV2a составила 98,44 %, а гомология нуклеотидной последовательности штамма PCV-2b и эталонного штамма PCV2b составила 99,53 %. Идентичность нуклеотидной последовательности между штаммом PCV2d и эталонным штаммом PCV2d составила 99,32 % (Рисунок 1).
Характер дрейфа генотипов PCV2 в Китае был следующим: PCV2a был преобладающим генотипом до 2004 года, PCV2b был преобладающим генотипом с 2004 по 2008 год, PCV2d стал преобладающим генотипом после 2009 года, что показывает тенденцию увеличения распространенности PCV2d и тенденцию уменьшения распространенности PCV2a и PCV2b. В ходе эволюции PCV2 генотипы претерпели два дрейфа: от PCV2a к PCV2b до 2004 г. и от PCV2b к PCV2d в 2009 г. Таким образом, эти результаты позволяют наглядно представить генотипический дрейф, как показано на рисунке 2.
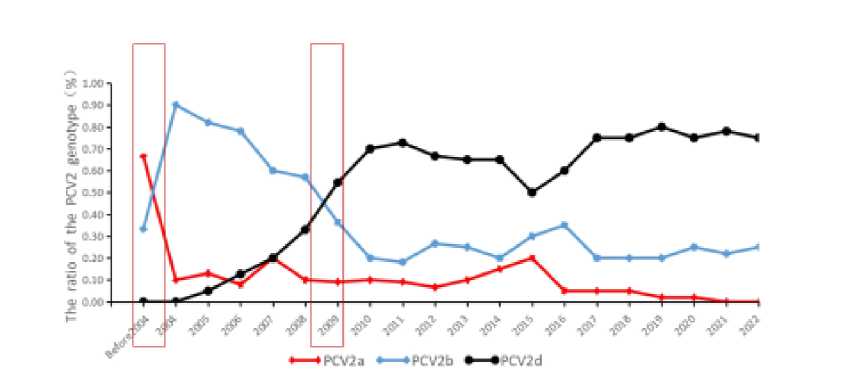
Рисунок 2 – Дрейф генотипов в процессе эволюции китайских штаммов PCV2
Было показано, что генетические различия между разными изолятами PCV2 могут напрямую влиять на вирулентность и антигенность, оказывая влияние на эффективность вакцинации, патогенез и диагностику заболевания [17]. Поэтому необходимо изучить вариабельность последовательностей изолятов PCV2, распространенных в Китае.
Для уточнения генетической изменчивости штамма PCV2 было проведено аминокислотное выравнивание белка Rep, кодируемого геномом ORF1, и белка Cap, кодируемого геномом ORF2, в полученной последовательности штамма. Результаты показали, что нуклеотидная мутация последовательности ЦВС-2 привела к образованию 14 белков Cap и 22 белков Rep. Гомология этих 14 белков Cap составляет 89,7-99,5 %; 22 белка Rep имеют высокую гомологию (98,0 ~ 99,6 %) друг с другом. В полученной последовательности штамма было мутировано 16 аминокислотных сайтов в белке Rep. Выравнивание аминокислот белка Cap, кодируемого геномом ORF2 в последовательности штамма, показало, что в белке Cap были мутированы 45 аминокислотных сайтов.
PCV2 кодирует капсидный белок Cap, который является единственным структурным белком PCV2 и поэтому важен для антигенности вируса. Он также подходит в качестве филогенетического и эпидемиологического маркера. Сравнение аминокислотной последовательности белка Cap штаммов в Северо-Восточном Китае показало, что 8 из 43 штаммов имели типичный мотив TNKISI, присутствующий в PCV2a. Двенадцать из 43 штаммов имели типичные мотивы SNPRSV, присутствующие в PCV2b, а 23 из 43 штаммов имели большинство мотивов SNPLTV, присутствующих в PCV2d (Рисунок 1). Однако область Cap ЦВС-2 сильно варьирует. Результаты показали, что среди штаммов существует 5 основных областей изменчивости белка Cap. Эти остатки включают 51-91, 121-151, 183-191, 206-217 и 232-234. Некоторые из этих вариабельных областей соответствуют четырем антигенным доменам (аминокислотные положения 6587, 113-139, 193-207 и 227-233), которые ранее были описаны как возможные иммунореактивные области.
Специфические замены аминокислот также были обнаружены в положениях 78, 200 и 222. Полные последовательности ORF2 43 штаммов ЦВС2 в этом исследовании имели длину 702 (20 штаммов) и 705 (23 штамма) п.н. соответственно. Последовательность ORF2 имеет длину 705 п.н. и кодирует белок Cap, состоящий из аминокислотных остатков 234. Он имеет кодоновый сдвиг с TTA на CTT в ORF2, что приводит к мутации стоп-кодона (с UAA на AAG) в ORF2, что приводит к удлиненным остаткам лизина (К), кодируемые AAG или AAA.
Заключение. Таким образом, распространенность PCV2 в Китае за последние 20 лет является сложной проблемой из-за сосуществования нескольких генотипов PCV2 в китайской популяции свиней. Анализ данных за последние 20 лет показал, что в северовосточном регионе Китая у цирковируса в данных популяциях производственного свиноводства среди 43 последовательностей PCV2 генотип PCV2a составлял 18,6 %, генотип PCV2b – 27,9 %, а генотип PCV2d – 53,4 %. Это указывает на то, что генетическое разнообразие PCV2 расширилось, а PCV2b и PCV2d стали преобладающими генотипами, распространенными в период с 2004 по 2023 гг.
Высокое генетическое разнообразие PCV2 в Китае может быть обусловлено рядом факторов, включая давление вакцинации, естественный отбор и международные перемещения свиней. Исследования показали, что PCV2 постоянно эволюционирует за счет точечных мутаций и геномной рекомбинации, что может привести к появлению ряда новых антигенных вариантов, и в настоящее время известно, что появляются новые варианты PCV2. Многие исследования показали, что быстрый переход генотипа от PCV2a к PCV2b связан с возникновением случаев послеотъемного мультисистемного истощения поросят (СПМИ) на уровне ферм по доращиванию и откорму свиней и сопутствующим увеличением тяжести клинических проявлений. В отличие от этого, патогенность нового генотипа PCV2d для свиней и связь между увеличением его распространенности и клиническими проявлениями в полевых условиях требуют дальнейшего изучения. Данное исследование предоставляет ценную информацию об эпизоотологии, генетике и эволюции цирковируса PCV2.
Список литературы Эпизоотология, генетика и эволюция цирковируса свиней типа 2 в Китае
- Обзор по цирковирусной инфекции свиней / В. М. Попова, В. А. Богомолова, Е. В. Маркова, Е. Н. Фелоров, Л. С. Люлькова // Свиноводство. – 2020. – № 3. – С. 8-10.
- Орлянкин, Б. Г. Цирковирусные болезни свиней: распространение, диагностика и специфическая профилактика / Б. Г. Орлянкин // Ветеринария. – 2013. – № 8. – С. 3-9.
- Стаффорд, В. В. Цирковирусная инфекция свиней. Обзорные данные / В. В. Стаффорд, Я. Б. Стрельцова, М. А. Аноятбеков // Труды ВИЭВ. – Т. 80. – № 1. – С. 324-330. - DOI: 10.30917/ATTPRINT-2018-1.
- Фу Юйцзе, Ф. Ю. Цирковирусная инфекция свиней / Ф. Ю. Фу Юйцзе, С. В. Теребова // Аграрный вестник Приморья. – 2021. – № 3(23). – С. 44-49.
- Цирковирусные инфекции возникают во всех свиноводческих хозяйствах: по материалам компании MSD Animal Health. - Текст электронный // Agrovesti.net [сайт]. – 2016. – URL: https://agrovesti.net/lib/tech/pig-breedingtech/tsirkovirusnye-infektsii-voznikayut-vovsekh-svinovodcheskikh-khozyajstvakh.html
- Differences in virulence among porcine circovirus type 2 isolates are unrelated to cluster type 2a or 2b and prior infection provides heterologous protection / T. Opriessnig, S. Ramamoorthy, D. M. Madson [et al.]. // J/ Gen Viro. – 2008. – V. 89. – P. 2482-2491. – doi: 10.1099/vir.0.2008/001081-0.
- Differential recognition of the ORF2 region in a complete genome sequence of porcine circovirus type 2 (PCV2) isolated from boar bone marrow in Korea / C. H. Kweon, L. T. Nguyen, M. S. Yoo, S. W. Kang // Gene. – 2015. – V.569. – P. 308-312. – doi: 10.1016/j.gene.2015.04.055.
- Genetic analysis of porcine circovirus type 2 in China / C. G. Jiang, G. Wang, Y. B. Tu [et al.]. // Arch Virol. – 2017. – V.162. – P. 2715-2726. – doi: 10.1007/s00705-017-3414-1.
- Genetic characterisation of Porcine circovirus type 2 (PCV2) strains from feral pigs in the Brazilian Pantanal: an opportunity to reconstruct the history of PCV2 evolution / G. Franzo, M. Cortey, A. M. de Castro [et al.] // Vet Microbiol. – 2015. – V.178. – P. 158-162. – doi: 10.1016/j.vetmic.2015.05.003.
- Genetic diversity of porcine circovirus type 2 (PCV2) in Thailand during 2009-2015 / N. Thangthamniyom, P. Sangthong, P. Poolperm [et al.] // Vet Microbiol. – 2017. – V. 208. – P. 239-246. – doi: 10.1016/j.vetmic.2017.08.006.
- Genetic variation analysis of Chinese strains of porcine circovirus type 2 / F. Wang, X. Guo, X. Ge [et al.] // Virus Res. – 2009. – V.145. –P. 151-156. – doi: 10.1016/j.virusres.2009.05.015.
- Genotypic shift of porcine circovirus type 2 from PCV-2a to PCV-2b in Spain from 1985 to 2008 / M. Cortey, E. Pileri, M. Sibila [et al.] // Vet J. – 2011. – V. 187(3). – P. 363-368. – doi: 10.1016/j.tvjl.2009.12.023.
- Larochelle, R. Genetic characterization and phylogenetic analysis of porcine circovirus type 2 (PCV2) strains from cases presenting various clinical conditions / R. Larochelle, R. Magar, S. D’Allaire // Virus Res. – 2002. – V. 90. – P. 101-112. – doi: 10.1016/s0168-1702(02)00141-7.
- Olvera, A. Molecular evolution of porcine circovirus type 2 genomes: phylogeny and clonality / A. Olvera, M. Cortey, J. Segales // Virology. – 2007. – V.35. – P. 175-185. – doi: 10.1016/j.virol.2006.07.047.
- PCV2d-2 is the predominant type of PCV2 DNA in pig samples collected in the U.S. during 2014-2016 / C. T. Xiao, K. M. Harmon, P. G. Halbur, T. Opriessnig. // Vet Microbiol. – 2016. – V.197. – P. 72-77. – doi: 10.1016/j.vetmic.2016.11.009.
- Phylodynamic analysis of porcine circovirus type 2 reveals global waves of emerging genotypes and the circulation of recombinant forms / G. Franzo, M. Cortey, J. Segales [et al.] // Mol Phylogenet Evol. – 2016. – V.10. – P. 269-280. – doi: 10.1016/j.ympev.2016.04.028.
- Pigs naturally exposed to porcine circovirus type 2 (PCV2) generate antibody responses capable to neutralise PCV2 isolates of different genotypes and geographic origins / S. Kurtz, L. Grau-Roma, M. Cortey [et al.] // Vet Res. – 2014. – V.45 (1). – P. 29. – doi: 10.1186/1297-9716-45-29.
- The emergence of a new strain of porcine circovirus-2 in Ontario and Quebec swine and its association with severe porcine circovirus associated disease-2004-2006 / S. Carman, H. Y. Cai, J. DeLay [et al.] // Can J Vet Res. – 2008. – V. 72(3). – P. 259-68.
- The emergence of porcine circovirus 2b genotype (PCV-2b) in swine in Canada / C. A. Gagnon, D. Tremblay, P. Tijssen [et al.] // Can Vet J. – 2007. – V. 48(8). – P. 811-819.
- Trible, B. R. Genetic variation of porcine circovirus type 2 (PCV2) and its relevance to vaccination, pathogenesis and diagnosis / B. R. Trible, R. R. Rowland. // Virus Res. – 2012. – V. 164. – P. 68-77. – doi: 10.1016/j.virusres.2011.11.018.
- Xiao, C. T. Global molecular genetic analysis of porcine circovirus type 2 (PCV2) sequences confirms the presence of four main PCV2 genotypes and reveals a rapid increase of PCV2d / C. T. Xiao P. G. Halbur, T. Opriessnig // J Gen Virol. – 2015. – V.96(Pt7). – P.1830-41. – doi: 10.1099/vir.0.000100. Epub 2015 Feb 23.


