К вопросу о маркировании локусов Pl, контролирующих устойчивость подсолнечника к возбудителю ложной мучнистой росы
Автор: Рамазанова С.А., Антонова Т.С.
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 1 (177), 2019 года.
Бесплатный доступ
Одной из самых вредоносных болезней подсолнечника является ложная мучнистая роса. К настоящему времени в мире обнаружено более 45 физиологических рас патогена. Эффективным методом контроля над возбудителем болезни является введение доминантных генов устойчивости к нему в растение-хозяина. Современные технологии генотипирования с помощью ДНК-маркеров позволяют контролировать наличие этих генов на каждом этапе селекции. Апробированы известные из литературных источников девять STS и три SSR-маркера генов Pl5 , Pl6 и Pl8 , контролирующих устойчивость к расам P. halstedii , для идентификации этих генов у линий-дифференциаторов устойчивости подсолнечника, входящих в международный тест-набор для идентификации рас P. halstedii . Был отобран и апробирован молекулярный STS-маркер НАР3, пригодный для идентификации локуса Pl6 , контролирующего устойчивость подсолнечника к возбудителю ложной мучнистой росы.
Днк-маркеры, мас, r-гены, устойчивость, подсолнечник
Короткий адрес: https://sciup.org/142220356
IDR: 142220356 | УДК: 631.523:633.854.78 | DOI: 10.25230/2412-608X-2019-1-177-17-23
Текст научной статьи К вопросу о маркировании локусов Pl, контролирующих устойчивость подсолнечника к возбудителю ложной мучнистой росы
Введение. Болезнь подсолнечника – ложная мучнистая роса, вызываемая облигатным паразитом – оомицетом Plasmopara halstedii (Farl.) Berl. et de Toni, является одной из самых вредоносных. При благоприятных условиях для развития возбудителя, болезнь вызывает значительное сокращение урожая семян подсолнечника и содержания в них масла [1]. К настоящему времени в мире обнаружено более 45 физиологических рас патогена и наблюдается постоянное возникновение новых, более вирулентных. Так, если до 2007 г. в Европе насчитывалось 13 рас патогена, в Америке – 20, в России в Краснодарском крае – 7, то уже к 2015 г. их число возросло до 24 в Европе и до 40 в Америке [1; 2]. В Краснодарском крае за последние 5 лет идентифицированы три новых расы: 334, 713 и 733 [4].
Наиболее эффективным методом контроля над возбудителем болезни является введение доминантных генов устойчивости к нему в растение-хозяина. Устойчивость к P. halstedii контролируется генами Pl. Впервые они были идентифицированы в культурном подсолнечнике еще в 70-х годах прошлого столетия, обозначены как Pl 1 и Pl 2 , контролировали устойчивость к расам 100 и 300 соответственно [5; 6]. Позднее они были картированы как относящиеся к восьмой группе сцепления LG на генетической карте SSR-локусов (Simple Sequence Repeat) [7]. Гены Pl 3 и Pl 4 контролируют устойчивость к этим же расам, локализация их на генетической карте подсолнечника пока не известна [8]. Гены Pl 5 и Pl 8 контролируют устойчивость к 16-и расам P. halstedii [9]. Эти два гена сцеплены и относятся к тринадцатой группе сцепления на генетической карте SSR-локусов. Ген Pl 6 так же, как Pl 1 и Pl 2 , расположен в восьмой группе сцепления LG и контролирует устойчивость к расам 100, 300, 700, 703, 710, 330, 770 и 730. К этой же группе сцепления относятся и кластеры генов Pl 7 , Pl 9 , Pl 10 [10]. Анализ публикаций, посвященных картированию генов устойчивости подсолнечника к P. halstedii , показал, что тип их расположения является одиночным диаллельным, то есть один локус имеет множество кодоминантных аллелей, контролирующих устойчивость к разным расам патогена [8; 9; 13; 16; 28]. Сгруппированы они, как минимум, в четырех геномных комплексах в трех группах сцепления (LG) [16].
Цель исследования – поиск ДНК-маркеров для идентификации генов, контролирующих устойчивость к P. halstedii , в селекционном материале ВНИИМК и создание на их основе системы маркеров с последующим использованием ее в селекционных программах, направленных на создание генотипов подсолнечника с комплексной устойчивостью к разным расам возбудителя ложной мучнистой росы.
Материалы и методы Объектом исследования послужили 14 линий-дифференциаторов устойчивости подсолнечника, входящих в международный тест-набор для идентификации рас P. halstedii (таблица).
Таблица 1
Линии-дифференциаторы устойчивости подсолнечника к поражению ложной мучнистой росой, содержащие известные гены Pl
|
Линия-дифференциатор |
Pl -ген |
Группа сцепления |
Литературный источник |
|
RHA-419 |
Pl arg |
LG1 |
Dussle et al., 2004; Wieckhorst et al., 2010; Imerovski et al., 2014 |
|
XRQ |
Pl 5 |
LG13 |
Bert et al., 2001; Radwan et al., 2004 |
|
83HR4RM |
Pl 7 |
LG8 |
Jocić et al., 2012 |
|
HIR-34 |
Pl 4 |
Не определена |
Jocić et al., 2012 |
|
PSC8 |
Pl 2 |
LG8 |
Gascuel et al., 2014 |
|
YVQ |
Pl 8 |
LG13 |
Gascuel et al., 2014 |
|
HA-335 |
Pl 6 |
LG8 |
Bouzidi et al., 2002; Panković et al., 2007 |
|
HA-R5 |
Pl 13 |
LG1 |
Mulpuri et al., 2009; Bertero de Romano et al., 2010 |
|
HA-R4 |
Pl 16 |
LG1 |
Liu et al., 2012 |
|
803-1 |
Pl 5 |
LG13 |
Gascuel et al., 2014 |
|
PM-17 |
Pl 5 |
LG13 |
Gascuel et al., 2014 |
|
DM-2 |
Pl 5, Pl 11, Pl 12 |
LG13 |
Rahim et al., 2002 |
|
RHA-274 |
Pl 2 , Pl 9, Pl 10 |
LG8 |
Gulya et al., 1991; Rahim et al., 2002; Molinero-Ruiz et al., 2002; Romano et al., 2010 |
|
RHA-265 |
Pl 1 |
LG8 |
Gedil et al., 2001; Rahim M., 2002 |
Для выделения ДНК применяли метод, основанный на использовании лизирующего буфера, содержащего гексадеци-лтриметиламмоний бромид (СТАВ) с модификациями [11; 12]. Концентрацию ДНК в полученном препарате определяли визуально по интенсивности свечения пробы объемом 10 мкл в ультрафиолетовом свете в 1 %-ном агарозном геле с добавлением 2 мкл бромистого этидия. Электрофорез растворов ДНК проводили при напряжении 100 V в течение 30 мин в трис-ацетатном буферном растворе (ТАЕ).
Для ПЦР-анализа использовали 12 праймеров (HAP1, HAP2 HAP3, Ha-P1, Ha-P2, Ha-P3, Ha-P4, Ha-P5, Ha-P6, ORS 37, ORS 166 и ORS 1043), разработанных для маркирования локусов Pl 5 Pl 6 Pl 8 [9; 13; 14; 15]. Полимеразную цепную реакцию выполняли в реакционной смеси (25 мкл) следующего состава: 67 мМ Трис-HCl (рН 8,8); 16,6 мM сульфата аммония; 1,5–3,0 мM MgCl 2 ; 0,01 % Tween 20; по 0,2
мM дезоксирибонуклеозидфосфатов; по 10 пМ праймеров; 10 нг матричной ДНК и 1 ед. рекомбинантной термостабильной ДНК-полимеразы (НПО «СибЭнзим», Россия). Реакции проводили в термоциклере S1000тм (BioRad, США) при следующих температурных режимах: начальная денатурация при 95 °С в течение 3 мин, далее 35 циклов с последовательной сменой температур: денатурация при 94 ºС в течение 10 сек, отжиг праймера при 58–63 ºС (в зависимости от праймера) – 30 сек, элонгация при 72 ºС – 1 мин 30 сек и заключительная элонгация 10 мин.
Электрофорез продуктов амплификации проводили в геле, содержащем 2 % агарозы и SВ-буфер, с использованием камеры для горизонтального электрофореза SE-2 (Хеликон, Россия) при напряжении ≈ 200 В, силе тока ≈ 100 мА, в течение 30 мин. Гели окрашивали бромистым этидием. Для визуализации и документирования результатов электрофореза применяли систему цифровой документации видеоизображения BIO-PRINT (Vil-ber Lourmat, Франция).
Результаты и обсуждение. По сообщениям многих авторов, каждая линия-дифференциатор устойчивости из международного тест-набора для идентификации рас P. halstedii содержит один или несколько генов Pl, которые обеспечивают их устойчивость к определенным расам патогена. В таблице представлены 14 линий подсолнечника, соответствующие им гены и группы сцепления, в которых они картированы на генетической карте SSR-локусов.
Для поиска ДНК-маркеров генов устойчивости Pl5, Pl6 и Pl8 к P. halstedii исследовали молекулярно-генетический полиморфизм девяти STS (Sequence-Tagged Site) и трех SSR (Simple Sequence Repeat) локусов ДНК. Результаты ПЦР ДНК линий подсолнечника показали, что только с парой праймеров HAP1 не было получено амплифицированных фрагмен- тов. По остальным 11 локусам были подобраны оптимальные параметры реакции амплификации и получен отжиг соответствующих праймеров на матрице. По четырем локусам (ORS 166, Ha-P3, Ha-P5 и Ha-P6) не было выявлено полиморфных фракций ДНК, поэтому эти маркеры были исключены из дальнейшего исследования.
По пяти локусам (ORS 37, ORS 1043, Ha-P1, Ha-P2 и Ha-P4) выявлен полиморфизм в виде наличия–отсутствия фрагментов ДНК. Для примера на рисунках 1 и 2 представлены фореграммы продуктов амплификации ДНК с праймерами ORS 37 и ORS 1043. В результате ПЦР с праймерами, фланкирующими эти микроса-теллитные локусы, не было получено фрагментов ДНК, характерных для линии НA-335, в генотипе которой присутствует ген Pl 6 ; так же, как и для линий XRQ, YVQ, и 803-1, PM-17, DM-2 – носителей кластеров генов Pl 5 /Pl 8.
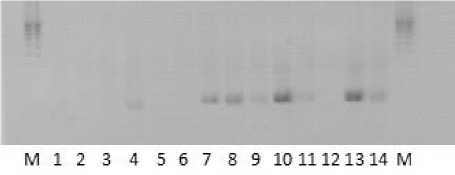
Рисунок 1 – Фореграмма продуктов амплификации ДНК линий-дифференциаторов подсолнечника с праймером ORS 1043. Дорожки:1 – RHA-419; 2 – XRQ;
3 – 83HR4RM; 4 – HIR 34; 5 – PSC8; 6 – YVQ; 7 – HA335; 8 – HAR-5; 9 – HAR-4; 10 – 803-1;
11 – PM-17; 12 – DM-2; 13 – RHA-274;
14 – RHA-265; M – маркер молекулярного веса

1 2 3 4 5 6 7 8 9 10 11 12 13 14 К" М
Рисунок 2 – Фореграмма продуктов амплификации ДНК линий-дифференциаторов подсолнечника с праймером ORS 37.
Дорожки: 1 – RHA-419; 2 – XRQ;
3 – 83HR4RM; 4 – HIR 34; 5 – PSC8; 6 – YVQ; 7 – Ha335; 8 – HAR-5; 9 – HAR-4; 10 – 803-1;
11 – PM-17; 12 – DM-2; 13 – RHA-274;
14 – RHA-265; M – маркер молекулярного веса
STS-локус HAP3 – один из трех, разработанных для маркирования локуса Pl 6 . Он представляет собой кластер из 13 генов, близко расположенных друг к другу, относящихся к TIR-NBS-LRR классу R-генов и картированных в группе сцепления LG8 на генетической карте SSR [13]. По данным авторов, эта праймерная пара дает четыре полиморфных фрагмента ДНК длиною от 988 до 1811 пар нуклеотидов (п.н.). Из них фракции длиною 988, 1119 и 1811 пар нуклеотидов выявлены у линий подсолнечника, устойчивых к расам 100, 300, 700, 703 и 710, а фрагмент длиною 1406 п. н. – у восприимчивого к этим расам образца. В нашем исследовании у линии НА-335 по локусу НАР3 получено два фрагмента ДНК в указанном диапазоне длин (рис. 3, дорожка 7). У линии РМ-17 (рис. 3, дорожка 8) не выявлено амплифицированных фрагментов ДНК, что может свидетельствовать об отсутствии у неё локуса Pl 6 . У остальных изученных двенадцати линий получены фрагменты ДНК разных длин, отличающиеся от таковых у линии НА-335 (рис. 3).
Был изучен молекулярно-генетический полиморфизм локуса НАР3 у линий подсолнечника селекции ВНИИМК с целью маркирования кластера генов Pl 6 . Для исследования выбраны линии, отличающиеся по устойчивости к разным расам.
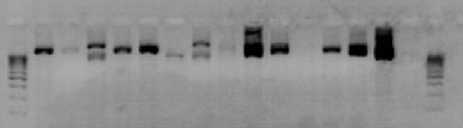
М 1 2 3 4 5 6 7 8 9 10 1112 13 14 К" М
Рисунок 3 – Фореграмма продуктов амплификации ДНК линий-дифференциаторов подсолнечника с праймером НАР3. Дорожки: 1 – RHA-419; 2 – XRQ;
3 – 83HR4RM; 4 – HIR 34; 5 – PSC8; 6 – YVQ; 7 – HА335; 8 – HAR-5; 9 – HAR-4; 10 – 803-1;
11 – PM-17; 12 – DM-2; 13 – RHA-274;14 – RHA-265; M – маркер молекулярного веса
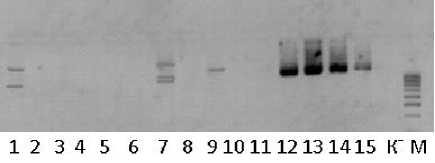
Рисунок 4 – Фореграмма продуктов амплификации ДНК линий подсолнечника селекции ВНИИМК с праймером НАР3.
Дорожки: 1 – НА-335; 2 – Л2018-1; 3 – Л689-15; 4 – Л693-15; 5 – Л697-16; 6 – Л678-15; 7 – Л665-15; 8 – Л700-15; 9 – Л699-15; 10 – ВК920; 11 – ВК917;
12 – СЛ 24 Б; 13 – СЛ 4 Б; 14 – ВК 935 Б;
15 – ВК 934 Б; К – отрицательный контроль; М – маркер молекулярного веса
На рисунке 4 показаны результаты амплификации ДНК этих линий с праймером НАР3. На первой дорожке фрагменты ДНК линии НА-335, далее на дорожках 2–11 представлены линии с разной устойчивостью к расам P. hals-tedii. На дорожке 2 – линия Л2018-1, устойчивая к расам 330, 334, 730 и 710. Далее на дорожках 3–9 представлены фрагменты ДНК линий, устойчивых к расам 330, 730 и 710, но восприимчивых к расе 334, а на дорожках 10–11 – устойчивых только к расе 330, на дорожках 12–14 – восприимчивых ко всем четырем расам. Результаты показали, что ни у одной изученной линии не выявлено точно таких фрагментов ДНК, как у линии НА-335. У линий Л2018-1, Л689-15, Л693-15, Л697-15, Л678-15, Л700-15, ВК 920, ВК 917 с устойчивостью к одной или нескольким расам (дорожки 2–6, 8, 10, 11) не получено амплифицированных фрагментов. Мы предполагаем, что в их геноме отсутствует локус Pl6, а устойчивость к P. halstedii контролируется генами, относящимися к другим группам сцепления. У линий
Л665-15 и Л699-15 получены амплифи-цированные фрагменты ДНК размером от 988 до 1811 п.н. (рис. 4; дорожки 7, 9). У четырех линий, восприимчивых ко всем четырем расам, получены четкие фрагменты одинакового размера, но отличающиеся длиной от линий НА-335, Л665-15 и Л699-15 (рис. 4). Это согласуется с данными из литературных источников, так как праймер НАР3 продуцирует четыре фрагмента ДНК, один из которых соответствует линиям, восприимчивым к расам 100, 300, 700, 703, 710, 330, 770 и 730.
Заключение. В результате проведенного исследования был подобран и апробирован молекулярный STS-маркер НАР3, пригодный для идентификации локуса Pl 6 , контролирующего устойчивость подсолнечника к расам P. halstedii 100, 300, 700, 703, 710, 330, 770 и 730. Этот маркер может стать одним из звеньев системы ДНК маркеров для идентификации тесно сцепленных генов и проведения маркер-вспомогательной селекции на устойчивость подсолнечника к возбудителю ложной мучнистой росы.
Список литературы К вопросу о маркировании локусов Pl, контролирующих устойчивость подсолнечника к возбудителю ложной мучнистой росы
- Virányi F., Gulya T.J., Tourvieille de Labrouhe D. Recent changes in the pathogenic variability of Plasmopara halstedii (sunflower downy mildew) populations from different continents//Helia. -2015 -V. 38 -P. 149-162.
- Gulya T.J. Distribution of Plasmopara halstedii races from sunflower around the world//Proc. 2nd Int. Downy Mildew Symposium «Advances in downy mildew research». Olomouc, Czech Republic, 2007. -V. 3. -P. 121-134.
- Spring O., Zipper R. New highly aggressive pathotype 354 of Plasmopara halstedii in German sunflower fields//Plant Protection Science. -2018. -V. 54 (2). -P. 83-86 DOI: 10.17221/99/2017-pps
- Iwebor M., Antonova T., Araslanova N., Saukova S. To the question of sunflower resistance to the downy mildew pathogen in the Krasnodar region of the Russian Fedeation//Proc. Intern. Congress on oil and protein crops. Chisinau, Republic of Moldova. -2018. -P. 133.
- Zimmer D.E., Kinman M.L. Downy mildew resistance in cultivated sunflower and its inheritance//Crop Sci. -1972. -V. 12 (6). -P.749-751. DOI: 10.2135/cropsci1972.0011183X001200060009x.
- Zimmer D.E. Physiological specialization between races of Plasmopara halstedii in America and Europe//Phytopathol. -1974. -V. 64. -P. 1465-1467
- DOI: 10.1094/Phyto-64-1465
- Liu Z, Gulya T.J., Seiler G.J., Vick B.A., Jan C-C. Molecular mapping of the Pl16 downy mildew resistance gene from HA-R4 to facilitate marker-assisted selection in sunflower//Theor. Appl. Genet. -2012. -V. 125. -P. 121-131.
- Jocić S., Miladinović D., Imerovski I., Dimitrijević A., Cvejić S., Nagl N., Kondić-Špika A. Towards sustainable downy mildew resistance in sunflower//Helia. -2012. -V. 35. -No 56. -P. 61-72.
- Radwan O., Bouzidi M.F., Vear F., Philippon J., Tourvieille de Labrouhe D., Nicolas P., Mouzeyar S. Identification of non-TIR-NBS-LRR markers linked to the P15/P18 locus for resistance to downy mildew in sunflower//Theoretical and Applied Genetics. -2003. -V. 106. -P. 1438-1446
- DOI: 10.1007/s00122-003-1196-1
- Gulya T.J., Sackston W.E., Viranyi F., Masirević S., Rashid K.Y. New races of the sunflower downy mildew pathogen (Plasmopara halstedii) in Europe and North and South America//Journal of Phytopathology. -1991. -V. 132 (4). -P. 303-311
- DOI: 10.1111/j.1439-0434.1991.tb00125.x
- Zolan M.E., Pukkila P.J. Inheritance of DNA methylation in Corpinus cinereus//Mol. Cell Biol. -1986. -Т. 6. -No 1. -Р. 195-200.
- Saghai-Maroof M.A., Soliman K.M., Jorgensen R.A., Allard R.W. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics//PNAS USA. -1984. -V. 81. -P. 8014-8018.
- Bouzidi M.F, Badaoui S., Cambon F., Vear F., Tourvieille de Labrouhe D., Nicolas P., Mouzeyar S. Molecular analysis of a major locus for resistance to downy mildew in sunflower with specific PCR-based markers//Theoretical and Applied Genetics -2002. -V. 104. -P. 592-600.
- Panković D., Radovanović N., Jocić S., Satović Z., Škorić D. Development of co-dominant amplified polymorphic sequence markers for resistance to sunflower downy mildew race 730//Plant Breeding. -2007. -V. 126 (4) -P. 440-444
- DOI: 10.1111/j.1439-0523.2007.01376.x
- Tang S., Yu J.-K., Slabaugh M.B., Shintani D.K., Knapp, S.J. Simple sequence repeat map of the sunflower genome//Theoretical and Applied Genetics. -2002 -V. 105 (8) -P. 1124-1136
- DOI: 10.1007/s00122-002-0989-y
- Gascuel Q., Martinez Y., Boniface M.-C., Vear F., Pichon M., Godiard L. The sunflower downy mildew pathogen Plasmopara halstedii//Molecular Plant Pathology. -2014. -V. 16 (2). -P. 109-122
- DOI: 10.1111/mpp.12164
- Qi L.L., Talukder Z.I., Hulke B.S., Foley M.E. Development and dissection of diagnostic SNP markers for the downy mildew resistance genes Plarg and Pl8 and marker-assisted gene pyramiding in sunflower (Helianthus annuus L.)//Molecular Genetics and Genomics. -2017. -V. 292 (3). -P. 551-563
- DOI: 10.1007/s00438-017-1290-8
- Dußle C.M., Hahn V., Knapp S.J., Bauer E. Plarg from Helianthus argophyllus is unlinked to other known downy mildew resistance genes in sunflower//Theoretical and Applied Genetics. -2004. -V. 109 (5). -Р. 1083-1086
- DOI: 10.1007/s00122-004-1722-9
- Imerovski I., Dimitrijević A., Miladinović D., Jocić S., Dedić B., Cvejić S., Surlan-Momirović G. Identification and validation of breeder-friendly DNA markers for Plarg gene in sunflower//Molecular Breeding -2014. -V. 34 (3). -P. 779-788
- DOI: 10.1007/s11032-014-0074-7
- Wieckhorst S., Bachlava E., Dußle C.M., Tang S., Gao W., Saski C., Bauer E. Fine mapping of the sunflower resistance locus PlARG introduced from the wild species Helianthus argophyllus//Theoretical and Applied Genetics. -2010. -V. 121 (8). -Р. 1633-1644
- DOI: 10.1007/s00122-010-1416-4
- Bert P.F., Tourvieille de Labrouhe D., Philippon J., Mouzeyar S., Jouan I., Nicolas P., Vear F. Identification of a second linkage group carrying genes controlling resistance to downy mildew (Plasmopara halstedii) in sunflower (Helianthus annuus L.)//Theoretical and Applied Genetics. -2001. -V. 103 -P. 992-997.
- Bertero de Romano A., Romano C., Bulos M., Altieri E., Sala C. A new gene for resistance to downy mildew in sunflower//Proc. Intern. Symposium "Sunflower Breeding on Resistance to Diseases", Russia, Krasnodar, 2010, June 23-24. -P. 141-146.
- Mulpuri S., Liu Z., Feng J., Gulya T. J., Jan C.-C. Inheritance and molecular mapping of a downy mildew resistance gene, Pl13 in cultivated sunflower (Helianthus annuus L.)//Theoretical and Applied Genetics. -2009. -V. 119 (5). -P. 795-803
- DOI: 10.1007/s00122-009-1089-z
- Liu Z., Gulya T.J., Seiler G.J., Vick B.A., Jan C-C. Molecular mapping of the Pl16 downy mildew resistance gene from HA-R4 to facilitate marker-assisted selection in sunflower//Theoretical and Applied Genetics. -2012. -V. 125. -P. 121-131.
- Rahim M., Jan C.C., Gulya T.J. Inheritance of resistance to sunflower downy mildew races 1, 2 and 3 in cultivated sunflower//Plant Breed. -2002. -V. 121. -P. 57-60.
- Molinero-Ruiz M.L., Domínguez J., Melero-Vara J.M. Races of isolates of Plasmopara halstedii from Spain and Studies on their virulence//Plant Disease -2002. -V. 86 (7). -P. 736-740
- DOI: 10.1094/pdis.2002.86.7.736
- Gedil M.A., Slabaugh M.B., Berry S., Johnson R., Michelmore R., Miller J., Gulya T., Knapp S.J. Candidate desease resistance genes in sunflower cloned using consereved nucleotide-binding site motifs: Genetic mapping and linkage to the downy mildew resistence gene Pl1//Genom. -2001. -V. 44 (2). -Р. 205-212.
- Ahmed S., Tourvieille de Labrouhe D., Delmotte F. Emerging virulence arising from hybridisation facilitated by multiple introductions of the sunflower downy mildew pathogen Plasmopara halstedii//Fungal Genetics and Biology -2012. -V. 49 (10) -P. 847-855
- DOI: 10.1016/j.fgb.2012.06.012


