Люди из каменного саркофага № 11 Юрьева монастыря: генетическая история на основе митохондриальных геномов
Автор: Андреева Т.В., Добровольская М.В., Седов В.В., Вдовиченко М.В., Решетова И.К., Сошкина А.Д., Дудко Н.А., Быданов А.С., Адрианова И.Ю., Бужилова А.П., Рогаев Е.И.
Журнал: Краткие сообщения Института археологии @ksia-iaran
Рубрика: Естественнонаучные методы в археологических исследованиях
Статья в выпуске: 270, 2023 года.
Бесплатный доступ
Палеогенетические исследования открывают новые возможности в изучении состава населения средневекового Новгорода. Мы представляем первый опыт работы с полными митохондриальными геномами индивидов из саркофага с захоронениями знати Новгорода XII-XIII вв. Саркофаг № 11 наряду с другими «сборными» саркофагами был обнаружен Новгородским архитектурно-археологическим отрядом Института археологии РАН в 2019 г. Он содержал останки трех индивидов. В результате проведенного полногеномного секвенирования мы подтвердили половую принадлежность двух костяков (Индивид 1 и Индивид 3) женщине и мужчине соответственно, а также определили женский пол у ребенка (Индивид 2). Нами впервые определены митохондриальные гаплогруппы у знати домонгольского Новгорода. Предположение о том, что в верхнем ярусе саркофага одномоментно захоронены близкие родственники, например, мать и дочь или тетка и племянница, или далекие родственники по женской линии, не оправдалось. Наличие родственных связей других степеней требует проверки и остается перспективной задачей по работе с полногеномыми данными этих погребенных. Митохондриальный геном взрослой женщины относится к гаплогруппе, присутствующей в древнерусском «курганном» населении Русского Севера и средневекового Ярославля. Два другие индивида представлены редкими митохондриальными гаплогруппами, не выявленными ранее у средневекового населения Древней Руси.
Митохондриальные гаплогруппы, новгородская знать, xii-xiii вв
Короткий адрес: https://sciup.org/143180605
IDR: 143180605 | DOI: 10.25681/IARAS.0130-2620.270.418-437
Текст научной статьи Люди из каменного саркофага № 11 Юрьева монастыря: генетическая история на основе митохондриальных геномов
Исследование генетического состава древнего населения Новгорода – большая и актуальная научная задача, выполнение которой становится возможным благодаря развитию палеогеномики и биоинформатики. Исторический смысл этих исследований проявляется при взаимодействии с историческими, археологическими, палеоантропологическими данными, характеризующими этот крупнейший город севера Руси. Археологические и антропологические исследования неоднократно демонстрировали сложность состава населения новгородских земель, поэтому необходимым условием анализа палеогеномных данных является изученность археологического контекста. Этот небольшой сюжет основан на исследовании митохондриальной ДНК, которая была выделена из скелетных останков людей, погребенных в одном из каменных саркофагов некрополя Георгиевского собора Юрьева монастыря Великого Новгорода. Более ранними исследованиями обосновано, что погребения в саркофагах у стен Георгиевского собора связаны с новгородским боярством ( Седов , 2018. С. 155). Летописные упоминания касаются фактов смерти и погребения 1203 и 1209 гг.: «преставися Мирошька, посадник новъгородьскыи, постригъся у святого Георгия» (Новгородская первая летопись. С. 45); «привезоша Дмитра Мирошкиниця мьрътвого из Володимиря и погрѣбоша и у святого Георгия въ манастыри, подъле отчя; а новгородьци хотяху съ моста съврѣщи, нъ възбрани имъ архепископъ Митро-фанъ» (Там же. С. 51, 248). В 1230 г. «На ту же зиму поиде княжиць Ростислав съ посадникомь Вънездомь на Тържькъ, мѣсяця декабря, въ недѣлю; а заутра убиша Смена Борисовиця, а домъ его всь розграбиша и села, а жену его яша, а самого погрѣбоша у святого Гюргя въ манастыри» (Там же. С. 70, 277).
Население Новгорода домонгольского периода с точки зрения физической антропологии и генетики до сих пор остается terra incognita по причине отсутствия ранних городских кладбищ. Антропологические материалы из новгородских земель происходят из Старой Ладоги, Пскова и ряда могильников сельских территорий. В среде палеоантропологов вопрос о происхождении словен новгородских остается в поле дискуссии. Ведущий ученый в области антропологических аспектов изучения этногенеза славян – Т. И. Алексеева – высказывала крайне осторожное суждение по этому поводу: «Истоки ильменских словен, а именно с ними связывается культура новгородских сопок, пока еще недостаточно ясны. Есть предположение об их родстве с балтийскими славянами» (Алексеева, 2002. С. 160). С миграцией славян из южноприбалтийских земель связывает словен новгородских В. В. Седов (Седов, 1952; 1979; 1982). Н. Н. Гончарова, базируясь на результатах краниологических исследований, видит «генетические связи словен новгородских с балтийскими славянами и их относительную обособленность от восточнославянского мира» (Гончарова, 2003. С. 211). С. Л. Санкина по краниологическим признакам выделяет общность балтов, славян и ранних жителей Эстонии, обусловленную их происхождением от носителей традиций культуры шнуровой керамики раннего бронзового века (Санкина, 2000). Ею же отмечается неоднородность антропологического состава населения Новгородской земли. Таким образом, основываясь на палеоантропологических данных, мы имеем достаточно сложную картину, что убеждает нас в важности крайне осторожного отношения к обобщениям и повышенного внимания к культурно-хронологической ситуации.
Реконструкция геномного своеобразия средневекового населения Новгородчины, выполненная на основании широкогеномного анализа современных людей из этих территорий, недавно опубликована в журнале «Генетика» ( Балановская и др. , 2021). Авторы предполагают, что древний генетический пласт современной Новгородчины восходит к генофонду ильменских словен, который сохранил многие генетические черты местного дославянского населения, прежде всего, волжских и пермских финно-язычных групп. Результаты палеогене-тических исследований, проводимых на материалах из могильников сельских территорий новгородских земель (Ижорское плато) ( Стасюк и др. , 2022. С. 52– 53), дополняют наши знания новыми конкретными данными.
Следует иметь в виду, что население североевропейского средневекового города, находящегося на путях трансконтинентальных торговых путей, конечно же, может включать в свой состав мигрантов первого поколения. Показательным в этом отношении является исследование генетического состава и изотопных показателей мобильности жителей Сигтуны X–XII вв. ( Krzewińska et al. , 2018). Исследователи констатировали высокий уровень миграционной активности мужчин и женщин, который фиксируется палеогенетическими и изотопными маркерами. При этом обнаружены не следы переселения групп родственников из каких-либо центров, а миграция «сетевого типа», при которой близкими родственниками являются люди, живущие на дальних расстояниях друг от друга. Высокая мобильность свойственна мужчинам и женщинам, людям из различных по социальному статусу групп, различных кладбищ. Авторы приходят к гипотезе о формировании особой системы расселения в среде североевропейских городов, изучать которую нужно, объединяя массивы данных из многих урбанистических центров, а не какого-то одного региона.
Наши представления об истоках формирования населения Новгорода основываются на концепции объединения представителей различных этнических группировок в форматах городских концов. В. Л. Янин определенно высказывается о том, что ключевую роль в появлении города сыграли «три поселка родовой аристократии» ( Янин , 2008. С. 378). Из общих соображений можно предположить, что боярство, обладающее значительными материальными ресурсами, стремясь к их приумножению, конфликтуя и соперничая, регламентировало заключение браков внутри определенного социального слоя. Поэтому есть основания ожидать, что поколения социальной верхушки Новгорода были объединены кровными связями, которые способствовали сохранению их высокого социального и имущественного положения ( Янин , 1981. С. 213–229).
Эти наблюдения, безусловно, делают нашу конечную задачу – изучение происхождения и структуры населения средневекового Новгорода домонгольского времени – особо сложной, а работу максимально кропотливой. Поэтому оправдано обращение к локальному погребальному объекту, имеющему яркую историческую, культурную, социальную характеристику – каменному саркофагу у стены Георгиевского собора Юрьева монастыря на южной окраине Великого Новгорода.
Археологический контекст и скелетные останки погребенных
Архитектурно-археологические исследования Георгиевского собора (1119 г.) ведутся Новгородским архитектурно-археологическим отрядом Института археологии РАН с 2013 г. В 2019 г. исследуемый участок примыкал к южной стене собора. Во время работ непосредственно у южного портала обнаружились три каменных саркофага, расположенных «лесенкой», т. е. как будто спускающихся по склону. С севера на юг это саркофаги № 7, 8 и 11; все они принадлежат к типу «сборных» саркофагов из шести плит и датируются XII–XIII вв. ( Седов, Вдовиченко , 2021). Все погребенные, судя по положению их каменных гробниц, принадлежали к верхним социальным слоям Новгорода, вероятнее всего, боярам, бывшим вкладчиками важнейшего в городе монастыря (рис. 1).
Саркофаг № 11 относится к сборным саркофагам из шести плит, плиты очень хорошо обработаны, пазы очень тщательно пригнаны друг к другу. Расчищенный саркофаг производит сильное впечатление благодаря хорошей ремесленной работе каменщиков, правильности поверхностей и форм и тщательным их сочленением.
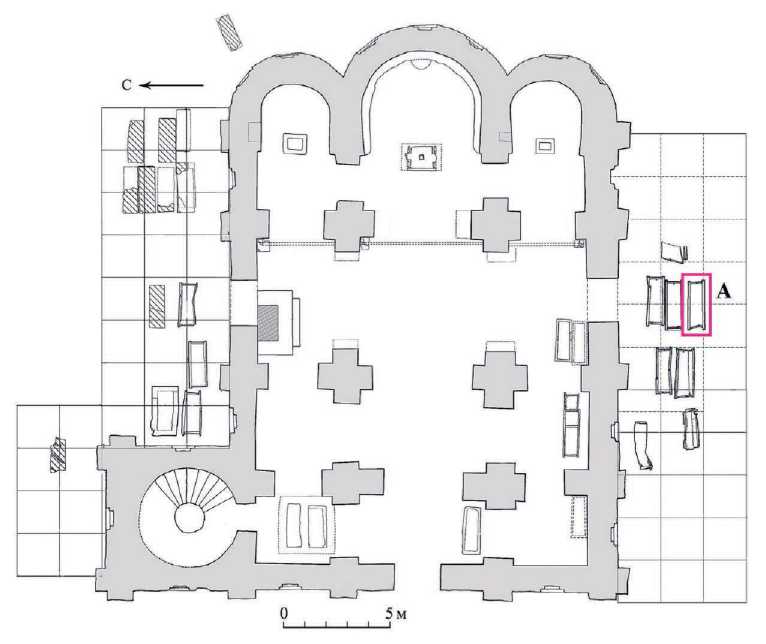
Рис. 1. Некрополь вокруг Георгиевского собора Юрьева монастыря.
План плит и саркофагов с выделением саркофага № 11.
Чертеж Е. Н. Пророковой, Ю. С. Фомичёвой, 2019
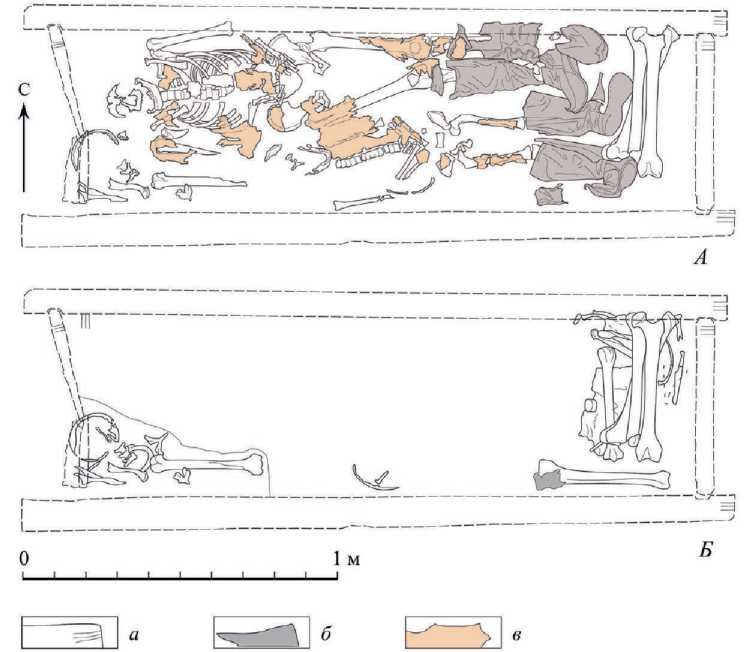
Рис. 2. Некрополь вокруг Георгиевского собора Юрьева монастыря.
Саркофаг № 11. Планы 1 (индивид 1, 2) и 2 уровней (индивид 3).
Чертежи Е. Н. Пророковой, 2019
Погребения в саркофаге № 11 были прикрыты сложенным вдвое покровом из бересты, который полностью перекрывал все внутреннее пространство. Очевидно, захоронение было неповрежденным, и все останки сохранили свое положение in situ (рис. 2).
После снятия бересты открылись останки первого уровня. Это погребение женщины более 50 лет (индивид 1). У нее в ногах был погребен ребенок 4–5 лет (индивид 2). На этом уровне в восточной части открыты кожаные сапоги, которые были надеты на ноги погребенных женщины и ребенка. Отчетливо проявлены контуры скелета погребенного: череп, ребра, левая рука сложена под тупым углом, так что ладонь покоилась на животе; ноги.
На следующем, нижнем уровне обнаружены кости с востока и кости (индивид 3, мужчина более 50 лет) в юго-западном углу; в восточной части зафиксированы фрагменты кожаной обуви индивида 3. Остальную поверхность занимал тлен от лежавших выше погребений. Важно отметить, что кости индивида 3
находились не в анатомическом сочленении. Часть скелета располагалась в югозападном углу (череп, шейные позвонки, ключицы, правая плечевая кость). Лопатки, ребра, тазовые кости, длинные кости конечностей компактно и аккуратно сложены в восточном углу. Отметим, что на скелетах не было обнаружено следов инфекционных заболеваний, травм, маркеров стрессов или анемических состояний. Мужчина и женщина достигли возраста, не столь часто встречавшегося в то время в обычных популяциях, сохранив хорошие формальные показатели здоровья. Наиболее вероятной, но гипотетической причиной смерти в этом контексте представляется скоротечное инфекционное заболевание.
Остановимся на двух обстоятельствах.
Традиция обращения с останками. Многократное использование саркофага предполагает наличие традиции обращения с останками, положенными с усыпальницу ранее. В том случае, если последующее захоронение приходится проводить через несколько месяцев или год-два, кости скелета еще соединены мягкими тканями, и остается лишь поместить еще одно тело поверх останков. В том случае, если мягкие ткани уже истлели, можно положить тело поверх, а можно переместить кости. Последнее, вероятно, производится с целью сохранения пространства (но это только догадка), чтобы увеличить срок возможности использования саркофага. Перемещение костей с подчеркнутым нарушением анатомической последовательности известно для христианских византийских захоронений в каменных саркофагах (например, могильник Горзувиты). Это позволяет эффективно использовать весь объем усыпальницы, превращая ее, отчасти, в костницу ( Добровольская, Мастыкова , 2020. С. 85–87). В этом случае нарушается локализация костей каждого скелета, кости от разных скелетов могут оказаться преднамеренно перемешанными, что подчеркивает их общность в этом саркофаге.
Продолжительность использования саркофага. Верхний слой останков производит впечатление одновременного захоронения двух людей (женщины и ребенка). Наиболее вероятной причиной смерти этих людей можно считать острое инфекционное заболевание, так как никаких следов тяжелых хронических недугов на скелетах не обнаружено. Чтобы совершить это двойное захоронение, были перемещены кости скелета мужчины. Повторим, что анатомические сочленения нарушены, однако в юго-западном углу около черепа с нижней челюстью обнаружены три шейных позвонка в сочленении. Это дает нам основание предполагать, что время между двумя эпизодами использования саркофага было не слишком большим. В самом общем виде, основываясь на опыте судебно-медицинских работ, можно предположить, что почти полностью завершенный процесс скелетирования останков проходит примерно за год ( Величко , 2000). Безусловно, важно иметь в виду, что есть стимулирующие и консервирующие факторы, поэтому год – очень примерная характеристика. Тем не менее два эпизода захоронения в саркофаг № 11, вероятно, отделены друг от друга небольшим временем. Хотя продолжительность использования каменных гробниц могла быть и многим больше, порою доходя до двух с половиной – трех столетий ( Добровольская, Мастыкова , 2020. С. 89).
Саркофаги рассматриваются как семейные усыпальницы, и палеогенетиче-ский анализ поможет подтвердить или опровергнуть эти гипотезу.
Образцы и методы палеогенетического исследования
Генетический анализ проводили в лабораторных помещениях, специально оборудованных для проведения работ с древней ДНК. Для выделения ДНК использовали около 100 мг костного порошка, полученного из фрагментов корней зубов трех исследованных индивидов (рис. 3), экстракцию проводили по описанному нами ранее протоколу ( Andreeva et al. , 2022). Выделенную ДНК использовали для приготовления фрагментных геномных библиотек на основе одноцепочечных фрагментов ДНК ( Gansauge et al. , 2017) и секвениро-вали на платформе Illumina. Полученные в результате секвенирования короткие прочтения после удаления адаптерных последовательностей с помощью AdapterRemoval ( Schubert et al. , 2016) картировали с помощью bwa ( Li, Durbin , 2009) на референсную митохондриальную последовательность человека (rCRS, NC_012920.1). Дополнительно для определения пола исследованных индивидов полученные прочтения были картированы на референсный геном человека. ПЦР-дупликаты были маркированы с помощью MarcDuplicate (Picard, 2019), аутентичность ДНК оценивали с помощью MapDamage ( Jónsson et al. , 2013). Генетический пол определяли по соотношению среднего покрытия половых хромосом (отдельно для Х- и Y-хромосом) к среднему покрытию аутосом, при этом использовали только прочтения длиной более 30 нт с качеством картиро-вани (MQ) более 30. Митохондриальные гаплогруппы определяли с помощью HaploGrep 2 ( Weissensteiner et al. , 2016). Филогенетический анализ проводили с использованием MEGA ( Kumar et al. , 2018). Для сравнительного анализа мтДНК использовали информацию и полные последовательности мтДНК древних индивидов, представленные в базах GenBank, YFull, AmtDB, Logan DNA Project и PhiloTree.
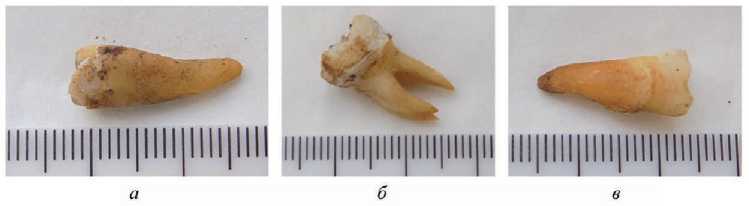
Рис. 3. Археологические образцы из саркофага 11 Георгиевского собора, использованные для проведения генетического анализа а – индивид 1 (женщина старше 50 лет); б – индивид 2 (ребенок 4–5 лет); в – индивид 3 (мужчина старше 50 лет)
Результаты
Геномная ДНК, выделенная из фрагментов корней зубов трех индивидов, захороненных в саркофаге № 11 была использована для приготовления фрагмент-ных геномных библиотек, статистика по результатам секвенирования представлена в табл. 1. Для всех трех образцов была подтверждена их аутентичность (рис. 4).
Таблица 1. Общая информация о результатах секвенирования ДНК трех индивидов*
|
% прочтений, картированных на геном |
Генетический пол |
Среднее покрытие мтДНК |
|
|
Индивид 1 |
18,71 % |
Ж |
32.99х |
|
Индивид 2 |
3,12 % |
Ж |
26.70х |
|
Индивид 3 |
1,83 % |
М |
8.96х |
Примечание : * после удаления адаптерных последовательностей.
В результате анализа геномных данных был подтвержден предполагаемый пол индивида 1 и 3 (женский и мужской соответственно), а также определен пол ребенка – женский (индивид 2).
Среднее покрытие митохондриальной ДНК, полученное в результате секвенирования, позволило определить митохондриальные гаплогруппы для всех трех людей из саркофага № 11 (табл. 2). Данные индивиды оказались носителями трех различных митохондриальных гаплогрупп (I1a1a1, H7b и W5a), что исключает их родство по материнской линии.
Таблица 2. Варианты, выявленные в последовательностях мтДНК трех индивидов из саркофага № 11 и их митохондриальные гаплогруппы
|
Гаплогруппа мтДНК |
Варианты, выявленные в исследованном образце (в соответствии с rCRS) |
|
Индивид 1 I1a1a1 |
ГВР2: 73G 199C 203A 204C 250C 263G 309.1C 315.1C 455.1T 573.1C Кодирующий регион: 750G 1438G 1719A 2706G 3447G 3990T 4529T 4769G 6734A 7028T 8251A 8616T 8860G 9053A 9947A 10034C 10238C 10398G 10915C 11719A 12501A 12705T 13780G 14766T 15043A 15326G 15547T 15924G ГВР1: 16129A 16172C 16223T 16311C 16391A 16519C |
|
Индивид 2 H7b4 |
ГВР2: 189G 263G 315.1C Кодирующий регион: 750G 1438G 4688C 4769G 4793G 5348T 8860G 13980A 14207A 15326G ГВР1: 16519C |
Окончание табл. 2
|
Гаплогруппа мтДНК |
Варианты, выявленные в исследованном образце (в соответствии с rCRS) |
|
Индивид 3 W5a |
ГВР2: 73G 189G 194T 195C 199C 204C 207A 263G 315.1C Кодирующий регион: 709A 750G 1243C 1438G 2706G 3505G 4769G 5046A 5460A 6040G 6528T 7028T 8251A 8705C 8860G 8994A 10097G 11674T 11719A 11947G 12414C 12705T 14766T 15326G 15775G 15884C ГВР1: 16223T 16292T 16362C 16519C |
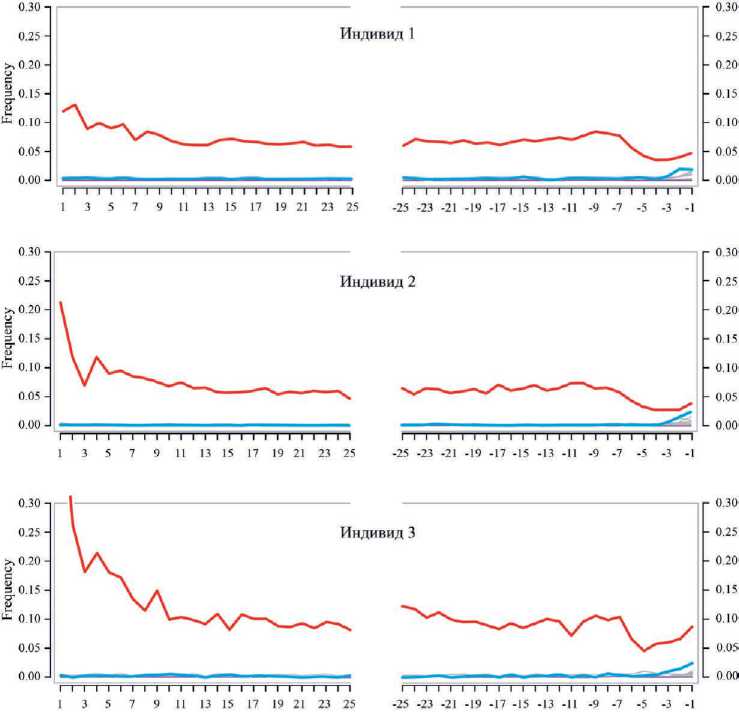
Рис. 4. Профили нуклеотидных замен в образцах ДНК трех индивидов из саркофага № 11 соответствуют профилю древней ДНК – наблюдается повышенная частота замен C > T (красный), связанная с постмортальными мутациями, по всей длине фрагментов ДНК и особенно к их концам
Мы провели поиск митохондриальных последовательностей, принадлежащих гаплогруппам I1a1a1, H7b и W5a в базах данных GenBank ( www.ncbi.nlm . nih.gov/genbank/), AmtDB ( https://amtdb.org/ ), Ian Logan mtDNA (http://www. ianlogan.co.uk) и YFull MTree 1.02 ( https://www.yfull.com/mtree/ ), и отобрали все древние последовательности мтДНК с известным этническим или географическим происхождением. Дополнительно была использована информация, представленная в базе www.haplogroup.info , а данные геномного секвенирования образцов, принадлежащих согласно этой базе к гаплогруппам I1a1a1, H7b и W5a, были использованы для реконструкции полных последовательностей мтДНК. Кроме того, в анализ были включены последовательности мтДНК древних образцов с территории России, из собственной базы митохондриальных геномов (лаборатории эволюционной геномики ИОГен РАН и НТУ Сириус), включающей более 400 образцов от палеолита до начала XX в.
Индивид 1
Митохондриальная ДНК женщины (индивида 1) принадлежит гаплогруп-пе I1a1a1, которая является одной из ветвей древней европейской гаплогруп-пы I, распространенной в настоящее время на территории современной Европы, Ближнего Востока и некоторых регионов Африки. Наибольшей частоты гапло-группы I в современной Евразии достигает в Иране и Турции (до 9,8 %), где также наблюдается наибольшее разнообразие ее подгрупп ( Olivieri et al. , 2013), а также в восточнославянской группе лемков (или русинов), проживающих в западной части Карпатского нагорья – 11,3 % ( Nikitin et al. , 2009). Клада I1a, являющаяся одной из дочерних клад гаплогруппы I, чаще всего встречается на северо-востоке современной Европы ( Olivieri et al ., 2013). Ее максимальная частота была выявлена в эпоху викингов у жителей Скандинавии ( Melchior et al. , 2010), где сейчас не превышает 5,9 % ( Mikkelsen et al. , 2010).
Для проведения сравнительного анализа в перечисленных выше базах данных нами были найдены 10 древних митохондриальных последовательностей, относящихся к кладе I1a1a и ее ветви I1a1a1, к которой принадлежит мтДНК индивида 1 (табл. 3).
Таблица 3. Митохондриальные последовательности, принадлежащие гаплогруппе I1a1a, выявленные в открытых базах данных древних образцов
|
GeneBank ID или шифр образца |
Происхождение, археологический возраст |
Число отличий от индивида 1 (нт)* |
Источник |
|
|
1 |
AB35 |
Вологодская обл., Новинки, XI–XII вв. |
1 |
Собственные данные |
|
2 |
WEZ64 |
Германия, бронзовый век, Толленс |
1 |
( Burger et al ., 2020) |
|
3 |
MHper87 |
Венгрия, 900 лет назад, сборная серия |
2 |
( Maár et al. , 2021) |
Окончание табл. 3
|
GeneBank ID или шифр образца |
Происхождение, археологический возраст |
Число отличий от индивида 1 (нт)* |
Источник |
|
|
4 |
VK458 |
Швеция, Готланд, эпоха викингов |
2 |
( Margaryan et al ., 2020) |
|
5 |
Индивид № 2, Захоронение № 76 |
Россия, Ярославль, 1238 г. |
3 |
( Андреева и др ., 2021) |
|
6 |
MHper28 |
Венгрия, 900 лет назад, сборная серия |
3 |
( Maár et al ., 2021) |
|
7 |
R57 |
Италия, средневековье |
3 |
( Antonio et al ., 2019) |
|
8 |
I4331 |
Хорватия, бронзовый век |
4 |
( Mathieson et al ., 2015) |
|
9 |
I6537 |
Польша, бронзовый век |
5 |
( Olalde et al ., 2018) |
|
10 |
I0234 |
Россия, Самара, срубная культура |
10 |
( Mathieson et al ., 2015) |
Примечание : * при анализе не учитывали позиции, в которых не определен нуклеотид (обозначен N в доступной последовательности из GenBank), а также варианты в полиС-трактах.
Обратим внимание на митохондриальные геномы, из которых отличаются 1–3 замены, чтобы определить максимально близких индивидов.
Различия по одной позиции мтДНК выявлены с индивидом № 1 из кургана древнерусского времени Вологодской области (памятник Новинки, XI–XII вв.). Кроме того, митохондриальная последовательность индивида 1 имеет отличия в 2–3 нуклеотидные замены с двумя персонами из обширной выборки населения Венгрии X–XI вв. Специфика этой выборки состоит в том, что она объединяет элиту и «простолюдинов» ( Maár et al. , 2021). Элита этого времени четко представлена мигрантным населением финно-угорского происхождения. Наши образцы (MHper87 и MHper28) происходят не из элитных захоронений и отражают специфику восточноевропейского населения.
Образец WEZ64 (Толленское поле биты) происходит из крупнейшего известного к настоящему времени «следа» битвы бронзового века в северной Германии (~ 3200 лет до н. э.). Это старейшее крупномасштабное конфликтное место к северу от Альп. Генетические данные показывают, что эти люди представляют единую неструктурированную популяцию Центральной/Северной Европы) ( Burger et al. , 2020).
Различия по двум позициям также выявлено с индивидом из кладбища эпохи викингов с территории Готланда (образец VK458).
И, наконец, три нуклеотидные замены отличают индивида 1 из саркофага № 11 от индивида мужского пола № 2 коллективного захоронения в Ярославле (сооружение 76) (Андреева и др., 2021), что позволяет предположить, что данная митохондриальная клада была распространена в средние века на территории Древней Руси.
Индивид 2
Гаплогруппа H7b4, выявленная у девочки (индивид 2), является одной из ветвей самой распространенной в Европе гаплогруппы H, которая составляет до 50 % от всех гаплогрупп в некоторых современных популяциях ( Roostalu et al ., 2007). Носители ее дочерней подгруппы H7 чаще всего встречаются сегодня на Ближнем Востоке, на Кавказе, а также в балто-славянском регионе. По результатам анализа открытых баз данных было обнаружено, что гаплогруп-па H7b4 является очень редкой как в современных, так и в древних популяциях, поэтому для сравнительного анализа нами были отобраны все древние индивиды (9 человек), митохондриальные последовательности которых относятся к га-плогруппе H7b4, а также к ее предковой кладе H7b (табл. 4).
Таблица 4. Митохондриальные последовательности, принадлежащие гаплогруппам H7b и H7b4, выявленные в открытых базах данных
|
GeneBank ID или шифр образца |
Происхождение, археологический возраст |
Число отличий от индивида 2 (нт)* |
Источник |
|
|
1 |
I4778 |
Казахстан, поздний бронзовый век, Карагаш 2 |
1 |
( Narasimhan et al ., 2019) |
|
2 |
BRE001 |
Казахстан, пазырыкская культура |
4 |
( Gnecchi-Ruscone et al ., 2021) |
|
3 |
I11214 |
Красноярск, бронзовый век |
4 |
( Narasimhan et al ., 2019) |
|
4 |
VK417 |
Норвегия, эпоха викингов |
4 |
( Margaryan et al ., 2020) |
|
5 |
VK461 |
Швеция, эпоха викингов |
4 |
(Ibid.) |
|
6 |
MK059574 |
Дания, средневековье |
5 |
( Klunk et al ., 2019) |
|
6 |
NLKJRT_M49 |
Китай, железный век |
5 |
( Wang et al ., 2021) |
|
8 |
VK370 |
Дания, эпоха викингов |
6 |
( Margaryan et al ., 2020) |
|
9 |
DA74 |
Казахстан, железный век |
12 |
( Damgaard et al. , 2018) |
Примечание : * при анализе не учитывали позиции, в которых не определен нуклеотид (обозначен N в доступной последовательности из GenBank), а также варианты в полиС-трактах.
Среди древних образцов был обнаружен только один, отличающийся на 1 замену от нашего, различия с остальными составили 4 нуклеотида и более, что говорит об относительно давнем разделении их митохондриальных линий (несколько тысяч лет назад). Наиболее близок образец из памятника Карагаш 2 с территории Казахстана, датировка 1728–1546 до н. э. Культурная атрибутика отсутствует. Индивид рассматривается авторами публикации как носитель значительной доли генофонда ямной культуры ( Narasimhan et al. , 2019). Следует, однако, отметить, что в этом образце не определены нуклеотиды в 26 позициях мтДНК, таким образом общее число различий может быть больше. В целом, полученные нами данные позволяют предположить, что редкая материнская линия девочки из саркофага № 11 (индивид 2), принадлежащая гаплогруппе H7b4, вероятно, не была распространена на территории Древней Руси в первом тысячелетии нашей эры.
Индивид 3
Анализ полной последовательности мтДНК индивида 3, для которого c использованием Haplogrep 2 была предсказана гаплогруппа W5a, показал отсутствие прочтений на участке, соответствующем позициям 186–214 rCRS. Согласно филогенетическому дереву митохондриальных гаплогрупп человека (PhyloTree.org), последовательности мтДНК, относящиеся к гаплогруппе W5a, содержат в этом локусе пять замен по сравнению с последовательностью rCRS (189G 194T 195C 204C 207A), что потенциально может затруднять картирование полученных в результате глубокого секвенирования коротких прочтений на этот участок. Для проверки этой гипотезы мы дополнительно картировали данные секвенирования индивида 3 на митохондриальную последовательность, принадлежащую гаплогруппе W5а из базы данных GeneBank (GU817015). В результате картирования на эту последовательность данных секвенирования ДНК индивида 3, участок, соответствующий позициям 186–214 rCRS, оказался полностью покрыт прочтениями, что позволило определить полный гаплотип мтДНК индивида 3 (табл. 2): помимо маркеров гаплогруппы W5a, в его геноме на данном участке присутствует дополнительный уникальный вариант 199C.
Для проведения сравнительного анализа мтДНК мужчины (индивид 3) из саркофага № 11 c последовательностями мтДНК из открытых баз данных нами были выявлены 9 полных митохондриальных генома, принадлежащих гаплогруппе W5a (табл. 5). Детальный анализ отобранных последовательностей показал, что в большинстве из них частично или полностью не определены нуклеотидные последовательности на участке, соответствующем позициям 186–214 rCRS, который содержит важные диагностические маркеры гаплогруппы W5а. Вероятно, в связи с этим не все последовательности в базах данных, относящиеся к этой гаплогруппе, могут быть выявлены. Тем не менее, наиболее близкие последовательности (различия в 3–5 нуклеотидных позициях) к мтДНК Индивида 3 были выявлены среди древних образцов с территории Польши (бронзовый век), Чехии (неолит) и Германии (медный век). Полученные результаты позволяют предположить, что гаплогруппа W5a, выявленная у мужчины (индивид 3), встречалась в древних европейских популяциях, но не на территории современной России.
Таблица 5. Митохондриальные последовательности, принадлежащие гаплогруппе W5a, выявленные в открытых базах данных
|
GeneBank ID или шифр образца |
Происхождение, археологический возраст |
Число отличий от индивида 3 (нт)* |
Источник |
|
|
1 |
I2405 |
Польша, бронзовый век |
3# |
( Tassi et al. , 2017) |
|
2 |
I7280 |
Чехия, неолит |
3 |
( Olalde et al ., 2018) |
|
3 |
I5834 |
Германия, медный век |
5# |
(Ibid.) |
|
4 |
LM51 |
Англия, средние века |
6 |
( Klunk et al ., 2019) |
|
5 |
MT588295 |
Польша, неолит |
6 |
( Juras et al ., 2021) |
|
6 |
G117 |
Дания, средние века |
7 |
( Krause-Kyora et al ., 2018) |
|
7 |
poz280 |
Польша, неолит |
8 |
( Juras et al. , 2021) |
|
8 |
MF114218 |
Польша, неолит |
11# |
( Tassi et al. , 2017) |
Примечания : * при анализе не учитывали позиции, в которых не определен нуклеотид (обозначен N в доступной последовательности из GenBank), а также варианты в полиС-трактах; # нуклеотидные последовательности в позициях 186-214 rCRS не определены.
Заключение
Первый опыт работы с полными митохондриальными геномами индивидов из саркофага с захоронениями знати Новгорода XII–XIII вв. показал отсутствие у погребенных родственных связей по женской линии. В результате проведенного полногеномного секвенирования мы подтвердили половую принадлежность двух костяков (индивид 1 и индивид 3) женщине и мужчине соответственно, а также определили женский пол у ребенка (индивид 2). Нами впервые определены митохондриальные гаплогруппы у знати домонгольского Новгорода. Предположение о том, что в верхнем ярусе саркофага одномоментно захоронены, например, мать и дочь или тетка и племянница, или даже далекие родственники по женской линии, не оправдалось. Наличие родственных связей других степеней требует проверки и остается перспективной задачей по работе с полногеномы-ми данными этих погребенных. Митохондриальный геном взрослой женщины относится к гаплогруппе, присутствующей в древнерусском «курганном» населении Русского Севера и средневекового Ярославля. Два другие индивида представлены редкими митохондриальными гаплогруппами, не выявленными ранее у средневекового населения Древней Руси.
Список литературы Люди из каменного саркофага № 11 Юрьева монастыря: генетическая история на основе митохондриальных геномов
- Алексеева Т. И., 2002. Антропологическая характеристика восточных славян эпохи средневековья в сравнительном освещении // Восточные славяне. Антропология и этническая история / Отв. ред. Т. И. Алексеева. М.: Научный Мир. С. 160–169.
- Андреева Т. В., Малярчук А. Б., Григоренко А. П., Кунижева С. С., Манахов А. Д., Эгноватова А. В., Рогаев Е. И., 2021. Археогенетический анализ индивида из захоронения с территории древнего Ярославского кремля // КСИА. Вып. 265. С. 209–308.
- Балановская Е. В., Черневский Д. К., Балановский О. П., 2021. Своеобразие Новгородского генофонда в контексте народонаселения европейской части России // Вестник Новгородского государственного университета. № 3 (124). Медицинские науки. С. 51–57.
- Величко Н. Н., 2000. Основы судебной медицины и судебной психиатрии: учебник. М.: Центр информ. и науч.-метод. обеспечения кадровой политики М-ва внутренних дел России. 325 с.
- Гончарова Н. Н., 2003. Антропология словен новгородских и вопросы их происхождения // Горизонты антропологии: тр. Междунар. науч. конф. памяти акад. В. П. Алексеева / Отв. ред. Т. И. Алексеева. М.: Наука. С. 206–211.
- Добровольская М. В., Мастыкова А. В., 2020. О датировке византийского храма в Горзувитах с помощью методов естественных наук // ΧΕΡΣΩΝΟΣ ΘΕΜΑΤΑ: Империя и полис: XII Междунар. Византийский семинар (Севастополь – Балаклава, 25–29 мая 2020 г.): материалы науч. конф. / Отв. ред. Н. А. Алексеенко. Симферополь: Колорит. С. 85–90.
- Новгородская первая летопись старшего и младшего изводов. М.; Л.: Изд-во АН СССР, 1950. 640 с.
- Санкина С. Л., 2000. Этническая история средневекового населения Новгородской земли. СПб.: Дмитрий Буланин. 110 с.
- Седов В. В., 1952. Антропологические типы населения северо-западных земель Великого Новгорода // Краткие сообщения Института этнографии. Вып. XV. С. 72–85.
- Седов В. В., 1979. Этнический состав населения Новгородской земли // Финно-угры и славяне / Отв. ред. Б. А. Рыбаков. Л.: Наука. С. 74–80.
- Седов В. В., 1982. Восточные славяне в VI–XIII вв. М.: Наука. 327 с. (Археология СССР.)
- Седов Вл. В., 2018. Княжеские саркофаги Георгиевского собора Юрьева монастыря // Вестник РФФИ. Гуманитарные и общественные науки. № 2. С. 142–158.
- Седов Вл. В., Вдовиченко М. В., 2021. Архитектурно-археологические работы в Юрьеве монастыре в 2019 г. // ННЗ. Вып. 34. С. 65–77.
- Стасюк И. В., Мустафин Х. Х., Альборова И. Э., 2022. Опыт сопоставления археологических и генетических данных в изучении средневековых некрополей Ижорского плато // Археология погребений: современные теоретические и методические подходы. Тез. докл. Всероссийской науч. конф. с международным участием, посвященной памяти Валерия Ивановича Гуляева / Отв. ред. Д. С. Коробов. М.: ИА РАН. С. 52–53.
- Янин В. Л., 1981. Новгородская феодальная вотчина (историко-генеалогическое исследование). М.: Наука. 296 с.
- Янин В. Л., 2008. Очерки истории средневекового Новгорода. М.: Языки славянских культур. 400 с.
- Andreeva T. V., Manakhov A. D., Gusev F. E., Patrikeev A. D., Golovanova L. V., Doronichev V. B., Shirobokov I. G., Rogaev E. I., 2022. Genomic Analysis of a Novel Neanderthal from Mezmaiskaya Cave Provides Insights into the Genetic Relationships of Middle Palaeolithic Populations // Scientific Reports. 12. 13016.
- Antonio M. L., Gao Z., Moots H. M., Lucci M., Candilio F., Sawyer S., Oberreiter V. et al., 2019. Ancient Rome: A Genetic Crossroads of Europe and the Mediterranean // Science. Vol. 366. Iss. 6466. P. 708–714.
- Barros Damgaard P. de, Marchi N., Rasmussen S., Peyrot M., Renaud G., Korneliussen T., Moreno-Mayar J. V. et al., 2018. 137 Ancient Human Genomes from across the Eurasian Steppes // Nature. 557, 7705. P. 369–734.
- Burger J., Link V., Blöcher J., Schulz A., Sell C., Pochon Z., Diekmann Y. et al., 2020. Low Prevalence of Lactase Persistence in Bronze Age Europe Indicates Ongoing Strong Selection over the Last 3,000 Years // Current Biology. Vol. 30. Iss. 21. P. 4307–4315 (e13).
- Gansauge M.-T., Gerber T., Glocke I., Korlevic P., Lippik L., Nagel S., Riehl L. M., Schmidt A., Meyer M., 2017. Single-Stranded DNA Library Preparation from Highly Degraded DNA Using T4 DNA Ligase // Nucleic Acids Research. 45, 10. e79.
- Gnecchi-Ruscone G. A., Khussainova E., Kahbatkyzy N., Musralina L., Spyrou M. A., Bianco R. A., Radzeviciute R. et al., 2021. Ancient Genomic Time Transect from the Central Asian Steppe Unravels the History of the Scythians // Science Advances. Vol. 7. № 13. P. 4414–4440.
- Jónsson H., Ginolhac A., Schubert M., Johnson P. L. F., Orlando L., 2013. MapDamage2.0: Fast Approximate Bayesian Estimates of Ancient DNA Damage Parameters // In Bioinformatics. Vol. 29. Iss. 13. P. 1682–1684.
- Juras A., Ehler E., Chyleński M., Pospieszny Ł., Spinek A. E., Malmström H., Krzewińska M. et al., 2021. Maternal Genetic Origin of the Late and Final Neolithic Human Populations from Present-Day Poland // American Journal of Physical Anthropology. Vol. 176. Iss. 2. P. 223–236.
- Klunk J., Duggan A. T., Redfern R., Gamble J., Boldsen J. L., Golding G. B., Walter B. S. et al., 2019. Genetic Resiliency and the Black Death: No Apparent Loss of Mitogenomic Diversity Due to the Black Death in Medieval London and Denmark // American Journal of Physical Anthropology. Vol. 169. Iss. 2. P. 240–252.
- Krause-Kyora B., Nutsua M., Boehme L., Pierini F., Pedersen D. D., Kornell S. C., Drichel D. et al., 2018. Ancient DNA Study Reveals HLA Susceptibility Locus for Leprosy in Medieval Europeans //Nature Communications. 9. 1569.
- Krzewińska M., Kjellström A., Günther T., Hedenstierna-Jonson C., Zachrisson T., Omrak A., Yaka R., Kilinc G., Somel M., Sobrado V., Evans J., Knipper C., Jakobsson M., Storå J., Götherström A., 2018. Genomic and Strontium Isotope Variation Reveal Immigration Patterns in a Viking Age Town // Current Biology. Vol. 28. Iss. 17. P. 2730–2738 (e10.)
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K., 2018. MEGA X: Molecular Evolutionary Genetics Analysis across Computing Platforms // Molecular Biology and Evolution. Vol. 35. Iss. 6. P. 1547–1549.
- Li H., Durbin R., 2009. Fast and Accurate Short Read Alignment with Burrows-Wheeler Transform // Bioinformatics. Vol. 25. Iss. 14. P. 1754–1760.
- Maár K., Varga G. I. B., Kovács B., Schütz O., Maróti Z., Kalmár T., Nyerki E. et al., 2021. Maternal Lineages from 10–11th Century Commoner Cemeteries of the Carpathian Basin [Электронный ресурс] // BioRxiv. URL: https://www.biorxiv.org/content/10.1101/2021.01.26.428268v1 (дата обращения: 26.03.2023).
- Margaryan A., Lawson D. J., Sikora M., Racimo F., Rasmussen S., Moltke I., Cassidy L. M. et al., 2020. Population Genomics of the Viking World // Nature. 585, 7825. P. 390–396.
- Mathieson I., Lazaridis I., Rohland N., Mallick S., Patterson N., Roodenberg S. A., Harney E. et al., 2015. Genome-Wide Patterns of Selection in 230 Ancient Eurasians // Nature. 528, 7583. P. 499–503.
- Melchior L., Lynnerup N., Siegismund H. R., Kivisild T., Dissing J., 2010. Genetic Diversity among Ancient Nordic Populations // PLoS ONE. Vol. 5. № 7. e11898.
- Mikkelsen M., Sørensen E., Rasmussen E. M., Morling N., 2010. Mitochondrial DNA HV1 and HV2 Variation in Danes // Forensic Science International: Genetics. 4. 4. P. 87–88.
- Narasimhan V. M., Patterson N., Moorjani P., Rohland N., Bernardos R., Mallick S., Lazaridis I. et al., 2019. The Formation of Human Populations in South and Central Asia // Science. Vol. 365. Iss. 6457.
- Nikitin A. G., Kochkin I. T., June C. M., Willis C. M., Mcbain I., Videiko M. Y., 2009. Mitochondrial DNA Sequence Variation in the Boyko, Hutsul, and Lemko Populations of the Carpathian Highlands // Human Biology. Vol. 81. № 1. P. 43–58.
- Olalde I., Brace S., Allentoft M. E., Armit I., Kristiansen K., Booth T., Rohland N. et al., 2018. The Beaker Phenomenon and the Genomic Transformation of Northwest Europe // Nature. 555, 7695. P. 190–196.
- Olivieri A., Pala M., Gandini F., Hooshiar Kashani B., Perego U. A., Woodward S. R., Grugni V. et al., 2013. Mitogenomes from Two Uncommon Haplogroups Mark Late Glacial/Postglacial Expansions from the Near East and Neolithic Dispersals within Europe // PLoS ONE. Vol. 8. № 7. e70492.
- Picard [Электронный ресурс] // Broad Institute. 2019. URL: http://broadinstitute.github.io/picard/ (дата обращения: 28.03.2023).
- Roostalu U., Kutuev I., Loogväli E. L., Metspalu E., Tambets K., Reidla M., Khusnutdinova E. K., Usanga E., Kivisild T., Villems R., 2007. Origin and Expansion of Haplogroup H, the Dominant Human Mitochondrial DNA Lineage in West Eurasia: The Near Eastern and Caucasian Perspective // Molecular Biology and Evolution. Vol. 24. Iss. 2. P. 436–448.
- Schubert M., Lindgreen S., Orlando L., 2016. AdapterRemoval v2: Rapid Adapter Trimming, Identification, and Read Merging // BMC Research Notes. 9. 1. 88.
- Tassi F., Vai S., Ghirotto S., Lari M., Modi A., Pilli E., Brunelli A. et al., 2017. Genome Diversity in the Neolithic Globular Amphorae Culture and the Spread of Indo-European Languages // Proceedings. Biological Sciences. Vol. 284. № 1867. 20171540.
- Wang W., Ding M., Gardner J. D., Wang Y., Miao B., Guo W., Wu X. et al., 2021. Ancient Xinjiang Mitogenomes Reveal Intense Admixture with High Genetic Diversity // Science Advances. Vol. 7. Iss. 14. eabd6690.
- Weissensteiner H., Pacher D., Kloss-Brandstätter A., Forer L., Specht G., Bandelt H.-J., Kronenberg F., Salas A., Schönherr S., 2016. HaploGrep 2: Mitochondrial Haplogroup Classification in the Era of High-Throughput Sequencing // Nucleic Acids Research. 44, W1. P. W58–63.


