Методы экстракции ДНК из костных образцов крупного рогатого скота, сохраняемых в краниологической коллекции
Автор: Абдельманова А.С., Мишина А.И., Волкова В.В., Чинаров Р.Ю., Сермягин А.А., Доцев А.В., Боронецкая О.И., Петрикеева Л.В., Костюнина О.В., Brem G., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: ДНК технологии
Статья в выпуске: 6 т.54, 2019 года.
Бесплатный доступ
Развитие молекулярно-генетических методов дает возможность уточнить происхождение и демографическую историю пород сельскохозяйственных животных. Сохранившиеся в краниологических коллекциях образцы костей и зубов служат источником ДНК для подобных исследований. Работа с историческими образцами осложняется наличием очень малых количеств ДНК, высокой степенью ее деградации и загрязнением образцов ингибиторами ПЦР. Целью настоящей работы было сравнение результативности различных методов экстракции ДНК из исторических черепов крупного рогатого скота, пригодной для проведения молекулярно-генети-ческих исследований. Материалом служили зубы, извлеченные из исторических черепов крупного рогатого скота ярославской и холмогорской пород, хранящихся в краниологической коллекции Музея животноводства им. Е.Ф. Лискуна РГАУ-МСХА им. К.А. Тимирязева. На первом этапе сравнивали различные методы выделения ДНК согласно протоколам к соответствующим коммерческим наборам, модифицируя используемое количества костного материала и условия лизиса: Prep Filer™ BTA Forensic DNA Extraction Kit («Thermo Fisher Scientific, Inc.», США), COrDIS «Экстракт» декальцин (ООО «ГОРДИЗ», Россия), М-сорб-кость (ООО «Синтол», Россия), QIAamp DNA Investigator Kit («Qiagen», США). По результатам предварительных исследований для более детального анализа отобрали QIAamp DNA Investigator Kit («Qiagen», США), в котором реализована технология выделения на колонках с силикагелевой мембраной, и Prep Filer™ BTA Forensic DNA Extraction Kit («Thermo Fisher Scientific, Inc.», США), основанный на использовании магнитных частиц. Количественные и качественные характеристики полученной ДНК оценивали измерением концентрации двухцепочечной ДНК на флуориметре Qubit™ («Invitrogen, Life Technologies», США) и по соотношению поглощения при l = 260 нм и l = 280 нм на приборе NanoDrop 8000 («Thermo Fisher Scientific, Inc.», США). Пригодность полученных препаратов ДНК для молекулярно-генетических исследований оценивали на основании мультиплексного анализа 11 микросателлитных локусов (TGLA227, BM2113, TGLA53, ETH10, SPS115, TGLA122, INRA23, TGLA126, BM1818, ETH225, BM1824), а также полногеномного генотипирования на ДНК-чипах высокой плотности, содержащих более 777 тыс. SNP (single nucleotide polymorphism) (Bovine HD BeadChip, «Illumina Inc.», США). Концентрации двухцепочечной ДНК, полученной с использованием наборов QIAamp DNA Investigator Kit и Prep Filer™ BTA Forensic DNA Extraction Kit, варьировали соответственно от 0,146 до 2,060 нг/мкл и от 0,110 до 13,600 нг/мкл, составив в среднем 0,83±0,23 и 2,75±1,33 нг/мкл. Коэффициент корреляции ( r ) между концентрацией двухцепочечной ДНК в препаратах, полученных двумя разными методами, составил 0,84. Анализ микросателлитов в образцах ДНК 10 исторических черепов скота холмогорской и ярославской пород показал, что каждый из образцов несет свой уникальный генотип, отличающий его от других исторических и современных особей. Результативность SNP-генотипирования исторических образцов составила 0,533-0,878 и 0,958-0,977 для препаратов ДНК, полученных с использованием соответственно QIAamp DNA Investigator Kit и Prep Filer™ BTA Forensic DNA Extraction Kit. Результаты анализа микросателлитов и генотипирования SNP, с одной стороны, показывают пригодность полученной ДНК для проведения исследований полиморфизмов, с другой - подтверждают соответствие лаборатории, в которой были проведены эти исселдования, критериям аутентичности для работы с древней ДНК. Проведение широкомасштабных исследований исторических образцов с использованием различных типов ДНК маркеров позволит уточнить происхождение и демографическую историю отечественных пород крупного рогатого скота и разработать эффективные программы их сохранения.
Историческая днк, краниологическая коллекция, выделение днк, микросателлитный анализ, snp-генотипирование, крупный рогатый скот, локальные породы
Короткий адрес: https://sciup.org/142226271
IDR: 142226271 | УДК: 636.2:577.21:577.088 | DOI: 10.15389/agrobiology.2019.6.1110rus
Текст научной статьи Методы экстракции ДНК из костных образцов крупного рогатого скота, сохраняемых в краниологической коллекции
* При проведении исследований было использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ВИЖ им. академика Л.К. Эрнста. Исследования выполнены при поддержке РНФ, проект 19-76-20012
Изучение эволюции и демографической истории пород сельскохозяйственных животных становится возможным благодаря вовлечению в исследования исторических и ископаемых образцов (1). Несмотря на то, что после смерти организма ДНК разрушается в процессе ферментативных реакций, при благоприятных условиях молекулы способны сохраняться в течение сотен и тысяч лет (2, 3).
Основная проблема при изучении ископаемой ДНК — загрязнение экстрактов или реагентов молекулами современной ДНК. Практически с самого начала работ с ископаемыми пробами вставал вопрос о том, действительно ли полученная ДНК принадлежит историческим образцам. В результате многочисленных исследований были сформулированы следующие так называемые критерии аутентичности: проведение в каждой партии образцов реакции с «чистым экстрактом», то есть без использования ископаемой ДНК; исследование каждого образца в дубликатах с последующим сравнением полученных результатов; контроль длины получаемых фрагментов (наличие продуктов ПЦР длиной более 500-700 п.н. может вызывать подозрение) (4-6). Соблюдение вышеназванных критериев — обязательное условие при работе с ископаемыми образцами. Минимально необходимыми требованиями при работе с древней ДНК считают наличие физически изолированной рабочей зоны, в которой проводятся все исследования до стадии амплификации (7); использование отрицательных контролей при амплификации и избегание (по возможности) положительного контроля, поскольку он несет опасность загрязнения (8, 9); повторяемость результатов, получаемых из разных экстракций ДНК одного и того же образца (10).
Поскольку мягкие ткани сохраняются очень плохо, для получения ДНК используются главным образом костные останки (11, 12). Лучшими субстратами для получения ДНК считают внутреннюю часть височной кости и слой цемента в корнях зубов, при этом систематической разницы в содержании ДНК между двумя субстратами не выявлено (13). Однако костная ткань чрезвычайно трудно поддается измельчению и растворению. Кроме того, древние кости и зубы часто содержат большое количество ингибиторов ПЦР, которые экстрагируются совместно с ДНК (12, 14, 15). Древняя ДНК достаточно сильно повреждена (16-18), поэтому следует избегать чрезмерно агрессивных обработок образца, таких как высокие температуры или использование сильных детергентов (19). Существует несколько способов выделения древней ДНК, включая осаждение этанолом или изопропанолом (12, 15), концентрирование ДНК с использованием мембран и связывание ДНК с диоксидом кремния (20-22).
Независимо от метода выделения ДНК, подготовка образца включает несколько этапов. Сначала кости (зубы) промывают детергентом и дистиллированной водой, а затем стачивают 2-3 мм ткани, чтобы удалить поверхностные загрязнения и современную ДНК. Очищенные костные фрагменты обрабатывают УФ-светом (λ = 254 нм) в течение 30 мин и механически измельчают в тонкую муку. Далее следуют этапы растворения костного порошка в лизирующем растворе, отмывание от примесей, ингибирующих ПЦР, получение очищенного экстракта ДНК. Лизирующие растворы могут различаться по составу, но, как правило, содержат протеиназу К, этилендиаминтетрауксусную кислоту (EDTA), натриевую соль N-лаурилсаркозина. Отличительная особенность процесса лизиса костного порошка — длительность инкубации. Для эффективного растворения клеток рекомендуется проводить лизис при заданной температуре и непрерывном перемешивании в течение не менее 24 ч. Увеличение времени ли- зиса до 48 ч и более целесообразно только при больших навесках образца и объемах раствора. В противном случае оно не приводит к значительному улучшению качества и повышению выхода конечного продукта. Далее следует очистка ДНК из лизатов, для выполнения которой находят применение различные методы.
Наиболее простой метод — осаждение ДНК этанолом или изопропанолом, промывка осадка с целью удаления примесей из раствора и растворение ДНК в бидистиллированной воде или буфере. Основные достоинства данного метода — быстрота получения результата и низкая стоимость. К недостаткам следует отнести высокую трудоемкость, повышенную вероятность потери образца или кросс-контаминации образцов, если несколько проб обрабатываются одновременно (23, 24).
Для получения чистых препаратов ДНК эффективно применение колонок с силикагелевой мембраной. Нуклеиновые кислоты избирательно связываются с мембраной, а примеси удаляются последовательным добавлением промывочных буферов и посредством центрифугирования. На завершающем этапе используется элюирующий буфер, который вымывает нуклеиновые кислоты из мембраны. К достоинствам метода относятся невысокая трудоемкость и низкая вероятность ошибок со стороны исследователя, хорошее качество получаемой ДНК. Также не требуется применять опасные для человека и окружающей среды реагенты, такие как фенол и хлороформ. Метод был реализован в ряде коммерческих наборов: Pure Link® Genomic DNA Mini Kit («Thermo Fisher Scientific, Inc.», США); Nu-cleo Spin® Tissue («MACHEREY-NAGEL GmbH & Co. KG», Германия); QIAamp DNA Investigator Kit («Qiagen», США) (25), что позволяет стандартизировать процесс выделения ДНК. Недостаток использования коммерческих наборов — их относительно высокая стоимость.
Перспективный метод очистки нуклеиновых кислот — использование в качестве сорбента магнитных частиц, которые изготавливаются из различных синтетических полимеров, биополимеров, пористого стекла или на основе неорганических магнитных материалов, таких как оксид железа (26). ДНК обратимо связывается с поверхностью магнитных частиц и после серии отмывок и удаления примесей легко снимается с сорбента при помощи элюирующего буфера. Метод удобен, технологичен, пригоден для подготовки образцов к ПЦР-амплификации. Однако возможны потери продукта вследствие необратимой сорбции на носителе, а также в процессе многочисленных отмывок, что критично при работе с малыми количествами ДНК в образце (27). Метод успешно использован в коммерческих наборах Prep Filer™ BTA Forensic DNA Extraction Kit («Thermo Fisher Scientific, Inc.», США); Quick-DNA/RNA MagBead («Zymo Research», США).
При изучении ископаемых образцов костей человека показано преимущество метода выделения ДНК с использованием силикагелевых колонок в отношении как количества получаемой ДНК (28, 29), так и размера фрагментов (29). Так, при применении силикагелевых колонок более высокая концентрация ДНК была получена в 68,4 % образцов, при использовании QIAquick PCR Purification Kit® («Qiagen», США) — в 21,05 % образцов (28).
В качестве материала для исследований демографической истории пород домашних животных интерес представляют исторические черепа и их части, сохраняемые в краниологических коллекциях. Краниологический метод, предложенный в 1865 году L. Rutimeyer, был основным при изучении происхождения домашних животных в конце XIX—первой по- ловине XX века (30). Это обусловило создание хорошо документированных краниологических коллекций, однако лишь несколько из них сохранились до наших дней. Одна из таких коллекций, собранная профессором L. Adametz и насчитывающая около 1300 черепов старых пород крупных домашних животных Центральной Европы, хранится в Музее истории естествознания в Вене (Natural History Museum Vienna, Naturhistorisches Museum Wien; .
Уникальная коллекция черепов отечественных и завозимых в старую Россию иностранных пород домашних животных (крупного рогатого скота, свиней, лошадей) собрана академиком Е.Ф. Лискуном и находится в РГАУ—МСХА им. К.А. Тимирязева . Коллекция содержит более 700 черепов животных, в том числе 350 — крупного рогатого скота 41 породы и породной группы (31).
Вместе с тем использование черепов из краниологических коллекций в качестве источника ДНК осложняется тем, что их подготовка к депонированию включает вываривание (в течение нескольких часов), а в ряде случаев отбеливание (в растворе аммиака с перекисью водорода) и обработку фосфорной кислотой или хлорамином для придания естественного оттенка. Такая обработка приводит к существенной деградации нуклеиновых кислот, поэтому при использовании исторических образцов в качестве источника ДНК необходима оптимизация методики, которая позволяет получать препараты ДНК, по количественным и качественным характеристикам пригодные для исследований различных типов полиморфизмов. Особенно критично получение достаточного количества ядерной ДНК, представленной всего двумя копиями на клетку, в отличие от митохондриальной, количество копий которой в одной клетке составляет от 100 до 10000 (32).
В настоящей работе мы впервые получили препараты ДНК, пригодные для проведения молекулярно-генетических исследований, из образцов черепов крупного рогатого скота, датированных первой половиной XX века и подвергшихся термической и химической обработке при подготовке для депонирования в коллекциях.
Целью работы было сравнение результативности различных методов экстракции ДНК из исторических черепов крупного рогатого скота.
Методика. Материалом служили зубы, извлеченные из исторических черепов крупного рогатого скота ярославской и холмогорской пород, хранящихся в краниологической коллекции Музея животноводства им. Е.Ф. Лискуна РГАУ—МСХА им. К.А. Тимирязева. При проведении исследований соблюдались все стандарты, установленные критериями подлинности древней ДНК (33).
Зубы очищали от механических загрязнений, промывали детергентом и дистиллированной водой, чтобы удалить поверхностные загрязнения и современную ДНК. Затем их облучали ультрафиолетовым светом (λ = 254 нм) в течение 30 мин. С использованием настольной электрической пилы MBS240/E («Proxxon», Германия), оборудованной биметаллическим полотном, зубы продольно распиливали на две части. После этого с помощью мини-дрели Dremel 3000-15 («Dremel», США) с алмазным бором из внутренней части зубов высверливали мелкодисперсный порошок. Высверливание проводили на минимальных оборотах (∼ 8000 об/мин) с перерывами для предупреждения чрезмерного нагрева дентина и уничтожения ДНК под действием высоких температур. Образующийся костный поро- шок пересыпали в предварительно облученные под ультрафиолетом стерильные пробирки типа эппендорф с системой Safe-Lock (1,5 мл).
На первом этапе сравнивали различные методы выделения ДНК, реализованные в форме коммерческих наборов, с модификацией используемого количества костного материала и условий лизиса: Prep Filer™ BTA Forensic DNA Extraction Kit («Thermo Fisher Scientific, Inc.», США), COrDIS «Экстракт» декальцин (ООО «ГОРДИЗ», Россия), М-сорб-кость (ООО «Синтол», Россия), QIAamp DNA Investigator Kit («Qiagen», США).
Для более детальных исследований были отобраны наборы QIAamp DNA Investigator Kit, в котором реализована технология селективного связывания ДНК на кремниевой мембране, и Prep Filer™ BTA Forensic DNA Extraction Kit, основанный на использовании магнитных частиц. С целью получения сопоставимых результатов протоколы выделения, рекомендованные производителями для каждого из наборов, были модифицированы. Навески образцов приводили к единой массе (100±3 мг), время лизиса увеличивали до 24 ч при температуре 56 °С, скорость ротации составила 1100 об/мин. Дальнейшее увеличение времени лизиса до 48 ч не оказывало значительного положительного влияния на качество и количество получаемой ДНК. Время элюирования при выделении набором QIAamp DNA Investigator Kit было увеличено до 30 мин.
Качество полученных препаратов ДНК оценивали следующим образом: измеряли концентрацию общей ДНК и соотношение поглощения препарата при X = 260 нм и X = 280 нм на приборе NanoDrop 8000 («Thermo Fisher Scientific, Inc.», США); определяли концентрацию двухцепочечной ДНК на флуориметре Qubit™ (1.0) («Invitrogen, Life Technologies», США).
Для оценки пригодности полученных препаратов ДНК для проведения молекулярно-генетических исследований был выполнен мультиплексный анализ по 11 микросателлитам (TGLA227, BM2113, TGLA53, ETH10, SPS115, TGLA122, INRA23, TGLA126, BM1818, ETH225, BM1824) с использованием панели, разработанной в ФНЦ животноводства — ВИЖ им. академика Л.К. Эрнста, а также проведено полногеномное генотипирование по примерно 777 тыс. SNP (single nucleotide polymorphism) на ДНК-чипах высокой плотности (Bovine HD BeadChip, «Illumina, Inc.», США). ПЦР-амплификацию осуществляли на термоциклере SimpliAmp Thermal Cycler («Life Technologies», США). Полиморфизм микросателлит-ных маркеров исследовали на капиллярном генетическом анализаторе ABI3500 («Applied Biosystems», США) с помощью программного обеспечения Gene Mapper v.5 («Applied Biosystems», США). Микросателлитные профили представителей современных пород были взяты из базы данных ФНЦ животноводства — ВИЖ им. академика Л.К. Эрнста.
SNP исследовали с помощью сканера iScan® System («Illumina, Inc.», США) и программного обеспечения, поставляемого с прибором.
Филогенетическое дерево для современных и исторических образцов крупного рогатого скота строили по методу UPGMA (Unweighted Pair Group Method with Arithmetic Mean) на основании генетических дистанций M. Nei (Ds) (34), рассчитанных по микросателлитам в программе Populations, 1.2.32 (35), и визуализировали в программе SplitsTree, 4.13.1 (36).
Результаты. Наборы, основанные на магнитных частицах (Prep Filer™ BTA Forensic DNA Extraction Kit, COrDIS «Экстракт» декальцин и М-сорб-кость), в целом позволяли получать препараты ДНК с более высокой концентрацией (2,94-9,80 нг/мкл двухцепочечной ДНК, величина OD260/280 = 1,00-1,64). Однако чистота экстрактов, полученных с исполь-1114
зованием спин-колонок с кремниевой мембраной (QIAamp DNA Investigator Kit), при меньшей концентрации выделяемой ДНК (0,56-6,42 нг/мкл) оказалась выше (OD260/280 = 1,47-1,90). Для более детального изучения были выбраны наборы QIAamp DNA Investigator Kit и Prep Filer™ BTA Forensic DNA Extraction Kit.
1. Концентрация и чистота препаратов ДНК, полученной из исторических образцов зубов крупного рогатого скота ярославской и холмогорской пород с использованием различных методов
|
¹ образца |
QIAamp DNA Investigator Kit |
Prep Filer™ BTA Forensic DNA Extraction Kit |
||||
|
Qubit, нг/мкл |
NanoDrop, нг/мкл |
OD 260 / 280 |
Qubit, нг/мкл |
NanoDrop, нг/мкл |
OD 260 / 280 |
|
|
1 |
0,434 |
141,50 |
0,82 |
0,246 |
5,60 |
1,70 |
|
2 |
2,060 |
23,02 |
1,71 |
13,600 |
79,40 |
1,87 |
|
3 |
0,308 |
9,12 |
1,46 |
0,110 |
70,68 |
1,96 |
|
4 |
0,316 |
33,80 |
1,83 |
1,260 |
26,51 |
1,67 |
|
5 |
0,318 |
14,70 |
1,58 |
0,454 |
17,38 |
0,67 |
|
6 |
0,334 |
16,44 |
1,54 |
0,540 |
18,75 |
1,59 |
|
7 |
1,590 |
19,13 |
1,73 |
2,400 |
40,09 |
1,74 |
|
8 |
0,884 |
15,19 |
1,72 |
2,600 |
42,48 |
1,64 |
|
9 |
0,146 |
12,40 |
1,53 |
0,378 |
19,65 |
1,54 |
|
10 |
1,910 |
16,57 |
1,90 |
5,880 |
64,23 |
1,88 |
П р и м е ч а н и е. На флуорометре Qubit («Invitrogen, Life Technologies», США) измеряли концентрацию двухцепочечной ДНК, на приборе NanoDrop 8000 («Thermo Fisher Scientific, Inc.», США) — концентрацию общей ДНК. OD260/280 — показатель чистоты препарата ДНК (соотношение поглощения при λ = 260 нм и λ = 280 нм), измеренная на приборе NanoDrop 8000.
Были выявлены существенные различия в концентрации ДНК как между образцами, так и между препаратами, полученными из одного образца различными методами (табл. 1). Концентрации двухцепочечной ДНК, выделенной с использованием наборов QIAamp DNA Investigator Kit и Prep Filer™ BTA Forensic DNA Extraction Kit, варьировала соответственно от 0,146 до 2,060 нг/мкл и от 0,110 до 13,600 нг/мкл, составив в среднем 0,83±0,23 и 2,75±1,33 нг/мкл. В 8 из 10 образцов концентрация двухцепочечной ДНК была в 1,43-6,60 раза выше при использовании набора Prep Filer™ BTA Forensic DNA Extraction Kit по сравнению с QIAamp DNA Investigator Kit, в то время как в двух оставшихся образцах — в 1,762,80 раза ниже. Коэффициент корреляции ( r ) между концентрацией двухцепочечной ДНК в препаратах, полученных двумя различными методами, составил 0,84.
Полученные данные показывают, что основной фактор, влияющий на успешность выделения ДНК, — это сохранность образца, что согласуется с результатами других исследований (28, 37, 38). Однако, в отличие от некоторых других работ (29), более высокие концентрации были получены нами при использовании метода на основе магнитных частиц. По всей видимости, это связано с сильной деградацией ДНК в процессе подготовки черепов для депонирования в коллекциях. В результате экстрагируемая ДНК представлена очень короткими фрагментами, которые лучше удерживаются магнитными частицами, чем силикагелевой мембраной в процессе промывки.
Для оценки пригодности выделенной ДНК для проведения молекулярно-генетических исследований и подтверждения соответствия лаборатории критериям аутентичности были получены микросателлитные профили исторических образцов животных ярославской и холмогорской пород. Определены генотипы всех 11 исследованных локусов микросателлитов (рис. 1). При этом следует отметить тенденцию снижения высоты пиков с увеличением длины аллеля микросателлитов, что указывает на сильную деградацию полученной ДНК, которая представлена в основном низкомолекулярными фрагментами.
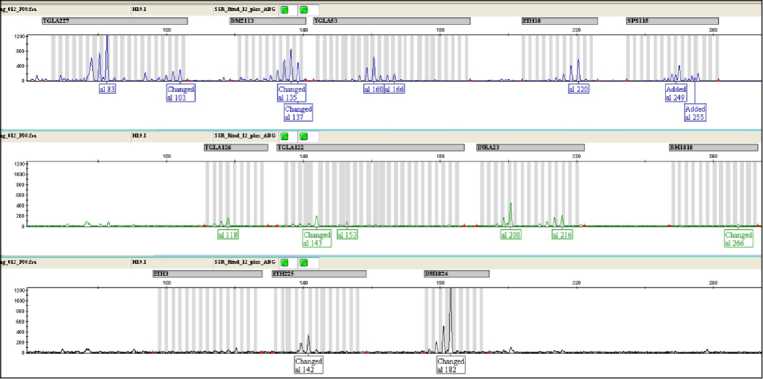
Рис. 1. Результаты капиллярного электрофореза при мультиплексном анализе микросателлитно-го профиля исторического краниологического образца (Н19.1) животного холмогорской породы крупного рогатого скота по 11 микросателлитным локусам: А — TGLA227 (аллели 83, 103), BM2113 (135, 137), TGLA53 (160, 166), ETH10 (220, 220), SPS115 (249, 255); Б — TGLA126 (118, 118), TGLA122 (143, 153), INRA23 (200, 216), BM1818 (266, 266); В — ETH225 (142, 142), BM1824 (182, 182). Ось Х — локусы и фрагменты (пики) которые в них детектированы; размер фрагментов (al 83, Changed al 103, Changed al 135 и т.д.) возрастает по мере продвижения по оси вправо). Ось Y — высота пика. Цветом отмечены разные метки для анализатора. Полностью рисунок см. на сайте
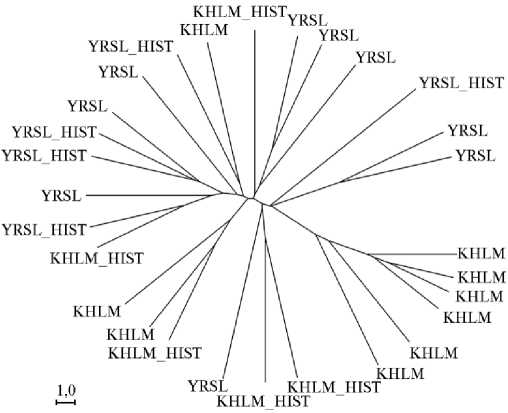
Рис. 2. Филогенетическое дерево для современных и исторических образцов крупного рогатого скота, построенное по методу UPGMA (Unweighted Pair Group Method with Arithmetic Mean) на основании генетических дистанций M. Nei (34), которые были рассчитаны по микросателлитам в программе Populations, 1.2.32 (35): KHLM — современный образец холмогорской породы, YRSL — современный образец ярославской породы, KHLM_HIST — исторический образец холмогорской породы, YRSL_HIST — исторический образец ярославской породы.
Анализ микросателлитных профилей исторических проб показал, что каждый из образцов несет свой уникальный генотип, отличающийся от генотипов других исторических и современных образцов. На филогенетическом дереве, построенном на основании генетических дистанций M. Nei (34), рассчитанных по микросателлитам, это проявлялось в фор- мировании каждым образцом своей самостоятельной ветви (рис. 2). Во всех пяти исторических образцах ярославской породы и в трех из пяти исторических образцов холмогорской породы были идентифицированы приватные аллели, отсутствующие в других изученных популяциях. Результаты анализа микросателлитов подтверждают соответствие лаборатории, в которой проводились эти исследования, критериям для работы с древней ДНК.
Результативность полногеномного генотипирования SNP при использовании ДНК, выделенной с помощью магнитных частиц, была существенно выше, чем при использовании ДНК, выделенной с помощью кремниевых спин-колонок (табл. 2). Это может быть связано с тем обстоятельством, что для анализа SNP размер фрагментов ДНК (в отличие от концентрации) не является критическим фактором. При анализе микроса-теллитных локусов успешность амплификации определяется степенью деградации ДНК (18).
2. Результативность полногеномного генотипирования ДНК, выделенной из исторических образцов черепов крупного рогатого скота холмогорской породы, с использованием ДНК-чипов высокой плотности Bovine HD BeadChip («Il-lumina, Inc.», США)
|
¹ образца |
Qubit, нг/мкл |
NanoDro, нг/мкл |
OD 260 / 280 |
Call Rate |
Способ выделения ДНК |
|
1 |
5,32 |
35,68 |
1,87 |
0,958 |
Prep Filer™ BTA Forensic DNA Extraction Kit |
|
2 |
4,52 |
26,19 |
1,96 |
0,970 |
Prep Filer™ BTA Forensic DNA Extraction Kit |
|
3 |
6,84 |
33,10 |
1,83 |
0,970 |
Prep Filer™ BTA Forensic DNA Extraction Kit |
|
4 |
4,94 |
65,71 |
1,90 |
0,977 |
Prep Filer™ BTA Forensic DNA Extraction Kit |
|
5 |
2,94 |
21,64 |
1,96 |
0,641 |
QIAamp DNA Investigator Kit |
|
6 |
1,18 |
21,15 |
1,86 |
0,533 |
QIAamp DNA Investigator Kit |
|
7 |
6,84 |
35,15 |
1,85 |
0,775 |
QIAamp DNA Investigator Kit |
|
8 |
4,48 |
24,75 |
2, 00 |
0,878 |
QIAamp DNA Investigator Kit |
П р и м е ч а н и е. На флуорометре Qubit («Invitrogen, Life Technologies», США) измеряли концентрацию двухцепочечной ДНК, на приборе NanoDrop 8000 («Thermo Fisher Scientific Inc.», США) — концентрацию общей ДНК. OD260/280 — чистота препарата ДНК (отношение степени поглощения при λ = 260 и 280 нм), измеренная на приборе NanoDrop 8000. Call Rate — доля генотипированных SNP от общего количества SNP на ДНК-чипе.
Основными проблемами при проведении молекулярно-генетических исследований древних и исторических образцов становятся низкие концентрации и высокая степень деградации ДНК в полученных экстрактах (13, 14, 18, 22). Небольшая длина сохранившихся целевых фрагментов может быть существенным ограничением для анализа микросателлитов с большей длиной фрагментов (200 п.н. и более) (41, 42). Для однозначной интерпретации полученных профилей в ряде работ (42, 43) рекомендуется проводить амплификацию каждого образца не менее трех раз. SNP маркеры менее требовательны к степени деградации ДНК. Как показали исследования (29, 44), высокопроизводительное секвенирование и полногеномное генотипирование SNP на ДНК-чипах позволят существенно расширить спектр применяемых процедур обработки данных и более полно использовать информацию, заключенную в исторической ДНК.
Таким образом, мы показали возможность успешного выделения ДНК, пригодной для проведения молекулярно-генетических исследований, из исторических черепов крупного рогатого скота, сохраняемых в краниологических коллекциях. Сравнение двух различных методов выделения ДНК, основанных на использовании колонок с силикагелевой мембраной (QIAamp DNA Investigator Kit) и магнитных частиц (Prep Filer™ BTA Forensic DNA Extraction Kit), выявило преимущество второго метода. В 8 из 10 исследованных образцов концентрация двухцепочечной
ДНК была выше при использовании метода на основе магнитных частиц, в двух образцах — при использовании силикагелевых колонок, при этом средние значения концентрации ДНК составили соответственно 2,75±1,33 и 0,83±0,23 нг/мкл. Анализ генотипов исторических образцов по 11 микросателлитным локусам показал, что каждый из 10 исследованных образцов холмогорской и ярославской породы имеет свой уникальный генотип, отличающийся от других образцов. Исследование результативности полногеномного генотипирования SNP показало преимущество набора, основанного на использовании магнитных частиц (Prep Filer™ BTA Forensic DNA Extraction Kit). При условии, что длина фрагментов в данном типе анализа не является критическим фактором, этот метод предпочтительнее за счет получения более высоких концентраций ДНК. В целом следует отметить необходимость проведения широкомасштабных исследований исторических образцов с использованием различных типов ДНК маркеров, что станет весомым дополнением результатов, полученных при исследовании современных представителей пород. Это позволит уточнить происхождение и демографическую историю отечественных пород крупного рогатого скота и разработать эффективные программы их сохранения.
Список литературы Методы экстракции ДНК из костных образцов крупного рогатого скота, сохраняемых в краниологической коллекции
- Zinovieva N.A., Sermyagin A.A., Dotsev A.V., Boronetskaya O.I., Petrikeeva L.V., Abdelmanova A.S., Brem G. Animal genetic resources: developing the research of allele pool of Russian cattle breeds - minireview. Sel'skokhozyaistvennaya Biologiya [Agricultural Biology], 2019, 54(4): 631-641 ( ). DOI: 10.15389/agrobiology.2019.4.631eng
- Lindahl T. Instability and decay of the primary structure of DNA. Nature, 1993, 362(6422): 709-715 ( ). DOI: 10.1038/362709a0
- Orlando L., Ginolhac A., Zhang G. et al. Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse. Nature, 2013, 99(7456): 74-78 ( ). DOI: 10.1038/nature12323
- Pääbo S., Higuchi R.G., Wilson A.C. Ancient DNA and the polymerase chain reaction: the emerging field of molecular archaeology (minireview). The Journal of Biological Chemistry, 1989, 264(17): 9709-9712.
- Ward R., Stringer C. A molecular handle on the Neanderthals. Nature, 1997, 388: 225-226 ( ). DOI: 10.1038/40746
- Cooper A., Poinar H.N. Ancient DNA: do it right or not at all. Science, 2000, 289(5482): 1139 ( ).
- DOI: 10.1126/science.289.5482.1139b
- Lindahl T. Recovery of antediluvian DNA. Nature, 1993, 365(6448): 700 ( ).
- DOI: 10.1038/365700a0
- Handt O., Krings M., Ward R.H., Pääbo S. The retrieval of ancient human DNA sequences. Am. J. Hum. Genet., 1996, 59: 368-376.
- Cooper A. DNA from museum specimens. In: Ancient DNA /B. Herrmann, S. Hummel (eds.). Springer-Verlag, NY, 1993: 149-165 ( ).
- DOI: 10.1007/978-1-4612-4318-2_10
- Greenwood A.D., Capelli C., Possnert G., Pääbo S. Nuclear DNA sequences from late Pleistocene megafauna. Molecular Biology and Evolution, 1999, 16(11): 1466-1473 ( ).
- DOI: 10.1093/oxfordjournals.molbev.a026058
- Hagelberg E., Sykes B., Hedges R. Ancient bone DNA amplified. Nature, 1989, 342: 485 ( ).
- DOI: 10.1038/342485a0
- Hänni C., Brousseau T., Laudet V., Stehelin D. Isopropanol precipitation removes PCR inhibitors from ancient bone extracts. Nucleic Acids Research, 1995, 23(5): 881-882 ( ).
- DOI: 10.1093/nar/23.5.881
- Hansen H.B., Damgaard P.B., Margaryan A., Stenderup J., Lynnerup N., Willerslev E., Allentoft M.E. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE, 2017, 12(1): e0170940 ( ).
- DOI: 10.1371/journal.pone.0170940
- Höss M., Pääbo S. DNA extraction from Pleistocene bones by a silica-based purification method. Nucleic Acids Research, 1993, 21(16): 3913-3914 ( ).
- DOI: 10.1093/nar/21.16.3913
- Kalmár T., Bachrati C.Z., Marcsik A., Raskó I. A simple and efficient method for PCR amplifiable DNA extraction from ancient bones. Nucleic Acids Research, 2000, 28(12): e67 ( ).
- DOI: 10.1093/nar/28.12.e67
- Pääbo S. Ancient DNA: extraction, characterization, molecular cloning, and enzymatic amplification. Proceedings of the National Academy of Sciences, 1989, 86(6): 1939-1943 ( ).
- DOI: 10.1073/pnas.86.6.1939
- Hofreiter M., Jaenicke V., Serre D., von Haeseler A., Pääbo S. DNA sequences from multiple amplifications reveal artifacts induced by cytosine deamination in ancient DNA. Nucleic Acids Research, 2001, 29(23): 4793-4799 ( ).
- DOI: 10.1093/nar/29.23.4793
- Hansen A.J., Mitchell D.L., Wiuf C., Paniker L., Brand T.B., Binladen J., Gilichinsky D.A., Rønn R., Willerslev E. Crosslinks rather than strand breaks determine access to ancient DNA sequences from frozen sediments. Genetics, 2006, 173(2) 1175-1179 ( ).
- DOI: 10.1534/genetics.106.057349
- Rohland N., Hofreiter M. Comparison and optimization of ancient DNA extraction. Biotechniques, 2007, 42(3): 343-352 ( ).
- DOI: 10.2144/000112383
- Leonard J.A., Wayne R.K., Cooper A. Population genetics of ice age brown bears. Proceedings of the National Academy of Sciences, 2000, 97(4): 1651-1654 ( ).
- DOI: 10.1073/pnas.040453097
- Hofreiter M., Rabeder G., Jaenicke-Després V., Withalm G., Nagel D., Paunovic M., Jambrĕsić G., Pääbo S. Evidence for reproductive isolation between cave bear populations. Current Biology, 2004, 14(1): 40-43 ( ).
- DOI: 10.1016/j.cub.2003.12.035
- Rohland N., Glocke I., Aximu-Petri A., Meyer M. Extraction of highly degraded DNA from ancient bones, teeth and sediments for high-throughput sequencing. Nat. Protoc., 2018, 13: 2447-2461 ( ).
- DOI: 10.1038/s41596-018-0050-5
- Bowtell D.D. Rapid isolation of eukaryotic DNA. Analytical Biochemistry, 1987, 162(2): 463-465 (
- DOI: 10.1016/0003-2697(87)90421-0)
- Mohammadi A., Ghorbani Alvanegh A., Khafaei M., Habibi Azarian S., Naderi M., Kiyani E., Miri A., Bahmani H., Ramezani M., Tavallaei M. A new and efficient method for DNA extraction from human skeletal remains usable in DNA typing. Journal of Applied Biotechnology Reports, 2017, 4(2): 609-614.
- Tolosa J.M., Schjenken J.E., Civiti T.D., Clifton V.L., Smith R. Column-based method to simultaneously extract DNA, RNA, and proteins from the same sample. Biotechniques, 2007, 43(6): 799-804 ( ).
- DOI: 10.2144/000112594
- Saiyed Z.M., Bochiwal C., Gorasia H., Telang S.D., Ramchand C.N. Application of magnetic particles (Fe3O4) for isolation of genomic DNA from mammalian cells. Analytical Biochemistry, 2006, 356(2): 306-308 ( ).
- DOI: 10.1016/j.ab.2006.06.027
- Suffys P., Vanderborght P.R., dos Santos P.B., Correa L.A.P., Bravin Y., Kritski A.L. Inhibition of the polymerase chain reaction by sputum samples from tuberculosis patients after processing using a silica-guanidiniumthiocyanate DNA isolation procedure. Memórias do Instituto Oswaldo Cruz, 2001, 96(8): 1137-1139 ( ).
- DOI: 10.1590/s0074-02762001000800019
- Palomo-Díez S., Martínez-Labarga C., Gomes C., Esparza-Arroyo Á., Rickards O., Arroyo-Pardo E. Comparison of two different DNA extraction methodologies for critical bone or teeth samples. Forensic Science International: Genetics Supplement Series, 2017, 6: Е359-Е361 ( ).
- DOI: 10.1016/j.fsigss.2017.09.110
- Gamba C., Hanghøj K., Gaunitz C., Alfarhan A.H., Alquraishi S.A., Al-Rasheid K.A., Bradley D.G., Orlando L. Comparing the performance of three ancient DNA extraction methods for high-throughput sequencing. Molecular Ecology Resources, 2016, 16(2): 459-469 ( ).
- DOI: 10.1111/1755-0998.12470
- Браунер А. Породы сельскохозяйственных животных. Крупный рогатый скот. Одесса, 1922.
- Боронецкая О.И., Барбосова М.Е., Никифоров А.И., Быкова А.В., Михеенков В.Е., Рабаданова Г.Ш., Петрикеева Л.В., Полуротова А.И., Рукавицина Е.А. Каталог краниологической коллекции академика Е.Ф. Лискуна /Под. ред. В.П. Панова. М., 2012.
- Robin E.D., Wong R. Mitochondrial DNA molecules and virtual number of mitochondria per cell in mammalian cells. J. Cell. Physiol., 1988, 136(3): 507-513 ( ).
- DOI: 10.1002/jcp.1041360316
- Pääbo S., Poinar H., Serre D., Jaenicke-Després V., Hebler J., Rohland N., Kuch M., Krause J., Vigilant L., Hofreiter M. Genetic analyses from ancient DNA. Annual Review of Genetics, 2004, 38: 645-679 ( ).
- DOI: 10.1146/annurev.genet.37.110801.143214
- Nei M. Genetic distance between populations. The American Naturalist, 1972, 106(949): 283-292.
- Langella O. Populations 1.2.32 (02/13/2011): а population genetic software. CNRS UPR9034. 1999. Режим доступа: http://bioinformatics.org/~tryphon/populations/. Без даты.
- Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution, 2006, 23(2): 254-267 ( ).
- DOI: 10.1093/molbev/msj030
- Vanek D., Silerova M., Urbanova V., Saskova L., Dubska J., Beran M., Genomic DNA extraction protocols for bone samples: the comparison of Qiagen and Zymo Research spin columns. Forensic Science International: Genetics Supplement Series, 2011, 3(1): e397-e398 ( ).
- DOI: 10.1016/j.fsigss.2011.09.060
- Scorrano G., Valentini F., Martínez-Labarga C., Rolfo M. F., Fiammenghi A., Lo Vetro D., Martini F., Casoli A., Ferraris G., Palleschi G., Palleschi A., Rickards O. Methodological strategies to assess the degree of bone preservation for ancient DNA studies. Annals of Human Biology, 2014, 42(1): 10-19 ( ).
- DOI: 10.3109/03014460.2014.954614
- Zinovieva N.A., Dotsev A.V., Sermyagin A.A., Wimmers K., Reyer H., Sölkner J., Deniskova T.E., Brem G. Study of genetic diversity and population structure of five Russian cattle breeds using whole genome SNP analysis. Agricultural Biology, 2016, 51: 788-800 ( ).
- DOI: 10.15389/agrobiology.2016.6.788eng
- Sermyagin A.A., Dotsev A.V., Gladyr E.A., Traspov A.A., Deniskova T.E., Kostyunina O.V., Reyer H., Wimmers K., Barbato M., Paronyan I.A., Plemyashov K.V., Sölkner J., Popov R.G., Brem G., Zinovieva N.A. Whole-genome SNP analysis elucidates the genetic structure of Russian cattle and its relationship with Eurasian taurine breeds. Genet. Sel. Evol., 2018, 50(1): 37 ( ).
- DOI: 10.1186/s12711-018-0408-8
- Petren K., Grant P.R., Grant B.R., Clack A.A., Lescano N.V. Multilocus genotypes from Charles Darwin's finches: biodiversity lost since the voyage of the Beagle. Phil. Trans. R. Soc. B, 2010, 365: 1009-1018 ( ).
- DOI: 10.1098/rstb.2009.0316
- Allentoft M., Heller R., Holdaway R., Bunce M. Ancient DNA microsatellite analyses of the extinct New Zealand giant moa (Dinornis robustus) identify relatives within a single fossil site. Heredity, 2015, 115: 481-487 ( ).
- DOI: 10.1038/hdy.2015.48
- Bourke B.P., Frantz A.C., Lavers C.P., Davison A., Dawson D.A., Burke T.A. Genetic signatures of population change in the British golden eagle (Aquila chrysaetos). Conserv. Genet., 2010, 11(5): 1837-1846 ( ).
- DOI: 10.1007/s10592-010-0076-x
- Knapp M, Hofreiter M. Next generation sequencing of ancient DNA: requirements, strategies and perspectives. Genes, 2010, 1(2): 227-243 ( ).
- DOI: 10.3390/genes1020227


