Микросателлиты и их использование для оценки генетического разнообразия животных (обзор иностранной литературы)
Автор: Марзанов Н.С., Озеров М.Ю., Насибов М.Г., Марзанова Л.К.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Наука за рубежом
Статья в выпуске: 2 т.39, 2004 года.
Бесплатный доступ
Представлен обзор зарубежной литературы, посвященный проблеме применения микросателлитов для исследований эволюции, происхождения и дивергенции сельскохозяйственных животных. На обширном материале рассматривается роль микросателлитов в организме человека и животных разных видов. Обсуждается возможность использования микросателлитов в качестве маркеров для оценки аллелофонда, достоверности происхождения и филогенеза локальных и широкораспространенных пород животных.
Короткий адрес: https://sciup.org/142132958
IDR: 142132958 | УДК: 636:577.2:575
Текст научной статьи Микросателлиты и их использование для оценки генетического разнообразия животных (обзор иностранной литературы)
Микросателлиты (microsatellites) — это наиболее распространенные тандемные непрерывные повторы участков ДНК, длина которых не превышает шести базовых пар, содержащих от одного до десяти нуклеотидов в одной единице (1-3). Исторически термин «микросателлиты» чаще всего использовали для обозначения динук-леотидных повторов типа СА (GT), а для других последовательностей применяли различные термины — от «простая последовательность» до STR (short tandem repeats), что привело к некоторой путанице. В настоящее время этим термином обозначают в основном короткие тандемные повторы, хотя было бы правильнее все короткие повторы, включая минисателлиты и простые кодирующие участки ДНК (cryptically simple regions), называть «простые последовательности» (4).
Микросателлиты подразделяют на пять основных групп по длине единицы повтора (5): моно-, ди-, три-, тетра-, пентануклеотидные и более (до десяти нуклеотидов включительно). Микросателлиты подразделяют также на основе их структуры на три класса (6): совершенные повторы мотивов (СА) n или (GT) n , которые не прерываются другими нуклеотидами и не расположены рядом с повторами других последовательностей; несовершенные (неполные) повторяющиеся последовательности с двумя или б о льшим числом блоков (CA) n , разделенных не более чем тремя следующими друг за другом неповторяющимися основаниями; сложные повторяющиеся последовательности, у которых блоки (CA) n отделены от других блоков более чем тремя неповторяющимися основаниями, представлены в виде одинаковых нуклеотидов в количестве 5-10 и более. Сложные повторы подразделяются в свою очередь на совершенные и несовершенные в зависимости от типа блоков (CA) n . Совершенные повторы составляют до 80 % микросателлитных последовательностей (табл. 1) (6-8).
Соотношение микросателлитов различных классов в геномах млекопитающих разных видов (%) (цит. по 6 и 7)
|
Класс микросателлитов |
Свинья |
Крупный рогатый скот |
Овца |
Куры |
Собака |
Человек |
|
Совершенные |
71 |
56 |
84 |
64 |
80 |
64 |
|
Несовершенные |
19 |
24 |
10 |
22 |
8 |
25 |
|
Сложные |
10 |
20 |
6 |
14 |
12 |
11 |
Р а с п р е д е л е н и е м и к р о с а т е л л и т о в п о г е н о-м у в наибольшей степени изучено у человека и мыши. Картирование на высоком разрешающем уровне предполагает более или менее равномерное распределение микросателлитов по всему геному обоих видов, однако даже такие карты содержат пробелы и участки с низкой плотностью, которые чаще встречаются вблизи теломер (9, 10).
Микросателлиты редко бывают локализованы в кодирующих участках генома и даже в непосредственной близости от них (11). Это частично относится к микросателлитам, повторяющиеся единицы которых содержат меньше трех оснований, например поли(СА). Тандемные повторы поли(A)/поли(T) наиболее часто встречаются в геноме человека. Однако не представляется возможным использовать микросателлиты для 104
генетического картирования или популяционного анализа в связи с их нестабильностью при проведении полимеразной цепной реакции (ПЦР). Показано, что наиболее распространенными являются динуклеотидные СА/ТG-повторы, которые встречаются в 2 и 3 раза чаще, чем соответственно АТ- и AG/TC-повторы (12). Среди тринуклеотидных повторов наибольшей частотой характеризуются CAG- и AAT-повторы (13). В геномах млекопитающих содержится такой же набор микросателлитов, как и в геноме человека, несмотря на небольшую разницу по частоте аллелей (12, 13). В то же время в геномах наиболее отдаленных видов (например человек и крыса) могут встречаться микросателлиты иной структуры (14).
В геноме дрожжей Saccharomyces cerevisiae выявлено б о льшее количество поли(AT)-повторов, чем в геноме человека (11). У растений GA- и AT-повторы встречаются чаще, чем CA-повторы, которые наиболее распространены в геноме Drosophila melanogaster (14, 15). В геноме бактерий число тандемно повторяющихся последовательностей меньше по сравнению с многоклеточными организмами, но и здесь наиболее распространены поли(A)/поли(T)-повторы (11, 16).
Происхождение этих различий до конца не выяснено. Hancock (1995) подсчитал, что большее количество вариаций по частоте простых последовательностей генома объясняется предполагаемыми механическими сдвигами при репликации ДНК (SSM — slipped strand mispairing), когда строящиеся цепи ДНК начинают соединяться в неправильном порядке. Различия между видами могут быть абсолютно случайными или отражать селективные процессы при отборе последовательностей, адаптированных к определенным функциям (4).
Ф у н к ц и о н а л ь н а я р о л ь м и к р о с а т е л л и т о в. В популяционной генетике микросателлиты используют как генетические маркеры с целью генетического картирования и оценки эволюционных связей. Однако в настоящее время накоплено много данных, свидетельствующих о регуляторной функциональной роли этих элементов в геноме (17). Микросателлиты были обнаружены повсеместно в участках, расположенных левее (upstream) промоторов. Во многих случаях у различных видов млекопитающих микросателлитные последовательности локализованы в одном и том же гене, в участках, близких к промоторам. Например, поли(TG)-повтор в геноме мыши, крысы и человека расположен слева от транскрипционной единицы рРНК, а у двух последних — слева от гена соматостатина (18-20). У кур и мыши цепь повторов (TCCC)2…(TCCC)2 обнаружена в участке, расположенном слева от промотора α 2(I)-гена, контролирующего образование коллагена (21). Последовательность CGG в 5'-области промотора гена FMR1 , связанного с синдромом хрупкости Х-хромосомы, была выявлена у 44 видов млекопитающих (от летучей мыши до дельфина); эта последовательность прошла 150 млн лет эволюции, что доказывает ее функциональную значимость (22). Данные многих исследований свидетельствуют о сохранении микросателлитов в геномах различных млекопитающих на протяжении десятков тысяч лет (13, 14, 23).
Некоторые микросателлиты, расположенные в участках промоторов могут влиять на функцию энхансера при экспрессии генов. Так, фрагмент ДНК, локализованный слева от гена Еβ иммуноглобулина и содержащий микросателлитные участки, служит в качестве позитивного энхансера (24). Однако имеются микросателлиты, которые ингибируют активность промотора, например расположенный слева от α 2(I)-гена коллагена кур длинный фрагмент ДНК, содержащий множество микроса-теллитных участков — (GGAA) 25 , (GGGA) 6 , (GGGAGGAA) 4 , (GGGGGGAA) 2 (25). Возможно, что в некоторых случаях неактивные участки микросателлитов все же несут какую-то функцию in vivo (4).
Показано, что короткие нуклеотидные последовательности, находящиеся вблизи генов, могут служить сайтами связывания различных белков, так же, как и промоторы, содержащие микросателлиты (26). Способность микросателлитов взаимодействовать с белками зависит от количества тандемных повторов на специфическом фрагменте последовательности, который может принимать участие в синтезе белков и особенно гомополимерных участков аминокислот (4).
Вариации повторяющихся повторов в микросателлитах оказывают влияние на фенотип, что наиболее наглядно можно проследить при генетических заболеваниях человека, вызванных увеличением тринуклеотидных повторов (27). Впервые взаимосвязь между заболеванием и увеличением числа повторов была отмечена в 1991 году при исследовании синдрома хрупкости Х-хромосомы (наиболее распространенная форма задержки умственного развития). К 1995 году было идентифицировано 11 локусов, в которых наблюдалось увеличение длины поли(CGG)-повторов (10-1000-кратное) и поли(CAG)-повторов (в 2-3 раза) по сравнению с длиной фрагментов у здоровых людей. Эти локусы обусловливают соответственно задержку умственного развития (пять сайтов) и нейродегенеративные заболевания (шесть сайтов). В настоящее время список заболеваний расширился и включает синполидакти-лию, атаксию Фридриха, аутосомальную доминантную спастическую параплегию и аутосомальную доминантную окулофарингеальную мышечную дистрофию, которые связаны с увеличением частоты следующих повторов: соответственно поли(CGG), по-ли(CAA), поли(CAG) и поли(GCG) (28-30).
М у т а ц и о н н ы е и з м е н е н и я м и к р о с а т е л л и - т о в. Частота мутаций микросателлитов сравнима с частотой точечных мутаций — 10–9-10–10. Предположительная частота мутаций на один локус in vivo составляет у Escherichia coli и дрожжей соответственно около 10–2 и 10–5 (31, 32). В работах по определению отцовства у людей установлена средняя частота мутаций на один локус (на одно поколение), которая равна 10–3 (33). Частота мутаций микросателлитов у мыши, овцы, свиньи и дрозофилы составляет соответственно 10–3-10–4, 1,3 ⋅ 10–4, 7 ⋅ 10–5 и 6 ⋅ 10–6 (34-38). Предполагают, что такая высокая частота мутаций микросателлитов возникает либо при ошибках репликации отдельной ДНК (31), либо в процессе рекомбинации между молекулами ДНК (39).
Ошибки во время репликации (SSM) появляются в то время, когда синтезирующаяся молекулярная цепь ДНК отделяется от матрицы. При репликации неповторяющихся последовательностей новая цепь присоединяется к матрице строго по последовательности нуклеотидов, а в случае повторяющихся фрагментов может произойти сбой при считывании, поэтому новая молекула ДНК будет короче или длиннее, чем матричная цепь; это зависит от того, где происходит сдвиг: если на матричной цепи, то продукт будет короче, если же на синтезирующейся цепи — длиннее.
Рекомбинация потенциально может повлиять на длину микросателлитов либо в результате неравного кроссинговера, либо конверсии генов. Неравный кроссин-говер обычно происходит между хромосомами, имеющими длинные тандемные повторы. При этом может возникнуть делеция в одной молекуле ДНК и вставка в другой, а также увеличиться или уменьшиться число вариантов в тандемно повторяющихся последовательностях (40). При конверсии генов происходит перенос последовательностей из одного аллеля в другой, то есть в локусах меняется порядок повторов в результате встраивания новых последовательностей. Большинство исследований все же свидетельствует о ведущей роли SSM в образовании мутаций в микросателлитах (32, 35).
Применение микросателлитов для иссле-дования э в о л ю ц и и с е л ь с к о х о з я й с т в е н н ы х ж и- в о т н ы х. Микросателлиты используют как для анализа эволюционной связи между различными породами сельскохозяйственных животных, так и для определения времени появления той или иной породы. Например, при исследовании шести пород овец (ромни-марш, авасси, бордер, суффолк, австралийские и новозеландские мериносы) при помощи восьми микросателлитов (MAF33, MAF48, MAF65, MAF209, OarFCB11, OarFCB128, OarFCB266 и OarFCB304) показано, что между австралийскими и новозеландскими мериносами генетическое расстояние было наименьшим (0,08), а между породами авасси и бордер — наибольшим (0,50) (41). Между остальными четырьмя породами и породой авасси этот показатель варьировал от 0,36 до 0,50, а между породами ромни-марш, суффолк и бордер — от 0,26 до 0,30.
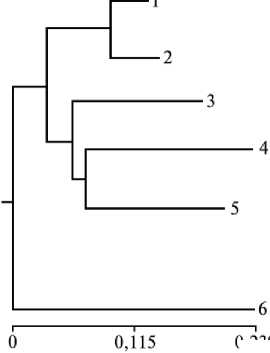
Дендрограмма взаимосвязи между шестью породами овец на основе анализа восьми микросателлитов: 1, 2, 3, 4, 5 и 6 — соответственно новозеландский меринос, австралийский меринос, ромни-марш, бордер, суффолк и авасси (цит. по 41).
0,230
Генетическое расстояние
На основе этих данных авторы построили дендрограмму, состоящую из трех ветвей, в одну из которых входили мериносы, в другую — породы ромни-марш, бордер и суффолк, а в третью — порода авасси, расположенная на большом расстоянии от остальных (рис.). Результаты этих исследований свидетельствуют о том, что расхождение между мериносами и британскими породами овец произошло не более чем 2000 лет назад (точнее 1094 года назад). На основании экспериментов с использованием 30 локусов белков крови эта дата определена как 69700 лет, а время расхождения между австралийскими и новозеландскими мериносами составляет 227 лет (42).
Микросателлиты (в отличие от белковых маркеров) позволяют более точно рассчитать время дивергенции популяций или пород, происходящих от общего предка. Так, в работе Diez-Tascon с соавт. были выявлены генетические связи между шестью популяциями мериносовых овец из Испании, Португалии и Новой Зеландии (испанский и новозеландский меринос, французский и немецкий мясной меринос, португальский черный и белый меринос) с помощью 20 микросателлитов: BM1824, BM4621, BM6444, BM6506, BM757,
ETH225, MAF209, MAF23, McM214, McM218, McM357, OarCP20, OarCP34,
OarCP49, OarFCB11, OarFCB128, OarFCB20, OarFCB304, OarFCB48, OarHH64 (43). При этом оказалось, что между испанскими и португальскими черными мериносами генетическое расстояние было наименьшим (0,086), между другими популяциями Пиренейского полуострова оно варьировало от 0,135 до 0,086. Генетическое расстояние между французскими и немецкими мериносами составляло 0,139, а между этими породами и новозеландскими мериносами — соответственно 0,356 и 0,328. Популяции, имеющие общую продуктивную направленность (мясная или шерстная), были объединены в общие кластеры.
Сравнительный анализ общности животных на основе оценки вероятности происхождения по микросателлитам позволяет определить родословную и принадлежность к той или иной породе. Например, анализ происхождения австрийских домашних овец ( Ovis aries ) проводили по шести микросателлитным локусам — McM42, McM527, OarFCB20, INRA49, TGLA53 и MAF65 (44). При этом рассматривалась возможность использования для микросателлитного анализа образцов различных тканей и жидкостей (кровь, ткани кожи, эпителий внутренней стороны щек, волосы и др.). Наиболее пригодным материалом оказались образцы волос, которые сохраняются при температуре окружающей среды без всякой предварительной консервации, что уменьшает расходы, а также они могут быть легко собраны.
Анализ происхождения пород крупного рогатого скота на основе микросател-литных маркеров представляет особенную ценность при формировании широкоразво-димых стад, в которых используют животных, полученных от нескольких производителей. Так, при определении родственных связей между несколькими группами крупного рогатого скота (по 50 гол. в каждой), включающими животных разных пород, оценивали эффективность 21 микросателлитного маркера (45). Для выявления отцовства анализировали 5960 телят (по 50-500 гол. в группе), полученных от производителей из 39 групп, включающих от 4 до 34 быков. В большинстве тестируемых групп количество телят, полученных от неизвестных быков, существенно различалось. При этом оказалось, что если доля телят, полученных от неизвестных производителей, составляет более 50,8 %, то точность определения отцовства возможна в 86 % случаев; в тех вариантах, где имелись сведения о быках-производителях, точность анализа составляла 100 %.
Polli с соавт. оценивали генетический полиморфизм семи аутохтонных (местных) итальянских пород крупного рогатого скота с целью определения их генетической структуры и филогенетического происхождения (46). Изучение генетического разнообразия аутохтонных пород имеет большое значение для программ сохранения биоразнообразия видов крупного рогатого скота. Породы Aosta Red Pied, Aosta Black Pied и Aosta Chestnut разводят в предгорьях Альп, а породы Oropa, серая альпийская, Rendena и Burlina — в других районах Италии. Для анализа использовали 15 высокополиморфных микросателлитов из набора, одобренного Международным обществом по генетике животных (МОГЖ), для изучения биоразнообразия крупного рогатого скота. ДНК выделяли из крови или спермы и амплифицировали посредством ПЦР. Аллели визуализировали автоматическим секвенатором в 4,2 % полиакриламидном геле. Оценивали гетерозиготность каждого локуса, частоту аллелей и PIC (polymorphism information content), рассчитывали генетические расстояния. При этом наименьшее генетическое расстояние выявлено между породами Aosta Red Pied и Aosta Black Pied, а наибольшее — Burlina и Aosta Black Pied.
Высокий уровень полиморфизма и случайное расположение в геноме позволяют использовать микросателлиты для идентификации отцовства, не генотипируя самок. Genzini с соавт. проанализировали 444 семьи итальянских буйволов (бык-производитель—дочь) с использованием шести микросателлитных локусов (DRB3, CSSM47, D5S2, MAF65, CSSM19, CYP21) и гена главного комплекса гистосовместимости класса II овец (47). По частоте аллелей общая вероятность исключения составила 0,98, то есть ошибка определения происхождения равна 2 %.
При исследовании происхождения финской айрширской и голштинской пород крупного рогатого скота были использованы девять микросателлитных локусов в различных комбинациях в трех реакциях мультиплекс-ПЦР, согласно стандартному протоколу: мультиплекс 1 — BM2133, BM1824, SPS115; мультиплекс 2 — ETH3, ETH225, ETH10; мультиплекс 3 — TGLA227, TGLA126, TGLA122 (48). В третью реакцию (мультиплекс 3) были включены дополнительные локусы — TGLA53 и INRA023 для анализа частоты аллелей и вероятности исключения. Высокий уровень полиморфности в популяции скота айрширской породы ( n = 50) выявлен по локусам BM2113, TGLA227, ETH10 и ETH225, голштинской породы ( n = 50) — по локусам TGLA227, TGLA122 и BM2113; вероятность исключения составляла соответственно 56 и 62 %. Общая вероятность исключения при использовании девяти локусов достигала 99,84 и 99,91 % соответственно у животных айрширской и голштинской пород; при использовании дополнительных локусов TGLA53 и INRA023 полученные ранее показатели возросли соответственно до 99,94 % и 99,98 %.
Для выявления полиморфизма у коз пяти китайских аборигенных популяций (льаонин кашмир, n = 33; внутренний монгольский кашмир, n = 35; тибетский кашмир, n = 37; вуан кашмир, n = 49 и мясные козы мату, n = 39) использовали микросателлиты крупного рогатого скота (BM1842, BM4621, BM6444, BM6506, BM757) и овец (OarCP20, OarCP34, OarCP49, OarFCB11, OarFCB11, OarFCB128, OarFCB20, OarFCB266, OarFCB304, OarFCB48, MAF33, McM218) (49). Более четырех аллелей были обнаружены в шести локусах (BM4621, OarCP34, OarFCB11, OarFCB20, OarFCB304, OarFCB48), другие девять микросателлитов оказались мономорфными. Определяли внутрипородное разнообразие, гетерозиготность и PIC. При этом рассчитывали общее генетическое разнообразие, среднюю гетерозиготность в пределах каждой популяции и коэффициенты дифференциации гена между породами; средние значения этих параметров составляли соответственно 0,831; 0,800; 0,038. По данным этих исследований, межпородное различие китайских аборигенных коз находится на низком уровне, тогда как внутрипородное разнообразие оказалось достаточно высоким.
Таким образом, представленный обзор свидетельствует о том, что микросателлиты обладают рядом свойств (высокий уровень полиморфизма и изменчивости, кодоминантное наследование, равномерное распределение по геному и др.), которые позволяют использовать их в качестве маркеров при оценке происхождения и эволю-108
ции различных видов и пород животных (50). С помощью микросателлитов можно идентифицировать все кодоминантно наследуемые аллели закрытого локуса, что не всегда возможно при анализе генетических маркеров I порядка, оценивать генетическое разнообразие популяций; они также подходят для теста на достоверность отцовства, оценки дивергенции пород, теста на диагностику явления «бутылочного горлышка» (bottleneck) в популяции. Недостатком этого метода является дороговизна, необходимость солидной материальной базы и специалистов, которых, к сожалению, пока в нашей стране мало.
Л И Т Е Р А Т У Р А
-
1. T a u t z D. Hypervariability of simple sequences as a general source for polymorphic DNA markers. Nucleic Acids Research, 1989, 17: 6463-6471.
-
2. T a u t z D., R e n z M. Simple sequences are ubiquitous repetitive components of eukariotic genomes. Nucleic Acids Research, 1984, 12: 4127-4138.
-
3. W r i g h t J.M. Mutation at VNTRs: are minisatellites the evolutionary progeny of microsatellites? Genome, 1994, 37: 345-347.
-
4. G o l d s t e i n D.B., S c h l o t t e r e r C. Microsatellites. Evolution and Applications. Oxford, 1999.
-
5. V o g t P. Potential genetic functions of tandem repeated DNA sequence blocks in human genome are based on
highly conserved chromatine folding code. Human Genetics, 1990, 84: 301-336.
-
6. W e b e r J.L. Informativeness of human (dC-dA) n ⋅ (dG-dT) n polymorphisms. Genomics, 1990, 7: 524-530.
-
7. W i n t e r o A.K., F r e d h o l m M., T h o m s e n P.D. Variable (dG-dT) n (dC-dA) n sequences in the
porcine genome. Genomics, 1992, 12: 281-288.
-
8. V a i m a n D., I m a m - C h a l i M., M o a z a m i - G o u d a r z i K. e.a. Conservation of a synthetic group of microsatellite loci between cattle and sheep. Mammalian Genome, 1996: 676-683.
-
9. D i b C., F a u r e S., F i z a m e s C. e.a. A comprehensive genetic map of the human genome based on 5264 microsatellites. Nature, 1996, 380: 149-152.
-
10. M o r a n C. Microsatellite repeats in pig ( Sus domestica ) and chicken ( Gallus domesticus ) genomes. Zoo Biology, 1993, 10: 341-352.
-
11. H a n c o c k J.M. The contribution of slippage-like processes to genome evolution. J. Molecular
Evolution, 1995, 41: 1038-1047.
-
12. B e c k m a n J.S., W e b e r J.L. Survey of human and rat microsatellites. Genomics, 1992, 12: 627-631.
-
13. S t a l l i n g s R.L. Distribution of trinucleotide microsatellites in different categories of mammalian genomic se
quence: implications of human genetic diseases. Genomics, 1994, 21: 116-121.
-
14. S t a l l i n g s R.L. CpG supression in vertebrale genomes does not account for the rarity of (CpG) n microsatellite repeats. Genomics, 1992, 17: 890-891.
-
15. S c h u g M.D., W e t t e r s t r a n d K.A., G a u d e t t e M.S. e.a. The distribution and frequency of microsatellite loci in Drosophila melanogaster . Molecular Ecology, 1998, 7: 57-69.
-
16. H a n c o c k J.M. Simple sequences in a «minimal» genome. Nature Genetics, 1996, 13: 14-15.
-
17. K a s h i Y., K i n g D., S o l l e r M. Simple sequence repeats as a source of quantitative genetic variation.
-
18. B r a a t e n D.C., T h o m a s J.R., L i t t l e R.D. e.a. Locations and contexts or sequences that hybridize to poly(dG-dT) n ⋅ (dC-dA) n in mammalian ribosomal DNAs and two X-linked genes. Nucleic Acid Research, 1988, 18: 865-881.
-
19. S h e n L.-P., R u t t e r W.J. Sequence of the human somatostatine gene. Science, 1984, 224: 168-170.
-
20. H a y e s T.E., D i x o n J.E. Z-DNA in the rat somatostatin gene. J. Biological Chemistry, 1985, 260:
-
21. M c K e o n C., S c h m i d t A., d e C r o m b r u g g h e B. A sequence conserved in both the chicken
and mouse α 2(I) collage promoter contains sites sensitive to S1 nuclease. J. Biological Chemistry, 1984,
259: 6636-6640.
-
22. E i c h l e r E.E., K u n s t C.B., L u g e n b e e l K.A. e.a. Evolution of the cryptic FMR1 CGG repeat.
-
23. M o o r e S.S., S a g e a n t L.L., K i n g T.J. e.a. The conservation of dinucleotide microsatellites among
mammalian genomes allows the use of heterologous PCR primer pairs in closely related species. Genomics, 1991, 10: 654-660.
-
24. G i l l i e s S.D., F o l s o m B., T o n e g a w a S. Cell-type specific enhancer element associated with a mouse MHC gene. Nature, 1984, 310: 594-597.
-
25. K o l l e r E., H a y m a n A.R., T r u e b B. The promoter of the chicken a2(VI) collagen gene has features characteristic of house-keeping genes and of proto-oncogenes. Nucleic Acids Research, 1991, 19: 485491.
-
26. L u e N.L., B u c h m a n A.R., K o r n b e r g R.D. Activation of yeast RNA polymerase II transcription by a thymidine-rich upstream element in vitro. Proceedings of the National Academy of Sciences of the USA, 1989, 86: 486-490.
-
27. S u t h e r l a n d G.R., R i c h a r d s R.I. Simple tandem DNA repeats and human genetic disease. Proceedings of the National Academy of Sciences of the USA, 1995, 92: 3636-3641.
-
28. C a m p u z a n o V., M o n t e r m i n i L., M o l t o M.E. e.a. Friedreich’s Ataxia: autosomal recessive disease caused by an intronic GAA triplet repeat expansion. Science, 1996, 271: 1423-1427.
-
29. N i e l s e n R. A likelihood approach to population samples of microsatellite alleles. Genetics, 1997, 146: 711-716.
-
30. B r a i s B., B o u c h a r d J.-P., X i e Y.-G. e.a. Short GCG expansions in the PABP2 gene cause oculo-pharingeal muscular distrophy. Nature Genetics, 1998, 18: 164-167.
-
31. L e v i n s o n G., G u t m a n G.A. High frequencies of short frameshifts in poly-CA/TG tandem repeats borne by bacteriophage M13 in Escherichia coli K-12. Nucleic Acid Research, 1987, 15: 5323-5328.
-
32. H e n d e r s o n S.T., P e t e s T.D. Instability of simple sequence DNA in Saccharomyces cerevisiae . Molecular and Cell Biology, 1992, 12: 2749-2757.
-
33. W e b e r J.L., W o n g C. Mutation of human short tandem repeats. Human Molecular Genetics, 1993, 2: 1123-1128.
-
34. D a l l a s J.F. Estimation of the microsatellite mutation rates in recombinant inbred strains of mouse. Mammalian Genome, 1992, 3: 452-456.
-
35. C r a w f o r d A.M., C u t h b e r t s o n R.P. Mutations in sheep microsatellites. Genome Research, 1996, 6: 876-879.
-
36. S c h u g M.D., M a c k a y T.F.C., A q u a d r o C.F. Low mutation rates of microsatellite loci in Drosophila melanogaster . Nature Genetics, 1997, 15: 99-102.
-
37. K w i a t k o w s k i D.J., H e n s k e E.P., W e i m e r K. e.a. Construction of GT polymorphism map of human 9q. Genomics, 1992, 12: 229-240.
-
38. E l l e g r e n H. Mutation rates at porcine microsatellite loci. Mammalian Genome, 1995, 6: 376-377.
-
39. J e f f r e y s A.J., T a m a k i K., M a c L e o d K. e.a. Complex gene conversion events in germline muta
tion at human minisatellites. Nature Genetics, 1994, 6: 136-145.
-
40. D o v e r G.A. Molecular drive: a cohesive mode of species evolution. Nature, 1982, 299: 111-117.
-
41. B u c h a n a n F.C., A d a m s L.J., L i t t l e j o h n R.P. e.a. Determination of evolutionary relationships
among sheep breeds using microsatellites. Genomics, 1994, 22: 347-403.
-
42. M a n w e l l C., B a c k e r C.M.A. Genetic distance between the Australian Merino and the Poll Dorset sheep. Genetics Research Cambridge, 1977, 29: 239-253.
-
43. D i e z - T a s c o n C., L i t t l e j o h n R.P., A m e i d a P.A.R. e.a. Genetic variation within the Merino sheep breed: analysis of closely related populations using microsatellites. Animal Genetics, 2000, 31: 243251.
-
44. A c h m a n n R., B r e m G. Parentage control in Austrian domestic mountain sheep ( Ovis aries ) using DNA microsatellite analysis. Animal Genetics, 1998, 29: 12-13.
-
45. V a n k a n D.M., H e f f o r d C.G., F a d d y M.J. Efficacy and reliability of paternity testing in cattle using DNA microsatellites. Animal Genetics, 1998, 29: 9.
-
46. P o l l i M., D e l B o L., C e r i o t t i G. e.a. Genetic distances of seven cattle breeds of the Italian Alpine area by microsatellites. Animal Genetics, 1998, 29: 9-10.
-
47. G e n z i n i E., B l a s i M., C a p u a n o M. e.a. Amplification of seven microsatellites for parentage identification in Italian buffalo. Animal Genetics, 1998, 29: 10.
-
48. B r e d b a c k a P., K o s k i n e n M.T. Polymorphism of microsatellite loci used in parentage testing in Finnish Ayrshire and Holstein populations. Animal Genetics, 1998, 29: 10-11.
-
49. Z h a o S., Y a n g L., L i K. e.a. Analysis of genetic variation among five Chinese indigenous goat breeds by using bovine and ovine microsatellites. In: Animal Genomics: Synthesis of Past, Present, and Future Directions. 27th Intern. Conf. on Animal Genetics. USA, 2000.
-
50. M a l e v i C i U t ё J., B a 11 r ё n a i t ё L., M i c e i k i e n ё I. Domestic cattle breed diversity in Lithuania. Veterinary Medicine and Zootechnics, 2002, 20: 87-91.
Trends in Genetics, 1997, 13: 74-78.
8145-8156.
Nature Genetics, 1995, 11: 301-307.
Всероссийский государственный НИИ животноводства,


