Молекулярно-генетическая характеристика изолятов вируса прагриппа-3
Автор: Галиуллин А.К., Гериш А., Гумеров В.Г., Шаева А.Ю., Магдеева Э.А.
Статья в выпуске: 4 т.248, 2021 года.
Бесплатный доступ
В данном исследовании сообщается о молекулярно-генетической характеристике полевого штамма вируса ПГ-3 крупного рогатого скота, выделенного в Республике Татарстан. Фрагменты М-гена российского изолята ЛД-9 и вакцинного штамма ПТК-45/86 были секвенированы и выравнены с соответствующими последовательностями штаммов вируса, представленными в GenBank. Результаты филогенетического анализа показали, что российский изолят ЛД-9 (MW52481) и вакцинный штамм ПТК45/86 относятся к генотипу А вируса парагриппа-3, как и референтный штамм SF-4. Российский изолят ЛД-9 показал 98,3 % сходство с референтным штаммом SF-4, а российский вакцинный штамм ПТК-45/86 был идентичен референтному штамму на 100 %.
Парагрипп-3, вирус парагриппа-3, крупный рогатый скот, генотип, филогенетический анализ
Короткий адрес: https://sciup.org/142231411
IDR: 142231411 | УДК: 619:616.9:616.24 | DOI: 10.31588/2413-4201-1883-248-4-42-47
Текст научной статьи Молекулярно-генетическая характеристика изолятов вируса прагриппа-3
Респираторные заболевания являются важной проблемой в молочном скотоводстве, которая в основном затрагивает молодое поголовье. Болезни органов дыхания крупного рогатого скота носят многофакторный и многоэтиологический характер, в котором, наряду с вирусами и бактериями, задействованы факторы риска, связанные с окружающей средой.
Вирус парагриппа-3 крупного рогатого скота является одним из основных возбудителей респираторных болезней, особенно у молодняка [1, 3, 9]. Это РНК-содержащий вирус, относящийся к роду Paramyxoviridae и поражающий центральную нервную и дыхательную системы, способствующий повреждению тканей у телят [2, 6]. Течение парагриппа-3 также может осложняться вторичной бактериальной инфекцией и приводить к тяжелой бронхопневмонии.
На основе филогенетического и генетического анализа зарубежными исследователями было выявлено три генотипа вируса парагриппа-3 крупного рогатого скота: A (BPIV3a), B (BPIV3b) и C (BPIV3c) [12]. Генотип А был впервые был выделен в США, а также был обнаружен в Японии, Китае, Аргентине и Египте [5, 10]. До 2012 года генотип В, был зарегистрирован только в Австралии, согласно Maidana и др., в 2012 году генотип
В был также выделен в Аргентине [7]. Генотип С был обнаружен в Южной Корее, Китае, Японии, Турции и Аргентине [4, 8, 10, 11]. Несмотря на то, что вирус был выделен в России еще в 1968 году, информация о генотипическом разнообразии этого инфекционного агента, циркулирующего на территории РФ, в частности, в Республике Татарстан отсутствует.
Целью данного исследования было выявление и молекулярная характеристика штамма парагриппа крупного рогатого скота типа 3 (BPIV-3), полученного из образцов легких и мазков из носа крупного рогатого скота в Лаишевском районе Республики Татарстан, а также вакцинного штамма ПTK 45/86.
Материал и методы исследований. В работе использовали следующие штаммы: 1. SF-4 – референтный штамм вируса ПГ-3, инфекционная активность на перевиваемой линии культуры клеток легкого эмбриона коровы (ЛЭК) – 7,25 lg ТЦД 50/мл, гемагглютинирующий титр 1:32 в РГА с 1 % эритроцитами морской свинки. Этот штамм был использован для контроля; 2. ПТК-45/86 – вакцинный штамм вируса ПГ-3, инфекционная активность на перевиваемой линии культуры клеток ЛЭК – 6,75 lg ТЦД 50/мл, гемагглютинирующий титр 1:16 в РГА с 1 % эритроцитами морской свинки (ЭМС).
Данный штамм был использован в исследовании в качестве контроля и для сравнения с референтным штаммом; 3. ЛД-9 - вирусный изолят, выделен из патологического материала от больного респираторным заболеванием теленка из Лаишевского района РТ (на основании изучения физико-химических и биологических свойств идентифицирован как вирус ПГ-3), инфекционная активность на перевиваемой линии культуры клеток ЛЭК – 6,25 lg ТЦД 50/мл.
Выделение нуклеиновой кислоты (РНК) вируса проводили из образцов тканей (носовая слизь, легочная ткань и лимфатические узлы) с использованием коммерческого набора РИБО-преп (AmpliSens, Россия), согласно инструкции производителя. Все выделенные пробы перед тестированием хранили при температуре минус 20 ° C.
Вирусная РНК подвергалась обратной транскрипции с помощью комплекта REVERТA-L (AmpliSens, Россия) в соответствии с инструкцией изготовителя. Образцы ДНК изолята ЛД-9, вакцинного штамма ПТК-45/86 и референтного штамма SF-4 были исследованы методом ПЦР со специфическими праймерами, кодирующими фрагмент матричного гена (М гена) вируса. В таблице 1 указаны область и размеры ампликона. Амплификацию проводили в твердотельном амплификаторе фирмы «Терцик» ДНК-технология (Россия).
Таблица 1 – Область и размеры ампликона
|
Вирус |
Ген |
Праймер |
Нуклеотидные последовательности праймеров |
Размер продукта |
|
ВПГ-3 |
М-ген |
M 1 |
AGTGATCTAGATGATGATCCA |
328 п.н. |
|
M 2 |
GTTATTGATCCAATTGCTGT |
Таблица 2 – Нуклеотидные последовательности штаммов вируса ПГ-3 крупного рогатого скота из GenBank, использованные для филогенетического анализа
|
Вирус |
Номер в GenBank |
Страна |
Генотип |
Длина генома |
|
SF-4 |
AF178655 |
USA |
a |
15456 |
|
NM09 |
JQ063064 |
China |
a |
15456 |
|
JCU |
EF108221 |
Australia |
a |
697 |
|
BN-1 |
AB770484 |
Japan |
a |
15480 |
|
ISU |
EU439428 |
USA |
a |
15480 |
|
Texas |
EU439429 |
USA |
a |
15456 |
|
910N |
D84095 |
JAPAN |
a |
15480 |
|
Kansas |
AF178654 |
USA |
a |
15454 |
|
LE017 |
MG976794 |
Uruguay |
a |
274 |
|
BP4158 |
EF108222 |
Australia |
b |
697 |
|
TVMDL15 |
KJ647284 |
USA |
b |
15474 |
|
SD0835 |
HQ530153 |
China |
c |
15498 |
|
NX49 |
KT071671 |
China |
c |
15474 |
|
TVMDL20 |
KJ647287 |
USA |
c |
15474 |
|
HS9 |
LD000638 |
Japan |
c |
15474 |
|
12Q061 |
JX969001 |
Korea |
c |
15474 |
|
GP |
AB012132 |
Japan |
/ |
15462 |
По 10 мкл полученных продуктов ПЦР были внесены в лунки 1,5 % агарозного геля, электрофорез проходил при 200 В в течение 40 мин. Детекцию результатов проводили на трансиллюминаторе с длиной волны 312
нм.
Продукты
амплификации
подвергались секвенированию.
Секвенирование проводили в
Междисциплинарном центре протеомных исследований Казанского федерального университета (КФУ) методом Сэнгера на секвенаторе NextSeq 500 (Illumina) с использованием тех же самых праймеров ПЦР и режима амплификации.
Результаты секвенирования были проанализированы с помощью программ UGENE и MegAlign Pro. Частичные геномные последовательности полевого изолята ЛД-9, вакцинного штамма ПТК-45/86 и референтного штамма SF-4 были выравнены с последовательностями штаммов вируса парагриппа-3, полученными из базы данных GenBank с использованием программы ClustalW системы MegAlign Pro (DNASTAR) (Таблица 2). Построение филогенетического древа также осуществлялось с применением системы MegAlign Pro.
Результат исследований. В результате полимеразной цепной реакции из всех трех образцов были получены специфичные продукты амплификации размером 328 п.н.
Специфический фрагмент матричного гена (М-гена) был визуализирован с помощью электрофореза в 1,5 % агарозном геле (Рисунок 1).
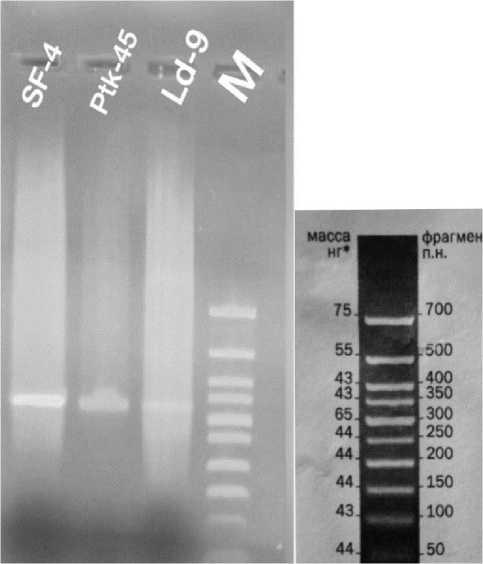
Рисунок 1 – Электрофорез продуктов амплификации: М – маркер молекулярных масс; 1, 2, 3 – положительные результаты амплификации фрагмента М гена штаммов SF-4, ПТК-45/86 и
ЛД-9 вируса парагриппа-3 крупного рогатого скота
В результате множественного выравнивания 17 нуклеотидных последовательностей, депонированных в GenBank, с последовательностями, полученными нами, российский изолят ЛД-9 показал 98,3 % идентичность с референтным штаммом SF-4, относящимся к генотипу А, тогда как российский вакцинный штамм ПТК 45/86 был на 100 % идентичен референтному штамму SF-4 (Таблица 3). Нуклеотидная последовательность фрагмента М-гена изолята ЛД-9 вируса ПГ-3 была размещена в базе данных GenBank под регистрационным номером MW524841.
Результаты филогенетического анализа показали, что российский изолят ЛД-9 (MW52481) и вакцинный штамм ПTK45/86 относятся к генотипу А вируса парагриппа-3, как и референтный штамм SF-4 (Рисунок 2).
Таблица 3 – Сравнительный анализ идентичности в нуклеотидных последовательностях области М гена (328 п.н.) изолята ЛД-9, вакцинного штамма ПTK45/86 и 17 штаммов вируса
ПГ-3 крупного рогатого скота
|
Штаммы |
Идентичность, % |
||
|
SF-4 |
ПТК45/86 |
ЛД-9 |
|
|
ПТК45/86 |
100 |
/ |
98,25 |
|
ЛД-9 |
98,3 |
98,25 |
/ |
|
SF-4 |
/ |
100 |
98,3 |
|
NM09 |
94,3 |
94,3 |
92,98 |
|
JCU |
92,5 |
92,5 |
90,8 |
|
BN-1 |
93,42 |
93,42 |
91,67 |
|
ISU* |
91,67 |
91,67 |
89,91 |
|
910N |
93,42 |
93,42 |
91,67 |
|
Kаnsas* |
99,12 |
99,12 |
97,35 |
|
BP4158 |
87,28 |
87,28 |
85,53 |
|
Texas |
100 |
100 |
98,25 |
|
TVMDL15 |
86,84 |
86,84 |
85,09 |
|
SD0835 |
87,72 |
87,72 |
86,4 |
|
NX49 |
87,72 |
87,72 |
84,4 |
|
TVMDL20 |
87,28 |
87,28 |
85,96 |
|
HS9 |
88,16 |
88,16 |
86,84 |
|
12Q061 |
87,28 |
87,28 |
85,96 |
|
LE017 |
99,12 |
99,12 |
97,37 |
|
GP** |
81,58 |
81,58 |
81,4 |
Примечание: *swine parainfluenza virus type 3 (SPIV-3), **Human parainfluenza type 3 (HPIV-3)
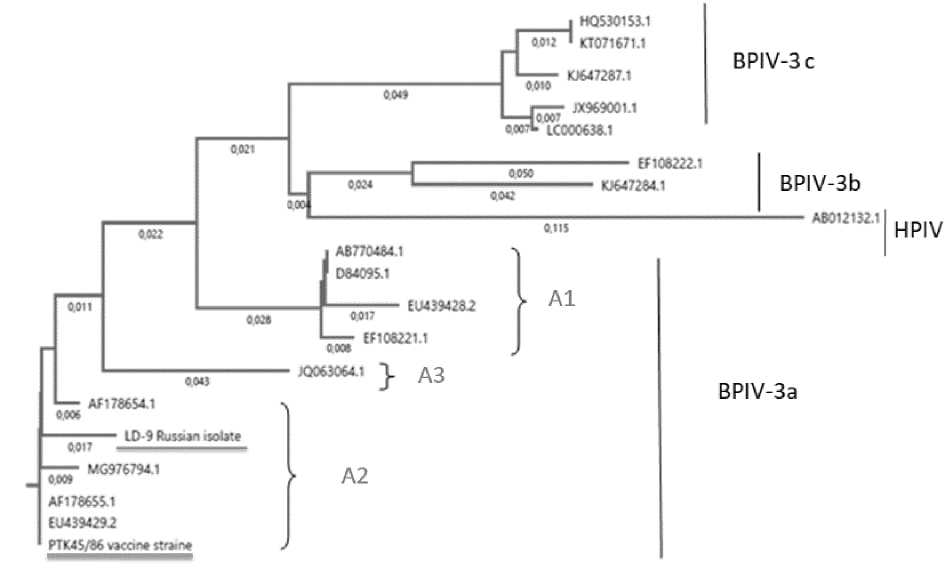
Рисунок 2 – Филогенетический анализ нуклеотидных последовательностей изолятов вируса ПГ-3 на основе фрагмента М гена
Заключение. Матричный белок вируса парагриппа-3 играет важную роль в сборке вирионов, установлении персистирующей инфекции и патогенезе вируса. Ген (М-ген), кодирующий этот белок, является подходящей мишенью для создания информативной филогенетической реконструкции вируса парагриппа-3 крупного рогатого скота.
Филогенетический анализ нуклеотидной последовательности фрагмента М-гена представителей BPIV-3 показал, что российский изолят ЛД-9 и вакцинный штамм ПTK-45/86 относятся к генотипу А. Вакцинный штамм ПTK-45/86, в свою очередь, присутствует в живой вакцине «Тривак», применяемой для профилактики инфекционного ринотрахеита, парагриппа-3, вирусной диареи крупного рогатого скота. Выделенный изолят ЛД-9 генотипически близок к американским и уругвайским штаммам.
Вакцины, используемые в России против вируса парагриппа крупного рогатого скота-3, были разработаны на основе референтного штамма, принадлежащего генотипу А. Перекрестная защита таких вакцин от парагриппа-3, вызванного генотипами В и С, остается неясной, поэтому следует оценить необходимость разработки альтернативных диагностических тестов и вакцин для профилактики. Генетическая характеристика циркулирующих штаммов возбудителя может способствовать разработке более эффективных методов диагностики и профилактики данной инфекции.
В данном исследовании сообщается о молекулярно-генетической характеристике полевого штамма вируса ПГ-3 крупного рогатого скота, выделенного в Республике Татарстан. Фрагменты М-гена российского изолята ЛД-9 и вакцинного штамма ПТК-45/86 были секвенированы и выравнены с соответствующими последовательностями штаммов вируса, представленными в GenBank. Результаты филогенетического анализа показали, что российский изолят ЛД-9 (MW52481) и вакцинный штамм ПTK45/86 относятся к генотипу А вируса парагриппа-3, как и референтный штамм SF-4. Российский изолят ЛД-9 показал 98,3 % сходство с референтным штаммом SF-4, а российский вакцинный штамм ПТК-45/86 был идентичен референтному штамму на 100 %.
Summarу
This study reports on the molecular-genetic characterization of a field strain of BPIV-3 isolated in the Republic of Tatarstan. M gene fragments of the Russian isolate LD-9 and the vaccine strain PTK/86 were sequenced and aligned with the corresponding sequences of the virus strains presented in GenBank. The results of the phylogenetic analysis showed that the Russian isolate LD-9 (MW52481) and the vaccine strain PTK45/86 belong to the genotype A of the parainfluenza virus-3, as well as the reference strain SF-4. The Russian isolate LD-9 showed 98.3 % similarity with the reference strain SF-4, and the Russian vaccine strain PTK-45/86 was 100 % identical to the reference strain.
Список литературы Молекулярно-генетическая характеристика изолятов вируса прагриппа-3
- Гумеров, В.Г. Эпизоотологический и серологический мониторинг смешанных респираторно-кишечных инфекций крупного рогатого скота / В.Г. Гумеров, В.В. Евстифеев, Х.Н. Макаев [и др.] // Ученые записки Казанской государственной академии ветеринарной медицины им. Н.Э. Баумана. – 2019. – № 237 (1). – С. 56-60.
- Тяпша, Ю.И. Полимеразная цепная реакция для детекции вируса парагриппа-3 / Ю.И. Тяпша, О.В. Дубаневич // Актуальные проблемы биотехнологии в аграрно-промышленном комплексе. – 2015. – С. 205-208.
- Adams, M.J. Ratification vote on taxonomic proposals to the international committee on taxonomy of viruses / M.J. Adams, E.J. Lefkowitz, A.M.Q. King [et al.] // Arch Virol. – 2016. – V. 160(7). – P. 2921-2949.
- Albayrak, H. Characterisation of the first bovine parainfluenza virus 3 isolate detected in cattle in Turkey / Z. Yazici, E. Ozan [et al.] / Vet. Sci. – 2019. – V. 6. – doi:10.3390/vetsci6020056
- Konishi, M. Complete genome sequence of the first isolate of genotype C bovine parainfluenza type 3 in Japan. / M. Konishi, T. Ohkura, M. Shimizu, et al. - Genome Announcements. – 2014. – V. 2(6). – doi: 10.1128/genomeA.01215-14.
- Lamoril, J.A. Les techniques de séquencage de l’ADN: une révolution en marche. Première partie DNA sequencing technologies: A revolution in motion. Part one/ J.A. Lamoril., B.P. Ameziane, M. Bouizegarènea // Immuno-analyse et biologie spécialisée. – 2008. – № 23. – P. 260-279.
- Maidana, S. Isolation and characterization of bovine parainfluenza virus type 3 from water buffaloes (Bubalus bubalis) in Argentina / S. Maidana, P. Lomonaco, G. Combessies [et al.] // BMC Veterinary Research. – 2012. – 8:83. – P. 1-9.
- Oem, J.K. Molecular characterization of a Korean bovine parainfluenza virus type 3 isolate / J.K. Oem, E.Y. Lee, K.K. Lee [et al.] // Veterinary Microbiology. – 2013. – V. 162. – P. 224–227.
- Ohkura, T. Complete genome sequences of bovine parainfluenza virus type 3 strain BN-1 and vaccine strain BN-CE / T. Ohkura, M. Konishi T. Kokuho [et al.] // Genome Announcements. – 2013. – V. 1(1). – DOI:10.1128/genomeA.00247-12
- Sobhy, N.M. Surveillance, isolation and complete genome sequence of bovine parainfluenza virus type 3 in Egyptian cattle / N.M. Sobhy, S.K Mor, М.В. Iman [et al.] // International Journal of Veterinary Science and Medicine. – 2017. – V. 5 (1). – Р. 8-13.
- Wen, X. Identification and genetic characterization of bovine parainfluenza virus type 3 genotype C strain isolated from cattle in Western China / X. Wen, Sun J., Zhang Y. [et al.] // Turkish Journal Of Veterinary And Animal Sciences. – 2017. – V. 41(2). – P. 180-186.
- Zhu, Y.M. Isolation and genetiс characterization of bovine parainfluenza virus type 3 from cattle in China / Y.M. Zhu, H.F. Shi, Y.R. Gao [et al.] // Veterinary Microbiology. – 2011. – V. 149(3-4). – P. 446-451.


