Молекулярно-генетический анализ трансгенных клеточных линий мягкой пшеницы с использованием IRAP ПЦР
Автор: Бавол Андрей Васильевич, Дубровная Оксана Васильевна
Журнал: Известия Самарского научного центра Российской академии наук @izvestiya-ssc
Рубрика: Биотехнология
Статья в выпуске: 3-5 т.15, 2013 года.
Бесплатный доступ
Методом IRAP ПЦР с праймером к LTR последовательностям ретротранспозона SIRE1, у трех трансгенных линий мягкой пшеницы обнаружено появление новых, относительно высокомолекулярных (длиной более 1000 п.н.) ампликонов, что свидетельствует о транспозиции данного мобильного элемента. Полученные данные могут служить доказательством того, что индукция транспозиции SIRE 1 обусловлена геномным стрессом, вызванным встраиванием чужеродной ДНК или стрессом, непосредственно связанным с процессом трансформации (микропоранение, культивирование на селективных средах).
Мягкая пшеница, биолистическая трансформация, каллусные линии, ретротранспозон sire 1, irap пцр
Короткий адрес: https://sciup.org/148202042
IDR: 148202042 | УДК: 581.143.6:58.085
Текст научной статьи Молекулярно-генетический анализ трансгенных клеточных линий мягкой пшеницы с использованием IRAP ПЦР
Технология генетической трансформации имеет большие перспективы в селекции растений для улучшения хозяйственно полезных признаков, в частности урожайности, повышения качества, устойчивости к патогенам и вредителям [1]. Для пшеницы перспективным методом генетической инженерии является биолистическая трансформация, поскольку ткани этой культуры имеют низкую чувствительность к агробактериальной инфекции [2]. На сегодняшний день большинство протоколов получения генетически модифицированных организмов предполагает использование культуры in vitro , в том числе этапы образования каллуса и регенерации растений. Следует учитывать, что при этом может возникать значительное число мутаций, известных под общим названием сомакло-нальной изменчивости [3]. Однако кроме мутагенного эффекта культуры тканей, процесс трансформации сопровождается дополнительным стрессом, связанным как с механическим поранением (при использовании бомбардировки микрочастицами), так и с встраиванием генетической конструкции в геном реципиента [4].
Несмотря на то, что темпы получения и выращивания генетически модифицированных культур постоянно возрастают, все еще остается вопрос насколько точно современные методы генетической инженерии могут обеспечить перенос в геном чужеродных генов и сколько непредсказуемых генетических и эпигенетических эффектов при этом может возникать [5]. Следствием переноса чужеродной ДНК может быть нарушение стабильности генома трансформированного растения, однако данной проблеме уделяется недостаточно внимания, хотя такие исследования связаны не только с фундаментальными вопросами организации и функционирования генома в целом, но также с проблемой безопасности употребления трансгенных растений.
Показано, что интеграция чужеродной ДНК в ядерный геном может вызвать как эпигенетические изменения, в частности изменения уровня метилирования ДНК, так и активацию транскрипции мобильных генетических элементов (МГЭ) [6]. Matzke и сотрудники [7] получили данные, которые убедительно свидетельствуют о том, что в присутствии трансгенов в геноме реципиента наблюдается повышение частоты генетических перестроек и активация МГЭ. Явление транспозиции представляет значительный интерес, поскольку показана его роль как в изменении экспрессии структурных генов, так и индукции хромосомных аберраций [8].
IRAP-анализ (Inter-Retrotransposon Amplified Polymorphism) - метод амплификации геномной ДНК между близко расположенными последовательностями ретротранспозонов. Одним из преимуществ этого метода является возможность одновременного анализа многих локусов в различных участках генома, что особенно важно при культивировании in vitro . Широкое присутствие в геноме растений мобильных генетических элементов и их способность к самостоятельному копированию позволяет методом IRAP-анализа идентифицировать относительно недавние изменения генома, в том числе вызванные действием стрессовых факторов. Одним из мобильных элементов, который используется для анализа генома растений является соевый ретротранспозон SIRE 1, относящийся к семейству Ty1-copia -подобных ретроэлементов. Он присутствует в геноме многих видов растений, в том числе и пшеницы, имеет длину примерно 12 т.п.н. В данной работе был использован праймер, родственный к длинным концевым повторам (LTR-последовательностям) SIRE-1. Этот участок содержит низкокопийные последовательности нуклеотидов и является одним из самых консервативных. Именно поэтому изменения в длине между последовательностями ретротранспозонов, которые можно зафиксировать, как появление/исчезновение или изменение размера IRAP-ампликона, могут служить объективным критерием изменчивости генома.
В связи с этим, целью данной работы был молекулярно-генетический анализ трансгенных клеточных линий мягкой пшеницы с использование IRAP ПЦР с праймером к LTR последовательностям ретротранспозона SIRE 1.
Каллусные культуры были получены из апекса побега 3-суточных стерильных растений пшеницы сорта Зимоярка по разработанной нами методике [9]. Исходный каллус разделяли на 2 части. Одна часть была использована для проведения генетической трансформации, другая параллельно пассировалась на питательной среде МС. Генетическую трансформацию осуществляли биолистическим методом, используя самодельную пушку типа particle inflow gun (PIG). В исследованиях была использована векторная конструкция pAHC25 [10], содержащая селективный ген фосфинотрицин-N-ацетилтрансферазы (bar), который обеспечивает устойчивость к фосфинотрицину и репортерный ген β-глюкуронидазы (GUS). Способ получение и анализ трансгенных клеточных линий описан в работе [11].
ДНК из каллусных культур выделяли по методике Делапорта [12] с определенными модификациями, концентрацию и чистоту определяли спектрофотометрически. Для амплификации ДНК конечный объем реакционной смеси составлял 25 мкл: 10 мM TRIS-HCl, 50 мM KCl, 1,5 мM хлорида магния, 2 мM каждого дезоксинуклеотидтрифосфата (дНТФ), 0,2 мкл праймера, 1 ед. акт. Taq ДНК полимеразы (Thermo Scientific) и 100-120 нг исследуемой ДНК. Амплификация проводилась по следующей программе: начальная денатурация при 94 °С 5 мин, 37 циклов (денатурация 94 °С - 30 с, отжиг 58 °С - 90 с, элонгация 72 °С - 2 мин) и финальная элонгация 72 °С 10 мин. Для проведения ПЦР был использован единичный праймер к LTR
M 1
ретротранспозона SIRE 1, имеющий следующую нуклеотидную последовательность: 5'-GCACTTATGCAAGTGGGATCAGC-3 [13]. Продукты амплификации разделяли методом электрофореза в 1,6% агарозном геле с добавлением бромистого этидия. Спектры продуктов ПЦР визуализировали под УФ лучами. Для оценки размеров ампликонов применяли маркер 100 bp DNA Ladder (Thermo Scientific). Размер продуктов ПЦР определяли с помощью пакета прикладных программ TotalLab v.2.01 (Nonlinear Dynamics) [14]. Для количественной оценки полученных результатов данные были представлены в виде бинарных матриц в которых наличие/отсутствие одинаковых по размеру ампликонов соответствовало значение 1 или 0. Проверку стабильности ПЦР реакции, осуществляли путем трехкратного повторения амплификации с одним и тем же праймером на том же растительном материале.
Известно, что в процессе совместной эволюции сформировались механизмы, защищающие геном хозяина от чужеродной транспозиции, в частности описано ее подавление на транскрипционном (сайленсинг путем метилирования ДНК) и посттранскрипционном (сайленсинг за счет РНК-интерференции) уровнях [15]. Поэтому в обычных условиях активность ретротранспозонов отсутствует или наблюдается на незначительном уровне [16]. В условиях эксперимента нам не удалось зафиксировать транспозицию у исходных и контрольных форм. В целом, у нетрансформированного первичного каллуса, а также каллуса 3-го и 6-го пассажей обнаружено 10 четких ампликонов, размером от 407 до 1360 п.н. У всех исследуемых объектов выявлен идентичный спектр продуктов ПЦР. На рис. 1 представлено по одному образцу из каждой исследуемой выборки.
-
2 3
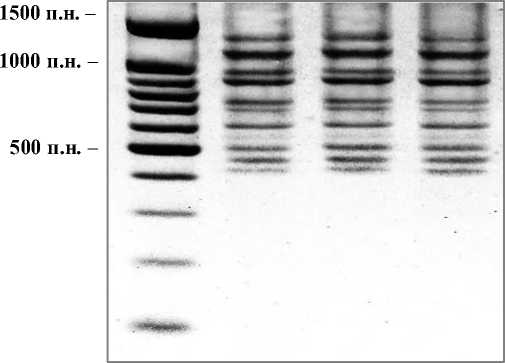
Рис. 1. Спектр продуктов амплификации ДНК контрольного нетрансформированного каллуса: М - маркер молекулярных масс (100 bp DNA Ladder), 1 – первичный каллус (0 пассаж); 2 - каллус 3 пассажа; 3 - каллус 6-го пассажа
Следующим этапом исследований было выявление полиморфизма ДНК у трансформированных форм. Для этого отбирали образцы каллусных линий LТ1-LТ7. Полученные результаты представлены на рис. 2.
М 1 2
3 4 5 6
1500 п.н. –
1000 п.н. –
500 п.н. –
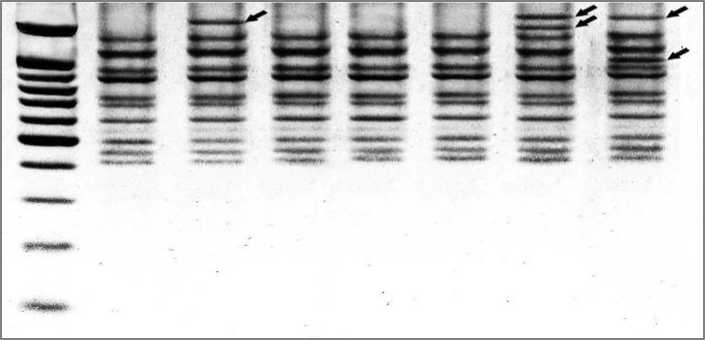
Рис. 2. Спектр продуктов амплификации ДНК трансформированных каллусных линий: М - маркер молекулярных масс (100 bp DNA Ladder), 1 - клеточная линия LТ1, 2 - клеточная линия LТ2, 3 - клеточная линия LТ3, 4 -клеточная линия LТ4, 5 - клеточная линия LТ5, 6 - клеточная линия LТ6, 7 - клеточная линия LТ7. Стрелками показаны новые ампликоны
На полученных электрофореграммах продуктов ПЦР каждый ампликон соответствует анонимному локусу между двумя близко расположенными копиями SIRE-1, поскольку применен праймер, родственный к LTR-последовательности, а общая длина этого ретротраспозона составляет около 12 тыс. п.н., что делает невозможным синтез фрагментов в пределах одной копии. Таким образом, каждый но- вый выявленный ампликон фиксирует возникновение нового межретранспозонного локуса, то есть свидетельствует о событии транспозиции. Нами отмечено появление новых, относительно высокомолекулярных (длиной более 1000 п.н.) ампликонов у трех трансгенных линий - LТ2, LТ6 и LТ7 (табл.). В частности, у двух последних обнаружено по два новых фрагмента.
Таблица. Длина новых, выявленых у трансгенных линий, ампликонов
|
Линия |
Количество новых ампликонов, шт. |
Длина амликона, п.н. |
|
LТ2 |
1 |
1567 |
|
LТ6 |
2 |
1491, 1660+* |
|
LТ7 |
2 |
1075, 1660+* |
Прим. * - При расчете длины ампликона на пакете прикладных программ TotalLab v.2.01 получено значение 1660 п.н. (линия LТ6) и 1651 п.н. (линия LТ7), однако поскольку разница между этими значениями меньше инструментальной погрешности метода (включающей разрешающую способность электрофоретического метода и погрешность вычисления), фрагменты нами условно обозначены как 1660+, что может свидетельствовать как об их идентичности, так и о несхожести по нуклеотидному составу (межретранспозонному локусу)
Появление нескольких новых ампликонов, а также то, что они были обнаружены у трех из семи исследуемых клеточных линий, может свидетельствовать об относительно высокой частоте события транспозиции в ДНК трансформированных клеточных линий. В исследуемых локусах, фланкированных LTR ретротранспозона SIRE-1, нами не выявлено точечных мутаций в сайтах связывания с праймером и/или делеций, которые могли бы приводить к исчезновению ампликона в ДНК- профилях ПЦР.
Следует отметить, что в литературе была описана транспозиционная активность ретротранспозонов Tos17 у трансгенных растений риса [4] и Tag1 у арабидопсиса [17]. Авторы также отмечают отсутствие такой активности у контрольных форм. Хотя использованный нами метод не позволяет непосредственно выявить транскрипционную и транспозиционную активность ретротранспозонов, однако он может быть быстрым и эффективным инструментом исследования изменений генома при генетической трансформации и подтверждения транспозиции МГЭ.
Таким образом, нами проведен молекулярногенетический анализ трансгенных клеточных линий мягкой пшеницы с использование IRAP ПЦР с праймером к LTR последовательности ретротранспозона SIRE 1. У трех трансформированных линий
-
- LТ2, LТ6 и LТ7 обнаружено появление новых, относительно высокомолекулярных (длиной более 1000 п.н.) ампликонов, что свидетельствует о транспозиции данного мобильного генетического элемента. Поскольку эти фрагменты отсутствовали в спектрах продуктов амплификации ДНК контрольного нетрансформированного каллуса, это дает основания считать индукцию транспозиции SIRE 1 связанной с геномным стрессом, вызванным встраиванием чужеродной ДНК (векторной конструкции pAHC25) или стрессом непосредственно связанным с процессом трансформации (микропоранение, культивирование на селективных средах).
-
1. Clive J. Global Status of Commercialized Biotech // GM Crops. 2011. ISAAA Brief. N. 43. P. 1-8.
-
2. Rakszegi M., Tamas S., Szucs P. et al. Current status of wheat transformation // J. Plant Biotechnology. 2001. V. 3. P. 67-81.
-
3. Gaspar T., Franck T., Bisbis B. et al. Concepts in plant stress physiology. Application to plant tissue cultures // Plant Growth Regul. 2002. V. 37. P. 263-285.
-
4. Wu R., Guo W.L., Wang X.R., et al. Unintended consequence of plant transformation: biolistic transformation caused transpositional activation of an endogenous retrotransposon Tos17 in rice ssp. japonica cv. Matsumae // Plant Cell Reports. 2009. V. 28. N 7. P. 1043-1051.
-
5. Lu B.R., Snow A.A. Gene flow from genetically modified rice and its environmental consequences // Bio. Sci. 2005. V. 55. P. 669–678.
-
6. Muller K., Heller H., Doerfier W . Foreign DNA integration. Genome-wide perturbations of methylation and transcription in the recipient genomes // J. Biol. Chem. 2001. V. 276. P. 14271-14278.
-
7. Matzke M.A., Mette M.F., Matzke A.J.M. Transgene silencing by the host genome defense: implications for the evolu-
tion of epigenetic control mechanisms in plants and vertebrates // Plant Mol. Biol. 2000. V. 43. P. 401-415.
-
8. Kumar A., Bennetzen J.L. Plant retrotransposons // Annu. Rev. Genet. 1999. V. 33. P. 479-532.
-
9. Бавол А.В., Дубровна О.В., Лялько І.І. Регенерація рослин із експлантів верхівки пагона проростків пшениці // Вісник Українського товариства генетиків і селекціоне-рів. 2007. Т .5. № 1-2. С. 3-10.
-
10. Christensen A.H., Quail P.H. Ubiquitin promoter-based vectors for high-level expression of selectable and/or screenable marker genes in monocotyledonous plants // Transgenic Res. 1996. V .5. N 3. Р. 213-218.
-
11. Бавол А.В., Моргун Б.В. Нітовська І.О., та ін. Генетична трансформація пшениці з використанням калюсних культур, отриманих з апікальної меристеми пагона // Дося-гнення і проблеми генетики, селекції та біотехнології: збірник наук. праць. 2012. Т .4. С. 411-416.
-
12. Delporte F., Mostadel O., Jacquemin J. Plant regeneration through callus initiation from thin mature embryo fragments of wheat // Plant Cell, Tissue and Organ Culture. 2001. V. 67. N 2. P. 73-80.
-
13. Календарь Р.В., Глазко В.И. Типы молекулярногенетических маркеров и их применение // Физиология и биохимия культурных растений. 2002. Т. 34. № 4. С. 279-296.
-
14. Nei M., Li W.H . Mathematical model for studying genetic variation in terms of restriction endonucleases // Proc. Natl. Acad. Sci. 1979. V. 76. Р. 5269-5273.
-
15. Casacuberta J.M., Santiago N. Plant LTR-retrotransposons and MITEs: control of transposition and impact on the evolution of plant genes and genomes // Gene. 2003. V. 311. P. 1-11.
-
16. Vicient C.M. Transcriptional activity of transposable elements in maize // BMC Genomics. 2010. V. 11. N. 601. P. 1-10.
-
17. Bhatt A.M., Lister C., Crawford N., Dean C. The transposition frequency of Tag1 elements is increased in transgenic Arabidopsis lines // Plant Cell. 1998. V. 10. P. 427–434.
ANALYSIS OF TRANAGENIC CELL LINES OF BREAD WHEAT USING IRAP PCR
Список литературы Молекулярно-генетический анализ трансгенных клеточных линий мягкой пшеницы с использованием IRAP ПЦР
- Clive J. Global Status of Commercialized Biotech//GM Crops. 2011. ISAAA Brief. N. 43. P. 1-8.
- Rakszegi M., Tamas S., Szucs P. et al. Current status of wheat transformation//J. Plant Biotechnology. 2001. V. 3. P. 67-81.
- Gaspar T., Franck T., Bisbis B. et al. Concepts in plant stress physiology. Application to plant tissue cultures//Plant Growth Regul. 2002. V. 37. P. 263-285.
- Wu R., Guo W.L., Wang X.R., et al. Unintended consequence of plant transformation: biolistic transformation caused transpositional activation of an endogenous retrotransposon Tos17 in rice ssp. japonica cv. Matsumae//Plant Cell Reports. 2009. V. 28. N 7. P. 1043-1051.
- Lu B.R., Snow A.A. Gene flow from genetically modified rice and its environmental consequences//Bio. Sci. 2005. V. 55. P. 669-678.
- Muller K., Heller H., Doerfier W. Foreign DNA integration. Genome-wide perturbations of methylation and transcription in the recipient genomes//J. Biol. Chem. 2001. V. 276. P. 14271-14278.
- Matzke M.A., Mette M.F., Matzke A.J.M. Transgene silencing by the host genome defense: implications for the evolution of epigenetic control mechanisms in plants and vertebrates//Plant Mol. Biol. 2000. V. 43. P. 401-415.
- Kumar A., Bennetzen J.L. Plant retrotransposons//Annu. Rev. Genet. 1999. V. 33. P. 479-532.
- Бавол А.В., Дубровна О.В., Лялько I.I. Регенерацiя рослин iз експлантiв верхiвки пагона проросткiв пшеницi//Вiсник Українського товариства генетикiв i селекцiонерiв. 2007. Т.5. № 1-2. С. 3-10.
- Christensen A.H., Quail P.H. Ubiquitin promoter-based vectors for high-level expression of selectable and/or screenable marker genes in monocotyledonous plants//Transgenic Res. 1996. V.5. N 3. Р. 213-218.
- Бавол А.В., Моргун Б.В. Нiтовська I.О., та iн. Генетична трансформацiя пшеницi з використанням калюсних культур, отриманих з апiкальної меристеми пагона//Досягнення i проблеми генетики, селекцiї та бiотехнологiї: збiрник наук. праць. 2012. Т.4. С. 411-416.
- Delporte F., Mostadel O., Jacquemin J. Plant regeneration through callus initiation from thin mature embryo fragments of wheat//Plant Cell, Tissue and Organ Culture. 2001. V. 67. N 2. P. 73-80.
- Календарь Р.В., Глазко В.И. Типы молекулярно-генетических маркеров и их применение//Физиология и биохимия культурных растений. 2002. Т. 34. № 4. С. 279-296.
- Nei M., Li W.H. Mathematical model for studying genetic variation in terms of restriction endonucleases//Proc. Natl. Acad. Sci. 1979. V. 76. Р. 5269-5273.
- Casacuberta J.M., Santiago N. Plant LTR-retrotransposons and MITEs: control of transposition and impact on the evolution of plant genes and genomes//Gene. 2003. V. 311. P. 1-11.
- Vicient C.M. Transcriptional activity of transposable elements in maize//BMC Genomics. 2010. V. 11. N. 601. P. 1-10.
- Bhatt A.M., Lister C., Crawford N., Dean C. The transposition frequency of Tag1 elements is increased in transgenic Arabidopsis lines//Plant Cell. 1998. V. 10. P. 427-434.


