Молекулярно-генетический мониторинг ассоциированной микрофлоры лососевидных рыб: разнообразие и физиологический статус
Автор: Белькова Н.Л., Суханова Е.В., Деникина Н.Н., Русинек О.Т., Дзюба Е.В.
Журнал: Известия Самарского научного центра Российской академии наук @izvestiya-ssc
Рубрика: Мониторинг
Статья в выпуске: 1-4 т.12, 2010 года.
Бесплатный доступ
Актуальность внедрения молекулярно-генетического мониторинга ассоциированной микрофлоры рыб предусматривает развитие молекулярной диагностики заболеваний и обусловлена в первую очередь угрозой широкого распространения бактериальных, вирусных и грибковых инфекций в условиях искусственного содержания. Разработка и внедрение эффективной методики селективной детекции живых и жизнеспособных бактериальных клеток на основе направленного ингибирования внеклеточной ДНК позволит быстро и корректно выявлять возбудителя инфекционных заболеваний рыб и контролировать эффективность проведения санитарно- эпидемиологических мероприятий. Предложен и апробирован на лососевидных рыбах из аквариумных экспозиций Байкальского музея ИНЦ СО РАН метод селективной инактивации ДНК отмерших клеток организма-хозяина и бактерий с использованием моноазида этидиумбромида (ЕМА). Подобраны условия выделения ДНК, в групп- и видо-специфичных ПЦР получены наборы целевых ампликонов, проведена серия экспериментов с лабораторной культурой штамма E. coli. Установлено, что при низких концентрациях анавидин является эффективным лизирующим реагентом и на его основе возможно создание нового набора для выделения тотальной ДНК. Предлагаемая методика будет востребована при разработке современных наукоемких технологий молекулярно-генетического мониторинга водных экосистем.
Лососевидные рыбы, микрофлора рыб, бактериальные инфекции, молекулярно-генетические методы, аквариумные экспозиции
Короткий адрес: https://sciup.org/148199093
IDR: 148199093 | УДК: 576.8.095.38+504.062:639.2.03
Текст научной статьи Молекулярно-генетический мониторинг ассоциированной микрофлоры лососевидных рыб: разнообразие и физиологический статус
Использование новых, современных технологий в профилактике и терапии заболеваний рыб позволит существенно снизить экономические потери, вызванные их массовой гибелью. Методы диагностики бактерии-альных инфекций на основе видоспецифичной полимеразной цепной реакции (ПЦР-диагнос-тика) показали свое преимущество перед классическими микробиологическими методами [3] и могут стать основой молекулярно-генетического мониторинга ассоциированной микрофлоры водных организмов. Они требуют меньше времени и эффективны в детекции конкретных патогенов на всех стадиях заболевания. ПЦР-диагностика позволяет корректно идентифицировать микроорганизмы, культивирование и определение которых по классическим биохимическим тестам затруднительно. Эти методы активно используется в странах с развитой аквакультурой, постоянно совершенствуются и развиваются [5, 8, 9, 19, 20].
Известно, что бактериальные клетки имеют различный физиологический статус: живые; жизнеспособные, но некультивируемые;
резистентные – споры; мертвые. Определение жизнеспособности клеток с использованием молекулярно-генетических методов имеет принципиальное значение в медицинской, ветеринарной и пищевой микробиологии. Методы микроскопии и проточной цитометрии с красителями, позволяющими различать живые и мертвые клетки, такие как Syto 9 или BacLight [6, 13, 17], существенно менее чувствительны, чем полимеразная цепная реакция (ПЦР) или подсчет колониеобразующих единиц (КОЕ) [17]. Применение ОТ-ПЦР для детекции живых клеток [4, 16] также имеет ряд недостатков, как в корректности дифференцировки живых и мертвых клеток при использовании мРНК в качестве маркерных молекул, так и в воспроизводимости результатов анализа [12, 18]. В связи с этим становится актуальной разработка метода дифференцировки ДНК из живых и мертвых бактериальных клеток, что позволит полностью использовать потенциал ПЦР-диагнос-тики [11]. Лабораторные эксперименты по селективному различению ДНК из живых и мертвых бактериальных клеток в настоящее время проводятся с применением производных этидиумбромида [7, 10, 11, 15].
Цель работы – разработка эффективной методики селективной детекции живых и жизнеспособных бактериальных клеток на основе направленного ингибирования внеклеточной ДНК, позволяющей проводить молекулярногенетический мониторинг ассоциированной (в том числе и патогенной) микрофлоры рыб.
Материалы и методы. Исследования выполнены на здоровых и больных (с язвами на внешних покровах) байкальских рыбах: ленке ( Brachymystax lenok), байкальском сиге ( Corego-nus lavaretus baicalensis ) и черном байкальском хариусе ( Thymallus baicalensis ) из аквариумной экспозиции Байкальского музея ИНЦ СО РАН.
Отбор биологического материала и первичная обработка проб. Соскобы с внешних покровов живых рыб собирали стерильным скальпелем в пробирки эппендорф и добавляли от 100 мкл до 1 мл трис-солевого буфера в зависимости от объема биологического материала (ТСБ: 10 мМ трис-HCl, pH 7,5; 0,1 M NaCl; 2 мМ ЭДТА). Проводили три эксперимента по определению оптимальных условий первичной обработки бактериального материала.
-
(1) Определение оптимальных условий фиксации биологического материала для его транспортировки в лабораторию. Для этого суспензию клеток в ТСБ или фиксировали
этанолом до конечной концентрации 70%, или использовали без фиксации, после чего обрабатывали лизирующим раствором из коммерческих наборов (ДНК-сорб, АмплиСенс, Москва).
-
(2) Оценка возможности использования анавидина для эффективного лизиса клеточной биомассы. Для этого лизис образцов проводили в растворах с понижающими концентрациями анавидина, лизат использовали для выделения ДНК (табл. 1).
-
(3) Обработка биологического материала моноазидом этидиумбромида (ЕМА) для удаления внеклеточной ДНК. Для этого к 100 мкл суспензии добавляли 2 мкл ЕМА (50 мг/мл), инкубировали во льду 1 мин при освещении 650 вт. Выдерживали в темноте 20 мин.
Все эксперименты были проведены в трех повторностях. Суспензии клеток использовали для выделения тотальной ДНК методом ферментативного лизиса, а лизаты – с помощью коммерческих сорбентов.
Таблица 1 . Схема эксперимента по использованию раствора анавидина
|
Концентрация анавидина |
Дополнительный лизирующий агент |
Резуль таты амплифи кации |
|
0,01% Ана 1* |
– |
– |
|
0,02% Ана 1 |
– |
– |
|
0,05% Ана 1 |
– |
– |
|
0,1% Ана 1 |
– |
– |
|
0,01% Ана 1 |
1% SDS |
+ |
|
0,02% Ана 1 |
1% SDS |
+ |
|
0,05% Ана 1 |
1% SDS |
+ |
|
0,1% Ана 1 |
1% SDS |
– |
|
0,01% Ана 2** |
– |
+ |
|
0,02% Ана 2 |
– |
– |
|
0,05% Ана 2 |
– |
– |
|
0,1% Ана 2 |
– |
– |
|
0,01% Ана 2 |
1% SDS |
+ |
|
0,02% Ана 2 |
1% SDS |
+ |
|
0,05% Ана 2 |
1% SDS |
+ |
|
0,1% Ана 2 |
1% SDS |
– |
|
– |
1% SDS |
– |
|
– |
ферментативный лизис |
+ |
Примечания : * - Анавидин 1 – товарный продукт; ** - Анавидин 2 – № партии 16-07 МК-6
Выделение ДНК методом ферментативного лизиса проводили по [1].
Выделение ДНК с помощью коммерческих сорбентов. После обработки образцов лизирующими растворами, их прогревали 10 мин при 60°С, центрифугировали 15 мин на настольной центрифуге (12000 об/мин), надосадочную жидкость переносили в новую пробирку. К прозрачному лизату добавляли 25 мкл сорбента универсального (АмплиСенс, Москва). Суспензию тщательно перемешивали на вортексе, выдерживали при комнатной температуре 5 мин. Центрифугировали 30 сек при 12000 об/мин, супернатант удаляли, а осадок промывали 400 мкл 70% этанола и подсушивали 30 мин при 60°С. Нуклеиновые кислоты элюировали в 25 мкл ТЕ буфера и использовали в качестве матрицы в ПЦР.
Полимеразная цепная реакция. В серии экспериментов подбирали режим амплификации, обеспечивающий высокое качество ПЦР-продукта требуемой длины на праймерах, комплементарных участкам гена 16S рРНК бактерий с разным уровнем специфичности (табл. 2) по [1]. Для этого проверяли температуру отжига в градиенте температур от 44 до 72°С в амплификаторе DNA Engine DYADTM. Анализ продуктов проводили разделением фрагментов ДНК в 1,5% агарозном геле.
Таблица 2. Структуры праймеров, использованных в работе
|
Название праймера |
Структура 5'–3' |
Детектируемая филогенетическая группа |
|
Консервативные прайме |
ры |
|
|
21L |
TTCCGGTTGATCCYGCCGGA |
архебактерии |
|
109L |
ACKGCTCAGTAACACGT |
архебактерии |
|
333L |
TCCAGGCCCTACGGG |
архебактерии |
|
344L |
ACGGGGYGCAGCAGGCGCGA |
архебактерии |
|
915R |
GTGCTCCCCCGCCAATTCCT |
архебактерии |
|
958R |
YCCGGCGTTGAMTCCAATT |
архебактерии |
|
1492R |
GGCTACCTTGTTACGACTT |
архебактерии |
|
500L |
CGTGCCAGCAGCCGCGGTAA |
эубактерии |
|
1350R |
GACGGGCGGTGTGTACAAG |
эубактерии |
|
338L |
ACTCCTACGGGAGGCAGCAG |
эубактерии |
|
500R |
TTACCGCGGCTGCTGGFACG |
эубактерии |
|
NS8 |
TCCGCAGGTTCACCTACGGA |
эукариоты |
|
Групп-специфичные праймеры |
||
|
35L |
CTGGCTCAGAYCGAACG |
альфа-протеобактерии |
|
681L |
AGTGTAGAGGTGAAATT |
альфа-протеобактерии |
|
663L |
GAATTCCATCCCCCTCT |
бета-протеобактерии |
|
680L |
CRCGTGTAGCAGTGA |
бета-протеобактерии |
|
319L |
GTACTGAGACACGGACCA |
цитофаги-флавобактерии |
|
342L |
CAGCAGTAGGGAATCTTC |
бациллы |
|
31L |
GATCCTGGCTCAGAATC |
ацидобактерии |
|
235L |
CGCGGCCTATCAGCTTGTTG |
актинобактерии |
|
930R |
CTCCACCGCTTGTGTGA |
планктомицеты |
|
NS1 |
GTAGTCATATGCTTGTCTC |
Saprolegniaceae |
|
Видо-специфичные праймеры |
||
|
АН-F |
GAAAGGTTGATGCCTAATACGTA |
Aeromonas hydrophila |
|
AH-R |
CGTGCTGGCAACAAAGGCCAG |
Aeromonas hydrophila |
|
PAF |
GACCTCGCGCCATTA |
Pseudomonas anguilliseptica |
|
PAR |
CTCAGCAGTTTTGAAAG |
Pseudomonas anguilliseptica |
|
BF1 |
TCACCAAGGCRACGATGCG |
Bacillus licheniformis |
|
BR2 |
CGTATTCACCGCGGCATG |
Bacillus licheniformis |
|
650L |
TAGAGTATGGGAGAGGAT |
Acinetobacter calcoaceticus |
Результаты и обсуждение. Для разработки эффективной методики, позволяющей проводить молекулярно-генетический мониторинг ассоциированной микрофлоры рыб, важно оценить применение различных способов фиксации бактериального материала и выделения тотальной ДНК. Нами были использованы как общепринятые методики: фиксация этанолом и выделение ДНК методом ферментативного лизиса, так и апробирована возможность применения новых химических реагентов, лизирующих клеточные стенки бактерий и денатурирующих внутриклеточные белки. Для этой цели впервые был использован эффективный антисептик – ана-видин (табл. 1), разработанный на основе производных гуанидина в Иркутском институте химии СО РАН (патент РФ N2144024). Качество полученной ДНК проверяли с помощью ПЦР на консервативных бактериальных праймерах (табл. 2). Установлено, что оптимальной для лизиса клеток является концентрация анавидина 0,01% с добавлением додецилсульфата натрия до конечной концентрации 1% (рис. 1).
Ранее нами показано, что при использовании рибосомной ДНК в качестве мишени в амплификации на консервативных бактериальных праймерах образуются неспецифичные ПЦР-продукты, соответствующие фрагменту гена 18S рРНК исследуемого вида рыб [2]. Загрязнение ДНК организма-хозяина при проведении групп- и видоспецифичной амплификации на бактериальных праймерах может приводить к ложно положительному результату. Поэтому для дальнейшей разработки методики использованы праймеры с широким уровнем специфичности (табл. 2). Установлено, что с матричных ДНК получается спектр целевых ампликонов, а неспецифичные могут синтезироваться с ДНК организма-хозяина (рис. 2). Повышение температуры отжига в некоторых случаях не увеличивает специфичность амплификации, а приводит к полному отсутствию ПЦР-продукта – ложно отрицательному результату. Поэтому на следующем этапе была предпринята попытка селективной амплификации ДНК из живых бактериальных клеток.
При проведении молекулярно-генетического мониторинга ассоциированной микрофлоры рыб важно иметь достоверную информацию: из живых или мертвых бактериальных клеток получен ампликон. Актуальность проведения такой диагностики вызвана следующими факторами: 1) в пробах, взятых с внешних покровов рыб, особенно с травмированных участков тела, присутствуют как бактериальные, так и мертвые (лизированные) клетки организма-хозяина; 2) специфика профилактики и лечения бактериальных инфекций заключается в регулярной обработке аквариумов дезинфицирующими средствами, разрушающими клетки микроорганизмов. Известно, что некоторые производные этиди-умбромида не способны проникать через клеточную стенку живых и жизнеспособных клеток, однако они встраиваются в двойную цепь внеклеточной и находящейся в мертвых или частично разрушенных клетках ДНК и способствуют ее разрушению при УФ-облучении или освещении с мощностью более 600 вт. Так-им образом, обработка биологического материала производным этидиумбромида – моноазидом (ЕМА) с последующим кратковременным световым воздействием приводит к селективной инактивации внеклеточной ДНК [14, 21].
М <1.01 02 0.05 0J М Р a.0t О1 0.05 0.1
* . ' ■■■■■■ Н l%SDS! ! BireenTHW 2
1400 п.н. .
1000 п.н. — w ■* — . ... -
500 п.н. , .
Рис. 1. Электрофоретический анализ результатов амплификации тотальной ДНК из соскоба внешних покровов с язвенными проявлениями байкальского сига: М – маркер молекулярного веса, Р – положительный контроль – ДНК выделена методом ферментативного лизиса, в остальных случаях указаны концентрации анавидина 1 и 2 (%, об/об)
Ml 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 М
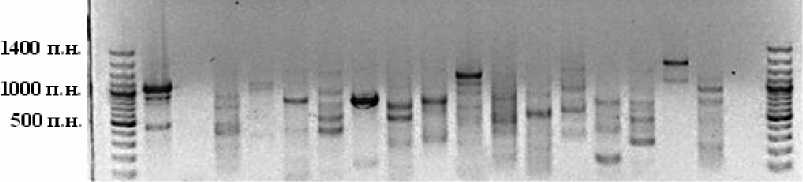
Рис. 2. Электрофоретический анализ результатов амплификации тотальной ДНК, выделенной из соскоба внешних покровов с язвенными проявлениями байкальского сига на парах праймеров:
1 – 500L-1350R, 2 – 21L-1492R, 3 – 109L-915R, 4 – 333L-958R, 5 – 344L-958R, 6 – 35L-500R, 7 – 681L-1350R, 8 – 663L-1350R, 9 – 680L-1350R, 10 – 319L-1350R, 11 – 338L-930R, 12 – 235L-1350R, 13 – 31L-1350R, 14 – BF1-BR2, 15 – 342L-1350R, 16 – NS8-NS1, 17 – 650L-1350R, 18 – безматричный контроль на паре 500L-1350R, М – маркер молекулярного веса
Мы использовали моноазид этидиум-бромида для селективной инактивации внеклеточной ДНК, а так же ДНК из мертвых клеток организма-хозяина и (или) бактерий. При этом вся процедура дополнительной обработки занимает несколько минут (обработка светом идет в течение 90 сек). В экспериментах использовали соскобы с внешних покровов рыб (рис. 3). Следует отметить не только полное исчезновение неспецифичного ПЦР-продукта и, следовательно, ложно положительного ампликона, с рибосомной ДНК организма-хозяина (рис. 3А), но и специфичную детекцию таких организмов как представители рода Bacillus и Acinetobacter calco-aceticus . ПЦР-продукты на этих специфичных праймерах удалось получить только с препаратов ДНК, полученных после дополнительной обработки ЕМА (рис. 3Б, В).
Очевидно, что соотношение ДНК: ЕМА на стадии пробоподготовки биологического материала необходимо оптимизировать для полного ингибирования ДНК как в мертвых клетках, так и внеклеточной. Проведены предварительные эксперименты по поиску оптимальных концентраций суспензии клеток для их разрушения или фиксации на примере культуры грамотрицательных бактерий Escherichia coli. Результаты показали, что фиксация или лизис клеточной суспензии, титр клеток в которой меньше 1–2×105 кл./мл, с последующей обработкой ЕМА приводят к полному ингибированию тотальной ДНК и ампликоны на праймерах с разным уровнем специфичности получить не удается. При титре клеток выше 1×106 кл./мл обработка суспензии клеток ЕМА после их лизиса не влияет на получение ПЦР-продукта. По-видимому, соотношение ДНК:ЕМА в последнем случае не является оптимальным для полного ингибирования ДНК в мертвых клетках. Это соотношение необходимо учитывать при оценке оптимального для проведения диагностического молекулярно-генетического анализа количества биологического материала. Для этого следует провести дополнительные эксперименты по определению концентрацией ЕМА, требуемых для ингибирования внеклеточной ДНК, при обработке микроорганизмов с разным типом клеточной стенки.
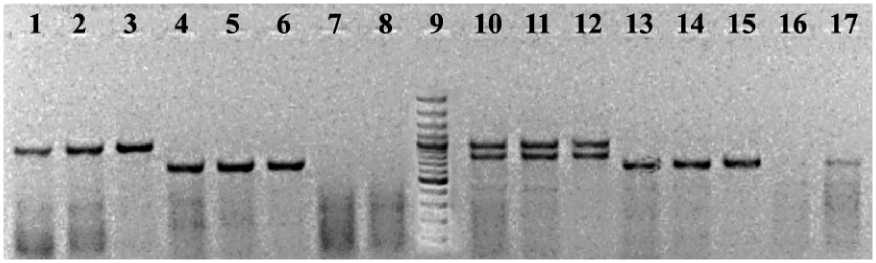
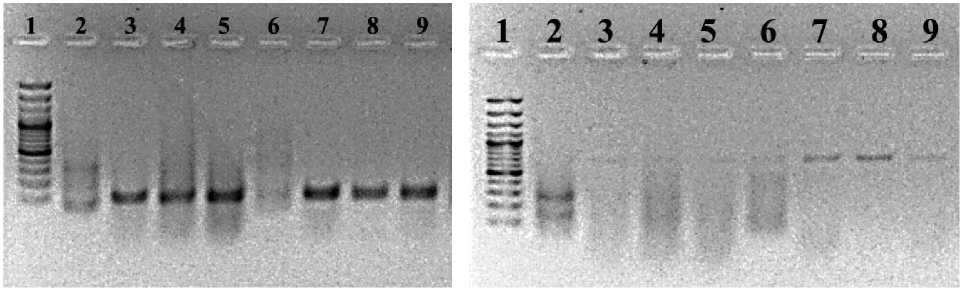
Рис. 3. Электрофоретический анализ результатов амплификации тотальной ДНК, выделенной из соскобов с внешних покровов байкальского сига:
(А) Пары праймеров 500L-1350R (1,2,3,10,11,12), ALF-1350R (4,5,6,13,14,15), BET-1350R (7,8,16,17), 9 – маркер молекулярного веса. 1-8 – суспензия клеток обработана ЕМА, 10-17 – суспензия клеток без обработки ЕМА. (Б) Пара праймеров специфичных для представителей рода Bacillus , 1 – маркер молекулярного веса, 2,6 – суспензия клеток без обработки, 3,4,5,7,8,9 – суспензия клеток обработана ЕМА. (В) Пара видоспецифичных праймеров для Acinetobacter calcoaceticus , 1 – маркер молекулярного веса, 2,6 – суспензия клеток без обработки, 3,4,5,7,8,9 – суспензия клеток обработана ЕМА
На следующем этапе была проведена диагностика бактериальных инфекций лососевидных рыб, вызываемых Aeromonas hydro-phila и Pseudomonas anguilliseptica , в условиях аквариумной экспозиции Байкальского музея ИНЦ СО РАН на основе видо-специфичной амплификации. Апробацию метода и подбор условий видо-специфичной амплификации проводили для анализа соскобов с внешних покровов исследуемых видов рыб. Ампликоны были получены с препаратов ДНК из образцов биологического материала, обработанного ЕМА, взятого от рыб с явными проявлениями язвенных заболеваний.
Выводы: разработанная методика позволит решить проблему селективной детекции живых бактериальных клеток для корректного выявления возбудителей инфекционных заболеваний рыб и найдет применение в системе молекулярно-генетического мониторинга водных экосистем.
Работа поддержана инновационным проектом «Тест-системы для диагностики бактериальных инфекций рыб» 2008 г. ИНЦ СО РАН и выполнена в рамках программы РАН №26, подпрограммы 1, проект 26.13.
Список литературы Молекулярно-генетический мониторинг ассоциированной микрофлоры лососевидных рыб: разнообразие и физиологический статус
- Белькова, Н.Л. Введение в молекулярную экологию микроорганизмов: Учебно-методическое пособие/Н.Л. Белькова, А.М. Андреева. -Ярославль: Изд-во ООО «Принтхаус», 2009. -91 с.
- Белькова, Н.Л. Адаптация методов молекулярно-генетического анализа для изучения микроорганизмов, ассоциированных с рыбами/Н.Л. Белькова, Е.В. Дзюба, Е.В. Суханова, Т.А. Ханаева//Биология внутренних вод. -2008. -№2. -С. 91-94.
- Белькова, Н.Л. Молекулярно-генетическая идентификация кишечной микрофлоры и протистов байкальских рыб/Н.Л. Белькова, Е.В. Дзюба, Е.В. Суханова//Аннотированный список фауны озера Байкал и его водосборного бассейна: в 2 томах. -Новосибирск: Наука, 2009. -Т.II: Водоемы и водотоки юга Восточной Сибири и Северной Монголии/О.А. Тимошкин, В.И. Провиз, Т.Я. Ситникова и др. -(Справочники и определители по фауне и флоре озера Байкал). -С. 957-980.
- Bentsink, L. Amplification of RNA by NASBA allows direct detection of viable cells of Ralstonia solanacearum in potato/L. Bentsink, G.O. Leone, J.R. van Beckhoven et al.//J. Appl. Microbiol. -2002. -V. 93. -P. 647-655.
- Buller, N.B. Bacteria from fish and other aquatic animals: a practical identification manual. -Oxfordshire: CABI publishing, 2004. -361 p.
- Burnett, S.L. Comparison of methods for fluorescent detection of viable, dead, and total Escherichia coli O157:H7 cells in suspensions and on apples using confocal scanning laser microscopy following treatment with sanitizers/S.L. Burnett, L.R. Beuchat//Int. J. Food Microbiol. -2002. -V. 74. -P. 37-45.
- Flekna, G. Insufficient differentiation of live and dead Campylobacter jejuni and Listeria monocytogenes cells by ethidium monoazide (EMA) compromises EMA/real-time PCR/G. Flekna, P. Stefanic, M. Wagner et al.//Res. Microbiol. -2007. -V. 158. -P. 405-412.
- Haygood, M.G. Light organ symbioses in fishes//Crit. Rev. Microbiol. -1993. -V. 19. -P. 191-216.
- Haygood, M.G. Polymerase chain-reaction and 16S-ribosomal-RNA gene-sequences from the luminous bacterial symbionts of 2 deep-sea anglerfishes/M.G. Haygood, D.L. Distel, P.J. Herring//J. Mar. Biol. Assoc. U.K. -1992. -V. 72. -P. 149-159.
- Lee, J.-L. Quantification of total viable bacteria on fish fillets by using ethidium bromide monoazide real-time polymerase chain reaction/J.-L. Lee, R.E. Levin//Int. J. Food Microbiol. -2007. -V. 118. -P. 312-317.
- Lee, J.-L. A comparative study of the ability of EMA and PMA to distinguish viable from heat killed mixed bacterial flora from fish fillets/J.-L. Lee, R.E. Levin//J. Microbiol. Methods. -2009. -V. 76. -P. 93-96.
- McKillip, J.L. rRNA stability in heat killed and UV-irradiated enterotoxigenic Staphylococcus aureus and Escherichia coli O157:H7/J.L. McKillip, L.A. Jaykus, M. Drake//Appl. Environ. Microbiol. -1998. -V. 64. -P. 4264-4268.
- Michel, C. Production of viable cultures of Flavobacterium psychrophilum: approach and control/C. Michel, D. Antonio, R.P. Hedrick//Res. Microbiol. -1999. -V. 150. -P. 351-358.
- Nocker, A. Selective removal of DNA from dead cells of mixed bacterial communities by use of ethidium monoazide/A. Nocker, A.K. Camper//Appl. Environ. Microbiol. -2006. -V. 72. -P. 1997-2004.
- Nocker, A. Selective detection of live bacteria combining propidium monoazide sample treatment with microarray technology/A. Nocker, A. Mazza, L. Masson et al.//J. Microbiol. Methods. -2009. -V. 76. -P. 253-261.
- Novak, J.S. Detection of heat injury in Listeria monocytogenes Scott A/J.S. Novak, V.K. Juneja//J. Food Prot. -2001. -V. 64. -P. 1739-1743.
- Rudi, K. Use of ethidium monoazide and PCR in combination for quantification of viable and dead cells in complex samples/K. Rudi, B. Moen, S.M. Dromtorp, A.L. Holck//Appl. Environ. Microbiol. -2005. -V. 71. -P. 1018-1024.
- Sheridan, G.E. Detection of mRNA by reverse transcription-PCR as an indicator of viability in Escherichia coli cells/G.E. Sheridan, C.I. Masters, J.A. Shallcross, B.M. MacKey//Appl. Environ. Microbiol. -1998. -V. 64. -P. 1313-1318.
- Spanggaard, B. The microflora of rainbow trout intestine: a comparison of traditional and molecular identification/B. Spanggaard, I. Huber, J. Nielsen et al.//Aquaculture. -2000. -V. 182. -P. 1-15.
- Van der Maarel, M.J.E.C. Detection of methanogenic archaea in seawater particles and the digestive tract of a marine fish species/M.J.E.C. Van der Maarel, W. Sprenger, R. Haanstra, L.J. Forney//FEMS Microbiol. Lett. -1999. -V. 173. -P. 189-194.
- Wagner, A.O. Removal of free extracellular DNA from environmental samples by ethidium monoazide and propidium monoazide/A.O. Wagner, C. Malin, B.A. Knapp, P. Illmer//Appl. Environ. Microbiol. -2008. -V. 74. -P. 2537-2539.


