Молекулярно-генетическое исследование рода heracleum: теоретические и прикладные аспекты
Автор: Шипилина Л. Ю., Чухина И.Г., Бажанова О.В., Семилет Т.В., Стрыгина К.В., Швачко Н.А., Хлесткина Е.К.
Журнал: АгроЗооТехника @azt-journal
Рубрика: Общее земледелие и растениеводство
Статья в выпуске: 4 т.8, 2025 года.
Бесплатный доступ
Цель работы – выявление филогенетических отношений внутри рода Heracleum, оценка возможности применения полученных знаний для решения практических задач, в частности уточнения объема сорных видов борщевиков. Материалом послужили 102 образца рода Heracleum, собранные в европейской части России (от территории Северного Кавказа до Заполярья), и 14 гербарных образцов различных видов борщевика. Анализ полиморфизма ДНК осуществляли при помощи ISSR-маркеров, RAPD-маркеров, а также на основе секвенирования района ITS-2. Данный анализ привнес ясность в аспекты видового разнообразия борщевиков на исследованных территориях. Благодаря анализу базы данных NCBI и секвенированию области ITS-2 образцов изучаемых популяций Heracleum установлено, что большинство образцов являются родственными группе гигантских борщевиков. Выделен кластер образцов, генетически близких к борщевику Сосновского, разделяющихся на два субкластера, из которых один, вероятно, содержит межвидовые гибриды, а другой – образцы, сходные именно с H. sosnowskyi. Оценочно, последние составляют менее 30%. В результате проведенного RAPD-анализа показано, что среди изучаемых представителей рода Heracleum наблюдается высокая генетическая разнородность гигантских борщевиков на Северо-Западе Европейской части России и склонность их к образованию межвидовых гибридов. Полученные данные указывают на то, что без тщательного анализа, вплоть до идентификации на уровне ДНК, участки засорения растениями гигантских борщевиков могут быть ошибочно отнесены к территориям распространения борщевика Сосновского. Для идентификации «спорных» образцов предложен метод экспресс-проверки (как возможный арбитражный метод) с использованием высоко полиморфного ISSR-маркера. Представлен конкретный кейс проверки на наличие засорения борщевиком H. sosnowskyi территории, подлежащей контролю распространения данного вида, показавший отсутствие H. sosnowskyi среди «спорных» образцов борщевика.
Heracleum, ISSR, ITS, RAPD, борщевик, кластерный анализ, молекулярные маркеры, филогенетический анализ, сорные растения
Короткий адрес: https://sciup.org/147252064
IDR: 147252064 | УДК: 632.51:58.002 | DOI: 10.15838/alt.2025.8.4.3
Текст научной статьи Молекулярно-генетическое исследование рода heracleum: теоретические и прикладные аспекты
Гигантские борщевики, в число которых входят борщевик Сосновского ( Heracleum sosnowskyi Manden.) и борщевик Манте-гацци ( H. mantegazzianum Somm. et Lev.), в последние десятилетия активно расселяются за пределы своего естественного ареала, находящегося на территории Кавказа, Закавказья, Дагестана. В настоящее время борщевик как адвентивное растение интенсивно распространяется во многих регионах страны, вытесняя естественную растительность (Сандина, 1959; Лунева, 2014; Черная книга флоры Сибири, 2016; Афонин и др., 2017; Озерова и др., 2018).
Для ликвидации инвазионных популяций борщевика и предотвращения его экспансии на территории России необходимо всестороннее изучение аборигенного и адвентивного генофонда данного рода, в том числе исследование его генома. Секвенирование и сборка полного генома инвазионной формы борщевика ( H. sosnowskyi ) были осуществлены только в 2024 году (Schelkunov et al., 2024), в геноме борщевика выявлено необычно большое количество генов (55106), в отличие от 25–35 тысяч (норма у большинства растений, в т. ч. у близкого к борщевику вида Daucus carota , имеющего ок. 30 тыс. генов (Iorizzo et al., 2012; Lorizzo et al., 2016) при отсутствии недавних полногеномных дупликаций) (Schelkunov et al., 2024). Вероятно, геномные данные о борщевике Сосновского помогут в ближайшем будущем дополнить существующие методы борьбы с ним (Мотыль и др., 2013; Khlestkina, 2012) новыми способами противодействия.
Однако распространение инвазионных видов зачастую сопровождается межвидовой гибридизацией, образованием новых форм. Об этом свидетельствует отмечаемая в популяциях борщевика большая фенотипическая разнородность (Сандина, 1959; Лунева, 2013; Черная книга флоры Сибири, 2016). В связи с этим одних лишь сведений о геноме конкретного вида недостаточно, необходимы популяционные и филогенетические исследования на основе анализа полиморфизма ДНК. Именно этому посвящена представленная работа. Ее цель – исследовать при помощи анализа полиморфизма ДНК филогенетические отношения внутри рода Heracleum с использованием образцов разных видов из Гербария Ботанического института РАН и образцов, собранных в европейской части России от Северного Кавказа до Заполярья, а также применить полученные знания для решения практических задач. Для анализа полиморфизма ДНК мало изученных видов распространены методы ДНК-фингерпринтинга (мультилокусные ДНК-маркеры), к которым относят, среди прочих, RAPD (Random Amplification of Polymorphic DNA – случайно амплифици-руемая полиморфная ДНК) и ISSR (Inter Simple Sequence Repeats – межмикросател-литный полиморфизм) (Khlestkina, 2012). Наличие секвенирования пластидного генома H. moellendorffii (Kang et al., 2019) позволяет использовать применяемый у других таксонов способ ДНК-штрихкодирования – ITS (Internal transcribed spacer – внутренний транскрибированный спей-сер, консервативный участок генов рибосомальной РНК) (Group et al, 2011) и для разных видов борщевика (Kang et al., 2019).
Материалы и методы
Материалом для проведения исследования стали 102 образца борщевика ( Heracleum L.), из них 87 образцов, собранных в Кингисеппском районе Ленинградской области, в пригороде г. Санкт-Петербурга, на территории Кольского полуострова и на территории Кавказского заповедника в рамках исследования по договору № PO19-5179 с Nord Stream 2 AG, а также 14 гербарных образцов четырех различных видов борщевика ( H. sphondylium , H. stevenii , H. sibiricum , H. sosnowskyi ), предоставленные Ботаническим институтом РАН ( табл. 1, Приложение ). Выделение геномной ДНК из образцов свежего и гербарного материала осуществляли при помощи набора DNeasy Plant Mini Kit (QIAGEN) согласно инструкции производителя. Препарат гербарных образцов подвергался инкубации с буфером следующего состава: 100 мМ трис-HCl, pH = 8,0; 0,35 М сорбитол; 5 мМ ЭДТА; 1% PVP.
Амплификацию геномной ДНК с праймерами ОРА проводили в 20 мкл ПЦР-сме-си, содержащей 30 нг ДНК-матрицы, по 4 нг праймера, 0,25 мМ каждого dNTP, 1x реакционного буфера (67 мМ TrisHCl, pH 8,8, 2,0 мМ MgCl 2 , 18 мМ (NH 4 ) 2 SO 4 , 0,01% Твин-20) и 1U Taq полимеразы. Продукты амплификации разделяли в 2% агарозном геле.
Амплификацию геномной ДНК с ISSR-праймером проводили в 20 мкл ПЦР-смеси, содержащей 30 нг ДНК-матрицы, 4 нг праймера, 0,25 мМ каждого dNTP, 1x реакционного буфера (67 мМ TrisHCl, pH 8,8, 1,3 мМ MgCl 2 , 18 мМ (NH 4 ) 2 SO 4 , 0,01% Твин-20) и 4U Taq полимеразы. Продукты амплификации разделяли в 2% агарозном геле. Для гербарных образцов перед постановкой ПЦР применили дополнительный этап рефрагментации с помощью набора Sigma-Aldrich GenomePlex Complete Whole Genome
Amplification (WGA) Kit (Великобритания) согласно протоколу производителя. Технология обогащения фрагментов ДНК путем целевой гибридизации позволяет накопить пул фрагментов ДНК для повышения точности сборки протяженных областей генома.
Амплификацию геномной ДНК c остальными праймерами проводили в 20 мкл ПЦР-смеси, содержащей 30 нг ДНК-матрицы, по 1 нг каждого праймера, 0,25 мМ каждого dNTP, 1x реакционного буфера (67 мМ TrisHCl, pH 8,8, 1,5 мМ MgCl 2 , 18 мМ (NH 4 ) 2 SO 4 , 0,01% Твин-20) и 1U Taq полимеразы. Продукты амплификации разделяли в 1% агарозном геле.
Используемые праймеры для ПЦР и протоколы приведены в табл. Праймеры были разработаны с использованием программы Oligo Primer Analysis Software v.7 . Выделение из геля и очистку ПЦР-фраг-ментов проводили набором diaGene для элюции ДНК из агарозного геля (Диаэм) согласно протоколу производителя. Секвенирование ПЦР-фрагментов проводили по Сэнгеру.
Филогенетический анализ на основании полученных данных молекулярногенетического анализа осуществляли при помощи программ NTSYS 2.0 (при сравнении аллелей маркерных локусов) и MEGA X (при сравнении нуклеотидных последовательностей). Помимо последовательностей ITS образцов, анализируемых в настоящей работе, при построении филогенетического древа были дополнительно использованы последовательности ITS представителей рода Heracleum, выявленные в базе данных NCBI (Coordinators, 2018). Кластерный анализ в программе MEGA X (Kumar et al, 2018) проводили с использованием алго-ритмома UPGMA (Unweighted Pair-Group
Таблица. Праймеры для ПЦР, используемые в работе
Method Using Arithmetic Averages – метод невзвешенного попарного среднего; восходящий метод иерархической кластеризации) при бутстреп-поддержке 1000. Генетическое сходство на основе данных генотипирования осуществляли при использовании программы NTSYS pc, ver. 2.2 cat/ntsyspc/.
Результаты и обсуждение
Результаты кластерного анализа на основе данных секвенирования ITS (выявлены в базе NCBI) указывают на формирова-
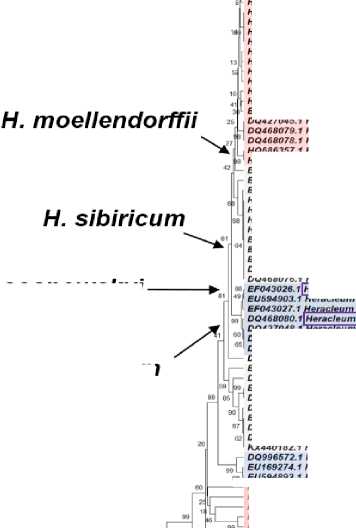
ние среди представителей рода Heracleum определенных групп. Наиболее интересной для настоящего исследования является Кавказская группа гигантских борщевиков, в которую входят H. sosnowskyi и H. mantegazzianum (рис. 1) .
Для секвенирования ITS-2 были отобраны случайным образом 23 собранных в 2019 году образца, а также 11 гербарных образцов (Приложение) . Благодаря проведенному анализу удалось установить, что ряд борщевиков, произрастающих на территории Кавказского заповедника, являются генетически близки-
|
Цель |
Структура праймеров |
Наименование гена или праймера [Ссылка (или номер последовательности, к которой подбирались праймеры)] |
Протокол |
|
Проверка качества ДНК для ПЦР |
5’CACACGGTGCCAATTTATGAA3’ / 5’GATCACGGCCAGCAAGGT3’ |
DcACT [GenBank: X17525] |
1 цикл 94 °C 2 мин; 13 циклов 94 °C 15 сек, 65 °C 30 сек (уменьшение с каждым циклом на 0.7 °C), 72 °C 45 сек; 24 цикла 94 °C 15 сек, 56 °C 30 сек, 72 °C 45 сек; 1 цикл 72 °C 5 мин. |
|
5’TCTGGTGCCATACCCAAGGA3’ / 5’ATAGGCCTTCTCAGCGGAGAT3’ |
DcTBA [GenBank: AY007250] |
||
|
5’GGGCTCTCTATGTCTTCCACATTC3’ / 5’AAACTGTTCACTAACTCGTCGAAACA3’ |
Dcβ-TUB [GenBank: AAB64308] |
||
|
5’TCTTCGCCGGCAAGCA3’ / 5’GTGGACTCCTTCTGGATGTTGTAGT3’ |
DcUBQ [GenBank: U68751] |
||
|
5’GACTACGTCCCTGCCCTTTG3’ / 5’TCACCGGACCATTCAATCG3’ |
Dc18S [GenBank: X17534] |
||
|
5’GGGAGGTGCAAAGAAAGTTATCA3’ / 5’TTCCTTTTCATTGACACCAACAA3’ |
DcGAPDH [GenBank: AY491512] |
||
|
5’TGGTGATGCTGGTTTCGTTAAG3’ / 5’AGTGGAGGGTAGGACATGAAGGT3’ |
DcEF-1A [GenBank: D12709] |
||
|
Филогенетический анализ |
5’CGATGAAGAACGTAGCGAAATG3’ / 5’AATGTGTGCCGCCTAAGGGTC3’ |
Heracleum ITS-2 [GenBank: MG220206, EF043026, AM167916] |
|
|
5’CAGGCCCTTC3’ |
OPA-01 |
1 цикл 94 °C 2 мин; 45 циклов 94 °C 45 сек, 37 °C 45 сек, 72 °C 2 мин; 1 цикл 72 °C 5 мин. |
|
|
5’AGTCAGCCAC3’ |
OPA-03 |
||
|
5’AATCGGGCTG3’ |
OPA-04 |
||
|
5’GGTCCCTGAC3’ |
OPA-06 |
||
|
5’CAATCGCCGT3’ |
OPA-07 |
||
|
5’GGGTAACGCC3’ |
OPA-09 |
||
|
5’GTGATCGCAG3’ |
OPA-10 |
||
|
5’GAAACGGGTG3’ |
OPA-11 |
||
|
5’CAAACGTCGG3’ |
OPA-19 |
||
|
Разработка экспресс-метода для идентификации образцов борщевика |
5’AGAGAGAGAGAGAGAGC3’ |
ISSR |
1 цикл 94 °C 2 мин; 45 циклов 94 °C 45 сек, 49 °C 45 сек, 72 °C 2 мин; 1 цикл 72 °C 5 мин. |
Источник: результаты исследований авторов.
53. DQ42 704 7.1 Heracleum ponticum DQ468075.1 Heracleum freynianum 211 DO468077.1 Heracleum asperum tJ HQ886328.1 Heracleum soutiei . HQ686338.1 Heracleum wenchuanense
HQ886510.1 Heracleum fargesii HQ686329.1 Heracleum franchetii HQ686330.1 Heracleum yungningense HQ68636 7.1 Heracleum henryi HQ886333.1 Heracleum scabridum HQ886381.1 Heracleum subtomentelium HQ686356.1 Heracleum rapuia HQ886325.1 Heracleum forrestii HQ886359.1 Heracleum creocharis HQ686355.1 Heracleum tiliifotium HQ686372.1 Heracleum wolongense EU169278.1 Heracleum maximum DQ42 7045.1 Heracleum antasiaticum Heracleum leskovii Heracleum platytaenium HQ68635 7.1 Heracleum dissectifoiium HQ586335. l \Heracleum moelfendorffii | EU594925.1 Heracleum tanatum EU594901.1 Heracleum orphanidis EU169283.1 Heracleum pyrenaicum HQ66638 7.1 Heracleum dissectum HQ686343.1 Heracleum hemsleyanum HQ685348.1 Heracleum stenopterum HQ686342.1 Heracleum_vreintjm_^
Heracleum sibiricum Heracleum sibiricum
Heracleum sibiricum
Heracleum sibiricum
Heracleum qrandifiorum
H. sosnowskyi
EFO43O32.1 №043031.1 EFO43O3O.1 OQ996576.1 DO488076.1
Н. mantegazzianum
Heracleum sosnowskyq Heracleum pubescens tra^iiYlgma_______ mantegazzianum | DQ42 7048.1 Heracleum lehmartnianum DQ468071.1 Heracleum afghanicum DQ42 7042.1 Heracleum rechingeri DQ42 7050.1 Heracleum apiifotlum EU594915.1 Heracleum sphondylium DQ42 7046.1 Heracleum scabrum EF043022.1 Heracleum pastinacifolium DQ427049.1 Heracleum chorodanum DQ4 68074.1 Heracleum roseum EF043025.1 Heracleum schelkovnihovii DQ4 58073.1 Heracleum transcaucasicum DQ427041.1 Heracleum argaeum KX440182.1 Heracleum crenatifolium
Heracleum minimum Heracleum alpinum as1 EU594892.1 Heracleum austriacum ------ HQ686483.1 Heracleum nyalamense FJ812153.1 Heracleum rigens HQ686349.1 Heracleum millefolium HQ686368.1 Heracleum candicans FJ312138.1 Heracleum obtusifotium ------ EU594894.1 Heracleum inexpectatum — HQ686474.1 Heracleum bivittatum »l HQ68G352.1 Heracleum kingdonii HQ885503.1 Heracleum xiaojinense
AY552527.1 Caucus carota
Евроазиатская группа
«медицинская группа»
_ Азиатская группа
«безопасные борщевики»
Кавказская группа
~ «гигантские борщевики»
Кавказская группа“ «медицинская группа»
— Альпийская группаАзиатская группа
-
— «медицинская группа»
014 0120 ОНО МЕО ОЛЕО 004 00» 00»
Рис. 1. Филогенетический анализ представителей рода Heracleum по данным ITS-последовательностей из NCBI
Программа MEGA X, алгоритм UPGMA. Пояснения даны в тексте. Источник: результаты исследований авторов.
ми к гербарным образцам H. stevenii (H3) и H. sosnowskyi (villosum) (H11), собранным в Крыму в 2015 и 1906 годах соответственно, что ставит под сомнение правильность видового определения растений. Также удалось выяснить, что борщевики, распространенные на Кольском полуострове и определяемые как борщевики Сосновского H. sosnowskyi , а также борщевики из Кингисеппского района, определенные как борщевики сибирские H. sibiricum , оказались близки к гербарным образцам борщевика обыкновенного H. sphondylium и, соответственно, должны относиться к этому виду или же являться с ним гибридами.
Часть секвенированных по региону ITS-2 последовательностей группировалась с гигантскими борщевиками, гене-
тически близкими к H. mantegazzianum и H. sosnowskyi ( рис. 2 , выделенная группа в фиолетовой рамке). В этой группе выделяются два кластера, нижний из которых разделяется на два субкластера. К первому субкластеру относятся борщевики, собранные на территории Кольского полуострова, два представителя Кавказского заповедника, а также один борщевик из Кингисеппского района. Интересным является тот факт, что в данную группу попал один гербарный образец H. stevenii (H13). Таким образом, эти образцы генетически крайне близки к H. stevenii , ареалом обитания которого является территория Кавказа. Второй субкластер включает преимущественно растения, собранные в Кингисеппском районе Ленинградской области и в пригороде Санкт-Петербур-
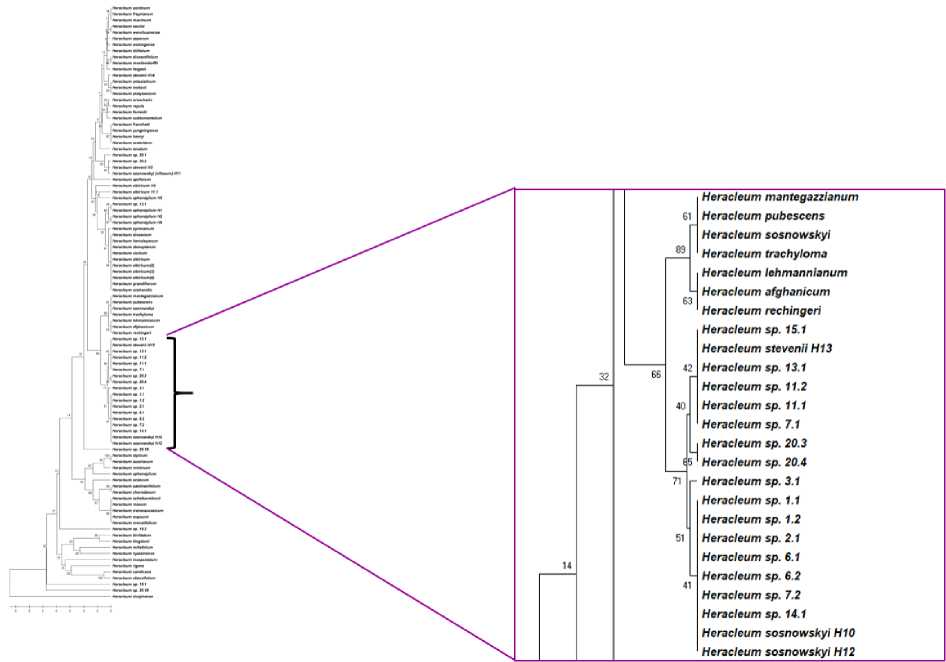
Рис. 2. Филогенетический анализ представителей рода Heracleum , секвенированных (по ITS-2) в настоящей работе
Программа MEGA X, алгоритм UPGMA. Фиолетовым выделен кластер образцов представителей рода Heracleum, проанализированных в настоящем исследовании и попавших в одну группу.
H – гербарные образцы; sp. – неопределенные до вида проанализированные представители рода Heracleum.
Источник: результаты исследований авторов.
га, а также один образец с территории Кольского полуострова. Именно этот субкластер включает и гербарные образцы H. sosnowskyi . Таким образом, встречаемость образцов, сходных с H. sosnowskyi , оценочно составляет менее 30%.
Некоторые образцы, собранные на Кольском полуострове и в Кавказском заповеднике, достоверно не группируются ни с одним из секвенированных или найденных через поиск в NCBI представителем рода Heracleum . Таким образом, для более точного определения видового состава исследованных нами популяций необходимо секвенировать более крупные участки генома.
В дополнение к секвенированию был проведен скрининг отобранных пред-
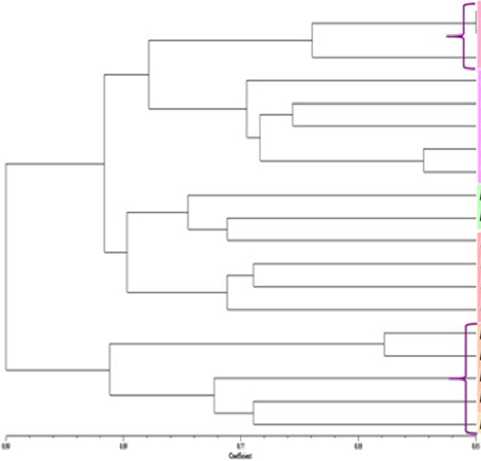
ставителей Heracleum посредством RAPD-анализа с 10 праймерами OPА (см. табл.). В результате выявлены и проанализированы суммарно 102 локуса. На основе полученных данных построены бинарные матрицы, которые затем использовались для создания филогенетического древа с применением программы NTSYS и алгоритма UPGMA. Полученное древо разделяется на три кластера (рис. 3) .
Данный анализ привнес ясность в аспекты видового разнообразия борщевиков на исследованных территориях. В кластер I попали образцы из Кавказского заповедника, а также часть образцов из Кингисеппского района, ранее попадавшие в одну группу с борщеви-
H. sibiricum 11.1
H. sibiricum 11.2
Heracleum sp. 12.1
Heracleum sp. 13.1
Heracleum sp. 15.1
Heracleum sp. 14.1
Heracleum sp.6A
Heracleum sp. 6.2
Heracleum sp. 7.1
Heracleum sp. 7.2
Heracleum sp. 11.2
Heracleum sp. 1.1 Кингисеппский район
Heracleum sp. 1.2 - (Вторичный ареал
Heracleum sp. 3.1.
Heracleum sp. 20.1 ‘
Heracleum sp. 20.2
Heracleum sp. 20.18
Heracleum sp. 20.3
Heracleum sp. 20.4
обитания)
Кавказский заповедник (Первичный ареал обитания)
Кластер
Кингисеппский район (Вторичный ареал обитания)
Кольский полуостров (Вторичный ареал обитания)
Пригород СПб (Вторичный ареал обитания)
. Кингисеппский район (Вторичный ареал обитания)
Кластер
Кластер III
Рис. 3. Филогенетический анализ представителей рода Heracleum
Программа NTSYS, алгоритм UPGMA на основе данных RAPD-анализа. Источник: результаты исследований авторов.
ком Сосновского. Таким образом, вероятно, мы имеем дело не только с борщевиком Сосновского, но и с гибридными формами данного растения на территории Кингисеппского района. Однако, судя по анализу двух деревьев, чистые негибридные формы борщевика Со-сновского встречаются в Кингисеппском районе, а также в пригороде Санкт-Петербурга (кластер III), чего нельзя сказать о борщевиках, собранных на территории Кольского полуострова, выделяющихся в кластер II вместе с безвредными формами борщевика из Кингисеппского района. Это подтверждает гипотезу о склонности борщевиков к образованию гибридов. Так, в ходе работы было продемонстрировано, что большинство проанализированных образцов борщевика являются генетически близкими к гигантским борщевикам, родственными H. sosnowskyi и H. mantegazzianum . Однако среди собранных и проанализированных представителей рода Heracleum наблюдается высокая генетическая разнородность и склонность к образованию
межвидовых гибридов, в том числе с безвредными борщевиками.
Идентификация борщевика Сосновско-го является практически значимой задачей, в том числе в свете возможных арбитражных споров. В связи с этим в 2024 году на территории Павловска были отобраны «спорные» образцы борщевика ( Приложение , образцы сбора 2024 года), отбор проводился случайным образом с участка, обозначенного как «засоренный борщевиком Сосновского». Была поставлена задача проведения экспресс-провер-ки, на основе которой можно было бы однозначно ответить, относятся ли данные образцы к H. sosnowskyi или нет. Для ее решения выбран высоко полиморфный ISSR маркер. Результаты анализа изучаемых образцов в сравнении с гербарным образцом H. sosnowskyi представлены на рис. 4.
По результатам анализа полиморфизма ДНК был сделан вывод о том, что спорные образцы, собранные в Павловске в 2024 году, не относятся к H. sosnowskyi . Данный метод может быть предложен в качестве арбитражного.
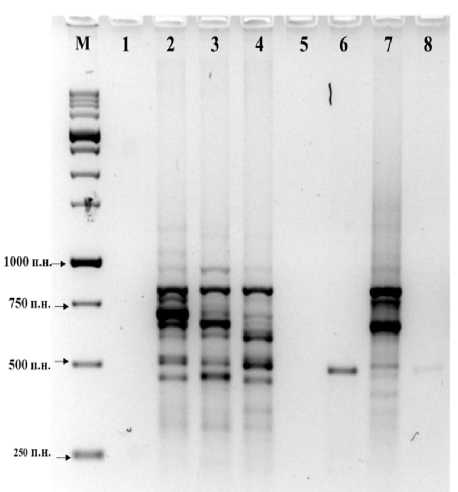
Рис. 4. Электрофореграмма продуктов амплификации образцов Heracleum
М – Маркер молекулярного веса; 1 – отрицательный контроль; 2, 3, 4, 7 – образцы, собранные в Павловске (2024 год); 5, 6, 8 – гербарные образцы Heracleum sosnowskyi Manden.
Источник: результаты исследований авторов.
Заключение
Молекулярно-генетический анализ позволил внести ясность в аспекты видового разнообразия борщевиков на исследованных территориях. Большинство образцов являются родственными группе гигантских борщевиков. На Северо-Западе образцы H. sosnowskyi оценочно составляют менее 30%, тогда как большая часть представляет собой межвидовые гибриды. Полученные данные указывают на то, что без тщательного анализа, вплоть до идентификации на уровне ДНК, участки засорения растениями гигантских борщевиков могут быть ошибочно отнесены к территориям распространения борщевика Сосновского, который утратил статус сельскохозяйственной культуры в 2015 году и был официально включен в российский реестр сорных растений. Предложен арбитражный экспресс-метод, позволяющий идентифицировать наличие или отсутствие борщевика Сосновского среди «спорных» образцов.


