Молекулярные маркеры генов PL6, PL13 и PLARG для использования в селекции подсолнечника на устойчивость к ложной мучнистой росе
Автор: Рамазанова С.А., Бадьянов Е.В., Гучетль С.З.
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 3 (183), 2020 года.
Бесплатный доступ
Одной из самых вредоносных болезней подсолнечника является ложная мучнистая роса, вызываемая оомицетом Plasmopara halstedii. Введение доминантных генов устойчивости к этому патогену в растение-хозяина является самым эффективным, экономичным и экологичным методом контроля над возбудителем болезни. Современные ДНК технологии позволяют контролировать наличие этих генов на любом этапе селекции. Одними из наиболее эффективных генов, контролирующих устойчивость к большинству рас P. halstedii, в настоящее время являются гены Pl6, РІ13 и Plarg. На линиях и селекционных образцах апробированы известные из литературных источников два STS и два SSR-маркера этих генов. Три изученных молекулярных маркера -HuP3 (локус Pl6), ORS 1008 (локус Pl13) и ORS509 (локус Plarg) позволили идентифицировать указанные гены в линиях и селекционных образцах ВНИИМК. Изученные ДНК-маркеры могут представлять особый интерес в маркер-ассоциированной селекции подсолнечника на устойчивость к возбудителю ложной мучнистой росы.
Днк-маркеры, мас, r-гены, устойчивость, подсолнечник
Короткий адрес: https://sciup.org/142224963
IDR: 142224963 | УДК: 633.854.78:577.2:632 | DOI: 10.25230/2412-608X-2020-3-183-20-26
Текст научной статьи Молекулярные маркеры генов PL6, PL13 и PLARG для использования в селекции подсолнечника на устойчивость к ложной мучнистой росе
One of the most dangerous diseases on sunflower is downy mildew caused by oomycete Plasmopara halstedii . Introduction of dominant genes of resistance to this pathogen into a host-plant is the most effective economic and ecologically safe method of this pathogen control. The current DNA-technologies allow controlling these genes presence at the any stage of breeding. Currently, the genes Pl6 , Pl13 and Pl arg are the most effective ones controlling resistance to the most races of P. halstedii . We approbated known from the literary sources two STS and two SSR-markers of these genes on the lines and breeding samples. Three studied molecular markers – HаP3 (locus Pl 6 ), ORS1008 (locus Pl 13 ) and ORS509 (locus Pl arg ) – allowed us identifying the mentions genes in lines and breeding samples of VNIIMK. The studied DNA-markers can be interested in marker-associated sunflower breeding on resistance to a downy mildew pathogen.
Как известно, устойчивость к ложной мучнистой росе подсолнечника контролируется генами Pl . Начиная с 60-х годов прошлого века, учеными всех стран, где возделывается подсолнечник, ведется поиск и идентификация новых генов, обеспечивающих устойчивость к новым расам P. halstedii . Впервые гены Pl 1 и Pl 2 были идентифицированы еще в 1972 г. [2]. В настоящее время их изучено и картировано уже более 30 [3]. Расположены они в шести группах сцепления: LG1 ( Pl arg , Pl 14 , Pl 13 , Pl 16 ), LG2 ( Pl 18 ), LG4 ( Pl 19 , Pl 17 ), LG8 ( Pl 1 , Pl 2 , Pl 7 , Pl 6 , Pl 15 , Pl 20 ), LG11 ( Pl 23 – Pl 32 ), LG13 ( Pl 5 , Pl 8 , Pl 21 , Pl 11 , Pl 12 ) генетической карты SSR (Simple Sequence Repeat) [4; 5; 6; 7; 8; 9; 10]. К тому же установлено, что устойчивость к болезни контролируют еще как минимум три локуса количественных признаков QTL (Quantitative Trait Loci), картированные в группах сцепления LG7, LG8 и LG10. Считается, что они объясняют 42 % изменчивости по данному признаку [11].
Ряд исследований показал, что самые эффективные гены Pl интрогрессированы в культурный подсолнечник из диких видов. Ген Pl 6 из дикорастущего Helianthus annuus обеспечивает устойчивость к восьми расам – 100, 300, 330, 700, 710, 730, 733, 770, ген Pl 5 – из H. tuberosus также контролирует устойчивость к восьми расам: 100, 300, 304, 310, 700, 710, 703, 714 , ген Pl 7 – из H. praecox , гены Pl 8 и Pl arg – из H. argophyllus [8; 10; 11]. Хотя оба гена – Pl arg и Pl 8 – были перенесены из H. argophyllus, они отличаются. Ген Pl 8 был получен из образца H. argophyllus 415 и обеспечивает устойчивость к 16 расам патогена [4], тогда как Pl arg происходит из образца H. argophyllus 1575 [11] и уже два десятилетия по-прежнему весьма эффективен против всех известных рас P. halstedii. Ген Pl 13 был обнаружен в местной популяции в Аргентине и контролирует устойчивость к 11 расам: 100, 300,
-
304, 330, 334, 700, 710, 714, 730, 734, 770 [17].
Использование всех этих генов является перспективным для селекции на устойчивость к P. halstedii . Поэтому разработка высокопроизводительной диагностической системы молекулярных маркеров, связанных с генами Pl, будет способствовать созданию новых генотипов подсолнечника с заданными параметрами комплексной устойчивости к ложной мучнистой росе.
Целью наших исследований было апробировать молекулярные маркеры генов Pl 6 , Pl 13 , Pl arg и изучить возможность их использования для определения наличия этих генов в линиях и селекционных образцах подсолнечника селекции ВНИИМК.
Материалы и методы. В этом исследовании были использованы 14 линий подсолнечника: RHA 419, XRQ, 83HR4RM, HIR34, PSC8, YVQ, Ha335, HA-R-5, HA-R-4, 803-1, PM-17, DM-2, RHA274, RHA265, включенных в стандартный международный тест-набор линий-дифференциаторов устойчивости подсолнечника для идентификации рас P. halstedii, а также 111 линий и селекционных образцов подсолнечника селекции ВНИИМК, предоставленных сотрудниками лаборатории селекции гибридного подсолнечника ВНИИМК.
Фитопатологическая оценка всех линий и селекционных образцов подсолнечника на устойчивость к ложной мучнистой росе проводилась в лабораторных условиях лаборатории молекулярного маркирования совместно с сотрудниками лаборатории иммунитета ВНИИМК методом искусственного заражения проростков зооспорами оомицета [12]. Образцы оценивались на устойчивость к расам 330, 334, 710 и 730.
Для выделения ДНК использовали 3– 7-дневные проростки подсолнечника. Экстракцию ДНК проводили методом, в основе которого лежит лизис клеток буфером, содержащим СТАВ (ЦТАБ – це-тилтриметиламмонийбромид), депротеи- низация хлороформом и осаждение ДНК изопропанолом [13; 14].
Концентрацию ДНК в полученных препаратах определяли визуально по интенсивности свечения пробы объемом 10 мкл в ультрафиолетовом свете в 1%-ом агарозном геле с добавлением 2 мкл бромистого этидия. Электрофорез препаратов ДНК проводили при напряжении 100 V в течение 30 мин.
Для ПЦР-анализа применили 4 пары праймеров, разработанных для маркирования локусов Pl 6, Pl 8, и Pl arg (табл. 1) [8; 17; 21]. Полимеразную цепную реакцию выполняли в реакционной смеси (25 мкл) следующего состава: 67 мМ Трис-HCl (рН 8,8); 16,6 мM сульфата аммония; 1,5– 3,0 мM MgCl 2 ; 0,01 % Tween 20; по 0,2 мM дезоксирибонуклеозидфосфатов; по 10 пМ праймеров; 10 нг матричной ДНК и 1 ед. рекомбинантной термостабильной ДНК-полимеразы (НПО «СибЭнзим», Россия). Реакции проводили в термоциклере S1000тм (Bio-Rad, США) при следующих температурных режимах: начальная денатурация при 95 °С в течение 5 мин, далее 37 циклов с последовательной сменой температур: денатурация при 95 ºС, в течение 30 сек, отжиг праймера при 63 ºС – 30 сек, элонгация при 72 ºС – 1 мин 24 сек, и заключительная элонгация при 72 ºС в течение 10 мин.
Электрофорез продуктов амплификации проводили в геле, содержащем 2 % агарозы и SВ-буфер, с использованием камеры для горизонтального электрофореза SE-2 (Хеликон, Россия) при напряжении 200 В, силе тока 100 мА, в течение 30 мин. Для некоторых праймеров электрофорез дополнительно проводили в 8%-ном акриламидном геле в камере для вертикального электрофореза (Хеликон, Россия). Гели окрашивали бромистым этидием. Для визуализации и документирования результатов электрофореза применяли систему цифровой документации видеоизображения BIO-PRINT (Vilber Lourmat, Франция).
Таблица 1
Нуклеотидные последовательности праймеров, использованных для идентификации локусов Pl 6 , Pl 13 и Pl arg
|
Праймер |
5’-3’ последовательность |
Локус |
|
HaP3 |
F: GTT TGT GGA TCA TCT CTA TGC G R: TGC TTC TTC CTT CTA TCT CAC TC |
Pl 6 |
|
STS10D6 |
F: AAC TAC GAC CCA CAA AAG GAC AAG R: TTA GAC CAG GGC CCA ACA AAC |
Pl 13 |
|
ORS1008 |
F: GAT CAC CTT CAC TAT CCA CAA CC R: CAT GAG GGC ATT CTT GTC ATT T |
Pl 13 |
|
ORS509 |
F: CAA CGA AAA GAC AGA ATC GAA A R: CCG GGA ATT TTA CAA GGT GA |
Plarg |
Результаты и обсуждение. Исследование по маркированию генов устойчивости проводилось на линиях и селекционных образцах ВНИИМК, контрастно различающихся по степени устойчивости к возбудителю ложной мучнистой росы подсолнечника. Поэтому на первом этапе работы проводилась оценка образцов подсолнечника на устойчивость к расам 330, 334, 710 и 730 ложной мучнистой росы в лабораторных условиях методом искусственного заражения проростков зооспорами оомицета. Результаты теста показали, что все изученные образцы разделились на четыре группы: устойчивые ко всем расам; устойчивые к 330, 730, 710, но восприимчивые к 334 расе; устойчивые только к 330 расе; восприимчивые ко всем расам.
STS-локус HаP3 – один из трех, разработанных для маркирования локуса Pl6 [21]. Этот локус представляет собой кластер из 13 генов, относящихся к TIR-NBS-LRR классу R-генов, картированных в группе сцепления LG8 на генетической карте SSR. Праймерная пара HаP3 ам-плифицирует четыре маркерных фрагмента длиной от 988 до 1811 пар нуклеотидов. Три из них, обозначенных как HaP3/R1 (1811 п.н.), HaP3/R2 (1119 п.н.) и HaP3/R3 (988 п.н.), выявлены у линий подсолнечника, устойчивых к расам 100, 300, 700, 703, 710 и 730. Фрагмент длиной 1406 п.н., обозначенный как HаP3/S1, является общим для устойчивых и восприимчивых к этим расам образцов [21]. С 2018 г. нами изучался молекулярногенетический полиморфизм этого локуса и было показано, что он является весьма перспективным для его использования в маркер-ассоциированной селекции (МАС) [22]. В данном исследовании изучался молекулярно-генетический полиморфизм этого локуса у 111 линий подсолнечника и селекционных образцов ВНИИМК. На рисунке 1 представлена электрофореграмма продуктов амплификации ДНК селекционных образцов и линий с разной степенью устойчивости к P. halstedii с праймером НаРЗ.
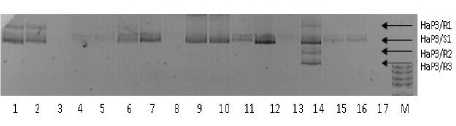
Рисунок 1 – Электрофореграмма продуктов амплификации ДНК линий подсолнечника с праймером HаP3. Дорожки:
1 – Л673-15; 2 – Л675-15; 3 – Л665-15;
4 – СЛ24; 5 – СЛ-4; 6 – Л689-15; 7 – ВК 788;
8 – Л665-15; 9 – ВК 934; 10 – ВК 732;
11 – ВК 680; 12 – ВК 905; 13 – ВК 653;
14 – ВК 101Б; 15 – ВК 276; 16 – ВК 585;
17 – отрицательный контроль; М – маркер молекулярного веса 100 bp. Стрелками обозначены четыре маркерных фрагмента ДНК
У образцов Л673-15, Л675-15 и Л689-15 (дорожки 1, 2 и 6) выявлены два маркерных фрагмента HaP3/R1 и HаP3/S1. Фитопатологическая оценка показала, что они устойчивы к трем расам: 330, 730, 710, и восприимчивы к расе 334. Аналогичный результат получен и для других образцов с устойчивостью к этим же расам. Образцы под номерами 3 и 8 были устойчивы ко всем четырем расам, однако у них не было выявлено амплифици-рованных фрагментов. Возможно, устойчивость у них контролируется другими генами. У образца под номером 14 выявлены все четыре маркерных фрагмента, и он также был устойчив ко всем расам. У остальных образцов, восприимчивых ко всем четырем расам (дорожки 4, 5, 7, 9–13, 15, 16), амплифицировался толь- ко один общий маркерный фрагмент HаP3/S1.
Известно, что ген Pl 13 – это доминантный ген или кластер из нескольких тесно связанных генов, расположенный в группе сцепления LG1 [17]. Для ПЦР-анализа ДНК линий-дифференциаторов устойчивости и селекционных образцов подсолнечника был выбран микросателлитный локус ORS1008, так как, по данным тех же авторов, он находится на расстоянии 0,9 сантиморганид (сМ) от данного гена. Исследование этого локуса позволило выявить аллель, характерный для линий НА-R-5 и НА-R-4, в генотипе которых присутствует этот ген [6; 7]. Только у этих линий в результате ПЦР с парой праймеров, фланкирующих локус ORS1008, амплифицирован фрагмент длиной 296 п.н. (рис. 2, дорожка 1).
1 2 3 4 5 6 7 8 9 10 11 12 1Э 14 15 16 17 18 19 М
Рисунок 2 – Электрофореграмма продуктов амплификации ДНК линий подсолнечника с праймером ORS1008. Дорожки: 1 – HA-R-5; 2–18 – селекционные образцы; 19 – отрицательный контроль; М – маркер молекулярного веса 100 bp. Стрелкой показан фрагмент ДНК 296 п.н.
Среди исследованных селекционных линий и образцов подсолнечника не было выявлено образца с точно таким же спектром фрагментов ДНК, как у линии HA-R-5 (рис. 2). Вероятно, изученные селекционные образцы не содержат ген Pl 13 .
Для маркирования этого же гена Pl13 применили также STS-маркер STS10D6, который является кодоминантным [17]. На рисунке 3 представлены результаты амплификации ДНК линий-дифференциаторов с этим праймером. Как видно из рисунка, для линий HA-R-5 и HA-R-4, носителей гена Pl13 (дорожки 8, 9), не полу- чено фрагментов, отличающих их от других.
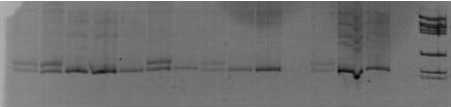
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 М
Рисунок 3 – Электрофореграмма продуктов амплификации ДНК линий-дифференциаторов подсолнечника с праймером
STS10D6. Дорожки: 1 – RHA 419; 2 – XRQ; 3 – 83HR4RM; 4 – HIR34; 5 – PSC8; 6 – YVQ; 7 – Ha-335; 8 – HA-R-5; 9 – HA-R-4; 10 – 803-1;
11 – PM-17; 12 – DM-2; 13 – RHA274;
14 – RHA265; 15 – отрицательный контроль;
М – маркер молекулярного веса 100 bp
Валидация этого маркера на 111 образцах подсолнечника показала такой же результат. У всех образцов были получены такие же спектры амплифицированной ДНК, как и у линий HA-R-5 и HA-R-4. Протестированный маркер не позволил определить устойчивые к ложной мучнистой росе генотипы, так как одинаково представлен у всех исследованных образцов подсолнечника.
Для маркирования гена Pl arg был применен микросателлитный локус ORS509 . На первом этапе исследования он был изучен на линиях-дифференциаторах. Из них только линия RHA 419 содержит этот ген. В результате исследований показано, что у этой линии амплифицирован фрагмент длиной 207 пар нуклеотидов. У линии На-335 обнаружен фрагмент длиной 190 п.н., у остальных – фрагменты длиной 200 п.н. Тестирование этого локуса показало, что фрагмент длиной 207 п.н., характерный для линии RHA 419, не выявлен у исследуемых образцов подсолнечника селекции ВНИИМК. Как видно из рисунка 4, у большинства генотипов амплифицировался фрагмент длиной 200 п.н. (дорожки 2, 4, 5, 7–17), у двух линий (дорожки 3 и 6) – 190 п.н.
2СГпн 2Хкк ІХкк

1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 М
Рисунок 4 – Электрофореграмма продуктов амплификации ДНК линий подсолнечника по локусу ORS509. Дорожки: 1 – RHA 419;
2–17 – селекционные образцы; М – маркер молекулярного веса 100 bp. Стрелками показаны фрагменты ДНК длиной 207, 200 и 190 п.н.
Это позволяет предположить, что в группе сортов данного исследования так же нет образцов, содержащих этот ген.
Выводы. Наши исследования показали, что три изученных молекулярных маркера – HаP3 (локус Pl 6 ), ORS1008 (локус Pl 13 ) и ORS509 (локус Pl arg ) позволили идентифицировать указанные гены в линиях и селекционных образцах ВНИИМК. Изученные ДНК-маркеры могут представлять особый интерес в маркер-ассоциированной селекции подсолнечника на устойчивость к возбудителю ложной мучнистой росы.
Список литературы Молекулярные маркеры генов PL6, PL13 и PLARG для использования в селекции подсолнечника на устойчивость к ложной мучнистой росе
- Ивебор М.В., Антонова Т.С., Саукова С.Л., Арасланова Н.М. Ложная мучнистая роса на юге России // Защита и карантин растений. - 2019. - № 10. - С. 29-33.
- Zimmer D.E., Kinman M.L. Downy mildew resistance in cultivated sunflower and its inheritance // Crop Sci. - 1972. - V. 12 (6). - P. 749-751. DOI: 10.2135/cropsci1972.0011183X001200060009x
- Pecrix Y., Penouilh-Suzette C., Munos S., Vear F. and Godiard L. Ten Broad Spectrum Resistances to Downy Mildew Physically Mapped on the Sunflower Genome // Front. Plant Sci. - 2018. -V. 9. - P. 1780. DOI: 10.3389/fpls.2018.01780
- Miller J.F., Gulya T.J. Inheritance of resistance to race 4 of downy mildew derived from interspecific crosses in sunflower // Crop Sci. -1991. - V. 31. - P. 40-43.
- Dußle C.M., Hahn V., Knapp S.J., Bauer E. РIаrg from Helianthus argophyllus is unlinked to other known downy mildew resistance genes in sunflower // Theor Appl Genet. - 2004. - V. 109. -P. 1083-1086. DOI: 10.1007/s00122-004-1722-9
- Liu Z., Gulya T.J., Seiler G.J., Vick B.A., Jan C-C. Molecular mapping of the Pl16 downy mildew resistance gene from HA-R4 to facilitate marker-assisted selection in sunflower // Theor. Appl. Genet. -2012. - V. 125. - P. 121-131.
- Bertero de Romano A., Romano C., Bulos M., Altieri E., Sala C. A new gene for resistance to downy mildew in sunflower // Proc. Intern. Symposium "Sunflower Breeding on Resistance to Diseases". - Russia, Krasnodar, June 23-24, 2010. -P.141-146.
- Wieckhorst S., Bachlava E., Dußle C.M., Tang S., Gao W., Saski C., Bauer E. Fine mapping of the sunflower resistance locus PlArg introduced from the wild species Helianthus argophyllus // Theor. Appl. Genet. - 2010. - V. 121 (8). - Р. 1633-1644.
- DOI: 10.1007/s00122-010-1416-4
- Gascuel Q., Martinez Y., Boniface M-C., Vear F., Pichon M., Godiard L. The sunflower downy mildew pathogen Plasmopara halstedii // Mol. Plant Path. - 2015. - V. 16. - P. 109-122.
- Bachlava E., Radwan O.E., Abratti G., Tang S.X., Gao W.X., Heesacker A.F. [et al.]. Downy mildew (Pl (8) and Pl (14) and rust (R (Adv)) resistance genes reside in close proximity to tandem-ly duplicated clusters of non-TIR-like NBS-LRR-encoding genes on sunflower chromosomes 1 and 13 // Theor. Appl. Genet. - 2011. - V. 122. - P. 1211-1221.
- DOI: 10.1007/s00122-010-1525-0
- Vear F., Serieys H., Petit A., Serre F., Boudon J.-P., Roche S. [et al.]. Origins of major genes for downy mildew resistance in sunflower // Proceedings of the 17th International Sunflower Conference, Cordoba, Spain, 2008a.
- Saghai-Maroof M.A., Soliman K.M., Jorgensen R.A., Allard R.W. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics // PNAS USA. - 1984. - V. 81. - P. 8014-8018.
- Zolan M.E., Pukkila P.J. Inheritance of DNA methylation in Corpinus cinereous // Mol. Cell Biol. - 1986. - V. 6. - No 1. - Р. 195-200.
- Pankovic D., Jocic S., Lacok N., Sakac Z., Skoric D. The use of PCR-based markers in the evaluation of resistance to downy mildew in NS-breeding material // Helia. - 2004. - No 27. - P. 149-158.
- Vincourt P, Assadi F, Bordat A, Langlade N.B., Gouzy J., Pouilly N. [et al.]. Consensus mapping of major resistance genes and independent QTL for quantitative resistance to sunflower downy mildew // Theor. Appl. Genet. - 2012. - V. 125. - P. 909-920.
- DOI: 10.1007/s00122-012-1882-y
- Mulpuri S., Liu Z., Feng J., Gulya T.J., Jan C.-C. Inheritance and molecular mapping of a downy mildew resistance gene, Pl13 in cultivated sunflower (Helianthus annuus L.) // Theor. Appl.Genet. - 2009. - V. 119 (5). - P. 795-803.
- DOI: 10.1007/s00122-009-1089-z
- Brahm L., Röcher T., Friedt W. PCR-based markers facilitating marker assisted selection in sunflower for resistance to downy mildew // Crop Sci. - 2000b. - V. 40. - P. 676-682.
- Yu J.-K., Tang S., Slabaugh M.B., Heesacker A., Cole G., Herring M. [et al.]. (2003) Towards a saturated molecular genetic linkage map for cultivated sunflower // Crop Sci. - 2003. - V. 43. - P. 367-387.
- Gilley M.A., Misar C.G., Gulya T.J., Markell S.G. Prevalence and virulence of Plasmopara halstedii (downy mildew) in sunflowers // In: Proceeding of 38th Sunflower Research Forum. -Fargo, ND, USA. - 2016, January 12-13.
- Bouzidi M.F., Badaoui S., Cambon F., Vear F., De Labrouhe D. Tourvielle, Nicolas P., Mouzeyar S. Molecular analysis of a major locus for resistance to downy mildew in sunflower with specific PCR-based markers // Theor. Appl. Genet. -2002. - V. 104. - P. 592-600.
- Рамазанова С.А., Антонова Т.С. Маркирование локусов использование Pl5, Pl6 и Pl8, контролирующих устойчивость к Plasmopara halstedii у линий подсолнечника селекции ВНИИМК // Масличные культуры. Науч.-тех. бюл. ВНИИМК. - 2018. - Вып. 3 (175). - С. 19 DOI: 10/25230/2412-608X-2018-3-175-19-27.


