Оценка бактериального состава сметаны с помощью высокопроизводительного секвенирования
Автор: Е.Ю. Нестерова, М.Ю. Сыромятников, В.Н. Попов
Журнал: Вестник Воронежского государственного университета инженерных технологий @vestnik-vsuet
Рубрика: Пищевая биотехнология
Статья в выпуске: 3 (89), 2021 года.
Бесплатный доступ
Молочнокислые бактерии активно применяются в качестве заквасок для различных продуктов питания, и, в том числе, при производстве молочной продукции. От качества закваски зачастую зависит качество конечного продукта. Сметана является традиционным продуктом для многих стран. Её изготовление основывается на ферментации сливок при добавлении определенных видов заквасочных культур, относящихся к таким родам, как Streptococcus, Lactobacillus, Lactococcus, Pediococcus и Enterococcus. С помощью метода высокопроизводительного секвенирования были исследованы бактериальные составы образцов сметаны различных производителей доступные на российском рынке. Секвенирование проводилось на секвенаторе Illumina MiSeq. Учитывались бактериальные таксоны, чьё процентное соотношение превышало 1% относительно всех идентифицированных микроорганизмов в образце. В результате анализа полученных данных было установлено, что доминирующими бактериями во всех образцах 20%-ой сметаны оказались представители родов Streptococcus и Lactococcus. Образцы №3 и №10 содержали Lacticaseibacillus. Их процентное содержание в данных образцах не превышало 2%. В образце сметаны №1 были выявлены представители рода Enterococcus (3% от общего числа бактерий, обнаруженных в образце). Видовой анализ микробного сообщества исследованных образцов с помощью системы Nucleotide BLAST позволил определить, что в качестве заквасочных культур производителями использовались Lactococcus lactis, Streptococcus thermophilus, Lacticaseibacillus rhamnosus (сметана под номерами 3 и 10) и Enterococcus faecium.(сметана №1) Данные бактерии традиционно используются при производстве кисломолочной продукции за счёт способности формировать специфические вкусы, ароматы и текстуры в процессе ферментации.
Молочнокислые бактерии, ферментирование, микробное сообщество, высокопроизводительное секвенирование, сметана
Короткий адрес: https://sciup.org/140259872
IDR: 140259872 | УДК: 577.29 | DOI: 10.20914/2310-1202-2021-3-129-134
Текст научной статьи Оценка бактериального состава сметаны с помощью высокопроизводительного секвенирования
Сметана – кисломолочный продукт, изготавливаемый на основе сливок и стартовых культур молочнокислых бактерий [1]. Это традиционный продукт во многих странах, который ценится за свои вкусовые характеристики и состав, насыщенный питательными веществами [2]. Многочисленные исследования показали положительное влияние молочнокислых продуктов на здоровье человека, как за счёт самих бактерий, входящих в состав, так и за счёт биоактивных элементов, выделяемых в процессе ферментации [3–5]. Было доказано положительно влияние молочнокислых бактерий на состояние желудочно-кишечного тракта человека [6].
В качестве основных микроорганизмов, применяемых при производстве ферментированных молочных продуктов, в том числе и сметаны, используются бактерии родов Streptococcus, Lactobacillus, Lactococcus, Pedi-ococcus и Enterococcus [7]. Для того чтобы
This is an open access article distributed under the terms of the Creative Commons Attribution 4.0 International License
определить бактериальный состав молочнокислой продукции, сегодня используются не только классические методы, основанные на культивировании на питательных средах [8, 9], но и современные быстрые и точные методики, к которым относится высокопроизводительное секвенирование. Эта методика значительно облегчила изучение сложной микробиоты продуктов питания [10].
Работы, посвященные изучению микробиологического состава сметаны, не многочисленны. Как правило, это комплексные исследования микробных сообществ в различных кисломолочных продуктах, динамики их состава в процессе ферментации [11–13]. Так, например, в 2018 китайские исследователи анализировали традиционные сметану и масло, изготавливаемые коренными жителями Бурятии, на основе комбинации методик секвенирования SMRT и масс-спектрометрии [14].
В связи с этим, цель нашей работы заключалась в проведении анализа бактериального состава коммерчески доступной сметаны на основе высокопроизводительного секвенирования.
Материалы и методы
В качестве объектов исследования было использовано 12 образцов 20%-ой сметаны различных производителей, доступных на территории Российской Федерации. Для выделения ДНК был использован коммерческий набор FаstDNА™ Spin Kit (MP Biomedicals, США).
Изготовление библиотек осуществлялось на основе ПЦР с использованием универсальных праймеров [15, 16] к области V4 гена 16S рРНК на основе методики, описанной ранее [17]. Нуклеотидные последовательности праймеров указаны в таблице 1 .
Таблица 1. Универсальные бактериальные праймеры для проведения секвенирования
Table 1.
Universal bacterial sequencing primers
|
Праймер Primer |
Нуклеотидная последовательность Nucleotide sequence |
|
515F |
5'-GTGBCAGCMGCCGCGGTAA-3' |
|
Pro-mod-805R |
5'- GACTACNVGGGTMTCTAATCC-3' |
На каждый из 12 образцов приходилось по две библиотеки, секвенирование которых осуществлялось параллельно, с использованием набора реагентов MiSeq Reagent Micro Kit v2 (300 циклов) MS-103–1002 (Illumina, США)
на секвенаторе MiSeq (Illumina, США), считывающим 150 п.н. с каждого конца.
Биоинформатический анализ данных, полученных в результате секвенирования, позволил выяснить распределение последовательностей по рабочим таксономическим единицам (OTU) на основе их сходства, которое составляло более 97%. Идентификация OTU осуществлялось на основе программы SILVАngs 1.3 [18].
Результаты и обсуждение
Высокопроизводительное секвенирование позволило выявить данные о фактическом бактериальном составе всех анализируемых образцов. В работе были учтены только те таксоны бактерий, чьё процентное соотношение превышало 1% от общего числа идентифицированных бактерий. Результаты анализа представлены в таблице 2.
Таблица 2.
Идентифицированные роды бактерий в образцах сметаны
Table 2.
Identified genera of bacteria in sour cream samples
|
Образец сметаны Sour cream sample |
Выявленный состав Revealed composition |
Процентное содержание в образце Percentage in the sample |
|
1 |
Streptococcus |
97% |
|
Enterococcus |
3% |
|
|
2 |
Streptococcus |
80% |
|
Lactococcus |
19% |
|
|
3 |
Streptococcus |
95% |
|
Lactococcus |
3% |
|
|
Lacticaseibacillus |
2% |
|
|
4 |
Streptococcus |
83% |
|
Lactococcus |
17% |
|
|
5 |
Streptococcus |
38% |
|
Lactococcus |
61% |
|
|
6 |
Streptococcus |
56% |
|
Lactococcus |
44% |
|
|
7 |
Streptococcus |
85% |
|
Lactococcus |
14% |
|
|
8 |
Streptococcus |
100% |
|
9 |
Streptococcus |
53% |
|
Lactococcus |
46% |
|
|
10 |
Streptococcus |
70% |
|
Lactococcus |
28% |
|
|
Lacticaseibacillus |
2% |
|
|
11 |
Streptococcus |
85% |
|
Lactococcus |
15% |
|
|
12 |
Streptococcus |
78% |
|
Lactococcus |
22% |
По результатам анализа было установлено, что все 12 образцов сметаны содержали в своём составе бактерии рода Streptococcus . Их процентное соотношение варьировало от 38% (образец № 5) до 100% (образец № 8). В 9 из 12 образцов доминировали бактерии Streptococcus . Их содержание превышало 70%. Это образцы под номерами 1–4, 7, 8, 10–12. Необходимо отметить, что образец сметаны № 8 определился как однокомпонентный продукт, т. е. в его составе были обнаружены только представители рода Streptococcus (рисунок 1).
На втором месте по встречаемости в исследованной продукции располагались представители рода Lactococcus . 83% исследованных образцов включали в состав представителей данного рода. Так, например, в сметане под номерами 5, 6 и 9 содержание Lactococcus составляло 61%, 44% и 46% соответственно. Процентное соотношение бактерий Lactococcus в остальных образцах не превышало 28%.
Образцы № 3 и № 10 характеризовались трехкомпонентным составом. В данной сметане помимо Streptococcus и Lactococcus , было выявлено наличие представителей рода Lacticasei-bacillus , чьё содержание в обоих образцах составило 2%. На рисунке 2 изображено процентное соотношение бактерий в составе сметаны № 3.
Образец сметаны под номером 1 отличался от остальных образцов наличием в составе помимо Streptococcus бактерий рода Enterococcus (3%) (рисунок 3).
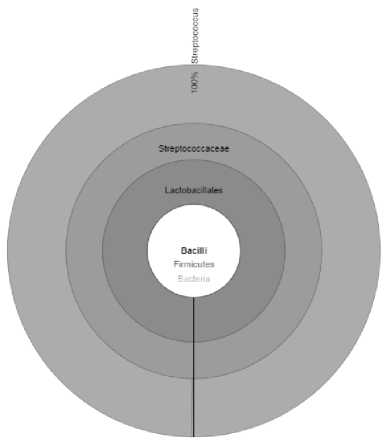
Рисунок 1. Процентное содержание бактерий в образце сметаны № 8
Figure 1. The percentage of bacteria in sour cream sample No. 8
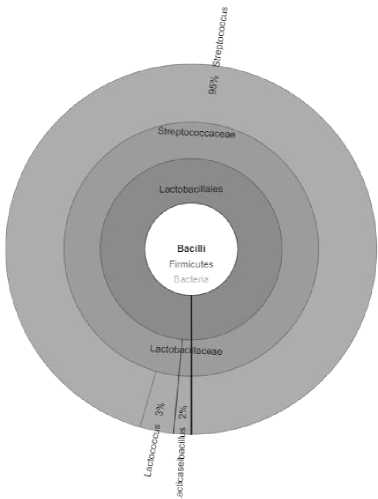
Рисунок 2. Процентное содержание бактерий в образце сметаны № 3
Figure 2. The percentage of bacteria in sour cream sample No. 3
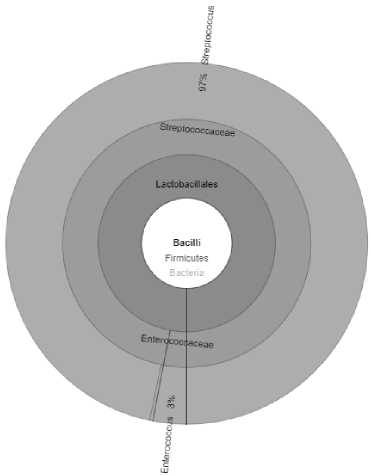
Рисунок 3. Процентное содержание бактерий в образце сметаны № 1
Figure 3. The percentage of bacteria in sour cream sample No. 1
Следующий этап исследования заключался в определении видовой принадлежности обнаруженных микроорганизмов. Нуклеотидные последовательности, полученные в результате секвенирования, были проанализированы с помощью системы Nucleotide BLAST. Такой анализ строится на сопоставлении нуклеотидных последновательностей с последовательностями, ранее занесенными в международную базу данных (таблица 3).
Таблица 3.
Видовая принадлежность обнаруженных бактерий в образцах сметаны
Table 3.
Species of bacteria detected in sour cream samples
|
Образец сметаны Sour cream sample |
Вид Species |
Совпадение в GenBank, % Compliance with GenBank, % |
|
1 |
Enterococcus faecium |
100,00 |
|
Streptococcus thermophilus |
99,26 |
|
|
2 |
Lactococcus lactis |
100,00 |
|
Streptococcus thermophilus |
99,58 |
|
|
3 |
Streptococcus thermophilus |
99,25 |
|
Lactococcus lactis |
98,80 |
|
|
Lacticaseibacillus rhamnosus |
98,41 |
|
|
4 |
Streptococcus thermophilus |
98,40 |
|
Lactococcus lactis |
100,00 |
|
|
5 |
Streptococcus thermophilus |
98,74 |
|
Lactococcus lactis |
100,00 |
|
|
6 |
Streptococcus thermophilus |
98,80 |
|
Lactococcus lactis |
99,60 |
|
|
7 |
Streptococcus thermophilus |
98,80 |
|
Lactococcus lactis |
99,60 |
|
|
8 |
Streptococcus thermophilus |
98,40 |
|
9 |
Streptococcus thermophilus |
98,80 |
|
Lactococcus lactis |
100,00 |
|
|
10 |
Lactococcus lactis |
98,80 |
|
Streptococcus thermophilus |
98,00 |
|
|
Lacticaseibacillus rhamnosus |
98,01 |
|
|
11 |
Lactococcus lactis |
99,20 |
|
Streptococcus thermophilus. |
97,80 |
|
|
12 |
Lactococcus lactis |
100,00 |
|
Streptococcus thermophilus |
98,80 |
Было установлено, что в состав заквасочных культур входили Lactococcus lactis, Streptococcus thermophilus, Lacticaseibacillus rhamnosus (для образцов № 3 и № 10) и
Enterococcus faecium (для образца № 1). Все бактерии, идентифицированные в образцах сметаны, входили в состав культур молочнокислых бактерий, используемых при производстве кисломолочной продукции. Так, Lactococcus lactis активно применяется в ферментации за счёт высокой метаболической стабильности и способности образовывать кислоты в молоке, тем самым влиять на вкусовые характеристики конечного продукта [19]. Одной из коммерчески важнейших заквасочных культур явялется Streptococcus thermophilus . Эти бактерии участвуют в метаболизме белков и сахаров, а также формировании ароматов [20]. Использование в производстве Lacticaseibacillus rhamnosus связано с его пробиотическими свойствами и со способностью подавлять рост и развитие плесени в готовой молочной продукции [21]. Бактерии Enterococcus faecium обладают протео-, эстеро- и липолитечской активностью, что обеспечивает формирование специфических текстурных, ароматических и вкусовых характеристик [22].
Заключение
В ходе проведения высокопроизводительного секвенирования 12 образцов сметаны было выяснено, что доминирующим компонентами микробных сообществ каждого из них являлись бактерии двух родов – Streptococcus и Lactococcus . Бактерии рода Streptococcus встречались во всех образцах сметаны. Что касается рода Lactococcus , 11 из 12 образцов сметаны содержали в своём составе представителей данного таксона. Процентное соотношение Lactococcus относительно других компонентов в образцах не превышало 28%. Сметана под номерами 3 и 10 помимо представителей вышеуказанных родов содержала Lacticaseibacillus (по 2% для каждого из образцов). В сметане № 1 по результатам анализа 3% приходилось на представителей рода Enterococcus .
Полученные результаты свидетельствуют о наличие в бактериальном составе исследованных образцов сметаны представителей культур молочнокислых бактерий, традиционно используемых в качестве заквасок при производстве кисломолочной продукции. Составы были представлены Lactococcus lactis, Streptococcus thermophilus, Lacticaseibacillus rhamnosus и Enterococcus faecium .
Список литературы Оценка бактериального состава сметаны с помощью высокопроизводительного секвенирования
- Danylenko S.G., Bodnarchuk O.V., Ryzhkova T.M. et al. The effects of thickeners upon the viscous properties of sour cream with a low fat content // Acta Scientiarum Polonorum, Technologia Alimentaria. 2020. V. 19. P. 359-368. https://doi.org/10.17306/J.AFS.0836
- Yu J., Wang H.M., Zha M.S. et al. Molecular identification and quantification of lactic acid bacteria in traditional fermented dairy foods of Russia // Journal of Dairy Science. 2015. V. 98. P. 5143-5154. https://doi.org/10.3168/jds.2015-9460
- Mathur H., Beresford T.P., Cotter P.D. Health Benefits of Lactic Acid Bacteria (LAB) Fermentates // Nutrients. 2020. V. 12. P. 1679. https://doi.org/10.3390/nu12061679
- Linares D.M., G?mez C., Renes E. et al. Lactic Acid Bacteria and Bifidobacteria with Potential to Design Natural Biofunctional Health-Promoting Dairy Foods // Frontiers in Microbiology. 2017. V. 8. P. 846. https://doi.org/10.3389/fmicb.2017.00846
- Arqu?s J.L., Rodr?guez E., Langa S. et al. Antimicrobial activity of lactic acid bacteria in dairy products and gut: effect on pathogens // BioMed Research International. 2015. V. 2015. P. 584183. https://doi.org/10.1155/2015/584183
- Marco M.L., Heeney D., Binda S. et al. Health benefits of fermented foods: microbiota and beyond // Current Opinion in Biotechnology. 2017. V. 44. P. 94-102. https://doi.org/10.1016/j.copbio.2016.11.010
- Hayek S., Gyawali R., Aljaloud S. et al. Cultivation media for lactic acid bacteria used in dairy products // Journal of Dairy Research. 2019. V. 86. P. 490-502. https://doi.org/10.1017/S002202991900075X
- Elionora Hantsis-Zacharov M.H. Culturable psychrotrophic bacterial communities in raw milk and their proteolytic and lipolytic traits. Applied and Environmental Microbiology. 2007. V. 73. P. 7162-7168. https://doi.org/10.1128/AEM.00866-07
- Duniere L., Xu S., Long J. et al. Bacterial and fungal core microbiomes associated with small grain silages during ensiling and aerobic spoilage // BMC Microbiology. 2017. V. 17. P. 50. https://doi.org/10.1186/s12866-017-0947-0
- Luzzi G., Brinks E., Fritsche J. et al. Microbial composition of sweetness-enhanced yoghurt during fermentation and storage // AMB Express. 2020. V. 10. P. 131. https://doi.org/10.1186/s13568-020-01069-5
- Zamfir M., Vancanneyt M., Makras L. et al. Biodiversity of lactic acid bacteria in Romanian dairy products // Systematic and Applied Microbiology. 2006. V. 29. P. 487-95. https://doi.org/10.1016/j.syapm.2005.10.002
- Terzi?-Vidojevi? A., Veljovi? K., Tolina?ki M. et al. Diversity of non-starter lactic acid bacteria in autochthonous dairy products from Western Balkan Countries - Technological and probiotic properties // Food Research International. 2020. V. 136. P. 109494. https://doi.org/10.1016/j.foodres.2020.109494
- Xu W.L., Li C.D., Guo Y.S. et al. A snapshot study of the microbial community dynamics in naturally fermented cow's milk // Food Science & Nutrition. 2021. V. 9. P. 2053-2065. https://doi.org/10.1002/fsn3.2174
- Yu J., Mo L., Pan L. et al. Bacterial Microbiota and Metabolic Character of Traditional Sour Cream and Butter in Buryatia, Russia // Frontiers in Microbiology. 2018. V. 9. P. 2496. https://doi.org/10.3389/fmicb.2018.02496
- Fadrosh D.W., Gajer B.M.P., Sengamalay N. et al. An improved dual-indexing approach for multiplexed 16S rRNA gene sequencing on the Illumine MiSeq platform // Microbiome. 2014. V. 2. P. 6. https://doi.org/10.1186/2049-2618-2-6
- Hugerth L.W., Wefer H.A, Lundin S. et al. Dege Prime, a program for degenerate primer design for broad-taxonomic-range PCR in microbial ecology studies // Applied and Environmental Microbiology. 2014. V. 80. P. 5116-5123. https://doi.org/10.1128/AEM.01403-14
- Merkel A.Y., Tarnovetskii I.Y., Podosokorskaya O.A. et al. Analysis of 16S rRNA primer systems for profiling of thermophilic microbial communities // Microbiology. 2019. V. 88. P. 671-680. https://doi.org/10.1134/S0026261719060110
- Quast C., Pruesse E., Yilmaz P. et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools // Nucleic Acids Research. 2013. V. 41. P. D590D596. https://doi.org/10.1093/nar/gks1219
- Bintsis T. Lactic acid bacteria as starter cultures: An update in their metabolism and genetics // AIMS Microbiology. 2018. V. 4. P. 665-684. https://doi.org/10.3934/microbiol.2018.4.665
- Tidona F., Francolino S., Ghiglietti R. et al. Characterization and pre-industrial validation of Streptococcus thermophilus strains to be used as starter cultures for Crescenza, an Italian soft cheese // Food Microbiology. 2020. V. 92. P. 103599. https://doi.org/10.1016/j.fm.2020.103599
- Siedler S., Rau M.H., Bidstrup S. et al. Competitive exclusion is a major bioprotective mechanism of Lactobacilli against fungal spoilage in fermented milk products // Applied and Environmental Microbiology. 2020. V. 86. P. e02312-19. https://doi.org/10.1128/AEM.02312-19
- Ben Bra?ek O., Smaoui S. Enterococci: Between Emerging Pathogens and Potential Probiotics // BioMed Research International. 2019. V. 2019. P. 5938210. https://doi.org/10.1155/2019/5938210


