Оценка биоразнообразия у межвидовых гибридов рода Ovis с использованием STR- и SNP-маркеров
Автор: Денискова Т.Е., Доцев А.В., Багиров В.А., Виммерс К., Рейер Х., Брем Г., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Молекулярные и клеточные технологии
Статья в выпуске: 2 т.52, 2017 года.
Бесплатный доступ
Интрогрессия диких и домашних родственных видов рассматривается как перспективный способ повышения генетического разнообразия в популяциях сельскохозяйственных животных. Нашей целью стало изучение влияния интрогрессии дикого вида (архара) на генетическое разнообразие межвидовых гибридов с домашними овцами с использованием STR- и SNP-маркеров. Объектом исследований были исходные родительские формы - овцы романовской породы Ovis aries (ROM, n = 35, материнская «домашняя» форма) и архары O. ammon polii (OAM, n = 10, отцовская «дикая» форма); гибридный самец F1 от скрещивания романовской овцы и архара (50 % крови архара); возвратные кроссы, полученные скрещиванием ярок романовской породы с гибридными самцами F1 (BC1, 25 % крови архара, n = 38) и BC1 (BC2, 12,5 % крови архара, n = 14). Полиморфизм 11 STR-локусов (BLT001B, CSRD247, FCB20, CSAP36, MAF65, McM147, OarCP49, D5S2, HSC, BMS2213 и INRA23) определяли на генетическом анализаторе ABI PRISM 3130xl («Applied Biosystems», США). Для генотипирования SNP использовали ДНК-чип Ovine SNP50K BeadChip («Illumina Inc.», США). После контроля качества для анализа были сформированы панели из 9 локусов STR и 8591 локусов SNP. Статистические расчеты проводили в программах GenAIEx 6.5, PLINK v1.07, HP-Rare 1.1, GENETIX 4.05 и STRUCTURE 2.3.4. Вне зависимости от используемого типа ДНК-маркеров у ROM по сравнению с OAM установлена более высокая степень генетического разнообразия, оцененного по показателям наблюдаемой гетерозиготности (Ho) и аллельного разнообразия (Ar). Гибридизация приводила к увеличению этого показателя у F1. В группах BC1 и BC2 значения Ho, рассчитанные по STR и SNP, превышали таковые у исходных родительских форм. Ar, рассчитанное по SNP, в группах BC1 и BC2 снижалось по сравнению с F1 и характеризовалось промежуточными значениями по сравнению с родительскими формами. При использовании STR четких изменений Ar в группах BC1 и BC2 не выявили. Результаты анализа главных компонент (PCA) более объективно описали распределение исследуемых животных в пространстве координат (согласно происхождению) при использовании SNP-маркеров. Уже PC1 позволяла четко дифференцировать группы OAM, ROM, F1 и BC1 + BC2. Суммарно первые две компоненты (PC1 и PC2) отвечали за 25,87 % изменчивости SNP-маркеров и лишь за 12,46 % изменчивости STR-маркеров. Компонента PC3, которая отвечала за 6,16 % изменчивости SNP-маркеров, позволяла дифференцировать группы BC1 и BC2, в то время как при использовании STR эти группы локализовались в виде общего массива. Результаты анализа в STRUCTURE показали, что объединение изучаемых особей в кластеры по STR-профилям не отражало реальной популяционной принадлежности животных, в то время как формирование кластеров на основании SNP-маркеров соответствовало фактическому происхождению индивидуумов. Таким образом, оба типа изучаемых ДНК-маркеров подходят для детектирования изменений генетического разнообразия в поколениях гибридов. Тем не менее, выявлено значительное преимущество множественных SNP-маркеров при дифференциации гибридных животных от родительских форм.
Межвидовые гибриды, интрогрессия, генетическое разнообразие, молекулярные маркеры, род ovis
Короткий адрес: https://sciup.org/142214022
IDR: 142214022 | УДК: 636.32/.38:575.113:577.2.08:51-76 | DOI: 10.15389/agrobiology.2017.2.251rus
Текст научной статьи Оценка биоразнообразия у межвидовых гибридов рода Ovis с использованием STR- и SNP-маркеров
Основные эволюционные рычаги, такие как рекомбинации и естественный отбор, действуют медленно и за редким исключением приводят к определенному эффекту по прошествии длительного времени, однако посредством интрогрессивной гибридизации эти события могут быть значительно ускорены (1-5). Интрогрессивная гибридизация представляет собой процесс включения аллелей одного вида в генофонд другого на основе повторяющегося возвратного скрещивания межвидового гибрида с одной из родительских форм (6-9). В природе интрогрессия, как правило, происходит между близкородственными видами (10, 11), а также характерна для гибридных зон, или зон контакта двух видов (1). Тем не менее, наиболь-
Работа выполнена при финансовой поддержке Российского научного фонда, проект ¹ 14-36-00039.
ший интерес представляет отдаленная, или межвидовая, гибридизация.
Интенсификация животноводства и длительная однонаправленная селекция, в том числе использование родственных линий или кроссов, приводят к снижению генетического разнообразия и усилению негативных последствий инбридинга, в частности к ухудшению адаптационных способностей животных. В этом случае дикие формы рассматриваются как уникальные резервуары генетической изменчивости для своих домашних сородичей (12).
В большинстве случаев межвидовые гибриды бесплодны и не способны передать свои уникальные свойства потомству (13). Однако существуют исключения. Так, потомки пиренейского козерога ( Capra pyrenaica ) и домашней козы ( Capra hircus ) сохраняют способность к размножению (5). Гибриды домашнего крупного рогатого скота ( Bos taurus ) с индийским скотом ( Bos indicus ) (14), американским бизоном ( Bison bison ), зубром ( Bison bonasus ) (13, 15), бантенгом ( Bos javanicus ) и гауром ( Bos gaurus ) фер-тильны (16, 17). Потомство от скрещивания домашней овцы с дикими видами — архаром ( Ovis ammon ), муфлоном ( Ovis orientalis musimon ), снежным бараном ( Ovis nivicola ) плодовито и может быть основой для создания новой селекционной формы овец, отличающейся превосходной адаптацией к сложным рельефным и природно-климатическим условиям и служащей источником диетического мяса (18, 19).
Плодовитость потомства от скрещивания овец разных пород с муфлонами была показана в многочисленных экспериментах (20, 21) и в природных условиях, что ставит под сомнение существование генетически чистых популяций этого копытного (22). В 1934-1950 годах в Казахстане на основе межвидовой гибридизации архара и домашних овец породы казахский меринос была выведена порода казахский архаромеринос, которая оказалась хорошо приспособлена к разведению в горных районах (19, 23).
Получение объективных данных о степени интрогрессии и изменении аллелофонда пород под влиянием гибридизации определяется прежде всего адекватным выбором типа ДНК-маркеров. Одним из наиболее популярных маркеров для детектирования межвидовых гибридов до недавнего времени служил полиморфизм митохондриальной ДНК (мтДНК) (1, 24-26).
Однако у этого подхода имеются существенные недостатки. Во-первых, не учитывается генетический вклад самца в интрогрессию. Во-вторых, анализ мтДНК не позволяет оценить размер и характер изменений, затрагивающих ядерную, или нуклеарную, ДНК (яДНК) гибридов. Контрастные генетические закономерности для ядерных и митохондриальных маркеров, как правило, объясняются высоким генетическим дрейфом мтДНК за счет уменьшения эффективного размера мтДНК по сравнению с яДНК (24, 25).
Микросателлиты, или STR-маркеры (short tandem repeats), — широко распространенный тип ядерных ДНК-маркеров (27). Кодоминантные, имеющие менделевский характер наследования и пригодные для анализа в автоматизированном режиме микросателлиты хорошо зарекомендовали себя для идентификации, характеристики аллелофонда и генетического разнообразия гибридов близкородственных видов (28-32). В то же время M.G. Sovic с соавт. (33) ставят под сомнение применение микросателлитов для оценки гибридизации более отдаленных видов.
С развитием современных высокопроизводительных технологий генотипирования SNP-маркеры (single nucleotide polymorphisms) становятся многообещающими инструментами для исследования интрогрессивных 252
изменений в геноме (33-35). Преимущество SNP — возможность идентификации локусов с фиксированными аллелями, специфическими для каждого из родительских видов (36, 37). Детектирование таких локусов, как правило, — несложный и быстрый процесс. Применение полногеномных ДНК-платформ позволяет найти несколько сотен локусов (38). Результаты, получаемые при сравнении двух таксонов и их гибридов с использованием диагностических локусов, однозначны и высокодостоверны (39).
Во Всероссийском НИИ животноводства им. Л.К. Эрнста создана уникальная модельная популяция межвидовых гибридов домашних овец ( O. aries ) романовской породы и архара памирской популяции ( O. ammon polii ), которая на сегодняшний день включает две генерации потомков от возвратного скрещивания романовских овец с гибридными самцами. В представленной работе впервые были детально изучены изменения генетических параметров у межвидовых гибридов по сравнению с обеими родительскими формами в популяции, полученной в искусственных, а не природных условиях.
Нашей целью стала оценка влияния интрогрессии дикого вида (архара) на генетическое разнообразие межвидовых гибридов с домашними овцами, выполненная с использованием SNP- и STR-маркеров.
Методика. Объектом исследований были исходные родительские формы — овцы романовской породы O. aries (ROM, n = 35, материнская «домашняя» форма) и архары O. ammon polii (OAM, n = 10, отцовская «дикая» форма); гибридный самец F1 от скрещивания романовской овцы и архара (50 % крови архара); возвратные кроссы, полученные скрещиванием ярок романовской породы с гибридными самцами F1 (BC1, 25 % крови архара, n = 38) и BC1 (BC2, 12,5 % крови архара, n = 14). Биоматериалом служили пробы ткани (выщип).
ДНК выделяли с помощью колонок Nexttec («Nexttec Biotechnology GmbH», Германия) и набора ДНК-Экстран (ЗАО «Синтол», Россия) согласно протоколам производителей.
Генетические исследования по STR-маркерам проводили на основе ПЦР-амплификации 11 микросателлитных локусов овец, рекомендованных ISAG (International Society for Animal Genetics) и объединенных в две мультиплексные панели — панель 1 (BLT001B, CSRD247, FCB20, CSAP36, MAF65, McM147) и панель 2 (OarCP49, D5S2, HSC, BMS2213 и INRA23). Амплификацию осуществляли в реакционной смеси (конечной объем — 20 мкл), содержащей 1 х ПЦР буфер, в состав которого входит 16,6 мМ (NH4)2SO4, 67,7 мМ Трис-HCl (pH = 8,8), 0,1 (v/v) Tween 20, а также 1,5 мМ MgCl2, 200 мкМ дНТФ, 20 пмоль каждого из праймеров, 1 U Taq ДНК-полимеразы («Диалат Лтд», Россия) и 1 мкл тестируемой ДНК (50-100 нг). После начальной денатурации (95 °С, 5 мин) проводили 35 циклов амплификации (95 °С, 20 с; 55 °С, 30 с; 72 °С, 1 мин) для панели 1 и 41 цикл амплификации (95 °С, 20 с; 63 °С, 30 с; 72 °С, 1 мин) — для панели 2. Продукты ПЦР разделяли на генетическом анализаторе PRISM 3131xl («Applied Biosystems», США). Размеры фрагментов определяли с использованием программного обеспечения GeneMapper® Software v4 («Thermo Fisher Scientific, Inc.», США).
Для полногеномного SNP-сканирования использовали ДНК-чип средней плотности Ovine SNP50K BeadChip («Illumina Inc.», США). С помощью программного обеспечения PLINK v1.07 (40) выполняли контроль качества, который включал четыре этапа. На первом этапе отбирали маркеры по показателям GenCall (GC) и GenTrain (GT), используя для каждого из SNP параметры отсечения, равные 0,5 (для GC) и 0,3 (для GT).
SNP, локализованные на половых хромосомах, и с неизвестной локализацией исключали из анализа. На втором этапе определяли полиморфные SNP для родительских форм с использованием следующих фильтров: частота минорных аллелей менее 10 % (--maf 0.01), результативность гено-типированния не менее 90 % животных (--geno 0.1), тест на равновесие по Харди-Вайнбергу p < 10-6 (--hwe 1e-6). После фильтрации отбирали только SNP, общие для обеих родительских групп. На третьем этапе были идентифицированы уникальные для каждой группы мономорфные SNP, поскольку в гибридных формах они могут стать полиморфными. После этого мономорфные SNP добавляли к отобранным на первом этапе полиморфным. На четвертом этапе к родительским формам были добавлены гибридные. Фильтрация проходила по критериям GENO (--geno 0.1) и тесту на неравновесие по сцеплению LD (--indep-pairwise 50 5 0.5): в окне из 50 SNP удалялся один из пары маркеров, для которых показатель LD был выше 0,5, после чего окно смещалось на 5 SNP. Фильтры MAF и HWE на этом этапе не применяли. После проведения LD-теста был сформирован набор SNP, используемый для статистических расчетов.
Для определения основных популяционных показателей — наблюдаемой (Ho) и ожидаемой (He) гетерозиготности, коэффициента инбридинга (FIS), индекса фиксации FST (41) и генетических дистанций M. Nei (DN) (42) по STR-маркерам использовали программное обеспечение GenAIEx 6.5.1 (43), по SNP-маркерам — GENETIX 4.05 (44). Аллельное разнообразие (Аr) для каждой группы животных по STR- и по SNP-маркерам вычисляли в программе HP-Rare 1.1 (45). Матрица дистанций идентичности по состоянию (IBS, identical-by-state), используемая для проведения анализа главных компонент (principal component analysis, PCA), была построена в программе PLINK v1.07. Для создания исходных файлов и визуализации полученных данных применяли R пакет версии 3.2.3 (46). В программе STRUCTURE 2.3.4 (47) анализировали популяционную структуру со следующими настройками калькуляций: длина burn-in периода — 50000, модель марковских цепей Монте-Карло (MCMC) — 50000 повторов, 10 итераций. Расчет проводили для коэффициента k = 2 (число предполагаемых популяций). Для каждой из групп определяли среднее значение коэффициента членства Q в i-м кластере для общего числа кластеров k (Qi/k).
Результаты. Из набора STR, используемых для анализа, были исключены локусы BLT001B и CSAP36 вследствие отсутствия информации по ним у 70 % животных в группе OAM. Финальный набор STR включал 9 локусов. Первичный анализ SNP-профилей родительских форм показал наличие 45155 полиморфных SNP у романовских овец и 9816 полиморфных SNP — у архара. После фильтрации по показателям MAF, GENO, HWE были отобраны общие для обеих групп 9002 полиморфных SNP, которые дополнили 231 уникальным для каждой группы мономорфным SNP. В итоге полученный набор состоял из 9233 маркеров. В результате контроля качества, проведенного на всей выборке c применением фильтра GENO и LD-теста, были исключены соответственно 61 и 581 SNP. Финальный набор SNP, используемый для анализа, включал 8591 маркер.
Вне зависимости от типа ДНК-маркеров у ROM по сравнению с OAM была установлена более высокая степень генетического разнообразия, оцененного по Ho и Ar (табл.). Скрещивание приводило к увеличению этого показателя у гибридов F1. В обеих группах возвратных кроссов (BC1 и BC2) значения Ho, рассчитанные по STR и SNP, превышали 254
показатели у исходных родительских форм. Величина Ar, рассчитанная по SNP, в группах BC1 и BC2 снижалась по сравнению с таковой у животных F1 и принимала промежуточные значения относительно показателей у родительских форм. При использовании STR четких изменений Ar в группах BC1 и BC2 мы не выявили.
Параметры генетического разнообразия родительских и межвидовых гибридных форм рода Ovis по STR- и SNP-маркерам
|
Маркер |
Группа |
n |
] Ho 1 |
H e 1 |
F IS |
1 Ar |
|
STR |
OAM |
10 |
0,544±0,107 |
0,683±0,058 |
0,266 |
1,719±0,061 |
|
ROM |
35 |
0,656±0,040 |
0,775±0,028 |
0,154 |
1,778±0,147 |
|
|
F 1 |
1 |
0,778 |
1,786±0,028 |
|||
|
BC 1 |
38 |
0,837±0,026 |
0,680±0,020 |
- 0,242 |
1,689±0,021 |
|
|
BC 2 |
14 |
0,745±0,074 |
0,757±0,025 |
0,027 |
1,785±0,027 |
|
|
SNP |
OAM |
10 |
0,270±0,002 |
0,280±0,002 |
0,038 |
1,296±0,002 |
|
ROM |
35 |
0,369±0,002 |
0,368±0,001 |
- 0,004 |
1,458±0,005 |
|
|
F 1 |
1 |
0,458 |
1,373±0,001 |
|||
|
BC 1 |
38 |
0,418±0,002 |
0,357±0,002 |
- 0,172 |
1,362±0,002 |
|
|
BC 2 |
14 |
0,433±0,003 |
0,344±0,002 |
- 0,260 |
1,357±0,002 |
П р и м е ч а н и е. Hо — наблюдаемая гетерозиготность, Hе — ожидаемая гетерозиготность, FIS — индекс фиксации, Ar — аллельное разнообразие; значения Ho и Ar для SNP-маркеров были достоверны при p > 0,999. OAM — архары (Ovis ammon polii), ROM — овцы романовской породы (O. aries), F1 — гибридный самец от скрещивания романовской овцы и архара (50 % крови архара), BC1 и BC2 — возвратные кроссы, полученные скрещиванием ярок романовской породы с гибридными самцами F1 (25 % крови архара) и BC1 (12,5 % крови архара). Данные для F1 не приводятся, поскольку группа F1 представлена одной особью.
Сходные тенденции по STR- и SNP-маркерам отмечали при оценке динамики гетерозогот в исследуемых генерациях. Так, в группе OAM был выявлен дефицит гетерозигот — значительный по STR-маркерам (13,9 %) и небольшой по SNP-маркерам (1,0 %). Группа ROM по SNP-маркерам находилась практически в равновесии, в то время как анализ по STR показал снижение дефицита гетерозигот до 11,9 %. Оба типа маркеров указывали на избыток гетерозигот у BC1, в то время как в BC2 наблюдались обратные тенденции: дефицит гетерозигот, оцененный по STR, и увеличение избытка гетерозигот, оцененного по SNP.
Результаты анализа главных компонент (рис. 1), проведенного на основании ДНК-профилей по двум типам генетических маркеров, показали более объективную картину распределения исследуемых животных в пространстве координат согласно их происхождению при использовании SNP-маркеров. Суммарно первые две компоненты (PC1 и PC2) отвечали за 25,87 % изменчивости SNP-маркеров и лишь за 12,46 % изменчивости STR-маркеров. Третья компонента (PC3) позволяла дополнительно объяснить соответственно 6,16 и 4,45 % изменчивости.
Формирование кластеров по STR-маркерам носило нечеткий характер, были выявлены животные, локализованные в «чужом» кластере (рис. 1, А, Б). При использовании SNP-маркеров уже PC1 позволяла четко дифференцировать группы OAM, ROM, F1 и BC1 + BC2 (см. рис. 1, В). Интересным было равноудаленное положение от обеих родительских форм гибрида F1, что полностью соответствует его происхождению. PC3 позволила четко разделить возвратные кроссы BC1 и BC2 (см. рис. 1, Г).
Сравнительный анализ степени генетической дифференциации изучаемых групп по FST и DN показал четкое соответствие характера генетических связей происхождению животных при использовании SNP-маркеров: максимальные различия наблюдались между исходными видами (FST = 0,280; DN = 0,250). У гибридных особей по мере снижения кровно-сти архара наблюдалось увеличение генетических различий по отношению к архару (FST = 0,223, DN = 0,174 для BC1; FST = 0,272, DN = 0,218 для
BC2), что приближало их к домашней форме. Характер генетических связей между группами, установленный по STR-маркерам, не соответствовал происхождению животных. Возможно, это объясняется недостаточной информационной емкостью девяти STR-локусов для исследовательских работ такого направления.
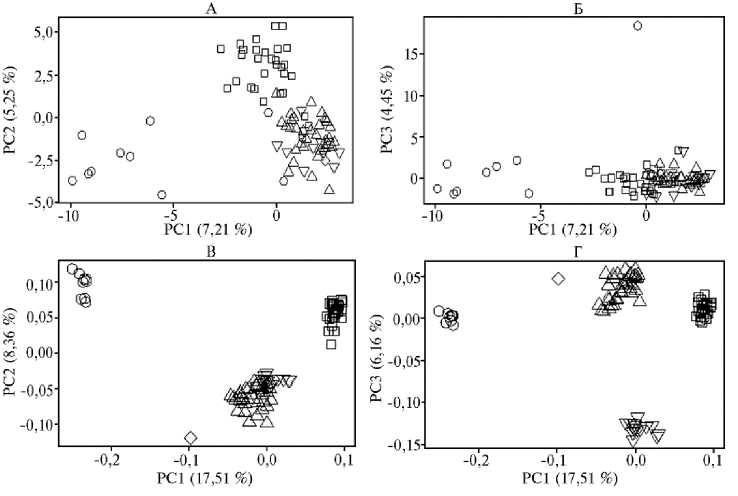
Рис. 1. Результаты PCA-анализа ( principal component analysis ) по 9 STR-локусам (А, Б) и по 8591 SNP-маркерам (В, Г) в пространстве главных компонент 1 и 2 (А, В) , а также 1 и 3 (Б, Г) для родительских и межвидовых гибридных форм рода Ovis : о — OAM, архары ( Ovis ammon polii ), о — ROM, овцы романовской породы ( O. aries ), о — F 1 , гибридный самец от скрещивания романовской овцы и архара (50 % крови архара), АиV — BC 1 и BC 2 , возвратные кроссы, полученные скрещиванием ярок романовской породы с гибридными самцами F 1 (25 % крови архара) и BC 1 (12,5 % крови архара).
Наиболее эффективным на сегодняшний день методом выявления гибридов многие авторы (29, 47-49) считают кластерный анализ адмикс-моделей на основе данных по мультилокусным маркерам, реализуемый в программе STRUCTURE. В нашей работе объединение изучаемых особей в кластеры на основании STR-профилей не соответствовало их происхождению как при k = 2 (рис. 2, А), так и при k = 3 (данные не приведены). Это объясняется двумя основными причинами. Во-первых, используемые микросателлиты открыты и рекомендованы для генетического анализа домашних овец, а не архара. Во-вторых, информационной емкости девяти маркеров могло не хватить для проведения подобных исследований. Так, по расчетам J.P. Vaha и C.R. Primmer (29), для адекватного отделения возвратных кроссов от чистопородных родительских особей требуется не менее 48 локусов.
STRUCTURE анализ по 8591 отобранному SNP-маркеру показал, что формирование кластеров соответствовало фактическому происхождению индивидуумов (см. рис. 2, Б). Родительские формы (дикая и домашняя) сгруппировались в два строго консолидированных кластера. Значение членства в собственном кластере для архаров составило Q1/2 = 0,94±0,04, для романовских овец — Q2/2 = 0,99±0,01. Гибрид F1, несущий по 50 % крови дикой и домашней форм, характеризовался приблизительно равными значениями членства в каждом из двух кластеров — Q1/2 = 0,54 и 256
Q2/2 = 0,46. В поколениях возвратных кроссов происходило снижение доли членства в кластере, специфическом для OAR (Q1/2 = 0,27±0,05 в группе BC1 и Q1/2 = 0,22±0,04 в группе BC2) и увеличение доли членства в кластере, специфическом для ROM (Q2/2 = 0,73±0,05 в группе BC1 и Q2/2 = 0,78±0,04 в группе BC2).
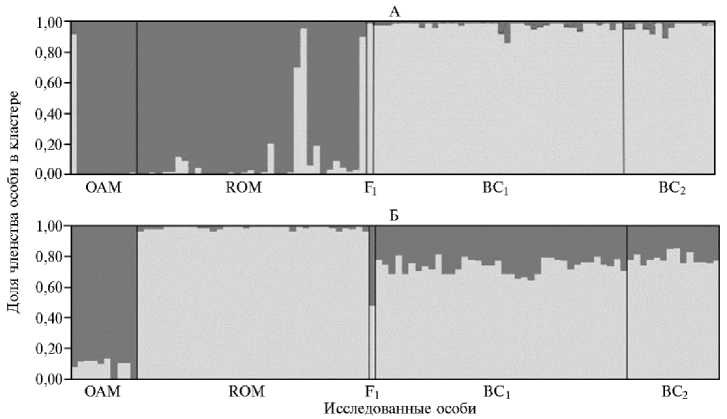
Рис. 2. Популяционная принадлежность родительских и межвидовых гибридных форм рода Ovis на основании анализа 9 STR-локусов (А) и 8591 SNP-маркера (Б) в программе STRUCTURE при k = 2: OAM — архары ( Ovis ammon polii ), ROM — овцы романовской породы ( O. aries ), F 1 — гибридный самец от скрещивания романовской овцы и архара (50 % крови архара), BC 1 и BC 2 — возвратные кроссы, полученные скрещиванием ярок романовской породы с гибридными самцами F 1 (25 % крови архара) и BC 1 (12,5 % крови архара).
Таким образом, изменение генетического разнообразия в поколениях межвидовых гибридов вследствие влияния интрогрессии архара может быть установлено как по STR-, так и по SNP-маркерам. Тем не менее, выявлено значительное преимущество использования множественных SNP-маркеров для дифференциации гибридных животных от родительских форм. В дальнейшем планируется более детальный поиск диагностических SNP локусов, необходимых для детального изучения интрогрессии в более отдаленных от дикого родоначальника поколениях.
Список литературы Оценка биоразнообразия у межвидовых гибридов рода Ovis с использованием STR- и SNP-маркеров
- Harrison R.G. Hybrid zones: windows on evolutionary process. Oxford Surveys in Evolutionary Biology, 1990, 7: 69-128.
- Arnold M.L. Evolution through genetic exchange. Oxford University Press, Oxford, UK, 2006.
- Martinsen G.D., Whitham T.G., Turek R.J., Keim P. Hybrid populations selectively filter gene introgression between species. Evolution, 2001, 55(7): 1325-1335 (doi: 10.1554/0014-3820(2001)0552.0.CO;2).
- Schwenk K., Brede N., Streit B. Introduction. Extent, processes and evolutionary impact of interspecific hybridization in animals. Philosophical Transactions of the Royal Society B: Biological Sciences, 2008, 363: 2805-2811 ( ) DOI: 10.1098/rstb.2008.0055
- Reza M.Md.T., Lee S.-J., Singh N.K. Interspecies hybridization in animals: An overview. Annals of Animal Resource Sciences, 2012, 23(2): 149-163 ( ) DOI: 10.12718/AARS.2012.23.2.149
- Anderson E., Hubricht L. Hybridization in tradescantia. III. The evidence for introgressive hybridization. Am. J. Bot., 1938, 25(6): 396-402 ( ) DOI: 10.2307/2436413
- Anderson E. Introgressive hybridization. Wiley & Sons, NY, 1949.
- Harrison R.G., Larson E.L. Hybridization, introgression, and the nature of species boundaries. J. Hered., 2014, 05(S1): 795-809 ( ) DOI: 10.1093/jhered/esu033
- Falniowski A. Introgression: in a contact zone of two closely related species? Not necessarily. International Journal of Pure and Applied Zoology, 2016, 4(3): 289-290.
- Katoh M., Ribi G. Genetic evidence for natural hybridization and apparent introgression between freshwater snail species (Viviparusater and V. contectus). J. Evolution. Biol., 1996, 9: 67-82.
- Randi E. Detecting hybridization between wild species and their domesticated relatives. Mol. Ecol., 2008, 17(1): 285-293 ( ) DOI: 10.1111/j.1365-294X.2007.03417.x
- Насибов Ш.Н., Багиров В.А., Кленовицкий П.М., Иолчиев Б.С., Зиновьева Н.А., Воеводин В.А., Амиршоев Ф.С. Генетический потенциал дикой фауны в создании новых селекционных форм животных. Достижения науки и техники АПК, 2010, 8: 59-62. Режим доступа: http://elibrary.ru/item.asp?id=15198540. Без даты.
- Anderson G.B. Interspecific pregnancy: barriers and prospects. Biol. Reprod., 1988, 38: 1-15 ( ) DOI: 10.1095/biolreprod38.1.1
- Nahar T.N., Islam M., Hasnath M.A. A comparative study on the performances of F1 crossbred cows under rural conditions. Asian-Australas. J. Anim. Sci., 1992, 5(3): 435-438 ( ) DOI: 10.5713/ajas.1992.435
- Kochhar H.P.S., Appa Rao K.B.C., Luciano A.M., Totey S.M., Gandolfi F., Basrur P.K., King W.A. In vitro production of cattle-water buffalo (Bos taurus-Bubalus bubalis) hybrid embryos. Zygote, 2002, 10: 155-162 ( ) DOI: 10.1017/S0967199402002216
- Lanza R.P., Cibelli J.B., Diaz F., Moraes C.T., Farin P.W., Farin C.E., Hammer C.J., West M.D., Damiani P. Cloning of an endangered species (Bos gaurus) using interspecies nuclear transfer. Cloning, 2000, 2: 79-90 ( ) DOI: 10.1089/152045500436104
- Loi P., Modlinski J.A., Ptak G. Interspecies somatic cell nuclear transfer: a salvage tool seeking first aid. Theriogenology, 2011, 76: 217-228 ( ) DOI: 10.1016/j.theriogenology.2011.01.016
- Багиров В.А., Эрнст Л.К., Насибов Ш.Н., Кленовицкий П.М., Иолчиев B.C., Зиновьева Н.А. Сохранение биоразнообразия животного мира и использование отдаленной гибридизации в животноводстве. Достижения науки и техники АПК, 2009, 7: 54-56. Режим доступа: http://elibrary.ru/item.asp?id=13002971. Без даты.
- Багиров В.А., Кленовицкий П.М., Иолчиев Б.С. Отдаленная гибридизация в овцеводстве. Сборник научных трудов Всероссийского научно-исследовательского института овцеводства и козоводства, 2012, 2(1): 15-25. Режим доступа: http://elibrary.ru/item.asp?id=17833720. Без даты.
- Mavrogenis A.P., Herzog A. Interspecific hybridization of the Cyprus mouflon (Agrinon) with domestic sheep. Animal Genetic Resources Information (FAO/UNEP), 1990, 7: 24-29
- Loi P., Ptak G., Fulka J.J., Cappai P., Clinton M. Genetic rescue of an endangered mammal by cross-species nuclear transfer using post-mortem somatic cells. Nat. Biotechnol., 2001, 19: 962-964.
- Schröder O., Lieckfeldt D., Lutz W., Rudloff C., Frölich K., Ludwig A. Limited hybridization between domestic sheep and the European mouflon in Western Germany. Eur. J. Wildlife Res., 2016, 62(3): 307-314 ( ) DOI: 10.1007/s10344-016-1003-3
- Бутарин Н.С. Породы сельскохозяйственных животных, выведенные в Казахстане. Алма-Ата, 1960.
- Alves P.C., Melo-Ferreira J., Freitas H., Boursot P. The ubiquitous mountain hare mitochondria: multiple introgressive hybridization in hares, genus Lepus. Philosophical Transactions of the Royal Society B: Biological Sciences, 2008, 363: 2831-2839 ( ) DOI: 10.1098/rstb.2008.0053
- Vigfúsdóttir F., Pálsson S., Ingólfsson A. Hybridization of glaucous gull (Larus hyperboreus) and herring gull (Larus argentatus) in Iceland: mitochondrial and microsatellite data. Philosophical Transactions of the Royal Society B: Biological Sciences, 2008, 363: 2851-2860 ( ) DOI: 10.1098/rstb.2008.0042
- Cabria M.T., Michaux J.R., Gómez-Moliner B.J., Skumatov D., Maran T., Fournier P., López de Luzuriaga J., Zardoya R. Bayesian analysis of hybridization and introgression between the endangered European mink (Mustela lutreola) and the polecat (Mustela putorius). Mol. Ecol., 2001, 20(6): 1176-1190 ( ) DOI: 10.1111/j.1365-294X.2010.04988.x
- Tautz D., Renz M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes. Nucleic Acids Res., 1984, 12: 4127-4138 ( ) DOI: 10.1093/nar/12.10.4127
- Lecis R., Pierpaoli M., Birò Z. S., Szemethy L., Ragni B., Vercillo F., Randi E. Bayesian analyses of admixture in wild and domestic cats (Felis silvestris) using linked microsatellite loci. Mol. Ecol., 2006, 15(1): 119-131 ( ) DOI: 10.1111/j.1365-294X.2005.02812.x
- Vähä J.P., Primmer C.R. Efficiency of model-based Bayesian methods for detecting hybrid individuals under different hybridization scenarios and with different numbers of loci. Mol. Ecol., 2006, 15: 63-72 ( ) DOI: 10.1111/j.1365-294X.2005.02773.x
- Verardi A., Lucchini V., Randi E. Detecting introgressive hybridization between free-ranging domestic dogs and wild wolves (Canis lupus) by admixture linkage disequilibrium analysis. Mol. Ecol., 2006, 15(10): 2845-2855 ( ) DOI: 10.1111/j.1365-294X.2006.02995.x
- Oliveira R., Randi E., Mattucci F., Kurushima J.D., Lyons L.A., Alves P.C. Toward a genome-wide approach for detecting hybrids: informative SNPs to detect introgression between domestic cats and European wildcats (Felis silvestris). Heredity, 2015, 115: 195-205 ( ) DOI: 10.1038/hdy.2015.25
- Kharzinova V.R., Dotsev A.V., Kramarenko A.S., Layshev K.A., Romanenko T.M., Solovieva A.D., Deniskova T.E., Kostunina O.V., Brem G., Zinovieva N.A. Study of the allele pool and the degree of genetic introgression of semi-domesticated and wild populations of reindeer (Rangifer tarandus L., 1758) using microsatellites Russia. Agricultural Biology, 2016, 51(6): 811-823 ( ) DOI: 10.15389/agrobiology.2016.6.811eng
- Sovic M.G., Kubatko L.S., Fuerst P.A. The effects of locus number, genetic divergence, and genotyping error on the utility of dominant markers for hybrid identification. Ecology and Evolution, 2014, 4(4): 462-473 ( ) DOI: 10.1002/ece3.833
- Morin P.A., Luikart G., Wayne R.K. SNPs in ecology, evolution and conservation. Trends in Ecology and Evolution, 2004, 19(4): 208-216 ( ) DOI: 10.1016/j.tree.2004.01.009
- Kalinowski S.T. How to use SNPs and other diagnostic diallelic genetic markers to identify the species composition of multi-species hybrids. Conservation Genetics Resources, 2010, 2: 63-66 ( ) DOI: 10.1007/s12686-009-9146-0
- Zinovieva N.A., Dotsev A.V., Sermyagin A.A., Wimmers K., Rayer H., Sölkner J., Deniskova T.E., Brem G. Study of genetic diversity and population structure of five Russian cattle breeds using whole-genome SNP analysis. Agricultural Biology, 2016, 51(6): 788-800 ( ) DOI: 10.15389/agrobiology.2016.6.788eng
- Finger A.J., Stephens M.R., Clipperton N.W., May B. Six diagnostic single nucleotide polymorphism markers for detecting introgression between cutthroat and rainbow trouts. Mol. Ecol. Resour., 2009, 9(3): 759-763 ( ) DOI: 10.1111/j.1755-0998.2009.02532.x
- Stephens M.R., Clipperton N.W., May B. Subspecies-informative SNP assays for evaluating introgression between native golden trout and introduced rainbow trout. Mol. Ecol. Resour., 2009, 9(1): 339-343 ( ) DOI: 10.1111/j.1755-0998.2008.02407.x
- Goedbloed D.J., van Hooft P., Megens H.-J., Langenbeck K., Lutz W., Crooijmans R. PMA, van Wieren S.E., Ydenberg R.C., Prins H.H.T. Reintroductions and genetic introgression from domestic pigs have shaped the genetic population structure of Northwest European wild boar. BMC Genetics, 2013, 14: 43 ( ) DOI: 10.1186/1471-2156-14-43
- Purcell S., Neale B., Todd-Brown K. Thomas L., Ferreira M.A.R., Bender D., Maller J., Sklar P., de Bakker P.I.W., Daly M.J., Sham P.C. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am. J. Hum. Genet., 2007, 81: 559-575 ( ) DOI: 10.1086/519795
- Weir B.S., Cockerham C.C. Estimating F-Statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358-1370 ( ) DOI: 10.2307/2408641
- Nei M. Genetic distance between populations. American Naturalist, 1972, 106: 283-392 ( ) DOI: 10.1086/282771
- Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research -an update. Bioinformatics, 2012, 28: 2537-2539 ( ) DOI: 10.1093/bioinformatics/bts460
- Belkhir K., Borsa P., Chikhi L., Raufaste N., Bonhomme F. GENETIX 4.05, Population Genetics Software for Windows TM. Université de Montpellier II, Montpellier, France, 2004.
- Kalinowski S.T. HP RARE 1.0: a computer program for performing rarefaction on measures of allelic richness. Mol. Ecol. Notes, 2005, 5: 187-189 ( ) DOI: 10.1111/j.1471-8286.2004.00845.x
- R Development Core Team. R: A language and environment for statistical computing. 2009. R Foundation for Statistical Computing, Vienna, Austria. ISBN 3-900051-07-0. Режим доступа: http://www.Rproject.org. Без даты.
- Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155: 945-959.
- Anderson E.C., Thompson E.A. A model-based method for identifying species hybrids using multilocus genetic data. Genetics, 2002, 160: 1217-1229.
- Corander J., Marttinen P. Bayesian identification of admixture events using multilocus molecular markers. Mol. Ecol., 2006, 15(10): 2833-2843 ( ) DOI: 10.1111/j.1365-294X.2006.02994.x


