Оценка генетического разнообразия образцов капусты кочанной (Brassica oleracea L.) с использованием SSR маркеров
Автор: Домблидес А.С., Бондарева Л.Л., Пивоваров В.Ф.
Журнал: Сельскохозяйственная биология @agrobiology
Статья в выпуске: 5 т.55, 2020 года.
Бесплатный доступ
Из всех разновидностей капусты в Российской Федерации наиболее распространены традиционные сорта и гибриды капусты кочанной Brassica oleracea L . сonvar. capitata (L.). Классификация селекционного материала с использованием ДНК маркеров позволяет выделить ценные генотипы и установить между ними генетические взаимосвязи для последующей селекции на генетически отличимые формы. Микросателлитные маркеры (simple sequence repeats, SSR) широко используют для генетической идентификации и генотипирования сельскохозяйственных культур. Работы на B. oleracea L. показали их высокую эффективность по выявлению полиморфизма между разновидностями, сортами и внутри сортов. В настоящем исследовании мы впервые установили генетические взаимосвязи между селекционными образцами капусты кочанной отечественной селекции на основе полиморфизма микросателлитных локусов. При сравнении трех разновидностей обнаружена тесная генетическая близость между генотипами савойской и белокочанной капусты. Целью работы было выявление генетических взаимоотношений между селекционными образцами Brassica oleracea L. сonvar. capitata (L.) Alef. var. capitata L. f. alba , var. capitata L. f. rubra и var. sabauda L. на основе типирования и генетической классификации с помощью SSR маркеров, а также сопоставление данных ДНК-анализа с принадлежностью изученных генотипов к соответствующими сортотипам и группами спелости. Объектом исследования были 24 селекционных образца капусты кочанной из коллекции Федерального научного центра овощеводства (ФГБНУ ФНЦО), включая краснокочанную и савойскую разновидности, выведенные в ФГБНУ ФНЦО. Геномную ДНК выделяли из молодых листьев растений в фазу 2-3-го листа с использованием набора реагентов Сорб-ГМО-Б (ООО «Синтол», Россия). Для оценки чистоты и концентрации выделенной ДНК использовали спектрофотометр SmartSpec Plus («Bio-Rad», США). Для осуществления SSR-анализа отобрали 21 микросателлитный локус с известными последовательностями праймеров. Амплификацию осуществляли на приборе С1000 Touch («Bio-Rad», США). ПЦР-продукты разделяли методом вертикального электрофореза с использованием системы Sequi-Gen GT («Bio-Rad», США) в 6 % полиакриламидном секвенирующем геле. Размеры амплифицированных фрагментов определяли в сравнении с маркером молекулярных масс GeneRuler100 bp plus DNA ladder («Thermo Fisher Scientific», США). Полученные цифровые фотографии электрофореграмм анализировали в программе Image Lab 3.0 («Bio-Rad», США). Структуру популяции изучали в программе STRUCTURE 2.3.4 (https://web.stanford.edu/group/pritchardlab/home.html). Расчет генетических расстояний проводили в программе GenAlEx 6.5 для Microsoft Excel по методу M. Nei. Для построения UPGMA дендрограммы использовали алгоритм программы MEGA5.2. В результате анализа получили 103 аллеля со средним показателем 4,9 на локус. Размеры продуктов амплификации составляли 130-410 п.н. Величина информационного полиморфизма (PIC) для праймеров варьировала от 0,3 до 0,9. Анализ популяции выявил, что все образцы распределились по шести основным кластерам. Генетические дистанции варьировали от 0,060 до 0,186. Дендрограмма UPGMA, построенная на основе генетических дистанций, отражала происхождение исследуемых селекционных образцов. Так, сорта Белорусская 455, Подарок 2500, Амагер 611 и Зимовка 1474 с происхождением из Северо-Западной Европы, были объединены в общий кластер, где также находился гибрид Северянка F1, созданный с использованием этих сортов. Сорта, принадлежащие к сортотипу Дитмарская ранняя, - Июньская 3200, Стахановка 1513, Номер первый грибовский 147 образовали отдельный кластер, куда также входил раннеспелый гибрид Аврора F1, имеющий в своей родословной сорт Июньская 3200. Две селекционные линии, выделенные из гибрида Аврора F1, были генетически отдалены и располагались в другом подкластере. Сорта Слава 1305 и Слава грибовская 231, относящиеся к отдельному сортотипу Слава, располагались на отдельной ветви дендрограммы. Относительно недавно полученные сорт Парус и два гибрида Зарница F1 и Мечта F1 генетически отличались от остальных образцов. Московская поздняя 15, стародавний сорт местного происхождения, также образовал отдельную ветвь дендрограммы. Три сорта капусты савойской сгруппировались вместе, причем новый раннеспелый сорт Московская кружевница был генетически отдален от двух других. Образцы капусты краснокочанной образовывали свою группу с достаточным генетическим отдалением между образцами. Полученные результаты на основе изменчивости SSR локусов совпадали с данными о происхождении образцов капусты кочанной, подтверждая их принадлежность к определенным сортотипам и группам спелости, что позволит использовать этот селекционный материал в дальнейшем для получения новых форм.
Brassica oleracea l, капуста кочанная, ssr маркеры, генетическая идентификация, сортовой полиморфизм, сортотип
Короткий адрес: https://sciup.org/142229433
IDR: 142229433 | УДК: 635.34:577.2 | DOI: 10.15389/agrobiology.2020.5.890rus
Текст научной статьи Оценка генетического разнообразия образцов капусты кочанной (Brassica oleracea L.) с использованием SSR маркеров
Китай, Индия и Российская Федерация — основные производители капусты в мире, причем Россия оставляет за собой лидерство в ее потреблении (1). Капуста обладает антиоксидантными свойствами благодаря содержанию полифенолов, антоцианов, галловой, ванилиновой и кумаровой кислот. В ее биохимический состав входят такие антивоспалительные вещества, как глутамин и флавоноиды. Капуста также служит прекрасным источников витамина С и глюкозинолатов (2).
Из вида Brassica oleracea L. (CC, 2 n = 18) наиболее известна капуста белокочанная B. oleracea L. сonvar. capitata L. Alef. var. capitata (L.) f. alba DC. как наиболее употребляемая в России. В меньшей степени известны краснокочанная B. oleracea L. сопуаг. capitata (L.) Alef. var. capitata (L.) f. rubra (L.) Thell., савойская B. oleracea L. convar. capitata (L.) Alef. var . sabauda L., цветная B. oleracea L. convar. botrytis (L.) Alef. var. botrytis L., брокколи B. oleracea L. convar. botrytis (L.) Alef. var. italica Plenck, брюссельская B. oleracea L. convar. gemmifera (DC.) Gladis var. gemmifera DC., кольраби B. oleracea L. convar. acephala (DC.) Alef. var. gongylodes L. и декоративная листовая B. oleracea L . convar. acephala (DC.) Alef. var. sabellica L. Сорта и высокопродуктивные гибриды — ожидаемый результат селекционной работы с видом B. oleracea L. Отличительная особенность кочанных капуст сonvar. capitata — наличие форм с наиболее длительным периодом яровизации и морозоустойчивостью. Первоначальное формирование биоразнообразия капусты кочанной происходило в Западном Средиземноморье, а также в Западной и Северной Европе, откуда она распространилась в Восточную Европу, Америку, страны Азии (Китай, Япония), Австралию и Африку. Ее широкому распространению способствовали такие свойства, как высокая урожайность и транспортабельность. Капуста кочанная характеризуется значительным сортовым разнообразием по скороспелости, не требовательна к теплу, обладает положительной реакцией на длинный световой день и отзывчива на применение технологий возделывания, позволяющих увеличить период вегетации (3).
Для рационального использования генетических ресурсов и получения новых форм с высоким показателем гетерозиса и стабильности проявления хозяйственно ценных признаков необходимо знать генетическую основу используемого материала. Кроме того, с появлением нового селекционного материала нужно различать все генотипы и идентифицировать среди них одинаковые или близкородственные. Использование ДНК маркеров на основе микросателлитных повторов (simple sequence repeats, SSR) облегчает задачу точной генетической классификации селекционных образцов. Преимущества микросателлитных маркеров над другими заключаются в надежности и выявлении высокого генетического полиморфизма, что делает их наиболее популярными и универсальными в генетических исследованиях сельскохозяйственных культур (4-6).
Накопление информации по изменчивости микросателлитных локусов привело к созданию базы данных, где общее количество микросателлитных маркеров для видов рода Brassica L., включая B. oleracea, достигало 398 (7). С использованием этих данных были установлены и подтверждены генетические взаимосвязи между видами рода Brassica (8-10). Проведен генетический анализ внутри вида B. oleracea и получены сведения о взаимосвязях как между разновидностями капусты, так и среди сортов и селекционных линий (11-14). Филогенетические исследования вида B. oleracea также проводили на основе изменчивости микросателлитов (15). Коллек- цию из 91 коммерческого образца капусты белокочанной проанализировали по 69 микросателлитным маркерам, которые выявили 359 аллелей, разделенных на 6 кластеров. В первые два входили генотипы капусты белокочанной, в остальные — генотипы разновидностей брокколи, цветной, кольраби и листовой капусты (16). Среди различных селекционных образцов капусты белокочанной была определена высокая генетическая изменчивость с возможностью точного типирования каждого растения на основе полиморфизма микросателлитов (17).
Обобщая результаты предыдущих исследований, нужно отметить, что число работ по изучению генетического разнообразия традиционных отечественных сортов и гибридов капусты кочанной ограничено. Сорта, выведенные и районированные с участием Е.М. Поповой еще в 1940-х годах на Грибовской опытной станции, и сегодня используются как исходный селекционный материал для получения новых форм.
В настоящей работе мы впервые установили генетические взаимосвязи между селекционными образцами капусты кочанной отечественной селекции на основе полиморфизма микросателлитных локусов. При сравнении трех разновидностей обнаружена тесная генетическая близость между генотипами савойской и белокочанной капусты.
Нашей целью было выявление генетических взаимоотношений между селекционными образцами Brassica oleracea L. сonуar. capitata (L.) Alef. уаг. capitata L. f. alba , var. capitata L. f. rubra и var. sabauda L. на основе типирования и генетической классификации с помощью SSR маркеров, а также сопоставление данные ДНК-анализа с принадлежностью изученных генотипов к соответствующими сортотипам и группами спелости.
Методика . Объектом исследования были 24 селекционных образца капусты кочанной из генетической коллекции (Уникальная научная установка, ВНИИССОК) Федерального научного центра овощеводства (ФГБНУ ФНЦО). Геномную ДНК выделяли из молодых листьев растений в фазу 23-го листа методом на основе СТАВ буфера с использованием набора реагентов Сорб-ГМО-Б (ООО «Синтол», Россия) согласно прилагаемому протоколу производителя. Перед выделением ткань молодых листочков (от пяти растений каждого образца) подвергали гомогенизации в экстракционном буфере на приборе TissueLyser II («Qiagen», Германия) (частота 26 Гц, 1560 колебаний/мин, продолжительность 1,7 мин). Конечную чистоту и концентрацию выделенной ДНК определяли на спектрофотометре SmartSpec Plus («Bio-Rad», США). Для проведения амплификации использовали растворы ДНК с OD 260/280 = 1,6-1,8.
Для осуществления SSR-анализа был отобран 21 микросателлитный локус с известными последовательностями праймеров (11, 12), которые в предыдущих исследованиях показывали значения PIC (рolymorphism information content, величина информационного полиморфизма) не менее 0,5. Базовую постановку ПЦР проводили в реакционной смеси (объем 25 мкл), которая включала 2,5 мкл 10 х ПЦР буфера, 2,5 мМ MgCl 2 , 0,25 мМ каждого dNTP, 0,3 мкМ каждого праймера, 1,5 ед. Taq ДНК-полимеразы (ООО «Синтол», Россия) и 3 мкл ДНК каждого исследуемого образца. Основной протокол амплификации включал следующие этапы: 45 с при 92-95 ° С (денатурация), 30 с при температуре от 52 до 58 ° С в зависимости от пары праймеров (отжиг), от 30 с до 1 мин при 72 ° С (элонгация). Амплификацию, рассчитанную на 35 циклов, осуществляли на приборе С1000 Touch («Bio-Rad», США).
ПЦР-продукты разделяли методом вертикального электрофореза с использованием системы Sequi-Gen GT («Bio-Rad», США) в 6 % поли-892
акриламидном секвенирующем геле при напряжении 1600 В в течение 1,52 ч. После электрофореза гели окрашивали красителем для гелей SYBR™ Safe DNA Gel Stain («Invitrogen», США), результаты документировали с использованием системы ChemiDoc XRS+ («Bio-Rad», США). Размеры ампли-фицированных фрагментов определяли в сравнении с маркером молекулярных масс GeneRulerlOO bp plus DNA ladder («Thermo Fisher Scientific», США). Полученные цифровые фотографии электрофореграмм анализировали в программе Image Lab 3.0 («Bio-Rad», США).
Для каждого SSR-локуса учитывали присутствие и отсутствие продукта (соответственно 1 и 0) для составления итоговой бинарной матрицы. Для каждого праймера рассчитывали значение PIC (18). Структуру популяции изучали в программе STRUCTURE 2.3.4 (18), для определения значения ДК использовали программу STRUCTURE HARVESTER v0.6.1 (20). Анализ данных в STRUCTURE не предусматривал информации о числе кластеров, поэтому их вероятное число определяли от 1 до 12 с последующим сравнением К для каждого из вариантов. Генетические расстояния рассчитывали в программе GenAlEx 6.5 (21) для Microsoft Excel по методу M. Nei (22, 23). Для построения UPGMA дендрограммы использовали алгоритм программы MEGA5.2 (24).
Результаты. Изученные образцы и разновидности капусты кочанной приведены в таблице 1, характеристики полиморфных микросателлитных локусов для вида B. oleracea , использованных в работе, указаны в таблице 2.
1. Селекционные образцы, отобранные для оценки генетического разнообразия капусты кочанной ( Brassica oleracea L.) с использованием SSR маркеров (коллекция ФГБНУ Федерального научного центра овощеводства)
Название | Сортотип | Обозначение
Образцы eonvar. capitata (L.) Alef. var. capitata L. f. alba DC
|
Амагер 611 |
Амагер |
Ama |
|
Аврора F 1 |
Дитмарская ранняя |
Avr |
|
Аврора F 1 линия 1 |
Дитмарская ранняя |
AvL1 |
|
Аврора F 1 линия 2 |
Дитмарская ранняя |
AvL2 |
|
Белорусская 455 |
Белорусская |
Bel |
|
Зарница F 1 |
Дитмарская ранняя |
Zar |
|
Зимовка 1474 |
Лангендейская зимняя |
Zim |
|
Июньская 3200 |
Дитмарская ранняя |
Iju |
|
Московская поздняя 15/репродукция Московская поздняя |
MoPo/MoPo1 |
|
|
Мечта F 1 |
Лангендейская зимняя |
Mech |
|
Номер первый грибовский 147 |
Дитмарская ранняя |
N1 |
|
Парус/репродукция |
Голландская группа |
Par/Par4 |
|
Подарок 2500 |
Белорусская |
Pod |
|
Северянка F 1 |
Белорусская |
Sev |
|
Слава 1305 |
Слава |
Sl13 |
|
Слава грибовская 231 |
Слава |
Sl23 |
|
Стахановка 1513 |
Дитмарская ранняя Образцы var. sabauda L. |
Sta |
|
Вертю 1340 |
Вертю |
Ver |
|
Московская кружевница |
Розеточная |
MosKr |
|
Юбилейная 2170 |
Ульмская Образцы var. capitata L. f. rubra |
Ubi |
|
Гако 741 |
Гако |
Gak |
|
Каменная головка 447 |
Эрфуртская |
KamG |
|
Рубин |
Турновская |
Rub |
|
Краснокочанная линия I3 |
Эрфуртская |
RedL1 |
|
Примечание. Репродукция - |
- тот же образец, полученный из элитных семян. |
|
При амплификации 21 микросателлитного локуса было получено 103 аллеля со средним числом 4,9 для каждого локуса. При этом для изученных образцов выявили 13 специфичных аллелей. Наибольшее число специфичных аллелей (семь) было найдено у образцов Северянка F1, Бело- русская 455, Подарок 2500, Амагер 611 и Зимовка 1474. Размеры продуктов амплификации варьировали от 130 до 410 п.н. Наиболее информативные SSR-локусы AF458409 и BZ523957 с индексом информативности для праймеров соответственно 0,90 и 0,86 были представлены пятью аллелями (рис. 1). Однако менее информативные SSR-локусы с PIC праймеров 0,69 могли содержать большое число аллелей — до 13, как у CC969431, или 11, как у CC956699, CC969507 и AF113918. Наименьшее значение PIC для праймеров локуса AF180355 было 0,30, где обнаружили всего 2 аллеля.
2. Полиморфные микросателлитные локусы, использованные для оценки генетических взаимосвязей между генотипами капусты кочанной ( Brassica oleracea L.) (коллекция ФГБНУ Федерального научного центра овощеводства)
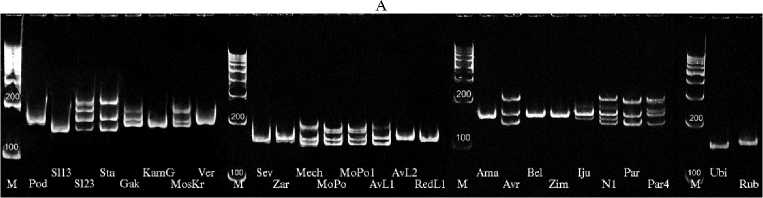
Б
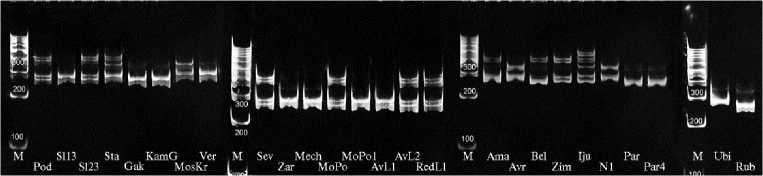
Рис. 1. Электрофореграммы продуктов ПЦР-амплификации микросателлитных локусов AF458409 (А) и BZ523957 (Б) у селекционных образцов капусты кочанной ( Brassica oleracea L.) (коллекция ФГБНУ Федерального научного центра овощеводства) . М — маркер молекулярных масс (GeneRuler100 bp plus DNA ladder, «Thermo Fisher Scientific», США). Расшифровку обозначений и описание образцов см. в таблице 1.
|
Название маркера в GenBank NCBI |
Гены с полиморфными микросателлит-ными последовательностями |
Мотив |
Число аллелей |
PIC |
|
AF051772 |
Reproductive meristem gene 1 (REM1) mRNA |
(gaa)5 |
3 |
0,81 |
|
AF051772(2) |
B3 domain-containing protein REM1 |
(ct)6-1(ct)4-1(tcc)3 |
4 |
0,78 |
|
AF458409 |
Deoxycytidine deaminase (DCTD1), mRNA |
(aga)6 |
5 |
0,90 |
|
AJ427337 |
mRNA for calmodulin 1 (cam1 gene) |
(ga)5 |
3 |
0,61 |
|
BZ523957 |
Genomic clone BOKAH45 |
(ttg)6 |
5 |
0,86 |
|
СС956628 |
Brassica oleracea genomic clone BOIAA94, |
|||
|
genomic survey sequence |
(tc)5 |
2 |
0,50 |
|
|
CC956699 |
Brassica oleracea genomic clone BOIAB20, |
|||
|
genomic survey sequence |
(cac)9 |
11 |
0,69 |
|
|
CC969431 |
Genomic clone BOIAB19 |
(ga)6 |
13 |
0,61 |
|
CC969459 |
Genomic clone BOIAB94 |
(cgg)5 |
2 |
0,31 |
|
CC969497 |
Genomic clone BOIAA26 |
(tgc)5 |
2 |
0,83 |
|
CC969507 |
Genomic clone BOIAB15 |
(ct)5 |
11 |
0,74 |
|
Х94979 |
mRNA for pollen coat protein |
(atg)5 |
2 |
0,44 |
|
AF241115 |
Isolate HRI/CGN 5688 cauliflower gene |
(at)5(ta)6 |
6 |
0,51 |
|
X92955 |
mRNA for pollen coat protein |
(tttta)2(ata)7 |
3 |
0,57 |
|
AF180355 |
Isolate B265 ABI1 protein (ABI1) gene |
(tc)16 |
2 |
0,30 |
|
AF113919 |
Phospholipase D2 (PLD2) gene |
(at)6(gt)5 |
3 |
0,58 |
|
AF273844 |
Thioredoxin-h-like protein 1 (THL1) mRNA |
(ctt)7 |
4 |
0,73 |
|
AF230693 |
Stearoyl-ACP desaturase (DELTA9-BO-1) gene |
(ctt)3(ct)6(cttg)6 |
4 |
0,81 |
|
AF113918 |
Phospholipase D1 (PLD1) gene |
(ct)7(at)7-1 |
11 |
0,60 |
|
U67451 |
Brassica oleracea homeotic protein boi1AP1 |
|||
|
(Boi1AP1) mRNA |
(at)9-1 |
6 |
0,81 |
|
|
AF241115(2) |
Isolate HRI/CGN 5688 cauliflower gene |
(ta)6-1 |
2 |
0,63 |
Рассчитанные данные для PIC в наших исследованиях согласуются со значениями, полученными для этих же маркеров другими авторами ( х 2 = 2,16 при p = 1,0) (11, 12). Маркеры для локусов, которые при анализе показывали значение PIC > 0,5, оказались эффективными при генетической дискриминации родственных генотипов (25). В итоге у изученных образцов с помощью этого набора маркеров мы выявили 76 % полиморфизма.
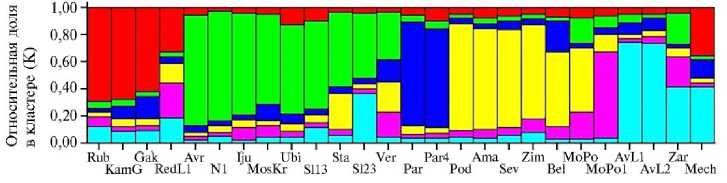
Рис. 2. Структура изученной выборки селекционных образцов капусты кочанной ( Brassica oleracea L.) (коллекция ФГБНУ Федерального научного центра овощеводства) , полученная с использованием программы STRUCTURE на основании байесовского анализа по шести кластерам (K = 6). Расшифровку обозначений и описание образцов см. в таблице 1.
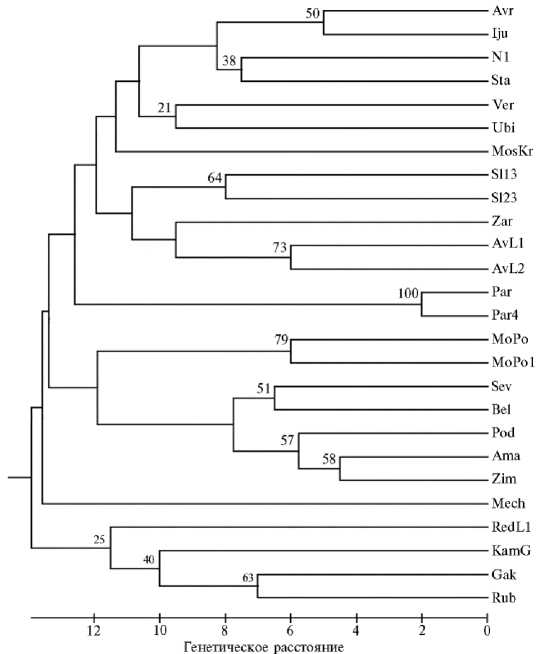
Рис. 3. UPGMA-дендрограмма генетического сходства между селекционными образцами капусты кочанной ( Brassica oleracea L.) (коллекция ФГБНУ Федерального научного центра овощеводства) , построенная на основе изменчивости микросателлитных локусов. Значения bootstrap получены для 1000 реплик. Расшифровку обозначений и описание образцов см. в таблице 1.
Результаты анализа данных в STRUCTURE показали, что все тестируемые образцы лучшим образом распределились по шести кластерам (AK = 2,05), где сгруппировались согласно происхождению (рис. 2). Подсчитанные генетические дистанции варьировали от 0,060 до 0,186, причем наибольшее генетическое расстояние было отмечено между сортом Парус и гибридом Мечта F1. Образцы капусты Северянка F1, Белорусская 455, Подарок 2500, Амагер 611 и Зимовка 1474 голландской группы с происхождением из Северо-Западной Европы объединились в общий кластер (рис. 3). Высокое генетическое сходство сортов Амагер 611, Белорусская 455 и Подарок 2500 подтверждает то, что последний был получен посредством сложной гибридизации двух первых. Относительно новый гибрид Северянка F1 с улучшенными показателями качества и устойчивости к фузариозному увяданию, киле и бактериозу был создан при участии сортов Белорусская 455, Зимовка 1474 и Подарок 2500, отселектированных на Грибовской опытной станции. Сорт Белорусская 455 имел происхождение из Витебской области и был получен с помощью отбора наиболее скороспелых форм и районирован с 1943 года. Районированный в том же году и обладающий высокой продуктивностью позднеспелый сорт Московская поздняя 15 находился в отдельном подкластере, ближе к более поздним по спелости образцам, что объясняет его местное происхождение из сорта Пышкинская (26). Генетическая отдаленность этого сорта также подтверждается его принадлежностью к отдельному сортотипу в Среднерусской группе сортов. Сорт Парус с высокими показателями качества образовывал свой отдельный кластер, что доказывает его генетическую отдаленность от общей группы сортов и сложное происхождение от различных линий с хозяйственно ценными свойствами.
Сорта сортотипа Дитмарская ранняя происхождением из Центральной Европы (Июньская 3200, Стахановка 1513, Номер первый грибовский 147 и скороспелый гибрид Аврора F 1 , имеющий в родословной сорт Июньская 3200) сгруппировались в отдельный кластер. Все эти образцы обладали скороспелостью. Сорт Номер первый грибовский 147, районированный с 1943 года, был использован для создания сорта Июньская 3200, что также согласуется с его расположением на дендрограмме. Линии, выведенные из гибрида Аврора F 1 , образовали отдельный подкластер. Такое их расположение подтверждает, что линейный материал из гибрида может быть достаточно отдален от него генетически. Выведенные на Грибовской опытной станции и районированные в 1940 году традиционные среднеспелые сорта Слава грибовская 231 и Слава 1305, представляющие сортотип Слава, находились в отдельном подкластере.
Новый гибрид Зарница F 1 среднераннего срока образовывал отдельную ветвь дендрограммы ближе к раннеспелым образцам, тогда как позднеспелый гибрид Мечта F 1 сформировал кластер, который находился на расстоянии 0,123 от общей группы капуст белокочанных.
В отдельный подкластер внутри кластера капуст белокочанных объединились сорта капусты савойской. Все они относились к разным сорто-типам, поэтому расположились генетически отдаленно друг от друга. Скороспелый сорт Юбилейная 2170 был получен в результате скрещивания капусты савойской Венская ранняя и сорта белокочанной капусты Номер первый 147. Новый сорт Московская кружевница среднераннего срока созревания с сильной пузырчатостью листа оказался наиболее генетически отдаленным от двух других сортов капусты савойской.
Традиционные сорта капусты краснокочанной Каменная головка 447 и Гако 741, выведенные на Грибовской опытной станции и районированные в 1943 году, относятся соответственно к сортотипам Эрфуртская и Гако, что подтверждается их генетической отдаленностью. В этом же кластере сгруппировались сорт Рубин и селекционная линия I3 424-17. Тради- ционные сорта хорошо адаптированы к местным условиям выращивания и представляют собой популяции, которые могут служить источниками генов для получения ценных линий, поскольку обладают генетической изменчивостью и пластичностью.
Нужно также отметить, что выведенные относительно недавно сорта Парус, Московская кружевница, гибриды Зарница F 1 и Мечта F 1 были достаточно отдалены от основных кластеров традиционных сортов. Кроме того, селекционные линии, выведенные из перспективных гибридов F 1 , в значительной мере отличались от изначальной формы, что позволяет контролировать процесс инбридинга при получении перспективных линий на основе гибрида с использованием ДНК маркеров.
В нашем исследовании относительно небольшой набор микросател-литных маркеров позволил классифицировать уникальные отечественные образцы капусты кочанной. В работе M.A. El-Esawi с соавт. (27) с использованием всего 12 микросателлитных локусов на 25 генотипах Brassica oleracea L., из которых 15 составляли капусты кочанные, было выявлено 75,7 % полиморфизма, что согласуется с полученными нами данными.
Несмотря на значительные достижения в генетике капустных культур, основные исследования, как правило, сконцентрированы на изучении устойчивости к патогенам (28-30), времени цветения (31, 32), морфологических показателей кочана (33), растрескивании кочана (34, 35), тогда как сведения по генетической идентификации сортов капусты кочанной, принадлежащих к различным группам спелости, практически отсутствуют. На основе генетической оценки изученных образцов мы выделили две основные группы генотипов капусты белокочанной: раннего срока созревания (кластер с образцами Аврора F 1 , Июньская, Номер первый грибовский 147, Стахановка 1513) и позднего срока созревания (Московская поздняя 15, Северянка F 1 , Белорусская 455, Амагер 611 и Зимовка 1474). Генотипы савойской разновидности в целом обладали большей скороспелостью, что может объяснить их близкое генетическое расположение к группе более скороспелых образцов. Из них сорт Вертю 1340 относился к наиболее позднеспелым с периодом созревания около 130 сут, тогда как поздние генотипы капусты белокочанной могут созревать более 140 сут.
В силу того, что морфологические признаки и характеристики известных сортов могут быть подвержены воздействию факторов внешней среды, которые вызывают фенотипическую изменчивость и делают оценку сортовых качеств затруднительной, ДНК маркеры оказываются единственным инструментом для точного определения, поддержания и сохранения необходимых для дальнейшей селекции генотипов (36). Селекционные образцы, принадлежащие к различным группам спелости и ценным отечественным сортотипам и пригодные для выращивания во всех регионах России, могут быть определены и отобраны на основании результатов ДНК-анализа с использованием набора известных SSR маркеров.
Таким образом, SSR маркеры показали эффективность в выявлении генетической изменчивости у 24 генотипов капусты кочанной, среди которых были близкие по происхождению и принадлежащие к одному сорто-типу. Результаты ДНК-анализа подтвердили все родственные связи среди традиционных сортов и новых гибридов. Следовательно, установленная генетическая основа изученных образцов может быть определяющей для возможных комбинационных скрещиваний при получении новых селекционных форм. Результаты SSR-анализа и данные о генетических дистанциях предоставляют информацию для отбора селекционного материала, контроля комбинаций среди форм различных групп спелости с признаками
èñõîäíûõ ñîðòîòèïîâ è âûäåëåíèÿ ñåëåêöèîííûõ ëèíèé, ïîëó÷åííûõ èç ãè-áðèäíûõ è/èëè ñîðòîâûõ ïîïóëÿöèé.
ËÈÒÅÐÀÒÓÐÀ
ФГБНУ Федеральный научный центр овощеводства , Поступила в редакцию
143080 Россия, Московская обё., Одищовский р-щ 16 июля 2020 года
ïîñ. ÂÍÈÈÑÑÎÊ, óë. Ñåëåêöèîííàÿ, 14,
Sel’skokhozyaistvennaya biologiya [ Agricultural Biology ], 2020, V. 55, № 5, pp. 890-900
ASSESSMENT OF GENETIC DIVERSITY AMONG HEADED CABBAGE ( Brassica oleracea L.) ACCESSIONS BY USING SSR MARKERS
A.S. Domblides В , L.L. Bondareva, V.F. Pivovarov
The authors declare no conflict of interests Received July 16, 2020
Список литературы Оценка генетического разнообразия образцов капусты кочанной (Brassica oleracea L.) с использованием SSR маркеров
- Chepkemoi J. The world leaders in cabbage production. WorldAtlas, 2017. Режим доступа: worldat-las.com/articles/the-world-leadeis-in-cabbage-production.html. Дата обращения: 15.03.2020.
- Novotny C., Schulzova V., Krmela A., Hajslova J., Svobodova K., Koudela M. Ascorbic acid and glucosinolate levels in new czech cabbage cultivars: effect of production system and fungal infection. Molecules, 2018, 23(8): 1855 (doi: 10.3390/molecules23081855).
- Лизгунова Т.В. Культурная флора СССР. Том 11. Капуста. Л., 1984.
- Formisano G., Roig C., Esteras C., Ercolano M.R., Nuez F., Monforte A.J., Pico M.B. Genetic diversity of Spanish Cucurbita pepo landraces: an unexploited resource for summer squash breeding. Genetic Resources and Crop Evolution, 2012, 59(б): 1169-1184 (doi: 10.1007/s10722-011-9753-y).
- Collard B.C.Y, Mackill D.J. Marker-assisted selection: аn approach for precision plant breeding in the twenty-first century. Philosophical Transactions of the Royal Society B: Biological Sciences, 2008, 363(1491): 557-572 (doi: 10.1098/rstb.2007.2170).
- Guichoux E., Lagache L., Wagner S., Chaumeil P., Léger P, Lepais O., Lepoittevin C., Ma-lausa T., Revardel E., Salin F., Petit R.J. Current trends in microsatellite genotyping. Molecular Ecology Resources, 2011, 11(4): 591-611 (doi: 10.1111/j.1755-0998.2011.03014.x).
- Lowe A.J., Moule C., Trick M., Edwards K.J. Efficient large-scale development of microsatellites for marker and mapping application in Brassica crop species. Theoretical and Applied Genetics, 2004, 108(6): 1103-1112 (doi: 10.1007/s00122-003-1522-7).
- Márquez-Lema A., Velasco L., Pérez-Vich B. Transferability, amplification quality, and genome specificity of microsatellites in Brassica carinata and related species. Journal of Applied Genetics, 2010, 51(2): 123-131 (doi: 10.1007/BF03195720).
- Thakur A.K., Singh K.H., Singh L., Nanjundanet J., Khan Y.J., Singh D. SSR marker variations in Brassica species provide insight into the origin and evolution of Brassica amphidiploids. Heredi-tas, 2018, 155(6): 1-11 (doi: 10.1186/s41065-017-0041-5).
- Raza A., Mehmood S.S., Ashraf F., Khan R.S.A. Genetic diversity analysis of Brassica species using PCR-based SSR markers. Gesunde Pflanzen, 2019, 71(1): 1-7 (doi: 10.1007/s10343-018-0435-y).
- Tonguc M., Griffiths P.D. Genetic relationships of Brassica vegetables determined using database derived sequence repeats. Euphytica, 2004, 137(2): 193-201 (doi: 10.1023/B:EUPH.0000041577.84388.43).
- Louarn S., Torp A.M., Holme I.B., Andersen S.B., Jensen B.D. Database derived microsatellite markers (SSRs) for cultivar differentiation in Brassica oleracea. Genetic Resources and Crop Evolution, 2007, 54(8): 1717-1725 (doi: 10.1007/s10722-006-9181-6).
- Saxena B., Kaur R., Bhardwaj S.V. Assessment of genetic diversity in cabbage cultivars using RAPD and SSR markers. Journal of Crop Science and Biotechnology, 2011, 14(3): 191-196 (doi: 10.1007/s12892-011-0018-2).
- Zhu S., Zhang X., Liu Q., Luo T., Tang Z., Zhou Y. The genetic diversity and relationships of cauliflower (Brassica oleracea var. botrytis) inbred lines assessed by using SSR markers. PLoS ONE, 2018, 13(12): e0208551 (doi: 10.1371/journal.pone.0208551).
- Артемьева А.М., Клоке Э., Чесноков Ю.В. Анализ филогенетических связей вида Brassica oleracea L. (капуста огородная). Информационный вестник ВОГиС, 2009, 13(4): 759-771.
- Izzah N.K., Lee J., Perumal S., Park J.Y., Ahn K., Fu D., Kim G.-B., Nam Y.-W., Yang T.-J. Microsatellite-based analysis of genetic diversity in 91 commercial Brassica oleracea L. cultivars belonging to six varietal groups. Genetic Resources and Crop Evolution, 2013, 60(7): 1967-1986 (doi: 10.1007/s10722-013-9966-3).
- Shapturenko M., Khotyleva L., Vakula S., Jakimovich A.V., Zabara Yu.M., Khotyleva L.V. Informative EST-SSR markers for genotyping and intraspecific differentiation of Brassica oleracea var. capitata L. Vavilov Journal of Genetics and Breeding, 2016, 20(1): 51-56.
- Чесноков Ю.В., Артемьева А.М. Оценка меры информационного полиморфизма генетического разнообразия. Сельскохозяйственная биология, 2015, 50(5): 571-578 (doi: 10.15389/agrobiology.2015.5.571rus).
- Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155(2): 945-959.
- Earl D.A., vonHoldt B.M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method. Conservation Genetics Resources, 2012, 4(2): 359-361 (doi: 10.1007/s12686-011-9548-7).
- Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 2012, 28(19): 2537-2539 (doi: 10.1093/bioinfor-matics/bts460).
- Nei M. Genetic distance between populations. American Naturalist, 1972, 106(949): 283-392 (doi: 10.1086/282771).
- Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 1978, 89(3): 583-590.
- Tamura K., Peterson D., Peterson N., Stecher G., Nei M., Kumar S. MEGA5: molecular evolutionary genetic analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 2011, 28(10): 2731-2739 (doi: 10.1093/mol-bev/msr121).
- DeWoody J.A., Honeycutt R.L., Skow L.C. Microsatellite markers in white-tailed deer. Journal of Heredity, 1995, 86(4): 317-319 (doi: 10.1093/oxfordjournals.jhered.a111593).
- Елена Михайловна Попова. Ученый, селекционер. Основоположник научной отечественной селекции по капустным культурам. Научное наследие — воспоминания, развитие научных идей /Под ред. В.Ф. Пивоварова. М., 2007.
- El-Esawi M.A., Germaine K., Bourke P., Malone R. Genetic diversity and population structure of Brassica oleracea germplasm in Ireland using SSR markers. Comptes Rendus Biologies, 2016, 339(3-4): 130-140 (doi: 10.1016/j.crvi.2016.02.002).
- Peng L., Zhou L., Li Q., Wei D., Ren X., Song H., Mei J., Si J. Qian W. Identification of quantitative trait loci for clubroot resistance in Brassica oleracea with the use of Brassica SNP microarray. Frontiers in Plant Science, 2018, 9(822): 1-8 (doi: 10.3389/fpls.2018.00822).
- Kifuji Y., Hanzawa H., Terasawa Y., Ashutosh, Nishio T. QTL analysis of black rot resistance in cabbage using newly developed EST-SNP markers. Euphytica, 2013, 190(2): 289-295 (doi: 10.1007/s10681-012-0847-1).
- Mei J.Q., Ding Y.G., Lu K., Wei D.Y., Liu Y., Disi J.O., Li J., Liu L., Liu S., McKay J., Qian W. Identification of genomic regions involved in resistance against Sclerotinia sclerotiorum from wild Brassica oleracea. Theoretical and Applied Genetics, 2013, 126(2): 549-556 (doi: 10.1007/s00122-012-2000-x).
- Okazaki K., Sakamoto K., Kikuchi R., Saito A., Togashi E., Kuginuki Y., Matsumoto S., Hi-rai M. Mapping and characterization of FLC homologs and QTL analysis of flowering time in Brassica oleracea. Theoretical and Applied Genetics, 2007, 114(4): 595-608 (doi: 10.1007/s00122-006-0460-6).
- Uptmoor R., Schrag T., Stutzel H., Esch E. Crop model based QTL analysis across environments and QTL based estimation of time to floral induction and flowering in Brassica oleracea. Molecular Breeding, 2008, 21(2): 205-216 (doi: 10.1007/s11032-007-9121-y).
- Lv H., Wang Q., Zhang Y., Yang L., Fang Z., Wang X., Liu Y., Zhuang M., Lin Y., Yu H., Liu B. Linkage map construction using InDel and SSR markers and QTL analysis of heading traits in cabbage. Molecular Breeding, 2014, 34(1): 87-98 (doi: 10.1007/s11032-014-0019-1).
- Pang W., Li X., Choi S.R., Nguyen V.D., Dhandapani V., Kim Y.Y., Ramchiary N., Kim J.G., Edwards D., Batley J., Na J., Kim H.R., Lim Y.P. Mapping QTLs of resistance to head splitting in cabbage (Brassica oleracea L. var. capitata L.). Molecular Breeding, 2015, 35(5): 1-12 (doi: 10.1007/s11032-015-0318-1).
- Su Y., Liu Y., Li Z., Fang Z., Yang L., Zhuang M., Zhang Y. QTL analysis of head splitting resistance in cabbage (Brassica oleracea L. var. capitata) using SSR and InDel makers based on whole-genome re-sequencing. PLoS ONE, 2015, 10(9): e0138073 (doi: 10.1371/jour-nal.pone.0138073).
- Korir N.K., Han J., Shangguan L., Wang C., Kayesh E., Zhang Y., Fang J. Plant variety and cultivar identification: advances and prospects. Critical Reviews in Biotechnology, 2013, 33(2): 111125 (doi: 10.3109/07388551.2012.675314).


