Олигонуклеотидные праймеры для детекции генов, кодирующих большую субъединицу бифенил 2,3-диоксигеназы, бактерий порядка Actinomycetales
Автор: Шумкова Екатерина Сергеевна, Плотникова Елена Генриховна
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 1, 2012 года.
Бесплатный доступ
Бифенил 2,3-диоксигеназа (БДО) является ключевым ферментом, участвующим в разложении бифенила и полихлорированных бифенилов (ПХБ) у бактерий. Структура ее активного центра определяет спектр утилизируемых конгенеров ПХБ. Нами были подобраны вырожденные праймеры для скрининга бактерий-деструкторов порядка Actinomycetales на наличие генов bphA1, кодирующих большую субъединицу бифенил диоксигеназ.
Полихлорированные бифенилы (пхб), бифенил 2, 3-диоксигеназа, бактерии, порядок actinomycetales, вырожденные праймеры, гены bpha1
Короткий адрес: https://sciup.org/147204575
IDR: 147204575 | УДК: 579.25
Текст научной статьи Олигонуклеотидные праймеры для детекции генов, кодирующих большую субъединицу бифенил 2,3-диоксигеназы, бактерий порядка Actinomycetales
Полихлорированные бифенилы (ПХБ) входят в число наиболее опасных абиотичесих веществ, созданных человеком. Это высокотоксичные соединения, мутагены, канцерогены. Благодаря исключительной стабильности, устойчивости к воздействию химических и физических факторов, ПХБ долго остаются в окружающей среде [Ross, 2004]. Несмотря на запрет производства ПХБ во многих странах, проблема их утилизации остается актуальной [Васильева, Стрижакова, 2007]. Всестороннее изучение микроорганизмов-деструкторов бифенила/ПХБ является перспективным для создания эффективных технологий очистки окружающей среды от данных соединений.
У ряда грамположительных и грамотрицатель-ных бактерий изучены ферментные системы, контролирующие разложение бифенила/ПХБ [Pieper, 2005]. Так, бифенил 2,3-диоксигеназа (БДО) является ферментом, катализирующим первую реакцию окисления одного из ароматических колец бифени-ла/ПХБ с образованием дигидродиольного производного. Изучению этого фермента уделяется особое внимание, так как он определяет спектр утилизируемых бактериями конгенеров ПХБ. Бифенил 2,3-диоксигеназа – мультикомпонентный фермент, состоящий из трех белков: терминальной оксигеназы (BphA1/BphA2), ферредоксина (BphA3) и ферредоксин редуктазы (BphA4). Терминальная оксигеназа содержит α- и β-субъединицы (кодируются генами bphA1 и bphA2 , соответственно) и является
α 3 β 3 гексамером [Seeger et al., 2001]. α-Субъединица БДО состоит из 458 – 468 АКО, которые образуют два домена [Zielinski et al., 2002]. N-концевой домен включает в себя железо-серный кластер Риске [2Fe-2S] (с 40 по 140 АКО у Spingobium yanoikyae B1). Два остатка гистидина и два цистеина координируют атомы железа и серы, входящие в этот кластер. С-концевой домен включает каталитический карман, в котором находится негемовое железо (II), координированное двумя остатками гистидина (His-207 и His-212) и одним остатком аспарагиновой кислоты (Asp-360) [Ferraro et al., 2007] (рис. 1). Предполагается, что размер и форма каталитического кармана напрямую влияют на спектр окисляемых ферментом субстратов и на эффективность взаимодействия [Jaconcic et al., 2007], а пространственная структура белка и, соответственно, структура активного центра, в свою очередь, определяется взаимодействием аминокислот. Известно, что замены определенных аминокислот, влекут за собой изменение структуры активного центра и, соответственно, спектра окисляемых субстратов [Kimura et al., 1997; Suenaga et al., 2002].
Большие субъединицы (α-субъединицы) негемовых железосодержащих диоксигеназ, способных осуществлять гидроксилирование бензольного кольца ароматических соединений, составляют большое семейство эволюционно родственных аминокислотных последовательностей, которые делятся на четыре подсемейства. Ферменты, осуществляющие гидроксилирование ароматического кольца бифенила/ПХБ, относятся к подсемейству бифе-
нил/толуол диоксигеназ (Б/Т ДО) [Gibson, Parales, 2000].
Однако есть данные о бифенил 2,3-диоксигена-зах бактерий родов Sphingomonas , Bacillus , Rhodo-coccus , существенно отличающихся от ферментов подсемейства Б/Т ДО и сходных с фенантрен- и нафталин диоксигеназами бактерий-деструкторов полициклических углеводородов [Romine et al., 1999; Mukerjee-Dhar et al., 2005; Taguchi et al., 2007; Yang et al., 2007]. Так, уровень сходства между недавно описанными генами, кодирующими БДО Rhodococcus sp. R04 и генами Rhodococcus sp. RHA1, БДО которого относится к ферментам подсемейства Б/Т ДО, составляет менее 44% [Yang et al., 2007].
Известно, что гены БДО грамположительных и грамотрицательных бактерий существенно отличаются. Так, по данным Е. Masai с соавторами [1995] БДО бактерий рода Rhodococcus более сходны с толуол диоксигеназами грамположительных бактерий, чем с бифенил диоксигеназами протеобакте-рий. Нами были выделены и охарактеризованы грамположительные бактерии порядка Actinomycetales , способные активно разлагать би-фенил/ПХБ [Плотникова и др., 1998; 2001; 2005;
Шумковa, 2009]. При изучении генетических систем деструкции данных соединений у ряда штаммов рода Rhodococcus , способных к активной деструкции бифенила/ПХБ, не были выявлены гены, присутствующие в геноме хорошо исследованных бактерий-деструкторов ПХБ. Так, отсутствовала специфическая амплификация гена bphA1 при использовании вырожденных праймеров, сконструированных на основе анализа известных последовательностей α-субъединиц гидроксилирующих диоксигеназ бифенила/толуола/бен-зола/изопропилбензола (подсемейство Б/Т ДО) [Witzig et al., 2006]. В то же время у ряда исследованных бактерий порядка Actinomycetales (рода Rhodococcus , Janibacter ), а также у грамотрицатель-ных бактерий-деструкторов рода Pseudomonas была амплифицирована область гена bphA1 , располагающаяся в районе 668–1152 п.н. Исходя из данных M. Zielinski с соавторами [2002], размер гена bphA1 у разных бактерий составляет 1374–1404 п.н.). Ам-плифицированный участок гена кодирует 222–384 АКО α-субъединицы БДО. Это АКО С-концевого домена, формирующие активный центр фермента (рис. 1) [Шумковa и др., 2008; Шумкова, 2009].
ll^T
II
N
[2Fe-2S]
и Кластер Риске
Активный центр
20 40 6
80 100 120 140 1 60 1 80 200 220 240 260 280
400 420 440 460
*
11^1_______________ у
Рис. 1 . Схема строения α-субъединицы бифенил 2,3-диоксигеназы [Zielinski et al., 2002] и исследуемые нами области:
заштрихованный и белый участки соответствуют каталитическому и Риске доменам, соответственно; короткие вертикальные линии, соединенные горизонтальной, показывают расположение лигандов железосерного кластера и мононуклеарного железа активного центра; I – исследованная нами ранее область, II – область, исследование которой планируется; длинными вертикальными линиями показано положение праймеров; внизу отображена нумерация АКО, соответствующая порядку их расположения в α-субъединице БДО Burkholderia xenovorans LB400
Таким образом, подбирая новые праймеры, мы ставили две цели:
-
1. Амплифицировать часть гена, кодирующую N-концевой домен α-субъединицы БДО, включающий железо-серный кластер Риске. Это позволит получить более полную информацию о генах тех бактерий, у которых ранее был амплифицирован участок гена bphA1 , кодирующий активный центр.
-
2. Получить ПЦР-продукт с другого участка гена тех бактерий, у которых ранее амплификации не наблюдалось. Это послужит основой для дальнейшего изучения полной нуклеотидной последо-
- вательности гена bphA1, а также позволит в дальнейшем проводить скрининг бактерий на присутствие генов, контролирующих деструкцию бифе-нила/ПХБ у бактерий порядка Actinomycetales.
Материалы и методы исследования
Программное обеспечение
Поиск генов bphA1 проводился с помощью программы BLAST в международной базе данных GenBank. Для выравнивания нуклеотидных последовательностей и поис- ка консервативных участков была использована программа VectorNTI 10 . Последовательности олигонуклеотидов подбирались вручную. Для анализа свойств праймеров использовалась программа OligoAnalyzer 3.1 (http:// com/analyzer/Applications/OligoAnalyzer/. Проверка комплементарности праймеров соответствующим генам проводилась с помощью программы Primer-BLAST
.
Результаты и обсуждение
Подбор праймеров
Поскольку bphA1 гены грамположительных и гра-мотрицательных бактерий имеют существенные отличия [Masai et al., 1995], подбор праймеров проводился на основе известных нуклеотидных последовательностей генов α-субъединиц БДО, депонированных в международной базе данных GenBank, грамположительных штаммов: Rhodo-coccus globerulus P6 [X80041], R. erythropolis TA421 [D88021], R. erythropolis TA421 [D88020], R. aetherivorans I24 cosmid 2G11 [AF452376], Rho-dococcus sp. RHA1 [D32142], Rhodococcus sp. R04 [DQ403247], Rhodococcus sp. HA99 [AB272986], R. erythropolis TA431 [AB272985], R. rhodochrous K37 [AB272984].
Было проведено множественное выравнивание нуклеотидных последовательностей (ген bphA1 и прилегающие к нему с краев участки длиной 230 п.н.) перечисленных выше бактерий. Найдено несколько консервативных областей без делеций, длина которых составляла 26–53 п.н., что достаточно для подбора праймера: 1) 377–430 п.н., 2) 452– 495 п.н., 3) 530–582 п.н., 4) 593–633 п.н., 5) 746–773 п.н., 6) 900–926 п.н., 7) 1410–1433 (рис. 2).
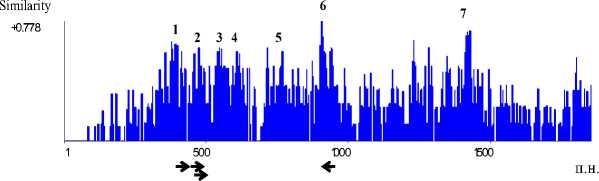
Рис. 2 . График сходства выравненных нуклеотидных последовательностей гена bphA1 :
по оси х – показатель сходства (Similarity) с консенсусной последовательностью – рассчитывался следующим образом: значения от 0 до 1, присвоенные каждому нуклеотиду (1 – идентичность, 0.5 – сходство, 0.2 – слабое сходство) суммированы и поделены на количество последовательностей в выравнивании; по оси у – порядковый номер нуклеотида от начала гена; стрелками показано положение праймеров; номерами (1–7) обозначены консервативные области
При подборе праймеров мы руководствовались рекомендациями [Kwok et al., 1994; Патрушев, 2004; Linhart, Shamir, 2005]. Кроме того, старались минимизировать вырожденность праймеров [Compton, 1990] и подобрать их таким образом, чтобы 3'-конец праймера не содержал нуклеотидных замен, т.е. максимально соответствовал участку, на который он будет отжигаться. На 5'-конце допускали несоответствия матрице. Необходимо было, чтобы амплифици-руемая область гена перекрывалась с ранее исследованным нами участком гена bphA1 , а положение прямого праймера было максимально близко к началу гена. С учетом поставленных задач для подбора праймеров подходили консервативные области № 1, 2 и 6. При соблюдении данных условий нам удалось подобрать несколько вариантов прямого, forvard(F) – праймера и один обратный revrse(R)-праймер (рис. 3): BphA1F409 5'-TTCTTGGCTGYTKHTSGSNCA-3', BphA1F450 5'-CCGGCGACTTYATSACSAMSTACAT-3', BphA1F462 5'-TGACCACGTACATGGGBGARGA-3', BphA1R900 5'-TCSGCDGCRAWYTTCCAGTT-3'.
Анализ термодинамических характерик и вторичной сруктуры подобранных праймеров
Далее мы провели анализ термодинамических параметров и вторичной структуры вышеперечисленных олигонуклеотидов (таблица). Forvard-праймер BphA1F409 позволяет амплифицировать максимально длинный участок гена bphA1, однако по сравнению с BphA1F450 и BphA1F462 обладает большой вырожденностью, худшими термодинамическими характеристиками. Вырожденный праймер – это смесь нуклеотидных последовательностей, каждая из которых представляет собой индивидуальный праймер. Чем выше «степень вырожденности» (число индивидуальных праймеров в смеси), тем сильнее падает эффективность ПЦР из-за снижения эффективной концентрации праймера. Рекомендуется, чтобы степень вырожденности не превышала 512 [Compton, 1990]. Также, в случае BphA1F409 разница между максимальной и минимальной температурой плавления индивидуальных вариантов праймера в общей смеси будет наибольшей: почти 11ºС по сравнению с 3.9ºC и 4.5ºC в случае BphA1F450 и BphA1F462, соответ- ственно (таблица), что может вызвать трудности при подборе температуры отжига в ПЦР.
AB 272985] _Rh о do с ос c us_eryt hro po lts_T A431 [DQ403247]_Rhodococcus_sp ,_R04 [D88021]_Rhodococcus_errthropois_TA421 [A F452376] _Rh о do с ос c us_ae t herivoran s_I24 [D 3 2142 ] _Rh od о co c cu s_sp ._RHA 1 [D8 80 20]_Rhodococcus_er>thropois_TA421 ГХ800411 Rjipdococcus oloberulus P6 [A В 27 29 8 6] _Rn о do с ос c us_sp, _H A 99 [A B2729 8 4] _Rh о do с ос c us_rh о do c hro us_K37

GTGGACC GAGATGTCTA C GA GCT CGAGС TC GA GO G CC ТС ТГС GC С АЛ ТА СC TG G CTGTTTCTC GG GСЛ GTGGACCGAGATGTCTACGAGCTCGAGCTCGAGCGCLTCTTCGCLA#TACCTGGCTGTTTCTCGGGC# i ACCGATCAGGCAATCTACGAGCGAGAGCTCGAGCAAGTTTTCGGGCC GAGTTGGCTGTTCCTCGCACJ I ACC^jACGAGGCGCTGTACGAACAGGAACTGGAGCGGATCTTCGGTC( CTCGTGGTTGCTGATGGGCCJ । ACCBACGAGGCGCTGTACGAACAGGAACTGGAGCGGATCTTCGGTC( CTCGTGGTTGCTGATGGGC CJ । A CgIaC GAGGCGCTTTACCAGCAGGAACTCGAGCTCATCTTCGGTCC GTCCTGGCTCCTGTTGGGGCJ ।
CGTGAGC CGTGAGC CGATTCG CGAGACG CGAGACG CGAGACG
GTGGACCGAGATGKTACC4GCTCGAGCTCGAGCGCCTCTTCGCCM TACCTGGCTGTTTCTCGGGCj CGTGAGC
(361) GTGGACCGAGATGTCTACGAGCTCGAGCTCGAGCGCCTCTTCGCCAj TACCTGGCTGTTTCTCGGGCj CGTGAGC
BphA1F409
[AB272985]_Rhodococcus_erythropolis_T A 431 (397) [DQ403247]_Rhod0CDCCUS_sp._R04 (385)
[D88021LRhodococcus_erythropolis_T A421 (331) [A F452376 ] _Rh od о co с c us_ae t herivoran s_I24 (401) [D32142]_Rhodococcus_sp ._RHA1 (363)
[D88020]_Rhodococcus_erythropolis_T A421 (380) [X 80041 ] _Rho d ос о cc и s_g Io be rulu s_P6 (380) [AB272986]_Rhodococcus_sp ,_HA99 (352) [AB272984]_Rhodococcus_rhodochrous_K37 (430)
GTGAGCLAGGTCGCCGlAtCGGGCGACTTTA
BTGAGCCAGGlCGCCGiAt CGGGCGACTTTA
TCACCACGTACAT
TCACCACGTACAT
GATTCGCAACTTCCGAAA( GCGGCAGCTTCGTGCAGACCTACAT
GAGAC GC AGArTC CGAAGlCC GGC GACTTCA GAGACGCAGATTC CGAAGt CCGGCGACTTCA GAGACGCAAATCCCGAAGt CCGGCGACTTCA gagacgIlaatcccgaagi CCGGCGACTTLA GTGAGCCAGGTCGCCGAAC CGGGCGACTTTA
ГGACGAAСТАCAT ГGACGAACTACAT ГGACCCAGTACAT ГGACCCAGTACAT TCACCACGTACAT
rGGTGAAGA [TCCGTTATCGTCTCCHCGGT rGGGGAAGA [TCCGTTATCGTCTCCCGCGGT jggtgagga :cctgtacttgttgtgcgacag rG G C GA GGA ГС С C GTG ATG GTC GTTC GT C A G rGG C GA GGA ГС С C GTG ATG GTC GTTC GT C A G rGGTGAGGA ^CCGGTTATCGTCTCGCGGCAG rG GT GA GGA JCCGGTTATCGTCTCGCGGCAG iGGTGAAC-A [TCCGTTATCGTCTCCCGCGGT
GTGAGCCAGGTCGCCGAAC CGGGCGACTTTA TZACCACGTACAT iGGTGAAGA'TCCGTTATCGTCTCCCGCGGT
BphA1F450
[AB272985]_Rhodococcus_e ryt hrupolis_T Д43 1 [DQ40 3 2 47]_Rhodococcus_sp._R]0 4 [D8 80 2 1 J_Rhodococcus_e ryt hropolis_T A42 1
[AF452376 ]_Rhodococcus_a et herivorans_I24 [D3 2 14 2 ]_R hodococcu s_s p ._RH Al
[D88020]_Rhodococcus_e ryt hropolis_T A421
ГХ800411 Rhodococcus doberulus P6 [ AB272986 ]_R hodococcu s_s p. _H A99 [AB272984]_Rhodococcus_rhodochrous_K37

BphA1F462
ACССТCGAGAС AACTGGAAGAT CGC C GCCGAGIAACTT CGCC-GCCGACATСТАCCACGTCGAАСАС TCGCACGC ACCGTCCAСАС( AACTGGAAGATCGCCGCCGAGAACTTCGCC-GCCGACATGTACCACGTCGAACACTCGCACGC GT TATC GACTG( AACTGGAДАТT T GCA TCCGAGCAGT-CTGCGAGCGACATGTA TCACGT GС-С TTТСTС СC
GT CAT T С C CT Gt ДАС T G GAAGT T C GC AG C GGAG CAAT T C TGC - A GC GA CAT GTA CCAC G C GG- G iZAC CA CAT С С C GTCATTССДТG( AACTGGAAGTTCGCAGCGGAGCAATTCTGC-AGCGACATGTAСCACGCGG-GCACCACATССC GTCAT CCAGTGiAACTGGAAGT TCGCAGCGGAGCAGTT CTGC-TCTGATATGTACCACGTCG-GCACGACGTCAC
GTCAT CCAGTG( AACTGGAAGT TCGCAGCGGAGCAGTTCTGC-TCTGATATGTACCACGTCG-GCACGACGTCAC ACC GT C GA CAC ( AAC T G GAAGATC GC C GC CGAG AAC T T C GC C - GCC GA CAT GTA CCAC G TCGAACAC TC GCAC G C ACC GT C GA CAC( AAC T G GAAGAT C GC C GC CGAG ДАС T T C GC C- GCC GA CAT GTA CCAC G TC GAACAC TC GCAC G C
BphA1R900
Рис. 3 . Фрагменты выравнивания нуклеотидных последовательностей генов bphA1 :
консервативные нуклеотиды выделены цветом, участки, комплементарные праймерам, выделены прямоугольниками и стрелками
Ни один из forvard-праймеров не образует тугоплавких шпилек (с ∆G меньше –7 kcal.mole -1 ). Однако для каждого из сконструированных праймеров имеются индивидуальные варианты, образующие тугоплавкие гомо- или гетеродимеры (таблица). Оптимальным в этом отношении является BphA1F462, который при степени вырожденности 6 образует 1 гетеродимер, характеризующийся свободной энергией Гиббса ∆G –10.13 kcal.mole -1 . BphA1F409 образует 11 вариантов (из 192) гомо- и гетеродимеров с ∆G меньше –7 kcal.mole -1 , что может вызвать затруднения на стадии отжига данных индивидуальных вариантов праймера. Однако BphA1F462, по сравнению с BphA1F450 и BphA1F462, позволяет амплифици-ровать наиболее короткий фрагмент гена bphA1 .
Проверка специфичности праймеров
В результате проверки комплементарности праймеров к имеющимся в базе GenBank генам bphA1 , было выявлено, что сконструированные нами олигонуклеотиды в разной степени специфичны генам, кодирующим α-субъединицы негемовых железосодержащих диоксигеназ.
Пара праймеров BphA1F409 и BphA1R900 в подавляющем большинстве случаев была компле- ментарна bphA1 генам грамположительных бактерий (рода Rhodococcus, Arthrobacter). Но среди найденных с их помощью нуклеотидных последовательностей, депонированных в GenBank, были соответствующие гены bphA1 грамотрицательных бактерий Comamonas testosteroni и Dyella ginsengi-soli, а также участок транспозона Tn4371, содержащего гены деструкции бифенила, грамотрица-тельной бактерии Ralstonia oxalatica. Длина ожидаемого ПЦР-продукта составляла 492 или 507 п.н.
Праймеры BphA1F450 и BphA1R900 также в большинстве случаев были комплементарны bphA1 генам грамположительных бактерий (рода Rhodococcus, Janibacter). Однако среди найденных в GenBank нуклеотидных последовательностей было несколько bphA1 генов грамотрицательных бактерий родов Pseudoxanthomonas и Dyella; гены α-субъединиц неизвестной гидроксилирующей диоксигеназы, фенилпропионат диоксигеназы и хлорбензол диоксигеназы грамотрицательных бактерий родов Rhizobium, Bordetella и Pseudomonas, соответственно. Размер ожидаемого ампликона составил 445 – 466 п.н. Вероятен также неспецифический отжиг на ДНК грамотрицательных бактерий рода Pirellula (с длиной ожидаемого про- дукта реакции 1432 п.н.), Starkeya (2691 п.н.) и ар- хей Methanocella (4614 п.н.).
Анализ термодинамических характеристик и вторичной структуры подобранных праймеров
|
Параметры |
BphA1F409 |
BphA1F450 |
BphA1F462 |
BphA1R900 |
|
Длина, п.н. |
21 |
25 |
22 |
20 |
|
Степень вырожденности |
192 |
32 |
6 |
48 |
|
Содержание GC, % |
51.6 |
52.0 |
55.3 |
51.7 |
|
T m (макс. – мин.) средн. ,ºC |
54.2 – 65.1 59.3 |
58.8 – 62.7 60.7 |
58.0 – 62.5 60.5 |
55.5 – 62.0 58.2 |
|
Разница между мин. и макс. T m , ∆T m , ºC |
10.9 |
3.9 |
4.5 |
6.5 |
|
Шпильки (шп.): количество вариантов, ∆G (kcal.mole -1 ) |
– |
– |
1 шп., ∆G -1.37 |
– |
|
Гомодимеры (д.), количество вариантов, ∆G (kcal.mole -1 ), кол-во спаренных оснований (сп. осн.) |
1 д.: ∆G -14.86, 7 сп. осн. 3 д.: ∆G -7.81 – -8.86
7 д.: ∆G -4.24 – -6.21,
2.42, 2 сп. осн. |
3 д.: ∆G -7.28 – -9.75, 4 – 6 сп. осн. 2 д., ∆G -4.68 – -5.52, 3 – 4 сп. осн. 11 д., ∆G -3.0 – -3.88, 2 – 3 сп. осн. 4 д., ∆G -1.47 – -1.76, 2 сп. осн. |
5 д.: ∆G -5.02 – -6.3, 3 – 4 сп. осн. 7 д.: ∆G -1.34 – - 2.88, 2 сп. осн. |
1 д.: ∆G -17, 7 сп. осн. 1 д.: ∆G -14.42, 6 сп. осн. 2 д.: ∆G -8.13 – -8.77,
5 д.: ∆G -3.34 – -3.52,
|
|
Димеры с BphA1R900, количество вариантов, ∆G (kcal.mole -1 ), кол-во спаренных оснований (сп. осн.) |
4 д.: ∆G -10.87 – -13.66, 5 – 7 сп. осн. 3 д.: ∆G -8.13 – -8.87, 4 сп. осн. 6 д.: ∆G -4.54 – -6.62, 3 – 4 сп. осн. 5 д.: ∆G -3.55 – -3.34, 2 – 3 сп. осн. 6 д.: ∆G -1.34 – -2.39, 2 сп. осн. |
1 д.: ∆G -7.28,
16 д.: ∆G -1.34 – -2.94, 2 – 3 сп. осн. |
1 д.: ∆G -10.13, 6 сп. осн. 3 д.: ∆G -4.45 – -5.4, 3 сп. осн. 10 д.: ∆G -3.07 – -3.91, 2 – 3 сп. осн. 14 д.: ∆G -1.34 – - 2.88, 2 сп. осн. |
– |
|
Разница T m c BphA1R900, ∆T m (макс. – мин.), ∆T m средн., ºC |
-1.5 – 3.1 1.1 |
3.3 – 0.7 2.5 |
2.5 – 0.5 2.3 |
– |
|
Порядковый № АКО (от N-конца белка BphA1), соответствующего 5'-концу праймера |
59 |
73 |
77 |
230 |
|
Порядковый № нуклеотида (от начала гена bphA1 ), соответствующего 5'-концу праймера |
179 |
220 |
232 |
690 |
Примечания. Степень вырожденности праймера рассчитывали, перемножая количество вариантов нуклеотидов в каждой позиции. T m - температура плавления.
Пара праймеров BphA1F462 и BphA1R900 оказалась наименее специфичной к bphA1 генам грамположительных бактерий. Среди данных, найденных с помощью нуклеотидных последовательностей сконструированных праймеров, было 7 генов α-субъединиц БДО бактерий рода Rhodococ-cus и 1 ген bphA1 бактерий рода Arthrobacter . По результатам проверки можно предположить, что с такой же долей вероятности праймеры с будут отжигаться и на гены bphA1 грамотрицательных бактерий: с помощью программы Primer-BLAST было найдено 9 bph -генов бактерий рода Pseudomonas, по 2 гена Burkholderia и Dyella , по 1 гену Comamo-nas , Pandoraea , Cupriavidus , Achromobacter , bphA1 ген некультивируемых бактерий и 9 генов, кодирующих синтетический конструкт BphA. Кроме того, возможен отжиг праймеров на гены больших субъединиц гидроксилирующих диоксигеназ кумола ( cumA1 ), этилбензола ( edoA1 ) и изопропилбензола грамотрицательных бактерий Pseudomonas fluorescens , гены α-субъединиц гидроксилирующих диоксигеназ Polaromonas naphthalenivo-rans и некультивируемой бактерии, на ДНК Pseu-doxanthomonas spadix, на интегративные и конъю-гативные элементы грамотрицательных бактерий Ralstonia и Acidovorax . Размер ожидаемого ПЦР-продукта составил 439 – 454. Вероятен неспецифический отжиг данной пары праймеров на ген рибосомального белка Pirellula staleyi с длиной ожидаемого продукта реакции 1432 п.н.
Заключение
Нами подобраны вырожденные праймеры для амплификации участка гена bphA1 , кодирующего N-концевой домен α-субъединицы БДО с расположенным в нем кластером Риске. Анализ термодинамических параметров и вторичной структуры олигонуклеотидов позволяет предположить, что амплификация фрагмента гена bphA1 с высокой вероятностью пройдет успешно. Все праймеры подходят для детекции генов, кодирующих каталитические субъединицы ферментов подсемейства Б/Т ДО у бактерий порядка Actinomycetales . Для скрининга генов bphA1 более всего подходит пара BphA1F409 и BphA1R900. Планируется экспериментальная проверка праймеров.
Работа выполнена при поддержке гранта РФФИ-Урал №11-04-96028 «Новые физиологические и молекулярно-биологические свойства бактерий-деструкторов (хлор)ароматических соединений».
Список литературы Олигонуклеотидные праймеры для детекции генов, кодирующих большую субъединицу бифенил 2,3-диоксигеназы, бактерий порядка Actinomycetales
- Васильева Г.К., Стрижакова Е.П. Биоремедиация почв и седиментов, загрязненных полихлорированными бифенилами//Микробиол. 2007. T. 76, № 6. С. 725-741.
- Патрушев Л.И. Искусственные генетические системы. М.: Наука, 2004. T. 1: Генная белковая инженерия. 526 с.
- Плотникова Е.Г. и др. Выделение и характеристика новых бактерий, разлагающих полихлорированные бифенилы//Сб. тез. IV Междунар. конф. «Проблемы загрязнения окружающей среды». Пермь, 1998. С. 160.
- Плотникова Е.Г. и др. Бактерии-деструкторы полициклических ароматических углеводородов, выделенные из почв и донных отложений района солеразработок//Микробиол. 2001. Т. 70,. № 1. С. 61-70.
- Плотникова Е.Г. и др. Штамм бактерий Rhodococcus ruber -деструктор полихлорированных бифенилов: пат. № 2262531 Рос. Федерации; опубл. 20.10.05. Бюл. № 29.
- Шумковa Е.С. и др. Скрининг и изучение ключевых генов катаболизма бифенила у бактерий//Вестн. Перм. ун-та. 2008. Вып. 9. Биология.С. 53-57.
- Шумкова Е.С. Генетические и ферментные системы деструкции ароматических соединений бактерий порядка Actinomycetales: автореф. дис. канд. биол. наук. Пермь, 2009. 27 с.
- Compton T. Degenerate primers for DNA amplification//PCR Protocols/Ediors M.A. Innis, D.H. Gelfand, J.J. Sninsky, T.J. White. New York: Academic Press, 1990. P. 39-45.
- Ferraro D. J. et al. Structural investigations of the ferredoxin and terminal oxygenase components of the biphenyl 2,3-dioxygenase from Sphingobium yanoikuyae B1//BMC Structural Biology. 2007. Vol. 7. P. 1170-1182.
- Gibson D.T., Parales R.E. Aromatic hydrocarbon dioxygenases in environmental biotechnology//Curr. Opin. Biotechnol. 2000. Vol. 11. P. 236-243.
- Jakoncic J. et al. The catalytic pocket of the ringhydroxylating dioxygenase from Sphingomonas CHY-1//Biochem. Biophys. Res. Commun. 2007. Vol. 352. P. 861-866.
- Kimura N. et al. Functional analyses of a variety of chimeric dioxygenases construated from two biphenyl dioxygenases that are similar structurally but different functionally//J. Bacteriol. 1997. Vol. 179. P. 3936-3943.
- Kwok S. et al. A guide to the design and use of mismatched and degenerate primers//PCR Methods and Appl. 1994. Vol. 3. P. 39-47.
- Linhart C., Shamir R. The degenerate primer design problem: theory and Applications//J. of Computational Biology. Vol. 12, N 4. 2005. P. 431-456.
- Masai E. et al. Characterization of biphenyl catabolic genes of gram-positive polychlorinated biphenyl degrader Rhodococcus sp. strain RHA1//Appl. Environ. Microbiol. 1995. Vol. 61. P. 2079-2085.
- Mukerjee-Dhar G. et al. bph Genes of the thermophilic PCB degrader, Bacillus sp. JF8: characterization of the divergent ringhydroxylating dioxygenase and hydrolase genes upstream of the Mn-dependent BphC//Microbiol. 2005. Vol. 151. P. 4139-4151.
- Pieper D.H. Aerobic degradation of polychlorinated biphenyls//Appl. Microbiol. Biotech. 2005. Vol. 67. P. 170-191.
- Romine M.F. et al. Complete sequence of a 184-kilobase catabolic plasmid from Sphingomonas aromaticivorans F199//J. Bacteriol. 1999. Vol. 181, № 5. P. 1585-1602.
- Ross G. The public health implications of polychlorinated biphenyls (PCBs) in the environment//Rev. Ecotox. Environ. Safety. 2004. Vol. 59. P. 275-291.
- Seeger M. et al. Dehalogenation, denitration, dehydroxylation, and angular attack on substituted biphenyls and related compounds by a biphenyl dioxygenase//J. Bacteriol. 2001. Vol. 183. P. 3548-3555.
- Suenaga H., et al. Alteration of regiospecificity in biphenyl dioxygenase by active-site engineering//J. Bacteriol. 2002. Vol. 184. P. 3682-3688.
- Taguchi К. et al. Polychlorinated biphenyl/biphenyl degrading gene clusters in Rhodococcus sp. K37, HA99, and TA431 are different from well-known bph-gene clusters of Rhodococci//Biosci. Biotechnol. Biochem. 2007. Vol. 71, № 5. P. 1136-1144.
- Witzig R. et al. Assessment of toluene/biphenyl dioxygenase gene diversity in benzene-polluted soils: links between benzene biodegradation and genes similar to those encoding isopropylbenzene dioxygenases//Appl. Environ. Microbiol. 2006. Vol. 72, N 5. P. 3504-3514.
- Yang X. et al. Characterization and functional analysis of a novel gene cluster involved in biphenyl degradation in Rhodococcus sp. strain R04//J. Appl. Microbiol. 2007. Vol. 103, № 6. P. 2214-2224.
- Zielinski M. et al. The principal determinants for the structure of the substrate-binding pocket are located within a central core of a biphenyl dioxygenase alpha subunit//Microbiol. 2002. Vol. 148. P. 2439-2448.


