Оптимизация метода выделения двуцепочечной РНК, гетерологично экспрессированной в Escherichia coli HT115(DE3)
Автор: Фадеев Р.Р., Кудрявцева Ю.С., Байазыт К.Д.К., Шухалова А.Г., Долгих В.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: ДНК-технологии
Статья в выпуске: 3 т.59, 2024 года.
Бесплатный доступ
Эффективность РНК-интерференции (РНКи) в подавлении транскрипционной активности генов-мишеней у насекомых-вредителей, фитопатогенных вирусов и грибов определяет интерес к биосинтезу двуцепочечной РНК (дцРНК) в бактериях Escherichia coli как менее затратной технологии по сравнению с синтезом in vitro. Упрощение процедуры выделения дцРНК из бактерий может снизить затраты на производство дцРНК и способствовать расширению применения РНКи в защите растений. В представленной работе впервые разработана достаточно простая и эффективная методика выделения и очистки дцРНК из бактериальной культуры Escherichia coli для дальнейшего изучения возможностей ее использования в сельскохозяйственной практике. Нашей целью было конструирование оригинального вектора для эффективной экспрессии молекул дцРНК в бактериях Escherichia coli HT115(DE3), а также оптимизация условий выделения нуклеиновых кислот из бактериальных клеток, обработки экстрактов РНКазой А и последующего выделения дцРНК с использованием кислого фенола. При создании нового вектора для получения дцРНК в бактериях E. coli HT115(DE3) мы использовали плазмиду pRSETа («Thermo Fisher Scientific», США) без участка, кодирующего N-концевой рекомбинантный таг. Чтобы встроить дополнительный промотор Т7 в состав вектора, плазмиду реамплифицировали с прямым праймером 5´-ССCTATAGTGAGTCGTATTAgaaaggaagctgagttggctgctg-3´ (промотор Т7 подчеркнут) и обратным 5´-CTTTGTTAGCAGCCGGATCAAG-3´, а также смесью для ПЦР Phusion Flash High-Fidelity PCR Master Mix («Thermo Fisher Scientific», США). Фосфорилирование концов ПЦР-продукта и их лигирование с помощью T4 полинуклеотидкиназы и Т4 ДНК-лигазы («Thermo Fisher Scientific», США) позволили получить оригинальный вектор для экспрессии дцРНК в бактериях. Для биосинтеза дцРНК в E. coli были использованы фрагменты генов, кодирующих субъединицу beta'-COP коатомерного комплекса колорадского жука Leptinotarsa decemlineata , одну из эндонуклеаз зеленой персиковой тли Myzus persicae , а также встроенные в один полилинкер фрагменты генов субъединицы А вакуолярной АТФазы, субъединицы 7 (Mov34) 26S протеасомы и актина L. decemlineata . Сконструированные плазмиды вводили в клетки E. coli HT115(DE3) с помощью электропоратора 2510 («Eppendorf», Германия) при 1700 В. Встраивание в новый вектор фрагментов генов L. decemlineata и M. persicae , последующая трансформация бактерий E. coli HT115(DE3) созданными конструкциями, экспрессия и выделение молекул дцРНК продемонстрировали высокую эффективность их биосинтеза, сравнимую с таковой для традиционного вектора L4440. Оптимизация условий экстракции нуклеиновых кислот из бактериальных клеток для последующей обработки экстрактов РНКазой А показала эффективность обработки ультразвуком, а также прогревания в воде при 95 °С. Для удаления одноцепочечной РНК оптимальным оказалось присутствие в реакционной смеси 0,4 М NaCl и 1,25 мкг фермента РНКазы А («Thermo Fisher Scientific», США) на 1 мл реакционной смеси. Обработка реакционной смеси кислым фенолом (рH 4,0), а затем хлороформом необходимы не только для избирательной экстракции дцРНК, но и для инактивации стабильной РНКазы А. Обработка реакционной смеси только хлороформом не приводила к полной инактивации фермента. Количество дцРНК, выделенной из бактерий, которые были обработаны ультразвуком перед инкубацией с РНКазой А, при подобранных нами условиях и использованном оборудовании составило 9,1±1,2 мкг/мл культуры, что оказалось соизмеримым с результатами более сложного метода экстракции, основанного на прогревании с детергентом додецилсульфатом натрия и последующей фенол-хлороформной экстракции (8,8±0,6 мкг/мл культуры). При прогревании бактерий в течение 20 мин в воде при 95 °С перед инкубацией с РНКазой А выход дцРНК составил приблизительно 55 % от значений, наблюдаемых при использовании двух других методов (5,0±1,5 мкг/мл культуры).
Насекомые-вредители, фитопатогены, рнк-интерференция, двуцепочечные рнк, экстракция, биосинтез, выделение
Короткий адрес: https://sciup.org/142242456
IDR: 142242456 | УДК: 632.9:577.2 | DOI: 10.15389/agrobiology.2024.3.460rus
Текст научной статьи Оптимизация метода выделения двуцепочечной РНК, гетерологично экспрессированной в Escherichia coli HT115(DE3)
В 1998 году у нематоды Caenorhabditis elegans впервые было описано явление РНК-интерференции (РНКи), представляющее собой посттранскрипционное подавление экспрессии генов, которое инициируется нали- чием в эукариотической клетке их фрагментов в виде молекул двуцепочечной РНК (дцРНК) (1). Поскольку дальнейшие исследования показали эффективное и специфичное подавление транскрипционной активности генов насекомых-вредителей (2, 3), фитопатогенных грибов (4, 5) и вирусов растений (6, 7) с помощью РНКи, были разработаны различные стратегии синтеза дцРНК в условиях in vivo и in vitro c выделением дцРНК для последующего изучения и практического использования.
Стратегия синтеза in vitro позволяет получить чистую дцРНК, но ее стоимость достаточно высока в связи с использованием очищенных ферментов, нуклеотидов и специально подготовленной ДНК-матрицы (8). Значительно ниже себестоимость биосинтеза дцРНК in vivo (9) в штамме бактерий Escherichia coli BL21(DE3) с искусственно встроенным геном РНК-полимеразы фага Т7. Для этого на основе бактериальной плазмиды pPD129.36 был сконструирован вектор L4440 с полилинкером, фланкированным двумя копиями T7 промотора, распознаваемыми РНК-полимеразой фага T7 и расположенными на разных цепях ДНК. Позднее эти же авторы показали, что использование штамма E. coli HT115(DE3), лишенного гена РНКазы III, позволяет значительно повысить эффективность накопления дцРНК в бактериях (10).
Несмотря на то, что синтез in vivo дает высокий выход относительно недорогой дцРНК (11), особенно в биореакторах при периодическом добавлении субстрата (12), такая стратегия требует последующей очистки продукта. Даже при эффективном использовании грубых, частично очищенных экстрактов для борьбы с фитопатогенами и вредителями (3, 6) сохраняется необходимость выделения и очистки дцРНК с целью оценки эффективности биосинтеза в бактериях и контрольных измерений точной концентрации в приготовленных пробах.
Первая методика выделения дцРНК из клеток E. coli HT115(DE3) была основана на фенол-хлороформной экстракции нуклеиновых кислот (НК) из бактерий при 65 ° С, их переосаждении спиртом с последующей обработкой ферментами ДНКазой I и бычьей панкреатической РНКазой А для удаления ДНК и молекул одноцепочечной РНК (оцРНК) (11). Впоследствии методику успешно использовали другие авторы (13). При этом следует отметить, что панкреатическая РНКаза А специфично расщепляет оцРНК (но не дцРНК) только в присутствии высоких концентраций соли (не менее 0,3 М NaCl) (14, 15). Поскольку при низком содержании соли этот фермент эффективно гидролизует и дцРНК, и оцРНК, в ряде публикаций подчеркивается, что обработка бактериальных экстрактов РНКазой A осуществлялась в присутствии 0,3 M NaCl (6, 16).
В некоторых работах схема очистки дцРНК из бактериальных экстрактов включала две экстракции фенолом и хлороформом (17) или такими содержащими фенол и гуанидин реагентами, как QIAzol («Qiagen», США) и TRIzol («Thermo Fisher Scientific», США) (16), до и после обработки НК ферментами РНКазой А и ДНКазой I. В последней работе также сравнили влияние предварительной обработки бактерий с помощью лизоцима, ультразвука (УЗ) и повышенной температуры (60 ° С) на эффективность последующей экстракции общей РНК в присутствии лизирующего реагента QI-Azol (16). Наибольшую эффективность экстракции показала УЗ-обработка бактерий. Последующая обработка полученных экстрактов РНКазой A в буфере, содержащем 0,3 М NaCl, или избирательное осаждение дцРНК в присутствии 4 М LiCl обеспечивали одинаковую степень очистки дцРНК. При этом обработка экстрактов РНКазой А не в присутствии 0,3 М NaCl, а в коммерческом буфере для TURBO™ ДНКазы («Thermo Fisher Scientific»,
США) приводила к значительной деградации дцРНК.
Упрощение процедуры выделения дцРНК из бактерий может снизить затраты на производство дцРНК и расширить применение РНКи в защите растений (18).
В представленной работе впервые предложена достаточно простая и эффективная методика выделения и очистки дцРНК из бактериальной культуры Escherichia coli для дальнейшего изучения возможностей использования дцРНК в сельскохозяйственной практике.
Нашей целью было конструирование оригинального вектора для эффективной экспрессии молекул дцРНК в бактериях Escherichia coli HT115(DE3), а также оптимизация условий выделения нуклеиновых кислот из бактериальных клеток, обработки экстрактов РНКазой А и последующего выделения дцРНК с использованием кислого фенола.
Методика. При создании нового вектора для получения экспрессии дцРНК в бактериях использовали плазмиду pRSETа («Thermo Fisher Scien-tific», США) без участка, кодирующего N-концевой рекомбинантный таг (19). Чтобы встроить дополнительный промотор Т7 в состав вектора, лишенного последовательности рекомбинантного тага между нуклеотидами в позициях 103 и 201 (19), плазмиду реамплифицировали с прямым праймером 5´-СС CTATAGTGAGTCGTATTA GAAAGGAAGCTGAGTTGGCTGCTG-3´ (в его составе подчеркнута последовательность Т7 промотора) и обратным праймером 5´-CTTTGTTAGCAGCCGGATCAAG-3´, используя смесь для ПЦР Phusion Flash High-Fidelity PCR Master Mix («Thermo Fisher Scientific», США). Для получения экспрессирующего дцРНК вектора проводили фосфорилирование концов ПЦР-продукта и их лигирование с помощью T4 по-линуклеотидкиназы и Т4 ДНК-лигазы («Thermo Fisher Scientific», США) в соответствии с протоколом производителя.
Для последующего биосинтеза дцРНК в E. coli в созданный вектор встраивали фрагменты генов, кодирующих субъединицу beta’-COP коатомер-ного комплекса колорадского жука Leptinotarsa decemlineata (номер в NCBI XM_023172101.1, Ld beta’-COP), одну из эндонуклеаз зеленой персиковой тли Myzus persicae (MN257576.1, Mp Nuc1), а также объединенные в одном полилинкере фрагменты генов субъединицы А вакуолярной АТФазы (XM_023156517.1), субъединицы 7 (Mov34) 26S протеасомы (XM_023171125.1) и актина (KJ577616.1) L. decemlineata (Ld V-ATPase A–Mov34–actin). Методика выделения общей РНК с помощью реагента TRIzol («Thermo Fisher Sci-entific», США), синтеза кДНК и ее использования в качестве матрицы для ПЦР-амплификации фрагментов генов описаны ранее (20), личинки насекомых гомогенизировали в жидком азоте. Каждый из праймеров для ПЦР-амплификации фрагментов генов насекомых-вредителей дополнительно содержал 5´-концевые сайты рестрикции для их клонирования в экспрессирующий дцРНК вектор. Фрагмент гена, кодирующего Ld beta’-COP, был встроен в полилинкер вектора по сайтам рестрикции EcoRI/HindIII, Mp Nuc1 — по сайтам BamHI/HindIII. Последовательности, кодирующие Ld V-ATPase A, Ld Mov34 и Ld актин, клонировали в одну и ту же плазмиду по сайтам BamHI/XhoI, XhoI/KpnI и KpnI/HindIII («Thermo Fisher Scientific», США) в соответствии с протоколом производителя. В трех независимо сконструированных плазмидах размеры фрагментов, фланкированных промоторами Т7, составили соответственно 399 (Ld beta’-COP), 677 (Mp Nuc1) и 867 (Ld V-ATPase A–Mov34–actin) п.н.
Сконструированные плазмиды вводили в клетки E. coli HT115(DE3) с помощью электропоратора 2510 («Eppendorf», Германия) при 1700 В. Свежие колонии трансформированных бактерий, выросшие на агаризованной 462
среде LB (10 г бакто-триптона, 5 г бакто-дрожжевого экстракта, 10 г NaCl в 1 л среды) с ампициллином (0,15 мг/мл), инокулировали в колбы с жидкой средой 2 УТ (16 г бакто-триптона, 10 г бакто-дрожжевого экстракта, 5 г NaCl в 1 л среды), содержащей ампициллин и тетрациклин в концентрациях соответственно 0,1 и 0,012 мг/мл, растили при 37 ° С с использованием термостатируемого орбитального шейкера Unimax 1010 («Heidolph Instruments GmbH & Co KG», Германия) до оптической плотности клеточной суспензии OD 600 = 0,6 и индуцировали одновременную экспрессию плюс- и ми-нус-цепей соответствующего фрагмента добавлением изопропил-бета-D-тиогалактопиранозида до конечной концентрации 0,5 мМ. После синтеза дцРНК в течение 5 ч при 37 ° С бактерии осаждали на центрифуге Universal 320R («Heidolph Instruments GmbH & Co KG», Германия) при 6000 g в течение 10 мин, дважды промывали очищенной с помощью установки Simplicity® («Merck», Германия) водой в объеме, равном объему среды для культивирования, и осадок хранили при - 80 ° С.
Для выделения общей РНК с помощью одного из стандартных методов клетки ресуспендировали в объеме очищенной на установке Simplicity® («Merck», Германия) воды, равном объему бактериальной культуры, прогревали при 95 ° С в присутствии 0,5 % (конечная концентрация) анионного детергента додецилсульфат натрия (SDS), экстрагировали общую РНК с помощью равного объема фенола, уравновешенного 0,1 М Na-ацетатным буфером (рН 4,0), и дважды таким же объемом хлороформа. К экстрагированной водной фазе добавляли равный объем изопропанола и после 3-кратного замораживания при - 80 ° С и оттаивания РНК осаждали на центрифуге Universal 320R в течение 30 мин при 14 000 g и 4 ° С. Осадок промывали 70 % этанолом и растворяли в очищенной с помощью установки Simplicity® («Merck», Германия) воде для измерения концентрации РНК с помощью нанофотометра NP80 («Implen GmbH», Германия) и подбора условий обработки ферментом РНКаза А («Thermo Fisher Scientific», США).
Одна из двух упрощенных схем экстракции НК из E. coli включала УЗ-обработку водной суспензии бактерий (0,6 мл) 3 раза в течение 10 с и с такими же по времени перерывами для ее охлаждения при 4 ° С и амплитуде 50 %. Мы использовали ультразвуковой дезинтегратор Q700MPX («QSonica», США), оснащенный зондом Cup Horn («QSonica», США) для бесконтактной обработки проб в микроцентрифужных пробирках. Еще более упрощенная процедура экстракции НК из бактерий включала прогревание водной суспензии при 80 или 95 ° С в течение 20 мин. В ряде экспериментов для повышения эффективности разрушения бактерий к суспензии предварительно добавляли SDS до конечной концентрации 0,02 или 0,1 %. После обеих процедур экстракции НК клеточный дебрис осаждали на центрифуге Universal 320R при 8000 g 10 мин, а отобранные супернатанты обрабатывали РНКазой А («Thermo Fisher Scientific», США) без выделения РНК кислым фенолом, хлороформом и спиртового переосаждения, как в более сложной стандартной схеме получения экстрактов.
Для оптимизации условий обработки РНКазой А использовали бактериальную РНК, выделенную с помощью описанной выше стандартной схемы, включающей предварительную экстракцию кислым фенолом и хлороформом с последующим спиртовым переосаждением. Выделенную РНК в концентрации 125 мкг/мл инкубировали 30 мин при 37 ° С в присутствии следующих компонентов (указаны конечные концентрации): 25 мМ Трис-HCl буфер (рН 7,4); 5 мМ EDTA (этилендиаминтетраацетат, рН 8,0); 0,15; 0,3 или 0,4 М NaCl; 1,25 мкг/мл реакционной смеси РНКазы А («Thermo Fisher Scientific», США). Во всех последующих экспериментах концентрация
NaCl в реакционной смеси составляла 0,4 M. При оптимизации количества РНКазы А, добавленной в реакционную смесь для обработки общей РНК, конечная концентрация фермента составляла 0,25; 1,25 или 6,25 мкг/мл.
Обработку РНКазой А экстрактов, полученных по упрощенной схеме (разрушение бактерий УЗ или прогреванием без экстракции фенолом и хлороформом), проводили при описанных выше условиях, но с использованием только оптимальных конечных концентраций NaCl (0,4 М) и РНКазы А (1,25 мкг/мл). После обработки ферментом дцРНК выделяли с помощью экстракции равным объемом кислого фенола (рН 4,0) и 2-кратно тем же объемом хлороформа. В сравнительном эксперименте была проведена лишь 1-кратная экстракция дцРНК равным объемом хлороформа. После добавления к полученным водным фазам 1/ 10 объема 3 М Na-ацетатного буфера (рН 5,0) и равного объема изопропанола смеси инкубировали в течение 1 ч при - 20 ° С, центрифугировали (Universal 320R) 20 мин при 14 000 g и 4 ° С, дважды промывали осадки 70 % этанолом и растворяли их в очищенной на установке Simplicity® воде для дальнейшего анализа.
Для инкубации с РНКазой III или ДНКазой I использовали дцРНК, выделенную после обработки бактерий УЗ. В 10 мкл реакционной смеси содержались 0,4 мкг дцРНК, 1 Ед. ДНКазы I или 2 Ед. ShortCut® РНКазы III (оба фермента производства «New England Biolabs», США) и соответствующий реакционный буфер того же производителя. Реакционная смесь для РНКазы III также содержала 20 мM MnCl 2 («New England Biolabs», США). Инкубацию проводили при 37 ° С в течение 30 мин.
Полученные пробы разделяли электрофорезом в 1 % агарозном геле с ТАЕ-буфером, содержащим флуоресцентный краситель бромистый этидий (3,8-диамино-5-этил-6-фенилфенантридиум бромид) для визуализации НК в геле при X = 312 нм (трансиллюминатор TCP-15.M, «Vilber Lourmat», Франция).
Содержание дцРНК в трижды независимо выделенных для каждой методики пробах определяли с помощью нанофотометра NP80 («Implen GmbH», Германия), используя настройки прибора для измерения концентрации молекул дцДНК.
Вычисляли средние значения количественных показателей ( M ) и их стандартные ошибки (±SEM). Статистическую значимость различий эффективности выделения дцРНК из клеток E. coli при использовании трех методик разрушения бактерий оценивали методом попарных сравнений с помощью t -критерия Стьюдента (21).
Результаты. Поскольку использованный нами вектор уже содержал высокоактивный промотор РНК-полимеразы фага Т7 на одной цепи плазмиды pRSETа за 65 п.н. от 5´-концевого сайта рестрикции BamHI в составе полилинкера, мы лишь добавили идентичную последовательность в состав комплементарной цепи (рис. 1, А).
Встроенная копия промотора Т7 в сконструированном векторе располагалась на расстоянии 21 п.н. от 3´-концевого сайта рестрикции HindIII полилинкера. Таким образом, экспрессия фрагмента ДНК, встроенного в этот вектор, добавляла к синтезируемой молекуле дцРНК от 86 до 139 п.н. в зависимости от выбранных для клонирования сайтов рестрикции. Как будет показано ниже, встраивание в новый вектор фрагментов генов L. decem-lineata и M. persicae (см. рис. 1, Б, В, Г), последующая трансформация бактерий E. coli HT115(DE3) созданными конструкциями, экспрессия и выделение молекул дцРНК продемонстрировали высокую эффективность их биосинтеза, сравнимую с таковой для традиционного вектора L4440 (16).
При выделении РНК из клеток E. coli мы взяли за основу стандарт- ную методику, включающую прогревание бактерий при 95 °С в присутствии SDS, экстракцию общей РНК кислым фенолом и хлороформом, ее спиртовое переосаждение с последующей инкубацией с РНКазой А. Для упрощения этой схемы мы исключили этапы экстракции РНК кислым фенолом и хлороформом, а также спиртового переосаждения перед обработкой ферментом. Водную суспензию бактерий после экспрессии дцРНК или обрабатывали УЗ, или прогревали при 80 и 95 °С в воде либо в присутствии анионного детергента SDS, а затем центрифугировали для дальнейшей обработки супернатанта РНКазой А.
А pRSETa Т7 промотор плюс-цепь —-
1GATCTCGATCCCGCGAAAT TAATACGACTCACTATAG GGAGACCACAACGGTTTCCCTCTA
62GAAATAATTTTGTTTAACTTTAAGAAGGAGATATACATATG CGGGGTTCTCATCATCATCATCAT
127 CATGGTATGGCTAGCATGACTGGTGGACAGCAA A TGGGTCGGGATCTGTACGACGATGACG
BamHI полилинхер Нтслп
ISSATAAGGAreGAEGGGGATCCGAGCTCGAGATCTGCAGCTGGTACCATGGAATTCGAAGCTr
249GATCCGGCTGCTAACAAAGCC CTATAGTGAGTCGTATTA GAAAGGAAGCTGAGTT ---встроенный Т7 промотор минус-цепь
Б
Ld beta’-COP
TA/I^CG^CrCACMT/iG GGAGACCACAACGGTTTCCCTCTAGAAATAAnTTGTTTAACTTTAAGAAGGAGATATACATATGGGAT ccgagctcgagatctgcagctggtaccatg GAATTCATTCCTTTGAGGCAC ATTCCGACTATGTGAGATGT ATAATAGTTCATCCAACCCAACCTTATATTCTGACAAGTAGTGATGACATGCTCATCAAATTGT GGAATTGGGATAAGGCTTGGGCTTGTCAGCAGGTCTTTGAAGGCCACTCTCATTATATCATG CAAATCGCCATCAATCCAAAAGACAATAACACATTTGCGAGTGCTTCACTGGACCGTACTTT GAAAGTGTGGCAACTTGGAGCAT CAACAGCGAACTTCACCCTTGAAGCTT gatccggctgctaaca AAGCCCTAraGIGACTCGMTM
В
Mp Nucl
ZMMcgactcacmmgggagaccacaacggtttccctctagaaataattttgtttaactttaagaaggagatatacatatgGGA TCCTCCGACGCGTTGGTCACGTGCGTCGGCGGCGACGTGCTCGCGTACCGAGGCCAGAC gtaccggtacgccgccttccagtgcgacggcatgcccaagtcggagctccgcgtcaccg acgaagtgtgccagccggcgaagtacaccgtggccgtggtcggtttccggacggatcgc gcgttcctccggctgtacggcatgtgcttcgacaagtcgaccaagaagagcctgtacactt ggtacgacgcccggtcgccgtactacgacaaccaccagaagtatagcaagcgaccgtcgt tcaacaaaaccaaagagctgtacggcaacacggacgtgaacaagaaatacactttcaaaga acagagaaacacggtggcgacaatactcagatcggacgaacttgcggataagtacataagg aatgataataaacacttcctgtcccggggccattacgcagccaaagttgactttttctttgct tatgaacaaacagcaacgttttactatgcgaacgttgccccgcagtggcagatcttcaacg GTGACATGTGGGCCGATCTGGAATCGACTACCCGGTCAAAGCTTcatccggctgctaacaaagcccth TAGTGAGTCGTATTA
г
Ld V-ATPase A—Mov34—actin mawcgactcactatagggagaccacaacgctttccctctagaaataattttgtttaactttaagaaggagatatacatatgGGA TCCCAACTTGATGTTGATTGGGTTGAATTCCCATTTGATAGTTCTGGAGAGTGAAGGCACGT tgacagccttgggaatgtagatactttccgtcatgtcacagatgtctttcagcggacgttgg ATACCATCAAAGATGGAACCCATAATACCTGGCCCAAGTTCCACAGATAGAGGTTTACCGGT acgcaacacaggatctcccaccgttactccagaagtttcttcatatacctgctcgaggcaac TGTGGTATCTTTGATATCCCTCAGAAGGTGCTCCACTCCCACCTCCTCTGCTTCTTCCGCCC CTATTTCGCTAGGTACATGCTCAAATGTTTTGGAAGTGGGCGATCCATCATCGTGGACTTCC TCCACAGCTTGATATGCCTCGGTTGGGAGGCCTAGATCCTTGGGTTTGGCATCAATTATTAC CGACACAGAATTGGGACAGTATCTCCTAATTAGTTCGTTTATTGCTATGTCATTCTGGTCTAAT TTTGGACCTGTGTGGTACCTCCAGAGAGGACGGTGTTGGCGTACAAGTCCTTACGGATATCA ACGTCACACTTCATGATGGAGTTGTAGACGGTTTCGTGGATACCGCTAGATTCCATACCCAA GAAGGAAGGTTGGAACAAGGCTTCTGGGCAACGGAACCTCTCGTTACCAATGGTGATGAC ctgtccatcgggaagttcgtagctcttttcgagggaggttgaagcagcagcggtggccat TTCCTGTTCGAAGTCGAGGGCAACATAGCAAAGCTTgatccggctgctaacaaagccctatagtgagtcgt ATTA
Рис. 1. Последовательности фрагментов четырех плазмид, сконструированных на основе вектора pRSETa для экспрессии молекул дцРНК в Escherichia coli HT115(DE3). A — полилинкер вектора pRSETa (в рамке), фланкированный Т7 промоторaми (обозначены жирным шрифтом и подчеркиванием). Участок, кодирующий рекомбинантный таг и ранее удаленный из состава вектора, обозначен серым цветом и зачеркнут; Б — фрагмент гена, кодирующего субъединицу beta’-COP коатомерного комплекса колорадского жука Leptinotarsa decemlineata (XM_023172101.1); В — фрагмент гена, кодирующего одну из эндонуклеаз зеленой персиковой тли Myzus persicae (MN257576.1); Г — встроенные в один полилинкер фрагменты генов субъединицы А вакуолярной АТФазы (XM_023156517.1), субъединицы 7 (Mov34) 26S протеасомы (XM_023171125.1) и актина (KJ577616.1) L. decemlineata (Ld V-ATPase A–Mov34–actin). Мелким шрифтом обозначены участки из состава вектора, добавляемые к синтезируемой дцРНК. Последовательности Т7 промотора показаны жирным курсивом и подчеркнуты. Сайты рестрикции, использованные для клонирования, обозначены серым цветом и жирным шрифтом. Праймеры для ПЦР-ампли-фикации фрагментов подчеркнуты.
Как показал электрофорез полученных НК в агарозном геле (рис. 2, А), наибольшая эффективность экстракции наблюдалась при УЗ-обработке водной бактериальной суспензии. При выбранных условиях и оборудовании 3-кратная обработка бактерий в течении 10 с при 4 ° С с такими же по продолжительности перерывами оказалась достаточной для эффективной экстракции дцРНК. Увеличение времени обработки до 10 циклов по 10 с приводило к разрушению экстрагированной дцРНК и существенным потерям. Прогревание бактериальной суспензии при 80 и 95 ° С в воде либо в присутствии SDS показало, что экстракция была эффективнее при повышенной температуре. При этом добавление детергента не приводило к существенному улучшению результата по сравнению с нагреванием бактерий в воде (см. рис. 2, А). Исходя из того, что денатурирующее действие SDS на белки хорошо известно (22), для дальнейших экспериментов мы выбрали супернатанты после центрифугирования бактериальных суспензий, прогретых в воде при 95 ° С без добавления этого детергента, а также обработанных в воде УЗ.
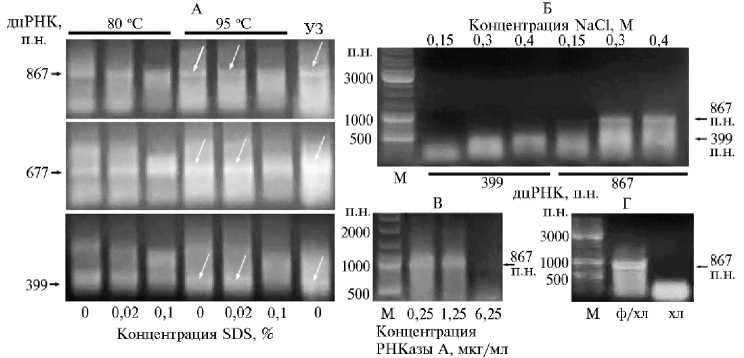
Рис. 2. Электрофореграммы (1 % агарозный гель, ТАЕ-буфер) выделенных разными методами препаратов дцРНК, экспрессированной в Escherichia coli HT115(DE3) после трансформации оригинальным вектором со встроенными фрагментами генов насекомых-вредителей. На основе вектора созданы и использованы три конструкции, содержащие фланкированные двумя разнонаправленными промоторами РНК-полимеразы фага Т7 фрагменты гена Leptinotarsa decemlineata beta’-COP (ожидаемый размер фрагмента 399 п.н.), гена нуклеазы тли Myzus persicae Nuc1 (677 п.н.) или объединенные в одном полилинкере фрагменты генов субъединицы А вакуолярной АТФазы, субъединицы 7 (Mov34) 26S протеасомы и актина L. decemlineata (867 п.н.).
А — УЗ-обработка и прогревание бактериальной суспензии при 80 и 95 ° С в присутствии разных концентраций анионного детергента додецилсульфат натрия (SDS). Белыми стрелками отмечена наиболее эффективная экстракция дцРНК из бактерий; Б — обработка экстрактов РНКазой А (1,25 мкг/мл реакционной смеси) в присутствии разных концентраций NaCl; В — обработка экстрактов дцРНК РНКазой А в концентрациях 0,25; 1,25 и 6,25 мкг/мл реакционной смеси; Г — экстракция обработанной ферментом дцРНК фенолом-хлорофор-мом (ф/хл) или только хлороформом (хл). M — маркер молекулярных масс GeneRuler DNA Ladder Mix («Thermo Fisher Scientific», США).
Прежде чем приступить к обработке полученных экстрактов РНКазой
A, мы оптимизировали этот процесс с использованием РНК, выделенной ранее с помощью более сложного стандартного метода, включающего прогревание бактерий с SDS, экстракцию РНК кислым фенолом и хлороформом, осаждение и промывание спиртом с последующим растворением в воде. Поскольку специфичный гидролиз оцРНК с помощью бычьей панкреатической РНКазы A в присутствии высокой концентрации соли — эффективный и часто используемый подход при выделении дцРНК (6, 14-16), полученную бактериальную РНК (125 мкг/мл реакционной смеси) инкубировали в присутствии РНКазы А (1,25 мкг/мл реакционной смеси) и разных концентраций NaCl. Показано, что при концентрации соли 0,15 M наблюдался гидролиз дцРНК под действием фермента (см. рис. 2, Б). При концентрации NaCl 0,3 M и выше фрагменты дцРНК разного размера стабильно сохранялись в реакционной смеси. Поскольку присутствие 0,4 M NaCl оказалось более надежным условием для предотвращения гидролиза дцРНК и обработанная проба содержала меньше деградированной РНК ниже целевой полосы по сравнению с 0,3 M NaCl (см. рис. 2, Б), эта концентрация была использована нами в дальнейших экспериментах.
На следующей стадии оптимизации условий обработки РНК ферментом в реакционную смесь добавляли разные количества РНКазы А в концентрациях 0,25; 1,25 и 6,25 мкг/мл реакционной смеси (см. рис. 2, В). Согласно результатам электрофоретического анализа проб, обработанных разным количеством фермента, использованная нами в предыдущем эксперименте концентрация 1,25 мкг/мл оказалась предпочтительной для дальнейших исследований. Увеличение содержания РНКазы А в реакционной смеси в 5 раз приводило к гидролизу дцРНК, а ее снижение — к менее качественному удалению оцРНК.
На заключительном этапе обработанная ферментом дцРНК была последовательно экстрагирована из реакционной смеси равным объемом кислого фенола и затем дважды равными объемами хлороформа с последующим переосаждением в присутствии 1/ 10 объема 3 М Na-ацетата (pH 5,0) и одного объема изопропанола (см. рис. 2, Г, дорожка ф/хл). Кроме того, обработанную РНКазой А дцРНК экстрагировали из реакционной смеси только равным объемом хлороформа с последующим переосаждением изопропанолом (см рис. 2, Г, дорожка хл). Полученные результаты показали, что применение фенола на заключительном этапе выделения дцРНК необходимо для инактивации РНКазы А, поскольку при использовании только хлороформной экстракции дцРНК в пробах разрушалась (см. рис. 2 , Г, дорожка хл). Вероятно, фермент, оставаясь активным, гидролизует дцРНК после ее переосаждения изопропанолом, обработки 70 % этанолом и растворения в воде.
Количественный анализ дцРНК, полученной при экстракции из бактериальной культуры с помощью стандартного и двух упрощенных методов, с последующими обработками РНКазой А, кислым фенолом и хлороформом, позволил сравнить эффективность трех изученных подходов. Обработка бактерий УЗ оказалась сравнимой по эффективности с более сложным подходом, основанным на прогревании бактерий в присутствии 0,5 % SDS, фенол-хлороформной экстракции и спиртовом переосаждении РНК перед обработкой РНКазой А (табл.). Выход дцРНК после прогревания бактерий в воде при 95 ° С перед обработкой РНКазой А составил приблизительно 55 % от значений, наблюдаемых при использовании двух других методов.
Электрофоретический анализ дцРНК, выделенной из обработанных
УЗ и прогретых при 95 ° С бактерий, подтвердил наличие в геле одиночных полос, соответствующих по подвижности размеру изучаемого фрагмента (рис. 3, А). Обработка дцРНК, выделенной и очищенной после УЗ-обра-ботки бактерий, ферментами РНКазой III и ДНКазой I подтвердила соответственно полное расщепление выделенного продукта специфичной для этой формы РНК нуклеазой (см рис. 3, Б, дорожка РIII) и отсутствие примеси ДНК в полученных препаратах (см. рис. 3, Б, дорожка ДI). Обработка пробы ДНК-специфичной ДНКазой I не приводила к снижению интенсивности свечения одиночной полосы дцРНК по сравнению с контролем (см. рис. 3, Б, дорожка К).
Эффективность выделения дцРНК из Escherichia coli HT115(DE3), трансформированной оригинальным вектором со встроенными фрагментами генов насекомых-вредителей, при использовании трех схем разной степени сложности
Схема выделения дцРНК
Количество дцРНК из 1 мл бактериальной культуры, мкг/мл
Прогревание бактерий в присутствии 0,5 % SDS при 95 °С, выделе- 8,8±0,6а ние РНК с помощью кислого фенола, хлороформа, переосаждение спиртом, обработка РНКазой А, выделение дцРНК с помощью кислого фенола и хлороформа, переосаждение спиртом
Обработка бактерий ультразвуком, РНКазой А, выделение дцРНК с 9,1±1,2а помощью кислого фенола и хлороформа, переосаждение спиртом
Прогревание бактерий в воде при 95 °С, обработка РНКазой А, вы- 5,0±1,5b деление дцРНК с помощью кислого фенола и хлороформа, перео- саждение спиртом
Пр им еч ан и е. Представленные в таблице средние значения ( M ) и их стандартные ошибки (±SEM) получены на основании трех независимых процедур очистки фрагмента гена нуклеазы тли Myzus persicae (MN257576.1) в форме дцРНК размером 677 п.н. Разные буквы обозначают статистически значимые различия (p < 0,05) эффективности выделения дцРНК с помощью разных подходов, проанализированные методом попарных сравнений с помощью t -критерия Стьюдента.
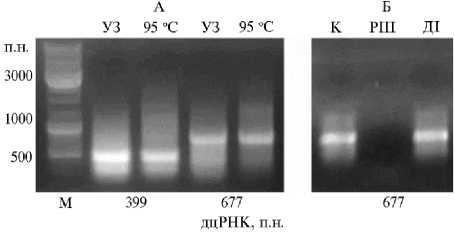
Рис. 3. Электрофоретический анализ (1 % агарозный гель, ТАЕ буфер)
дцРНК, выделенной с помощью предложенной методики после трансформации Escherichia coli HT115(DE3) оригинальным вектором со встроенными фрагментами генов насекомых-вредителей, последующей экспрессии и обработки бактерий ультразвуком или прогреванием. В векторе фрагменты генов Leptinotarsa decemlineata beta’-COP (ожидаемый размер фрагмента 399 п.н) или нуклеазы тли Myzus per- sicae Nuc1 (677 п.н.) фланкированы двумя разнонаправленными промоторами РНК-полимеразы фага Т7. А — разрушение бактериальных клеток посредством УЗ или прогревания в воде при температуре 95 °С (дорожки УЗ и 95 °С), обработка экстрактов РНКазой А (1,25 мкг/мл реакционной смеси), кислым фенолом, хлороформом с последующим спиртовым переосажде-нием. Б — обработка полученных препаратов ферментами РНКазой III и ДНКазой I (дорожки РIII и ДI). Дорожка К — ферменты не были добавлены в контрольную пробу. M — маркер молекулярных масс GeneRuler DNA Ladder Mix («Thermo Fisher Scientific», США).
Таким образом, задача настоящей работе заключалась в том, чтобы упростить методику выделения дцРНК, синтезированной в бактериях E. coli HT115(DE3). Поскольку РНКаза А широко используется для удаления молекул оцРНК из препаратов дцРНК, но необычайно устойчива к высокой температуре и детергентам (23, 24), в предложенной схеме нам пришлось сохранить процедуру экстракции дцРНК кислым фенолом после обработки бактериальных экстрактов этим ферментом для его необратимой денатурации. Как было показано (см. рис. 2, Г), экстракция пробы, обработанной РНКазой А, одним лишь хлороформом не инактивировала фермент и последующее снижение концентрации соли приводило к деградации дцРНК. Кроме того, экстракция фенолом, уравновешенным буферным раствором с низким рН 4,0, позволяла специфично выделить молекулы дцРНК, свободные от примесей ДНК, что подтвердилось при обработке полученных проб ферментом ДНКазой I (рис. 3, Б). Это позволило исключить из нашей методики этап удаления ДНК из препаратов дцРНК с помощью ДНКазы I. При выделении дцРНК мы также предлагаем исключить достаточно трудоемкий этап экстракции РНК из бактерий с помощью фенола-хлороформа или таких фенол-гуанидин содержащих реагентов, как QIAzol, TRIzol (11, 13, 16, 17), перед ее обработкой РНКазой А. Мы показали, что экстракция НК достаточно эффективна при простом прогревании бактерий в воде при 95 °С или их разрушении УЗ.
В заключение следует отметить, что использование предложенной схемы выделения дцРНК и сконструированного на основе плазмиды рRSETа вектора позволяет получить 5-9 мкг дцРНК из 1 мл бактериальной культуры (см. табл.). По данным литературы (13), при использовании традиционного вектора L4440 и разнообразных методов обработки бактериальных клеток с последующим выделением дцДНК выход дцРНК примерно такой же — 3-9 мкг дцРНК из 1 мл бактериальной культуры. Это указывает на сравнимую эффективность использованных подходов, несмотря на значительное упрощение схемы выделения дцРНК в нашем исследовании. Интересно отметить, что для синтезированных в E. coli молекул РНК, несущих на одной цепи и смысловую, и анти-смысловую последовательности для формирования шпилечной структуры, описан оригинальный подход для экстракции дцРНК (25). Метод основан на последовательной инкубации бактерий в течение 5 мин в 75 % этаноле, приготовленном на фосфатносолевом буферном растворе, и затем в течение 1 ч в 0,15 М NaCl с последующим удалением бактерий центрифугированием. В дальнейшем мы планируем изучить эффективность такого подхода для экстракции молекул дцРНК, синтезируемых, как в нашем случае, в клетках E. coli с использованием двух противоположно направленных промоторов без формирования шпилечных структур.
Итак, нами сконструирован оригинальный вектор, полилинкер которого фланкирован двумя разнонаправленными промоторами РНК-полимеразы фага Т7, для одновременной экспрессии в Escherichia coli HT115(DE3) комплементарных цепей РНК. Использование этого вектора для биосинтеза в бактериях молекул дцРНК позволило нам предложить относительно простую, малозатратную по времени и использованным реагентам схему выделения дцРНК. После экспрессии дцРНК в E. coli и осаждения бактерий с помощью центрифугирования их можно отмыть в дистиллированной воде и в виде замороженных осадков длительное время хранить при -80 °С. Для экстракции нуклеиновых кислот бактерии можно разрушить ультразвуком или просто прогреть в воде при 95 °С с последующим удалением дебриса центрифугированием. При использовании УЗ эффективность экстракции дцРНК возрастает приблизительно на 45 % по сравнению с нагреванием. Однако в этом случае необходимо иметь в распоряжении ультразвуковой дезинтегратор и подобрать условия обработки, оптимальные для разрушения бактерий и сохранения целостности дцРНК. Удаление молекул оцРНК из осветленных центрифугированием бактериальных экстрактов может быть достигнуто в процессе обработки в реакционной смеси, содержащей 0,4 М NaCl и 1,25 мкг/мл РНКазы А. Последующая экстракция дцРНК из реакционной смеси равным объемом кислого фенола, уравновешенного 0,1 М Na-ацетатным буфером (рН 4,0), и таким же объемом хлороформа (мы повторяем обработку хлороформом дважды) позволяет избавиться от присут- ствия в полученных пробах значительной части примесей ДНК. Осаждение выделенной дцРНК 1/10 объема 3 М Na-ацетатного буфера (рН 5,0) и одним объемом изопропанола, отмывка 70 % этанолом и растворение в воде позволили провести ее дальнейший анализ. После обработки полученного препарата ДНКазой I интенсивность свечения одиночной полосы соответствующего размера при электрофоретическом анализе в агарозном геле не снижается, тогда как дцРНК-специфичная РНКаза III полностью расщепляет полученный продукт. Спектрофотометрическое определение количества дцРНК, выделенной из единицы объема бактериальной культуры, позволяет точно оценить эффективность биосинтеза различных нуклеотидных последовательностей в клетках E. coli HT115(DE3).
Список литературы Оптимизация метода выделения двуцепочечной РНК, гетерологично экспрессированной в Escherichia coli HT115(DE3)
- Fire A., Xu S., Montgomery M.K., Kostas S.A., Driver S.E., Mello C.C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 1998, 391: 806-811 (doi: 10.1038/35888).
- Baum J.A., Bogaert T., Clinton W., Heck G.R., Feldmann P., Ilagan O., Johnson S., Plae-tinck G., Munyikwa T., Pleau M., Vaughn T., Roberts J. Control of coleopteran insect pests through RNA interference. Nature Biotechnology, 2007, 25: 1322-1326 (doi: 10.1038/nbt1359).
- Petek M., Coll A., Ferenc R., Razinger J., Gruden K. Validating the potential of double-stranded RNA targeting colorado potato beetle mesh gene in laboratory and field trial. Frontiers of Plant Science, 2020, 11: 1250 (doi: 10.3389/fpls.2020.01250).
- Koch A., Biedenkopf D., Furch A., Weber L., Rossbach O., Abdellatef E., Linicus L., Johanns-meier J., Jelonek L., Goesmann A., Cardoza V., McMillan J., Mentzel T., Kogel K.H. An RNAi-based control of Fusarium graminearum infections through spraying of long dsRNAs involves a plant passage and is controlled by the fungal silencing machinery. PLoS Pathogens, 2016, 12: e1005901 (doi: 10.1371/journal.ppat.1005901).
- Niño-Sánchez J., Chen L.H., De Souza J.T., Mosquera S., Stergiopoulos I. Targeted delivery of gene silencing in fungi using genetically engineered bacteria. Journal of Fungi, 2021, 7(2): 125 (doi: 10.3390/jof7020125).
- Tenllado F., Martínez-García B., Vargas M., Díaz-Ruíz J.R. Crude extracts of bacterially ex-pressed dsRNA can be used to protect plants against virus infections. BMC Biotechnology, 2003, 3: 3 (doi: 10.1186/1472-6750-3-3).
- Holeva M.C., Sklavounos A., Rajeswaran R., Pooggin M.M., Voloudakis A.E. Topical application of double-stranded RNA Targeting 2b and CP Genes of cucumber mosaic virus protects plants against local and systemic viral infection. Plants, 2021, 10(5): 963 (doi: 10.3390/plants10050963).
- Mu X., Greenwald E., Ahmad S., Hur S. An origin of the immunogenicity of in vitro transcribed RNA. Nucleic Acids Research, 2018, 46: 5239-5249 (doi: 10.1093/nar/gky177).
- Timmons L., Fire A. Specific interference by ingested dsRNA. Nature, 1998, 395: 854 (doi: 10.1038/27579).
- Timmons L., Court D.L., Fire A. Ingestion of bacterially expressed dsRNAs can produce specific and potent genetic interference in Caenorhabditis elegans. Gene, 2001, 263(1-2): 103-112 (doi: 10.1016/s0378-1119(00)00579-5).
- Abbasi R. Application of double-stranded RNA (dsRNA) produced by E. coli HT115 (DE3) and vector L4440 in reverse genetics studies in insects. Journal of Genetic Resources, 2023, 9(1): 41-47 (doi: 10.22080/jgr.2022.24326.1332).
- Papić L., Rivas J., Toledo S., Romero J. Double-stranded RNA production and the kinetics of recombinant Escherichia coli HT115 in fed-batch culture. Biotechnology Reports, 2018, 20: e00292 (doi: 10.1016/j.btre.2018.e00292).
- Saksmerprome V., Charoonnart P., Gangnonngiw W., Withyachumnarnkul B. A novel and inex-pensive application of RNAi technology to protect shrimp from viral disease. Journal of Virological Methods, 2009, 162(1-2): 213-217 (doi: 10.1016/j.jviromet.2009.08.010).
- Yakovlev G.I., Sorrentino S., Moiseyev G.P., Libonati M. Double-stranded RNA: the variables controlling its degradation by RNases. Nucleic Acids Symposium Series, 1995, 33: 106-108.
- Libonati M., Sorrentino S. Degradation of double-stranded RNA by mammalian pancreatic-type ribonucleases. In: Methods in enzymology, vol. 341 /A.W. Nicholson (ed.). Academic Press, 2001, 341: 234-448 (doi: 10.1016/s0076-6879(01)41155-4).
- Verdonckt T.-W., Broeck J.V. Methods for the cost-effective production of bacteria-derived dou-ble-stranded RNA for in vitro knockdown studies. Frontiers in Physiology, 2022, 13: 836106 (doi: 10.3389/fphys.2022.836106).
- Ahn S.-J., Donahue K., Koh Y., Martin R.R., Choi M.Y. Microbial-based double-stranded RNA production to develop cost-effective RNA interference application for insect pest man-agement. International Journal of Insect Science, 2019, 11: 1179543319840323 (doi: 10.1177/1179543319840323).
- Guan R., Chu D., 1 Han X., Miao X., Li H. Advances in the development of microbial double-stranded RNA production systems for application of RNA interference in agricultural pest control. Frontiers in Bioengineering and Biotechnology, 2021, 9: 753790 (doi: 10.3389/fbioe.2021.753790).
- Dolgikh V.V., Timofeev S.A., Zhuravlyov V.S. Senderskiy I.V. Construction and heterologous overexpression of two chimeric proteins carrying outer hydrophilic loops of Vairimorpha cer-anae and Nosema bombycis ATP/ADP carriers. Journal of Invertebrate Pathology, 2020, 171: 107337 (doi: 10.1016/j.jip.2020.107337).
- Dolgikh V.V., Zhuravlyov V.S., Senderskiy I.V., Ignatieva A.N., Timofeev S.A. Seliverstova E.V. Heterologous expression of scFv fragment against Vairimorpha (Nosema) ceranae hexokinase in Sf9 cell culture inhibits microsporidia intracellular growth. Journal of Invertebrate Pathology, 2022, 191: 107755 (doi: 10.1016/j.jip.2022.107755).
- Mishra P., Singh U., Pandey C.M., Mishra P., Pandey G. Application of student's t-test, analysis of variance, and covariance. Annals of Cardiac Anaesthesia, 2019, 22(4): 407-411 (doi: 10.4103/aca.ACA_94_19).
- Bhuyan A.K. On the mechanism of SDS-induced protein denaturation. Biopolymers, 2010, 93(2): 186-199 (doi: 10.1002/bip.21318).
- Miyamoto T., Okano S., Kasai N. Irreversible thermoinactivation of ribonuclease-A by soft-hy-drothermal processing. Biotechnology Progress, 2009, 25(6): 1678-1685 (doi: 10.1002/btpr.267).
- Bender A.T., Sullivan B.P., Lillis L., Posner J.D. Enzymatic and сhemical-based methods to inactivate endogenous blood ribonucleases for nucleic acid diagnostics. The Journal of Molecular Diagnostics, 2020, 22(8): 1030-1040 (doi: 10.1016/j.jmoldx.2020.04.211).
- Posiri P., Ongvarrasopone C., Panyim S. A simple one-step method for producing dsRNA from E. coli to inhibit shrimp virus replication. Journal of Virological Methods, 2013, 188(1-2): 64-69 (doi: 10.1016/j.jviromet.2012.11.033).


