ПЦР-анализ генов альдоксимдегидратаз нитрилутилизирующих бактерий
Автор: Кузнецова М.В., Максимова А.В., Овечкина Г.В., Максимов А.Ю.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 10, 2009 года.
Бесплатный доступ
Проведена детекция генов альдоксимдегидратаз у нитрилутилизирующих бактерий, выделенных на территории Пермского края. Установлено, что 14,3% бактерий лабораторной коллекции содержали гены альдоксимдегидратаз. У 12 штаммов рода Rhodococcus (20,3%) присутствовали фрагменты ДНК, соответствующие по размеру гену альдоксимдегидратазы. На матрице ДНК одного штамма из группы грамотрицательных микроорганизмов - Pseudomonas fluorescens 3220 - амплифицирована последовательность, соответствующая по размеру гену альдоксимдегидратазы P. chlororaphis B23.
Короткий адрес: https://sciup.org/147204446
IDR: 147204446 | УДК: 579.26:579.222.4
Текст научной статьи ПЦР-анализ генов альдоксимдегидратаз нитрилутилизирующих бактерий
-
a Пермский государственный университет, 614990, Пермь, ул. Букирева, 15
-
b Институт экологии и генетики микроорганизмов УрО РАН, 614081, Пермь, ул. Голева, 13
Проведена детекция генов альдоксимдегидратаз у нитрилутилизирующих бактерий, выделенных на территории Пермского края. Установлено, что 14,3% бактерий лабораторной коллекции содержали гены альдоксимдегидратаз. У 12 штаммов рода Rhodococcus (20,3%) присутствовали фрагменты ДНК, соответствующие по размеру гену альдоксимдегидратазы. На матрице ДНК одного штамма из группы грамотрицательных микроорганизмов – Pseudomonas fluorescens 3220 - амплифицирована последовательность, соответствующая по размеру гену альдоксимдегидратазы P. chlororaphis B23.
Альдоксимы – это интермедиаты биосинтеза некоторых биологически активных веществ, таких как индолилуксусная кислота, цианогенный гликозид и глюкозинолаты в растениях. Так, индолаце-тальдоксимгидролаза катализирует специфическую дегидратазную реакцию индолацетальдоксима в индолацетонитрил. Этот фермент можно обнаружить в некоторых родах грибов и высших растений, и впервые он выделен из Gibberella sp. (Asano et al., 1981).
У бактерий конверсия альдоксимов в нитрилы, а затем в соответствующие карбоновые кислоты происходит путем сочетания работы ферментов альдоксимдегидратазы и нитрилутилизирующих ферментов – нитрилгидратазы и амидазы и/или нитрилазы (Kato et al., 1998):
альдоксимдегидратаза нитрилгидратаза
R-CH=NOH ► R-CN + H2O --► R-CONH2 + H2O амидаза
R-CONH2 + H2O --► R-COOH +NH4+ альдоксимдегидратаза нитрилаза
R-CH=NOH ► R-CN + H2O --► R-COOH +NH4+
Штаммы – продуценты нитрилгидролитических ферментов имеют важное практическое применение: в крупнотоннажном промышленном производстве акриламида и солей акриловой кислоты (Kobayashi et al., 1992; Yanenko et al., 1995). Несмотря на постоянный поиск новых бактерий в природе, информация о распределении систем гидролиза нитрилов в группах микроорганизмов и их физиологическая роль еще не в полной мере определены. Поэтому внимание исследователей, изучающих нитрилутилизирующие ферменты микроорганизмов, привлекли альдоксимдегидратазы – ферменты, катализирующие альдоксимы до нитрильных соединений. В настоящее время выделены альдоксимтрансформирующие микроорганизмы Bacillus sp. OxB-1 и Rhodococcus sp. YH3-3.
Данные штаммы конвертировали альдоксимы через нитрилы в соответствующие карбоновые кислоты путем последовательной работы альдоксимдегидратазы и нитрилгидролизующих ферментов (Asano, Kato, 1998).
Процесс отбора штаммов, обладающих активностью альдоксимдегидратазы, может быть ускорен быстрым скринингом с помощью молекулярных методов детекции генов, кодирующих эти ферменты.
Работа посвящена детекции генов альдоксимдегидратаз у бактерий, способных утилизировать нитрилы карбоновых кислот, ранее выделенных из почвы на территории Пермского края.
Объекты и методы исследования
Объектами исследования являлись бактериальные культуры, утилизирующие нитрилы карбоновых кислот.
Культуры получены ранее в результате селекции на ацето- и/или изобутиронитриле из образцов почв и вод естественной среды и почвы промышленных предприятий (ФГУП "ПЗ им. С.М. Кирова" и ОАО "Бератон"). Всего проанализирована 91 культура микроорганизмов: 59 штаммов грамположительных бактерий (роды Rhodococcus , Arthrobacter , Miсrobacterium , Citreo-bacterium , Nocardia , Gordonia, Aureobacterium ) и 32 штамма грамотрицательных бактерий (роды Pseudomonas , Azomonas , Azotobacter и Acidovorax ).
Препараты хромосомной ДНК грамположи-тельных бактерий получали фенольным методом, модифицированным для выделения ДНК из акти-номицетов (Гловер и др., 1988). ДНК грамотрица-тельных микроорганизмов получали следующим образом: полную петлю биомассы бактериальной культуры инокулировали в 100 мкл стерильной
Таблица 1
Праймеры для ПЦР-анализа генов альдоксимдегидратаз
|
Праймер |
Последовательность |
Размер фрагмента, пар нуклеотидов |
№ в GenBank |
|
|
PsOxd |
F R |
ATGGAATCTGCGATCGACACGCATC TCAGGTGGGCGCGACAACGGCATC |
1058 |
АВ093544 |
|
RhOxd |
F R |
ATGGAATCTGCAATCGGCGAACAC TCAGTGCTCGGCGGTTGTCACCG |
1069 |
АВ094201 |
Н 2 О, выдерживали при 95°С в течение 15 мин в твердотельном термостате «Термит» (Россия) (Stone et al., 1994). Пробы охлаждали, центрифугировали 5 мин при 12 тыс. об./мин. Супернатант использовали в генетических исследованиях.
Детекцию генов альдоксимдегидратаз осуществляли посредством ПЦР с праймерами к генам, кодирующим известные альдоксимдегидратазы грамотрицательных и грамположительных микроорганизмов. Олигонуклеотидные праймеры были разработаны с использованием баз данных GENBANK и по заданным последовательностям синтезированы ООО «Евроген», Москва (табл. 1).
Амплификацию ДНК проводили с применением термостабильной Taq -полимеразы производства ООО «СибЭнзим» (Новосибирск) на термоциклере Т3 (Biometra, Германия). Режим амплификации для всех праймеров включал начальный цикл денатурации – 1 мин при 94°С и завершающий цикл 3 мин при 72°С. Для праймеров RhOxd F1/R1 режим амплификации составлял: 94°С – 20 сек; 64°С – 30 сек; 72°С – 60 сек (35 циклов). Для праймеров PsOxd F1/R1: 94°С – 20 сек; 54°С – 70 сек; 72°С – 60 сек (35 циклов). Электрофоретическое разделение ДНК проводили стандартным способом в 1,5% агарозном геле. Визуализацию результатов осуществляли с помощью системы гель-документации BioDocAnalyze (Biometra, Германия).
Результаты исследований
Известна только хромосомная локализация генов, кодирующих альдоксимдегидрогеназы у бактерий (Xie et al., 2003). Гены, контролирующие гидролиз нитрилов, также имеют хромосомную локализацию (Kobayashi et al., 1992).
Установлено, что у 12 штаммов (20,3%) грам-положительных нитрилутилизирующих бактерий присутствовали фрагменты ДНК, соответствующие по размеру гену альдоксимдегидратазы (рис. 1). Все штаммы принадлежали к роду Rhodococcus (из них 11 – R. erythropolis , 1 – R. rhodochrous ).
У штаммов, принадлежащих к родам Microbacterium , Citreobacterium , Nocardia , Gordonia, Aureo-bacterium , не обнаружены фрагменты генов альдоксимдегидратаз ни с одной парой праймеров к генам гидролитических ферментов.
На матрице ДНК одного штамма из группы грамотрицательных микроорганизмов ( Pseudomonas fluorescens 3220) амплифицирована последовательность, соответствующая гену альдоксимдегидратазы P. chlororaphis B23 (Oinuma et al., 2003).
Нужно подчеркнуть, что в большинстве случаев гены альдоксимдегидратазы были детектированы в штаммах (8), содержащих весь комплекс генов, кодирующих ферменты гидролиза нитрилов: нитрилгидратазу/амидазу и/или нитрилазу (табл. 2).
В трех случаях детектировали ген альдоксимдегидратазы и один из генов, ответственных за синтез гидролитических ферментов.
маркер
о _ —5 1
-§ | р Т
— 7^^:U —f- г | —f- —f- г | —f- к, к, г | “ к, —|-
1 kb-
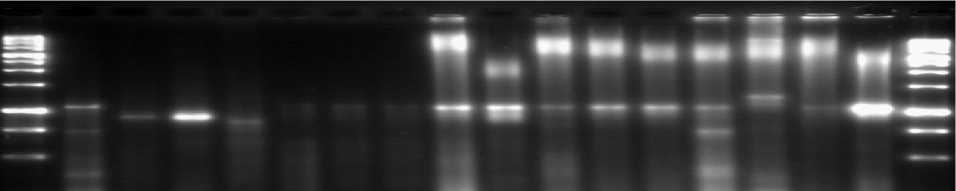
Рис. 1. Электрофорез ПЦР-продуктов, соответствующих генам альдоксимдегидратаз грамположи-тельных бактерий
Таблица 2
Распределение генов, кодирующих ферменты метаболизма нитрилов у некоторых штаммов изучаемой коллекции
|
Культура |
Гены, кодирующие ферменты, участвующие в метаболизме нитрилов |
||||
|
nhc Fe nhc Co |
nh α Fe nh β Fe |
ami |
nit |
oxd |
|
|
R. rhodochrous К17(Р) |
+ |
+/+ |
+ |
+ |
+ |
|
R. erythropolis Б2-4 |
+ |
+/+ |
+ |
+ |
|
|
R. erythropolis Б4-1 |
+ |
+/+ |
+ |
+ |
+ |
|
R. erythropolis Б4-3 |
+ |
+/+ |
+ |
||
|
R. erythropolis Б4-4 |
+ |
+ |
|||
|
R. erythropolis Б5-2 |
+ |
+/+ |
+ |
+ |
+ |
|
R. erythropolis Б5-5 |
+ |
+/+ |
+ |
+ |
|
|
R. erythropolis Б20-8 |
+ |
-/+ |
+ |
+ |
|
|
R. erythropolis ДО-1-1с |
+ |
+ |
|||
|
Pseudomonas sр. C3220 |
+ |
+ |
+ |
||
|
R. erythropolis П4-1-3 |
+ |
+ |
|||
|
R. erythropolis П11-2 |
+ |
+ |
+ |
+ |
|
|
Rhodococcus sр. П3 |
+ |
+ |
+ |
||
Заключение
На основе коллекции нитрилутилизирующих штаммов бактерий исследовано распространение генов альдоксимдегидратаз, кодирующих ферменты, участвующие в метаболизме нитрилов карбоновых кислот. Установлено, что только 14,3% штаммов нитрилутилизирующих бактерий лабораторной коллекции содержали гены альдоксимдегидратаз. Возможно, это связано с вариабельностью фланкирующих участков генов, кодирующих эти ферменты, и используемые праймеры не охватывают все возможные комбинации этих последовательностей.
При оценке распространения генов альдоксимдегидратаз среди микроорганизмов различных таксономических групп показано превалирование представителей рода Rhodococcus , продуцирующих железосодержащие нитрилгидратазы. Следует отметить, что при анализе и других генов, кодирующих гидролитические ферменты, доля этих бактерий была наибольшей.
Метод молекулярного скрининга при помощи ПЦР позволяет быстро идентифицировать альдоксимдегидратазы и может быть использован в процессе селекции альдоксим- и нитрилконвертирующих микроорганизмов.
Полученные результаты подтверждают важную роль альдоксимдегидратаз в метаболизме бактерий.
Работа выполнена при поддержке аналитической ведомственной целевой программы «Развитие научного потенциала высшей школы (2009– 2010 годы)» и программы Президиума РАН «Биологическое разнообразие»
Список литературы ПЦР-анализ генов альдоксимдегидратаз нитрилутилизирующих бактерий
- Гловер Д. Клонирование ДНК. Методы/Д. Гловер. М.: Мир, 1988. 538 с.
- Asano Y. Fungal degradation of triacrylonitrile/Y.Asano, S. Ando, Y. Tani, H. Yamada, T. Ueno//Agric. Biol. Chem. 1981. Vol. 45. P. 57-62.
- Asano Y. Z-Phenylacetaldoxime degradation by a novel aldoxime dehydratase from Bacillus sp. strain OxB-1/Y. Asanо, Y. Kato//FEMS Microbiol. Lett. 1998. Vol. 158. P. 185-190.
- Kato Y. et al. Isolation and characterization of a bacterium possessing a novel aldoxime-dehydration activity and nitrile-degrading enzymes/Y. Kato//Arch. Microbiol. 1998. Vol. 170. P. 85-90.
- Kobayashi M. Enzymatic synthesis of acrylamide: a success story not yet over …/M. Kobayashi, T. Nagasawa, H. Yamada//Trends Bioechnol. 1992. Vol. 10. P. 402-408.
- Oinuma K. et al. Novel aldoxime dehydratase involved in carbon-nitrogen triple bond synthesis of Pseudomonas chlororaphis B23. Sequencing, gene expression, purification, and characterization/K. Oinuma, Y. Hashimoto, K. Konishi, M. Goda et al.//J. Biol. Chem. 2003. Vol. 278. P. 29600-29608.
- Stone G.G. Detection of Salmonella serovars from clinical samples by enrichment broth cultivation-PCR procedure/G.G. Stone, R.D. Oberst, M.P. Hays, S. McVey, M.M. Chengappa//J. Clin. Microbiol. 1994. Vol. 32. P. 1742-1749.
- Xie S.X. A novel gene cluster responsible for alkylaldoxime metabolism coexisting with nitrile hydratase and amidase in Rhodococcus globerulus A-4/S.X. Xie, Y. Kato, H. Komeda et al.//Biochemistry. 2003. Vol. 42. P. 12056-12066.
- Yanenko A.S. Regulation of nitrile utilization in Rhodococcus/A.S. Yanenko, O.B. Astaurova, T.V. Gerasimova et al.//Biotechnologia. 1995. № 7-8. P. 139-144.


