ПЦР-анализ генов ферментов гидролиза нитрилов карбоновых кислот
Автор: Демаков В.А., Максимов А.Ю., Кузнецова М.В., Козлов С.В.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 10, 2009 года.
Бесплатный доступ
Разработаны эффективные способы селекции штаммов, способных к утилизации нитрилов карбоновых кислот. Проведена детекция генов метаболизма нитрилов (альфа- и бета- субъединиц железо- и кобальтзависимых нитрилгидратаз, а также нитрилаз) у микроорганизмов, изолированных на селективных нитрилсодержащих средах. Установлено взаимное расположение генов субъединиц нитрилгидратаз.
Короткий адрес: https://sciup.org/147204470
IDR: 147204470 | УДК: 579.26:579.222.4
Текст научной статьи ПЦР-анализ генов ферментов гидролиза нитрилов карбоновых кислот
-
a Пермский государственный университет, 614990, Пермь, ул. Букирева, 15
-
b Институт экологии и генетики микроорганизмов УрО РАН, 614081, Пермь, ул. Голева, 13
Разработаны эффективные способы селекции штаммов, способных к утилизации нитрилов карбоновых кислот. Проведена детекция генов метаболизма нитрилов (альфа- и бета-субъединиц железо- и кобальтзависимых нитрилгидратаз, а также нитрилаз) у микроорганизмов, изолированных на селективных нитрилсодержащих средах. Установлено взаимное расположение генов субъединиц нитрилгидратаз.
Успешный опыт использования для промышленного синтеза акриламида в Японии и России бактерий, способных к катаболизму нитрилов, а также перспектива биокаталитического получения других полезных соединений определяют расширение исследований бактерий-деструкторов нитрилов (Моисеева и др., 1991; Забазная, 1998; Kobayashi et al., 1992; Yanenko et al., 1995; Cowan et al., 1998; Asano, 2002). Большое значение в последнее время приобретает использование микроорганизмов для биоремедиации почв и сточных вод, загрязненных нитрилами.
Существуют различные пути метаболизма нитрилов у микроорганизмов: 1) двустадийный гидролиз, в котором участвуют два фермента – нитрилгидратаза и амидаза; 2) одностадийный гидролиз, осуществляемый нитрилазой; 3) окисление, катализируемое оксигеназой, с последующим гидролизом оксинитрилазой; 4) восстановление с участием нитрогеназы (Banerjee et al., 2002). У бактерий известны два альтернативных пути гидролиза нитрилов, дающие возможность получать химические продукты, в частности акриламид или акриловую кислоту, из акрилонитрила (Asano, 2002).
Традиционные методы селекции продуцентов промышленно значимых ферментов требуют значительных затрат времени и не позволяют проводить быстрый скрининг. В то же время молекулярные методы детекции генов являются эффективными, они ускоряют процесс отбора перспективных для биотехнологии штаммов. Поиск бактерий, обладающих соответствующими ферментны- ми системами, может быть ускорен с помощью ПЦР-анализа генов нитрилгидратаз или нитрилаз (Precigou et al., 2001).
Данная работа посвящена детекции генов нитрилаз, железо- и кобальтсодержащих нитрилгидратаз у бактерий, изолированных из почвы на территории Пермского края, способных утилизировать нитрилы карбоновых кислот.
Материалы и методы исследования
Объектами исследования являются полученные ранее в результате селекции на ацетонитриле штаммы R. erythropolis 84 и R. ruber gt1, обладающие высокой нитрилгидратазной активностью (Кузнецова и др ., 2001), а также штаммы микроорганизмов, вновь выделенные при проведении скрининговых исследований микрофлоры объектов окружающей среды (почва, вода) Пермского края. Всего в работе было проанализировано более 60 бактериальных культур с высокой активностью нитрилгидратазы и/или нитрилазы. Грамположи-тельные микроорганизмы в основном представлены родом Rhodococcus , среди грамотрицательных микроорганизмов преобладали представители рода Pseudomonas (табл.1).
Культуры выращивали на минеральной без-азотной солевой среде N, содержащей в качестве элективного фактора 10 мМ ацетонитрил. Активность нитрилгидратазы и нитрилазы оценивали так, как описано в работе А.Ю. Максимова и др. (2003).
Таблица 1
Характеристика объектов исследования
Препараты хромосомной ДНК грамположи-тельных бактерий получали фенольным методом, модифицированным для выделения ДНК из акти-номицетов (Гловер и др., 1988).
ДНК грамотрицательных микроорганизмов получали следующим образом: полную петлю биомассы бактериальной культуры инокулировали в 100 мкл стерильной Н 2 О, выдерживали при 95°С в течение 15 мин в твердотельном термостате «Термит». Пробы охлаждали на льду, центрифугировали 5 мин. при 12 тыс. об./мин. Супернатант использовали для ПЦР (Stoun et al., 1994).
Амплификацию ДНК проводили с применением термостабильной Taq -полимеразы производства ООО «СибЭнзим» (Новосибирск) на термоциклере Т3 (Biometra, Германия). Олигонуклеотидные праймеры к генам нитрилаз и субъединиц (СЕ) нитрилгидратаз (НГ) были разработаны нами с использованием базы данных GENBANK, синтезированы ООО «Евроген», Москва (табл. 1, 2).
Режим амплификации для всех праймеров включал начальный цикл денатурации – 1 мин при 94°С. Для праймеров АМ2/АМ7 и АМ3/АМ9 режим амплификации включал: 94°С (денатурация) – 60 с; 42°С (отжиг) – 60 с; 72°С (синтез) – 60 с (3 цикла); 94°С – 20 с; 56°С – 30 с; 72°С – 60 с (30 циклов) и завершающий этап – 3 мин при 72°С. Для праймеров АМ5/АМ8 и АМ4/АМ6 применялся следующий режим амплификации: 93°С – 40 с; 48°С – 60 с; 72°С – 50 с (5 циклов); 93°С – 50 с; 54°С – 70 с; 72°С – 60 с (30 циклов); 72°С – 180 с. ПЦР-анализ с праймерами FNH1/FNH2 и CNH1/ CNH2 проводился в аналогичном режиме с температурой отжига 60°С. Режим амплификации для праймеров J1, Comt и J03196 включал: начальный цикл денатурации – 60 с при 94°С; отжиг при 50°С – 60 с; элонгацию при 72°С – 80 с; (35 циклов) и завершающий этап – 60 с при 72°С. Для праймеров K22, JM3 и Acidv режим амплификации включал начальный этап денатурации – 60 с при 94°С; отжиг при 58°С – 60 с; элонгацию при 72°С – 80 с; (35 циклов) и завершающий этап – 60 с при 72°С.
Таблица 2
|
А |
|||
|
Штамм – источник гена nth |
Праймер |
Последовательность |
|
|
R. rhodochrous J1 |
α-СЕ Со-НГ |
F AM3 R АМ9 |
gtgatacatatgagcgagcacgtcaat atgcatcatacgatcacttcctg |
|
R. rhodochrous J1 |
β-СЕ Cо-НГ |
F АМ2 R АМ7 |
gtgatacatatggatggtatccacgac atgcatcacgcagagatcaggta |
|
R. rhodochrous N-774 |
α-СЕ Fe-НГ |
F АМ5 R АМ8 |
сatatgtcagtaacgatcgac atgcatcagacggtgggaacctg |
|
R. rhodochrous N-774 |
β-СЕ Fe-НГ |
F АМ4 R АМ6 |
сatatggatggagtacacgat atgcatcaggccgcaggctcgag |
|
R. rhodochrous N-771 |
Консервативная ПС Fe-НГ |
F FNH1 R FNH2 |
ctcggtctgccacccacctggtacaag cgggaggaccatgtagcgagtttcggc |
|
R. rhodochrous J1 |
Консервативная ПС Со-НГ |
F CNH1 R CNH2 |
aggtcgtggcgaagtcctgggtggaccctg cggccacggatagcacgaacacagagtgca |
Б
|
Штамм – источник гена nit |
Праймер |
Последовательность |
|
R. rhodochrous J1 (D11425) |
F J1-1 R J1-2 |
atggtcgaatacacaaacacattcaaag tcagatggaggctgtcgccc |
|
R. rhodochrous K22 (D12583) |
F К22-1 R 22-1 |
atgtccagcaatccagagctcaagtacac ctaggcctccgccttggccc |
|
Comamonas testosteroni (L32589) |
F Comt-1 R Comt-2 |
atgaaaaattatcctacagtcaaggtagc tcacgctgtggctacgcgcctc |
|
Klebsiella pneumoniae pBrx9 (J03196) |
F J03196-1 R J03196-2 |
atggacaccactttcaaagcagcc ttaggaatgtccgcaataacggg |
|
Alcaligenes faecalis JM3 (D13419) |
F AlcJM3-1 R AlcJM3-2 |
atgcagacaagaaaaatcgtccggg tcaggacggttcttgcaccagtagc |
|
Acidovorax facilis 72W (AX384691) |
F Acidv-1 R Acidv-2 |
atggtttcgtataacagcaagttcctc ctactttgctgggaccggttcttc |
Праймеры для амплификации генов нитрилгидратазы (А) и нитрилазы (Б )
Электрофоретическое разделение продуктов реакции проводили в 1,2-2,0 % агарозном геле в трис-боратном буфере при напряженности электрического поля 6 В/см. Для оценки молекулярной массы фрагментов ДНК использовали маркеры 1 kb и 100 b (ООО «СибЭнзим»).
Визуализацию полос и документирование данных осуществляли с использованием системы BioDocAnalyze (Biometra, Германия).
Результаты и их обсуждение
Детекция генов нитрилгидратаз
Гены, контролирующие гидролиз нитрилов у описанных в литературе штаммов – продуцентов нитрилгидратазы, имеют хромосомную локализацию (Kobayashi et al., 1992). Многие из изученных нитрилгидратаз содержат ионы железа: Agrobacterium tumefaciens d3, Rhodococcus sp. N-774, Pseudomonas chlororaphis B23 (Bauer et al., 1994; Endo, Watаnabe, 1989; Nagasawa et al., 1987). Кобальтсодержащие нитрилгидратазы встречаются реже, они были описаны у штаммов R. rhodochrous J1, R. rhodochrous М8, P. putida B5 и Bacillus smithii (Nagasawa et al., 1987, 1988; Ya-nenko et al., 1995; Payne et al., 1997; Takashima et al., 1998).
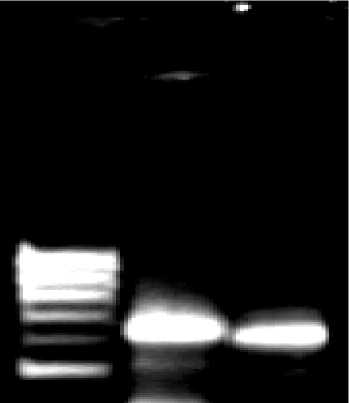
Путем амплификации на матрице хромосомной ДНК R. erythropolis 84 с праймерами, специфичными к генам α - и β - субъединиц железосодержащей нитрилгидратазы, получены фрагменты ДНК (рис. 1), соответствующие их полному размеру (Nagasawa et al., 1987).
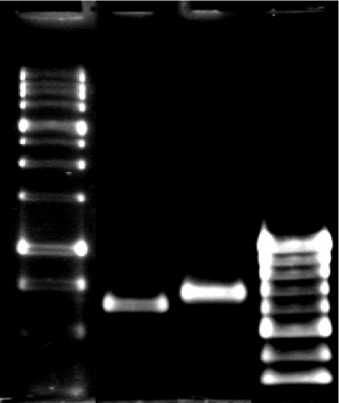
На матрице ДНК R. ruber gt1 с праймерами к генам α - и β - субъединиц кобальтсодержащего фермента амплифицированы фрагменты (рис. 2), соответствующие по размеру (680 пн - α -СЕ и 720 пн - β -СЕ) генам высокомолекулярной нитрилгид-ратазы штамма R. rhodochrous J1 (Nagasawa et al., 1988).
Идентичность ПЦР-фрагментов предполагаемым генам подтверждена методом секвенирования на установке MegaBACE1000.
Маркер 1300 пн
1000 п.н.-
100 b β-СЕ α-СЕ маркер
Рис. 1. Электрофорез ПЦР-продуктов ДНК R. erythropolis 84, соответствующих генам α- и β-субъединиц Fe-зависимой нитрилгидратазы
1000 п.н.-
1 kb α-СЕ β-СЕ 100 b маркер маркер
Рис. 2. Электрофорез ПЦР-продуктов ДНК R. ruber gt1, соответствующих генам α- и β-субъединиц Co-зависимой нитрилгидратазы
gt 1
А0
α-СЕ β-СЕ β+α α-СЕ β-СЕ
α+β α-СЕ β-СЕ β+α
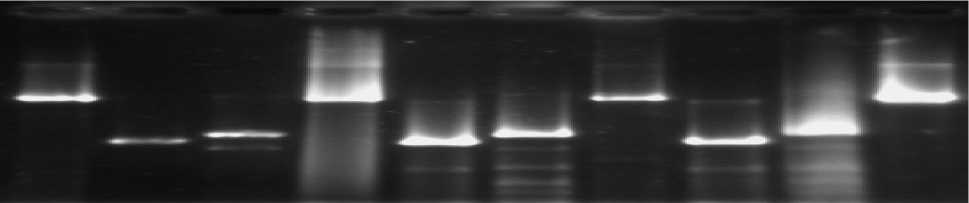
Рис. 3. Электрофорез ПЦР-продуктов ДНК R. ruber gt1, R. erythropolis 84 и А0 с комбинированными праймерами
Известно, что структурные гены α - и β -субъединиц нитрилгидратаз у всех ранее исследованных штаммов находятся в составе одного оперона, однако порядок их расположения различается. В частности, в каждом из генов низкомолекулярной и высокомолекулярной кобальтсодержащей нитрилгидратазы R. rhodochrous J1, М8 кодирующая последовательность β -субъединицы локализована выше гена α -субъединицы, в отличие от генов железозависимых нитрилгидратаз R. rhodochrous N-774 , P. chlororaphis В23 (Kobayashi et al., 1992; Вейко и др., 1995, Asano, 2002).
Комбинирование праймеров, комплементарных 5’ и 3’- концам близкорасположенных генов, позволяет определить в ПЦР-реакции их взаимное расположение и ориентацию. Таким путем исследовано взаимное расположение генов α- и β- субъединиц нитрилгидратаз штаммов R. ruber gt1 и R. erythropolis 84, А0 (рис. 3). При амплификации с матрицы ДНК R. ruber gt1 и R. erythropolis А0 ПЦР-продукт получен только с праймерами к 5’-концу гена β-субъединицы и к 3’-концу гена α-субъединицы нитрилгидратазы. Следовательно, за геном β-субъединицы однонаправленно расположен ген α-субъединицы, аналогично ориентации генов известных кобальтзависимых нитрилгидра-таз. У штамма R. erythropolis 84 гены субъединиц фермента были расположены в обратном порядке.
В ДНК почвенных культур, выделенных на изобутиронитриле, обнаружены фрагменты, соответствующие консервативному участку гена α-субъединицы Fe-зависимой нитрилгидратазы, а также полные последовательности генов α- и
β -субъединиц Fe-зависимой нитрилгидратазы
(рис. 4).
1,5 т.п.н.-
1 т.п.н. -
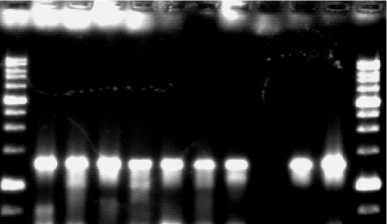
Рис. 4. Амплификация генов (αСЕ + βСЕ) Fe-зависимой нитрилгидратазы
В ДНК штаммов К12(ОМ) и К12ЖР с праймерами, специфичными к генам β -субъединицы кобальтсодержащей нитрилгидратазы детектированы фрагменты, соответствующие по размеру генам данных субъединиц известных штаммов.
Детекция генов нитрилаз
В результате амплификации образцов ДНК с праймерами к концевым участкам гена нитрилазы штамма R. rhodochrous J1 (D11425) в геномной ДНК 14 штаммов обнаружены нуклеотидные последовательности (1100 п.н.), соответствующие данному гену (рис. 5).
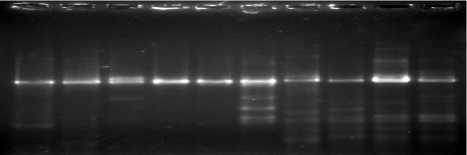
Рис. 5. ПЦР-фрагменты, соответствующие по размеру гену нитрилазы R. rhodochrous J1
При амплификации с праймерами, комплементарными концевым участкам нуклеотидной последовательности гена нитрилазы штамма Acidovorax facilis 72W (AX384691) в геномной ДНК восьми штаммов, включая высокоактивный штамм-продуцент нитрилазы Pseudomonas fluorescens С2, получены соответствующие этому гену ПЦР-фрагменты, имеющие размер около 1100 пар нуклеотидов. Также установлено, что четыре штамма бактерий содержат фрагменты ДНК, гомологичные гену нитрилазы штамма Rhodococ-cus rhodochrous K22. Фрагменты, соответствующие последовательности гена нитрилазы, ампли- фицированные с использованием праймеров к концевым участкам соответствующего гена, составляли около 1150 пар нуклеотидов.
Ни в одном из образцов геномной ДНК выделенных почвенных изолятов не обнаружено нуклеотидных последовательностей, гомологичных генам нитрилаз описанных ранее штаммов Com-amonas testosteroni sp. (L32589) и Klebsiella pneumoniae pBrx9 (J03196 ).
Заключение
На основе коллекции штаммов нитрилутилизирующих бактерий исследовано распространение генов, кодирующих ферменты гидролиза нитрилов карбоновых кислот. Показано, что подобные гены широко распространены среди микроорганизмов различных таксономических групп. Установлено, что среди штаммов, обладающих нитрилгидра-тазной активностью, преобладают представители рода Rhodococcus , гены нитрилазы чаще обнаруживались у грамотрицательных бактерий. Следует отметить, что гены кобальтзависимых нитрилгид-ратаз обнаружены только у четырех штаммов бактерий, ДНК большинства штаммов содержит гены железозависимых ферментов.
Известно, что экспрессия ферментов зависит от ряда специфических факторов, а традиционные методы скрининга не позволяют быстро дифференцировать нитрилазную активность от совместного действия нитрилгидратазы и амидазы. Разработанные протоколы ПЦР-анализа показали высокую эффективность при селекции штаммов, обладающих высокой активностью нитрилгидратазы и нитрилазы. Показано, что полимеразная цепная реакция дает возможность простой, быстрой, специфичной идентификации их генов и проведения скрининга микроорганизмов, способных метаболизировать нитрилы в соответствующие амиды или карбоновые кислоты.
Работа выполнена при поддержке программы Президиума РАН «Биологическое разнообразие» и гранта РФФИ-Урал-а 07-04-96071.
Список литературы ПЦР-анализ генов ферментов гидролиза нитрилов карбоновых кислот
- Вейко В.П. Клонирование и определение нуклеотидной последовательности гена Rhodococcus rhodochrous М8/В.П. Вейко, А.С. Яненко, М.Г. Алексеева и др.//Биотехнология. 1995. № 5-6. С. 3-5.
- Забная Е.В. Отбор штаммов, трансформирующих акрилонитрил и акриламид в акриловую кислоту/Е.В. Забная, С.В. Козулин, С.П. Воронин//Прикладная биохимия и микробиология. 1998. Т. 34, № 4. С. 377-384.
- Клонирование ДНК. Методы/пер. с англ.; под ред. Д. Гловера. М.: Мир, 1988. 538 с.
- Кузнецова М.В. Утилизация нитрилов штаммами-продуцентами нитрилгидратазы/М.В. Кузнецова, А.Ю. Максимов, Г.В. Овечкина и др.//Проблемы загрязнения окружающей среды-2001: материалы V междунар. конф. Волгоград-Пермь, 2001. С. 30.
- Максимов А.Ю. Влияние нитрилов и амидов на рост и нитрилгидратазную активность штамма Rhodococcus sp. gt1/А.Ю. Максимов, М.В. Кузнецова, Г.В. Овечкина и др.//Прикладная биохимия и микробиология. 2003. Т. 39, № 1. С. 63-68.
- Моисеева Т.Н. Скрининг штаммов-деструкторов акриловой кислоты и ее производных/Т.Н. Моисеева, С.В. Козулин, Л.К. Куликова, С.П. Воронин//Биотехнология. 1991. № 6. С. 79-83.
- Asano Y. Overview of screening for new microbial catalysts and their uses in organic synthesis -selection and optimization of biocatalysts/Y. Asano//J. Biotechnol. 2002. Vol. 94. P. 65-72.
- Banerjee A. The nitrile-degrading enzymes: current status and future prospects/A. Banerjee, R. Sharma, U.C. Banerjee//Appl. Microbiol. Biotechnol. 2002. Vol. 60. P. 33-44.
- Bauer R. Enantioselective hydrolysis of racemic 2-phenylpropionitrile and other (R,S)-2-arylpropionitriles by new bacterial isolate, Agrobacterium tumefaciens strain d3/R. Bauer, B. Hirrlinger, N. Layh et al.//Appl. Microbiol. Biotechnol. 1994. Vol. 42. Р. 1-7.
- Cowan D. Biochemistry and biotechnology of mesophilic and thermophilic nitrile metabolizing enzymes/D. Cowan, R. Cramp, R. Pereira et al.//Extremophiles. 1998. Vol. 2, № 3. P. 207-216.
- Endo I. Nitrile hydratase of Rhodococcus sp. N-774-purification and amino acid sequences/I. Endo, I. Watanabe//FEBS Lett. 1989. Vol. 243. P. 6461-6497.
- Kobayashi M. Enzymatic synthesis of acrylamide: a success story not yet over/M. Kobayashi, T. Nagasawa, H. Yamada//Trends Bioechnol. 1992. Vol. 10. P. 402-408.
- Nagasawa T. Nitrile hydratase of Pseudomonas chlororaphis B23/T. Nagasawa, H. Nanba, K. Ryuno et al.//Eur. J. Biochem. 1987. Vol. 162. P. 691-698.
- Nagasawa T. Occurrence of a cobalt-induced and cobalt containing nitrile hydratase in Rhodococcus rhodochrous J1/T. Nagasawa, K. Takeuchi, H. Yamada//Biochem. Biophys. Res. Commun. 1988. Vol. 155. P. 1008-1016.
- Payne M.S. A stereoselective cobalt-containing nitrile hydratase/M.S. Payne, S.J. Wu, R.D. Fallon et al.//Biochemistry. 1997. Vol. 36. Р. 5447-5454.
- Precigou S. Rapid and specific identification of nitrile hydratase (NHase)-encoding genes in soil samples by polymerase chain reaction/S. Precigou, P. Goulas, R. Duran et al.//FEMS Microbiology Letters. 2001. Vol. 204. Р. 155-161.
- Stone G.G. Detection of Salmonella serovars from clinical samples by enrichment broth cultivation-PCR procedure/G.G. Stone, R.D. Oberst, M.P. Hays//J. Clin Microbiol. 1994. Vol. 32, № 7 Р. 1742-1749.
- Takashima Y. Nitrile hydratase from a thermophilic Bacillus smithii/Y. Takashima, Y. Yamaga, S. Mitsuda//J. Indust. Microbiol. Biotechnol. 1998. Vol. 20. P. 220-226.
- Yanenko A.S. Regulation of nitrile utilization in Rhodococcus/A.S. Yanenko, O.B. Astaurova, T.V. Gerasimova et al.//Biotechnologia. 1995. № 7-8. P. 139-144.


