Поиск и анализ нуклеотидных последовательностей в геноме подсолнечника, ассоциированных с генами устойчивости к расам заразихи Orobanche cumana Wallr
Автор: Савиченко Д.Л., Гучетль С.З., Головатская А.В.
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 3 (183), 2020 года.
Бесплатный доступ
Исследование линий, устойчивых к расе G заразихи, созданных на центральной экспериментальной базе ВНИИМК, показало генетический контроль устойчивости как моногенный с неполным доминированием. Целью работы являлся поиск и анализ молекулярных маркеров для генов устойчивости к расе G заразихи на основе применения методов ПЦР. Материалом исследования служили созданные во ВНИИМК шесть линий-доноров, устойчивых к расе G заразихи: RGP1, RGP2, RGB, RGL1, RGL2, RGA, а также восприимчивые линии ВК 101, ВК 678, ВК 680. Выделение ДНК проводилось из листьев молодых растений с использованием CTAB-буфера. Для ПЦР анализа использовали 17 пар праймеров двух типов: SCAR (sequence characterized amplified region) и SSR (simple sequence repeat). Условия амплификации рекомендованные авторами маркеров с экспериментальным подбором температуры гибридизации праймеров. В рамках исследования используемые в работе SSR и SCAR локусы с помощью инструмента BLAST были сопоставлены со сборкой HanXRQr1.0. По этим данным состав лена физическая карта положения локусов. При оценке шести устойчивых и трех восприимчивых родительских линий полиморфизм по длине локуса ДНК показали семь маркеров, а по наличию/отсутствию амплифицированного фрагмента -два маркера. У маркеров ORS 683 и ORS 1112 была отмечена связь полиморфизма локусов с восприимчивостью растений родительских линий к заразихе. У ORS 683 это выражалось наличием аллеля длиной 364 п.н., а у ORS 1112 - отсутствием аллеля длиной 375 п.н. Данные маркеры отмечены как основные для гибридологического анализа в F1 и F2 с выбранными парами для скрещиваний. Дополнительные маркеры будут подобраны индивидуально на основании полученных данных по полиморфизму локусов. Таким образом, в результате исследования была составлена физическая карта для дальнейшего подбора маркеров, выбраны гибридные комбинации и маркеры из числа изученных для гибридологического анализа.
Подсолнечник, заразиха, устойчивость, днк, маркеры, пцр
Короткий адрес: https://sciup.org/142224955
IDR: 142224955 | УДК: 633.854.78:575:632.9 | DOI: 10.25230/2412-608X-2020-3-183-11-19
Текст научной статьи Поиск и анализ нуклеотидных последовательностей в геноме подсолнечника, ассоциированных с генами устойчивости к расам заразихи Orobanche cumana Wallr
Введение. Подсолнечник ( Helianthus annuus L.) является одной из важнейших масличных культур в мире. Посевы подсолнечника подвержены влиянию многих биотических лимитирующих факторов, среди которых одним из самых вредоносных является паразитический сорняк – заразиха ( Orobanche cumana Wallr.).
Заразиха кумская встречается во многих странах Европы и Азии, особенно в Центральной и Восточной Европе, Испании, Турции, Китае, Израиле, Иране и Казахстане [1; 2]. В среднем потери урожая подсолнечника, вызванные заразихой, могут превышать 50 % при выращивании восприимчивых гибридов [3; 4; 5]. В настоящее время обнаружено восемь рас, обозначенных буквами от А до Н [6; 7; 8]. Сегрегация O. cumana по вирулентности впервые была описана в СССР как разница между способностью заразихи поражать разные генотипы подсолнечника. Расы были описаны как A и комплекс рас B [9]. Генетическая резистентность устойчивого к этим расам материала в конечном итоге стала неэффективной, так как появилась новая раса С, сильно повлиявшая на производство подсолнечника [10]. В южных регионах Российской Федерации сообщается о расах F, G и H заразихи [11]. В Казахстане были выявлены 12
расы С и G [9]. Присутствие расы H наблюдалось на северо-востоке Украины, наряду с биотипом с вирулентностью выше, чем раса H [12]. Существует значительная разница в распределении вирулентности, так как наиболее вирулентные расы обычно встречаются в районах вблизи Черного моря [13]. Раса E является доминирующей в области, зараженной O. cumana в Сербии, а раса F была обнаружена локально [14; 15].
Использование устойчивых сортов и гибридов подсолнечника считается наиболее экологичным подходом к борьбе с паразитом. Селекция генотипов, устойчивых к заразихе включает использование доступных и эффективных источников резистентности, перенос генов устойчивости в существующий селекционный материал и практические процедуры оценки для обеспечения достаточного качества отбора. Данный процесс должен вестись непрерывно, так как появление устойчивости к новой расе приводит к ее преодолению и появлению более агрессивной расы. У подсолнечника в отношении O. cumana описаны как качественно, так и количественно наследуемые гены резистентности. Качественная устойчивость к заразихе контролируется главными генами и зависит от расы [7]. Учитывая, что гены устойчивости быстро преодолеваются, необходимо искать новые источники устойчивости и комбинировать имеющиеся гены [16].
Гены устойчивости к заразихе обозначаются как Or. А. Вранчану и соавторы [17] идентифицировали пять единичных доминантных генов (Or1 – Or5) для устойчивости к пяти расам (A – E) заразихи. Они определили набор из пяти линий-дифференциаторов, устойчивых к пяти последовательным расам. Появление расы F привело к идентификации устойчивых генотипов, контролируемых одним доминантным геном Or6 [18; 19], двумя рецессивными генами or6or7 [20; 21] и двумя частично доминантными генами Or6Or7 [22]. Таким образом, разница в способе наследования обусловлена различным происхождением генетического материала. Ранее в селекционных программах подсолнечника использовались доминантные гены устойчивости к заразихе, вплоть до обнаружения рецессивной устойчивости. Рецессивные гены могут повлиять на более длительную устойчивость к соответствующему паразиту, но приводят к необходимости включения генов устойчивости в обе родительские линии для развития резистентного гибрида [20]. Большинство моделей, которые пытаются объяснить устойчивость, предполагает, что доминантная устойчивость является активным процессом, когда растение синтезирует соединения, мешающие паразиту. И наоборот, рецессивная резистентность может быть результатом того, что в растительных клетках отсутствуют некоторые факторы, необходимые для жизненного цикла патогена [16].
Точное определение локализации гена устойчивости необходимо для выявления маркеров, которые могут быть использованы в маркер-ассоциированной селекции (MAС). Ген Or5, контролирующий устойчивость к расе Е заразихи, был картирован в теломерной области хромосомы 3 [23; 24; 25]. Кроме того, обнаружены пять QTL для устойчивости к расе E и шесть QTL для устойчивости к расе F в семи разных хромосомах [19]. Фенотипическая дисперсия устойчивости к расе E главным образом объяснялась основным QTL or3.1 на хромосоме 3, связанным с признаком устойчивости или восприимчивости. Ближайшим маркером к гену устойчивости к расе выше F, предварительно обозначенным как orab-vl-8, был ORS683 с генетическим расстоянием 1,5 сМ на хромосоме 3 [2]. Хотя этот ген был картирован на той же хромосоме, что и Or5, авторы доказали, что два гена устойчивости различны. В то время как ген Or5 находится в верхнем конце хромосомы 3 с ближайшим микросателлитным маркером ORS1036, находящимся на 7,5 сМ ниже по карте, orab-vl-8 картирован в нижней области хромосомы 3. Позже, I. Imerovski et al. [26] картировали два основных QTL на хромосоме 3. Обозначили QTL or3.1 и QTL or3.2. QTL or3.1 был расположен в геномной области, где картирован предыдущий ген устойчивости к заразихе Or5, в то время как QTL or3.2 был идентифицирован в нижней области той же хромосомы. Авторы провели четыре различных скрещивания с четырьмя различными источниками устойчивости к заразихе и смогли идентифицировать основной QTL. Обнаружено, что во всех скрещиваниях QTL устойчивости находились в 3-й хромосоме. Были картированы от 2 до 23 значимых QTL в геноме подсолнечника. Ген устойчивости к расе F O. cumana был картирован на хромосоме 7 [27].
Для развития селекционной программы по подсолнечнику необходим поиск генов устойчивости к новым расам и подбор ДНК-маркеров к ним для развития маркер-ассоциированной селекции. Данные маркеры ускорят и упростят отбор по интересующему признаку. В связи с этим цель работы – поиск молекулярных маркеров для генов, контролирующих устойчивость к расе G заразихи в материале подсолнечника отечественной селекции на основе применения методов ПЦР с известными SSR и SCAR праймерами.
Для достижения цели необходимо решить следующие задачи:
-
- поиск молекулярных маркеров, сцепленных с геном устойчивости Or 7 к расе G заразихи О. cumana в линиях подсолнечника селекции ВНИИМК;
-
- оценка коллекции линий подсолнечника ВНИИМК, устойчивых и восприимчивых к расе G заразихи по международной классификации, методом полимеразной цепной реакции амплификации ДНК по SSR- и SCAR-маркерам, сцепленным с разной частотой рекомбинации с Or 7 ;
-
- определение пар гибридизации для получения потомства F 1 и F 2 для дальнейшего гибридологического анализа.
Материалы и методы. Материалом исследования служили шесть линий-доноров, устойчивых к расе G заразихи:
RGP1, RGP2, RGB, RGL1, RGL2, RGA, которые были созданы на центральной экспериментальной базе ВНИИМК с использованием источников разного происхождения, резистентных к расе G и всем предыдущим [28], а также линии ВК 101, ВК 678, ВК 680, селекции ВНИИМК восприимчивые к расе G заразихи.
Выделение и очистка ДНК.
ДНК подсолнечника выделили из верхушечных листьев молодых побегов вегетирующих растений. Отборы проводились из пяти индивидуальных растений каждого образца. Выделение ДНК производилось по методу M. Saghai-Maroof et аl. [29]. Для получения качественных образцов на одном из этапов использовали активированный уголь [30]. Навеску 0,2 г растительной ткани гомогенизировали с использованием гомогенизатора Speed Mill plus (Analytic Jena, Германия). Концентрацию ДНК в полученном препарате определяли по интенсивности свечения пробы объемом 10 мкл в ультрафиолетовом свете после разгонки в 1%-ном агарозном геле, содержащем бромистый этидий.
Проведение реакции.
Для проведения полимеразной цепной реакции использовали 25 мкл реакционной смеси следующего состава: 67 мМ трис-HCl, рН 8,8; 16,6 мM сульфата аммония; 1,5–3,0 мM MgCl2; 0,01 %
Tween 20; по 0,2 мM дезоксирибонуклеозидфосфатов; по 10 пМ праймеров; 10 нг матричной ДНК и 1 ед. рекомбинантной термостабильной ДНК полимеразы (Сибэнзим, Россия). Для амплификации использовали термоциклер S1000тм (BioRad, США). Условия амплификации: начальная денатурация – 2 мин 96 °С, затем 30 циклов при соблюдении температурно-временного режима: денатурация при 94 °С – 30 сек, отжиг при 60 °С в течение 40 сек, элонгация – 1 мин при 70 °С, финальная элонгация – 2 мин.
Для ПЦР анализа использовали два типа праймеров:
-
- SCAR (sequence characterized amplified region) [24];
-
- SSR (simple sequence repeat) [25].
0.0
П
ORS1036
8.3
8.6
10.6
В общей сложности испытанию подверглись 17 пар праймеров. Нуклеотидные последовательности праймеров, фланкирующих ДНК-локусы, синтезированы фирмой «Синтол» (Россия).
Визуализация продуктов ПЦР.
Электрофорез продуктов амплификации проводили в агарозном геле (2 % агароза, 1хSB-буфер) с использованием камеры для горизонтального электрофореза (SE.2, Хеликон, Россия) в течение 1– 1,5 ч при силе тока 50–58 mA и напряжении 80–100, а также в полиакриламидном геле (8 %, 1хТБЕ) с использованием камеры VE-20 для вертикального электрофореза (Хеликон, Россия) в течение 2,5–3 ч при силе тока 40–50 mA и напряжении 200–230. Реакционную смесь (10–12 мкл) наносили на гель вместе с красителем бромфеноловым синим. Последующее окрашивание осуществляли бромистым этидием. Результаты электрофореза документировали при помощи гель-документи-рующей видеосистемы BIO-PRINT (Vilber Lourmat, Франция). Размер фрагментов ДНК определяли с использованием программного обеспечения Bio-Capture (Vilber Lourmat, Франция) относительно маркера длины фрагментов ДНК GeneRuler 100 bp DNA Ladder Thermo Scientific (Сибэнзим, Россия).
Результаты и обсуждение. Проведенный ранее анализ наследования признака устойчивости к расе G заразихи показал, что у линий устойчивость контролируется одним геном с неполным доминированием [31]. Для локализации гена используется анализ сцепления с молекулярным маркером. Для этой цели зарубежными исследователями были составлены молекулярно-генетические карты групп сцепления генома подсолнечника [25; 32]. На их основании проводился подбор генетических маркеров к гену Or 7 [32] (рис. 1).
ORS1112 ORS202 ■ORS665
15.1---ORS820
18.1---ORS683
24.3
ORS657
28.1
ORS1080
33.9---ORS1222
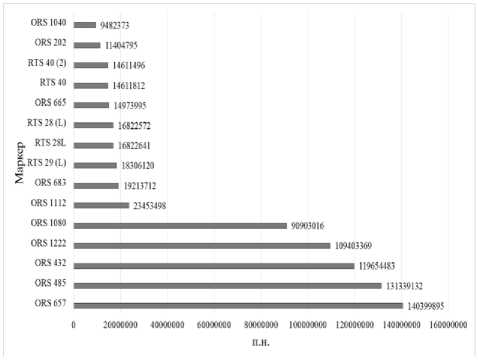
Рисунок 2 – Положение SSR и SCAR локусов, предположительно сцепленных с геном Or 7 в LG3 H. annuus
40.7---ORS485
43.5—U—ORS924
Рисунок 1 – Молекулярно-генетическая карта LG3 подсолнечника.
Микросателлитные маркерные локусы, амплифицированные полимеразной цепной реакцией и локализованные рядом с геном ora b -v l - 8
Картирование, основанное на анализе сцепления, показывает лишь относительные расстояния между исследуемыми локусами, однако на данный момент проведена сборка репрезентативного генома H. annuus – HanXRQr1.0, включающего в себя физическую карту группы сцепления на основании данных полногеномного секвенирования методом дробовика [33; 34]. С помощью инструмента Basic Local Alignment Search Tool (BLAST®) можно осуществлять поиск и сопоставление нуклеотидных последовательностей со сборкой репрезентативного генома, оценивать генетические расстояния изучаемых локусов в п.н., а в дальнейшем осуществлять поиск генов-кандидатов в искомой области [35] .
В рамках исследования используемые в работе SSR и SCAR локусы были сопоставлены со сборкой HanXRQr1.0. По этим данным составлена физическая карта положения локусов (рис. 2).
Локусы гибридизации для праймеров ORS 1036, RTS 05, ORS 1021 и ORS 777 не обнаружены в LG3, что может говорить об их положении в других группах или об отсутствии части последовательности в сборке репрезентативного генома HanXRQr1.0. Положения локусов на физической карте распределились на две условные группы, которые совпадали с составленной ранее молекулярно-генетической картой [32]. В верхней части LG3 располагается группа локусов, находящихся выше предполагаемого нахождения гена orab-vl-8 на молекулярно-генетической карте, а в нижней части
–
группа, помещенная ниже
данного гена. Также локусы верхней группы располагаются плотнее по отношению друг к другу, в отличие от локусов нижней группы, что тоже отмечено на молекулярно-генетической карте (рис. 1).
Таким образом, физическая карта локусов гибридизации может быть эффективным инструментом для более точного анализа и подбора маркеров. При появлении новых версий сборки генома карта будет актуализироваться.
Изучаемые линии подсолнечника были проанализированы на наличие полиморфизма по длине и наличие амплифи-цированных фрагментов ДНК при ПЦР с 15
используемыми праймерами (таблица).
Таблица
Амплифицированные фрагменты ДНК, полученные при проведении ПЦР с SSR-и SCAR-праймерами на ДНК родительских линий подсолнечника
|
Маркер |
Генотип |
||||||||
|
RGL1 |
RGL2 |
RGB |
RGА |
RGP1 |
RGP2 |
ВК678 |
ВК680 |
ВК101 |
|
|
ORS 665 |
294 |
294 |
294 |
294 |
294 |
294 |
294 |
294 |
294 |
|
ORS 485 |
324 |
324 |
324 |
324 |
324 |
324 |
324 |
324 |
324 |
|
ORS 683 |
400 |
400 |
400 |
400 |
400 |
400 |
364 |
364 |
364 |
|
ORS 1080 |
369 |
369 |
369 |
379 |
379 |
379 |
379 |
375 |
379 |
|
ORS 657 |
274 |
274 |
274 |
263 |
263 |
274 |
263 |
263 |
263 |
|
ORS 1222 |
490 |
490 |
490 |
490 |
490 |
490 |
490 |
490 |
490 |
|
ORS 1112 |
347 /375 |
347 /375 |
375 |
347 /375 |
375 |
347 /375 |
347 |
- |
347 |
|
ORS 202 |
309 |
309 |
309 |
333 |
333 |
333 |
309 |
309 |
309 |
|
ORS 1040 |
200 |
200 |
- |
200 |
200 |
200 |
192 |
200 |
200 |
|
ORS 777 |
315 |
315 |
315 |
315 |
315 |
315 |
315 |
315 |
315 |
|
ORS 1021 |
270 |
270 |
270 |
270 |
270 |
270 |
270 |
270 |
270 |
|
RTS 05 |
- |
- |
- |
+* |
+ |
+ |
+ |
- |
+ |
|
RTS 40 |
- |
+ |
- |
- |
- |
- |
- |
+ |
+ |
|
RTS 40 (2) |
- |
+ |
- |
- |
- |
- |
- |
+ |
+ |
|
RTS 28 (L) |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
ORS 432 |
163 |
163 |
163 |
163 |
163 |
163 |
163 |
163 |
163 |
|
RTS 29 (L) |
1 |
1 |
1 |
2 |
2 |
2 |
2 |
2 |
2 |
* +/– присутствие/отсутствие амплифициро-ванного фрагмента
При оценке шести устойчивых и трех восприимчивых родительских линий полиморфизм по длине локуса показали маркеры: ORS 683, ORS 1112, ORS 1080, ORS 657, ORS 202, ORS 1040, RTS 29 (L). По принципу наличия/отсутствия ампли-фицированного фрагмента полиморфизм наблюдался по локусам RTS 05 и RTS 40. Оставшиеся маркеры показали себя мономорфными для всех изучаемых линий, и дальнейшее их применение в исследовании нецелесообразно. Для гибридологического анализа необходим подбор наибольшего количества полиморфных маркеров и контрастных линий по искомому признаку для гибридизации и оценки в F 1 и F 2 . Для этих целей выделили маркеры ORS 683 и ORS 1112, у которых была замечена связь полиморфизма маркера с восприимчивостью растений родительских линий к заразихе. Восприимчивые линии характеризовались аллелем ORS 683 длиной 364 п.н. (таблица) и полным отсутствием аллеля длиной 375 п.н. у локуса ORS 1112 (рис. 3). Данные 16
маркеры находятся в относительной близости к гену orab-vl-8 на молекулярногенетической карте ORS 683 – 1,5 сМ, ORS 1112 – 11,3 сМ (рис. 1), а на физической карте ORS 683 является самым близкорасположенным локусом к ORS 1112 с расстоянием между ними порядка 4,25 млн п.н. (рис. 2).
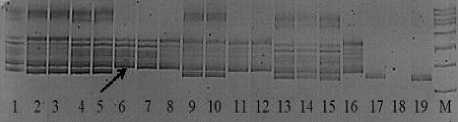
Рисунок 3 – Электрофоретические спектры локуса ORS 1112
Дорожки: 1, 4, 5 – RGL1; 2,3 – RGL2;
6–8 – RGВ; 9, 10 – RGА;
11, 12, 16 – RGP1; 13–15 – RGP2; 17 – ВК 678; 18 – ВК 680; 19 – ВК 101; М – маркер молекулярной массы 100 bp. Стрелкой обозначен фрагмент ДНК 375 п.н.
На основании вышеописанного, принято решение использовать маркеры ORS 683 и ORS 1112 как основные. Для гибридологического анализа выбраны пары для скрещиваний: RGP2 × ВК 680, ВК 678 × RGP2, ВК 680 × RGB, RGL1 × ВК 678, ВК 678 × RGP1, ВК 680 × RGL1 (RGL1 × ВК 680). Для гибридологического анализа будут использованы основные маркеры (для всех гибридных комбинаций), а также маркеры, показавшие полиморфизм между двумя родительскими формами.
Выводы. В результате проведенного исследования были определены маркеры для дальнейшей работы и выбраны пары для гибридизации и оценки в F 1 и F 2 . С помощью BLAST будет актуализироваться карта локусов и будут подобраны и созданы новые маркеры, расположенные в области поиска.
Список литературы Поиск и анализ нуклеотидных последовательностей в геноме подсолнечника, ассоциированных с генами устойчивости к расам заразихи Orobanche cumana Wallr
- Skoric D., Pacureanu M. Sunflower breeding for resistance to broomrape (Orobanche cumana Wallr.) // In: Proceedings of the International Symposium Sunflower Breeding on Resistance to Diseases, Krasnodar, Russia, 23-24 June, 2010. - P. 19-30.
- Molinero-Ruiz L., Delavault P., Pérez-Vich B., Pacureanu-Joita M., Bulos M., Altieri E., Dommguez J. History of the race structure of Orobanche cumana and the breeding of sunflower for resistance to this parasitic weed: A review // Span. J. Agric. Res. -2015. - V. 13. - 4 e10R01. 10.5424/sjar/ 2015134-8080. DOI: 10.5424/sjar/2015134-8080
- Dommguez J. R-41, a sunflower restorer inbred line, carrying two genes for resistance against a highly virulent Spanish population of Orobanche cernua // Plant Breed. - 1996. - V. 115. - P. 203-204.
- Jestin C., Lecomte V., Duroueix F. Current situation of sunflower broomrape in France // In: Proceedings of the 3rd International Symposium on Broomrape in Sunflower, Cordoba, Spain, 3-6 June, 2014. - P. 3-6.
- Perez-Vich B., Velasco L., Rich P.J., Ejeta G. Marker-assisted and physiology-based breeding for resistance to root parasitic Orobanchaceae // In: Parasitic Orobanchaceaem - Springer: Berlin/Heidelberg, Germany, 2013. - P. 369-391.
- Martm-Sanz A., Malek J., Fernândez-Martmez J.M., Pérez-Vich B., Velasco L. Increased virulence in sunflower broomrape (Orobanche cumana Wallr.) populations from Southern Spain is associated with greater genetic diversity // Front. Plant Sci. - 2016. -V. 7. - P. 589.
- Fernândez-Martmez J.M., Pérez-Vich B., Velasco L. Sunflower broomrape (Orobanche cumana Wallr.) // In: Sunflower. - AOCS Press: Urbana, IL, USA, 2015. - P. 129-155.
- Kaya Y. Current situation of sunflower broomrape around the world // In Proceedings of the 3rd International Symposium on Broomrape (Orobanche spp.) in Sunflower, Cordoba, Spain, 3-6 June, 2014. -P. 9-18.
- Antonova T.S. The history of interconnected evolution of Orobanche cumana Wallr. and sunflower in the Russian Federation and Kazakhstan // Helia. - 2014. - V. 37. - P. 215-225.
- Duca M., Port A., Boicu A. Molecular Characterisation of Broomrape Populations from Republic of Moldova using SSR Markers // Helia. - 2017. - V. 40. - P. 47-60.
- Antonova T.S., Araslanova N.M., Strelnikov E.A., Ramazanova S.A., Tchelustnikova T.A., Guchetl S.Z. Screening of wild Helianthus species for resistance to high virulent Orobanche cumana Wallr., affecting sunflower in the Rostov region of the Russian Federation // Helia. - 2011. - V. 34. - P. 115-123.
- Maklik E., Kyrychenko V.V., Pacureanu M.J. Race composition and phenology of sunflower broomrape (Orobanche cumana Wallr.) in Ukraine // In: Proceedings of the 4th International Symposium on Broomrape in Sunflower, Bucharest, Romania, 24 July, 2018. - P. 67-78.
- Pacureanu J.M., Raranciuc S., Sava E., Stanciu D., Nastase D. Virulence and aggressiveness of sunflower (Orobanche cumana Wallr.) populations in Romania // Helia. - 2014. - V. 32. - P. 111-118.
- Miladinovic D., Dedic B., Quiroz F., Alvarez D., Poverene M., Cantamutto M. Orobanche cumanaWallr. Resistance of commercial sunflower cultivars grown in Argentina // J. Basic Appl. Genet. - 2012. - V. 23. - P. 37-41.
- Dedic B., Miladinovic D., Jocic S., Cvejic S., Jockovic M., Miklic V. Increase in virulence of sunflower broomrape in Serbia // In: Proceedings of the 4th International Symposium on Broomrape in Sunflower, Bucharest, Romania, 2-4 July, 2018. - P. 27 35.
- Imerovski I., Dimitrijevic A., Miladinovic D., Dediv B., Jocic S., Köcis Tubic N., Cvejic S. Mapping of a new gene for resistance to broomrape races higher than F //Euphytica. - 2016. - Vol. 209. - Р. 281-289.
- Vranceanu A.V., Tudor V.A., Stoenescu F.M., Pirvu N. Virulence groups of Orobanche cumana Wallr. differential hosts and resistance sources and genes in sunflower // In: Proceedings of the 9th International Sunflower Conference, Torremolinos, Spain, 8-13 June, 1980. - P. 74-80.
- Pacureanu-Joita M., Vranceanu A.V., Soare G., Marinescu A., Sandu I. The evaluation of the parasite-host interaction system (Helianthus annuus L.) (Orobanche cumana Wallr.) in Romania // In: Proce-dings of the 2nd Balkan Symposium on Field Crops, Novi Sad, Yugoslavia, 16-20 June, 1998. - P. 1-20.
- Pérez-Vich B., Akhtouch B., Knapp S., Leon A., Velasco L., Fernândez-Martmez J., Berry S.T. Quantitative trait loci for broomrape (Orobanche Cumana Wallr.) resistance in sunflower // Theor. Appl. Genet. - 2004. - V. 109. - P. 92-102.
- Akhtouch B., Munoz-Ruz. J., Melero-Vara J.M., Fernândez-Martmez J.M., Dominguez J. Inheritance of resistance to race F of broomrape (Orobanche cumana Wallr.) in sunflower lines of different origin // Plant Breed. - 2002. - V. 121. - P. 266-268.
- Fernândez-Martmez J., Pérez-Vich B., Akhtouch B., Velasco L., Munoz-Ruz J., Melero-Vara J.M., Dommguez J. Registration of Four Sunflower Germplasms Resistant to Race F of Broomrape // Crop Sci. - 2004. - V. 44. - P. 1033-1034.
- Akhtouch B., del Moral L., Leon A., Velasco L., Fernândez-Martmez J.M., Pérez-Vich B. Genetic study of recessive broomrape resistance in sunflower // Euphytica. - 2016. - V. 209. - P. 419-428.
- Lu Y.H., Gagne G., Grezes-Besset B., Blanchard P. Integration of a molecular linkage group containing the broomrape resistance gene Or5 into an RFLP map in sunflower // Genome. - 1999. -V. 42. - P. 453-456.
- Lu Y., Melero-Vara J., Garcia-Tejada J., Blanchard P. Development of SCAR markers linked to the gene Or5 conferring resistance to broomrape (Orobanche cumana Wallr.) in sunflower // Theor. Appl. Genet. - 2000. - V. 100. - P. 625-632.
- Tang S., Heesacker A., Kishore V.K., Fernandez A., Sadik E.S., Cole G., Knapp S.J. Genetic mapping of the Or5 gene for resistance to Orobanche race E in sunflower // Crop Sci. - 2003. - V. 43. - P. 1021-1028.
- Imerovski I., Dedic B., Cvejic S., Miladinovic D., Jocic S., Owens G.L., Köcis Tubic N., Rieseberg L.H. BSA-seq mapping reveals major QTL for broomrape resistance in four sunflower lines // Mol. Breed. -2019. - V. 39. - P. 39-41.
- Duriez P., Vautrin S., Auriac M.C., Bazerque J., Boniface M.C., Callot C., Carrère S., Cauet S., Chabaud M., Gentou F. [et al.]. A receptor-like kinase enhances sunflower resistance to Orobanche cumana // Nat. Plants. - 2019. - V. 5. - P. 12111215.
- Антонова Т.С., Стрельников Е.А., Арасланова Н.М., Гучетль С.З., Челюстникова Т.А. Отбор на устойчивость к расе G заразихи из расщепляющихся популяций подсолнечника в искусственных условиях выращивания // Масличные культуры. Науч.-тех. бюл. ВНИИМК. - 2017. -Вып. 3 (171). - С. 18-22.
- Saghai-Maroof M.A., Soliman K.M., Jorgensen R A., Allard R. W. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics // PNAS. - 1984. - V. 81. - P. 8014-8018.
- Bi I.V. [et al.]. Improved RAPD amplification of recalcitrant plant DNA by the use of activated charcoal during DNA extraction // Plant Breeding. -1996. - V. 115. - V. 3. - P. 205-206.
- Гучетль С.З., Антонова Т.С., Арасланова Н.М., Челюстникова Т.А. Питинова Ю.В. Генетический анализ устойчивости к расе G Orobanche cumana Wallr. в F2 и ВС1 линий подсолнечника RGP1, RGP2, RGВ, RGL1, RGL2 // Масличные культуры. - 2019. - Вып. 4 (180). - С. 23-28. Х-2019-4-180-23-28.
- DOI: 10.25230/2412-608
- Imerovski I., Dimitrijevic А., Miladinovic D., Dedic B., Sinisa J., Kocis N. Cvejic S. Mapping of a new gene for resistance to broomrape races higher than F // Euphytica. - 2015. - V. 209. - P. 281-289.
- DOI: 10.1007/s10681-015-1597
- Badouin H., Gouzy J., Grassa C. [et al.]. The sunflower genome provides insights into oil metabolism, flowering and Asterid evolution // Nature. -2017. - V. 546. - P. 148-152. 10.1038/ Nature22380.
- DOI: 10.1038/nature22380
- https://www.ncbi.nlm.nih.gov/assembly/GCF_ 002127325.1.
- https://blast.ncbi.nlm.nih.gov/Blast.cgi.


