Поиск новых SSR-локусов ДНК для создания эффективной технологии генотипирования сои
Автор: Рамазанова Светлана Алексеевна, Савиченко Виолетта Георгиевна, Устарханова Эльмира Гереевна, Логинова Елизавета Дмитриевна, Рамазанов Руслан Нурмагомедович, Гучетль Аслан Хавиретович
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 4 (188), 2021 года.
Бесплатный доступ
Соя - основная белковомасличная культура, имеющая большое экономическое значение. В настоящее время для характеристики новых сортов, заявленных на выдачу патента, все больше применяют современные методы, основанные на анализе микросателлитных (SSR) локусов ДНК. Целью данной работы было провести поиск новых микросателлитных маркеров для оптимизации уже существующей технологии идентификации и паспортизации сортов сои. А так же подобрать условия проведения ПЦР с ними и апробировать на сортах коллекции ВИР. Из литературных источников и библиотечных баз данных были выбраны семь микросателлитных локусов, показавших высокий уровень полиморфизма на образцах сои и локализованных в разных хромосомах. Были подобраны экспериментальным путем оптимальные температуры отжига для всех пар SSR-праймеров. Результаты амплификации ДНК 20 генотипов сои показали, что все семь изученных SSR-локусов полиаллельны. В целом было выявлено 22 аллеля, что в среднем составило 3,1 на локус. Эффективное число аллелей Ne для изученных генотипов сои варьировало от 1,69 до 2,27 и в среднем составило 2,01. Среднее значение индекса полиморфного информационного содержания (PIC) было 0,50. Все изученные образцы сои имеют уникальные наборы аллелей по изученным локусам. Установлено, что семь апробированных нами локусов могут быть использованы при создании эффективной технологии для идентификации и паспортизации генотипов сои.
Соя, glycine max (l.) merr, ssr, микросателлиты, днк, паспортизация, генетическое разнообразие
Короткий адрес: https://sciup.org/142231746
IDR: 142231746 | УДК: 633.853.52:575 | DOI: 10.25230/2412-608X-2021-4-188-18-24
Текст научной статьи Поиск новых SSR-локусов ДНК для создания эффективной технологии генотипирования сои
Введение. При создании новых сортов сельскохозяйственных культур, сертификации и их коммерческом распространении генетическая паспортизация имеет большое значение. Довольно широкое применение для этих целей получили микросателлитные маркеры (SSR – Simple Sequence Repeats) [1; 2].
SSR-маркеры представляют собой простые повторяющиеся фрагменты ДНК, с единицей повтора 2-6 нуклеотидов. Их присутствие и равномерное распределение в геномах всех высших организмов, преимущественно кодоминантный характер наследования, высокий уровень полиморфизма, относительная простота детекции и высокая воспроизводимость результатов, а также возможность автоматизировать процесс анализа делает их одними из наиболее распространенных ДНК-маркеров [3].
В России и в мире проведено много исследовательских работ по идентификации генотипов растений с помощью мик-росателлитных маркеров. Полиморфизм микросателлитов был изучен у подсолнечника, ячменя, картофеля, риса, льна масличного, винограда, яблони и многих других сельскохозяйственных культур [4; 5; 6; 7; 8; 9;10].
Оценка генетического разнообразия и паспортизация сортов на основе SSR-маркеров также проводилась и на сое. B. Sun et al. проанализировали аллельные профили 149 образцов дикой сои из Китая с использованием 41 SSR-маркера, среднее значение индекса полиморфного информационного содержания PIC (poly- morphism information content) составило 0,825. Анализ выявил значительные различия среди популяций из провинций Хунань, Фуцзянь, Гуанси и Северного Китая. Также они предположили, что провинция Фуцзянь может быть основным центром генетического разнообразия однолетней дикой сои в Южном Китае [11].
Работы индийских ученых показали, что SSR-маркеры эффективны для оценки генетического родства сортов сои. J. Ghosh et al. проводили анализ 32 сортов с использованием 10 пар SSR-праймеров [12]. A. Bisen et al. проанализировали 38 сортов сои с помощью 16 микросателлит-ных локусов. Авторы предполагают, что SSR-маркеры эффективны для изучения генетического разнообразия, родства, а также идентификации разновидностей сои [13].
Сравнительная оценка полиморфизма 37 генотипов сои коллекции Казахстана была проведена с использованием 50 SSR-маркеров по всему геному сои. Было выявлено 167 аллелей. Индекс разнообразия проанализированной коллекции по Шеннону варьировал от 0,349 до 1,562. Среднее значение PIC составило 0,613. Установлены генетические расстояния между казахстанскими сортами, которые варьировали от 0,10 до 1,83. Для каждого коммерческого сорта сои Казахстана был разработан генетический паспорт на основе использования SSR-профилей [14].
Анализ вариабельности девяти микро-сателлитных локусов у 25 сортообразцов сои различного происхождения и четырех диких форм проводили П. Кезимана с соавторами. Индекс полиморфного информационного содержания PIC для изученных локусов составил в среднем 0,73 [15].
Идентификацию генотипов сои на основе микросателлитных маркеров проводили C. Mukuze et al. Они идентифицировали 34 сорта сои с использованием 21 пары SSR-праймеров [16]. В исследованиях M. Zulj Mihaljevic et al. анализи- ровали 97 сортов 42 парами микросател-литных праймеров [17].
Способ идентификации сортов и гибридных растений сои на основе микроса-теллитных маркеров был разработан в ФГБНУ ФНЦ ВНИИМК в 2008 г. [18; 19]. С помощью набора из девяти пар SSR-праймеров идентифицировали 72 генотипа сои российских и зарубежных селек-центров. Эту систему маркеров успешно применяли для идентификации сортов, линий и селекционных образцов. Однако с увеличением их количества этой системы маркеров стало недостаточно для идентификации некоторых генотипов. Поэтому появилась необходимость оптимизировать существующую технологию. Для идентификации генотипов сои использовали 13 пар полиморфных SSR-праймеров [20]. Но дальнейшие исследования показали, что примененная система молекулярных маркеров так же не позволяет различать некоторые генотипы. Поэтому целью исследования было подобрать новые информативные микро-сателлитные локусы ДНК и оценить возможность их использования для создания эффективной технологии для идентификации и паспортизации генотипов сои.
Материалы и методы. Материалом для исследования служили 20 сортов сои из коллекции ВИР, репродуцированной на Армавирской опытной станции – филиале ФГБНУ ФНЦ ВНИИМК: Лидия, Белор, Даурия, Соер-3, Северная 4, Сиб-НИИК 315, Окская, Ясельда, Витязь 50, СибНИИСХОЗ 6, Алтом, ВНИИОЗ-85, Лучезарная, Росинка, Венера (Приморская 915), Т-25, Альтаир, Брянская 11, Локус, Грибская-30.
Экстракцию ДНК осуществляли из фрагментов зеленых листьев из каждого растения сорта индивидуально и из смеси 10–15 растений по модифицированной методике с использованием СТАВ-буфе-ра [21]. Концентрацию ДНК определяли по интенсивности ее окрашивания бромистым этидием в 1%-ном агарозном геле.
Для исследования из литературных источников выбрали семь микросателлит-ных локусов (SCT413, SАT420, SАTT309, SАTT149, SАTT307, SАTT286, SАTT532). Экспериментально осуществили подбор температурных режимов для полимеразной цепной реакции с ними.
Полимеразную цепную реакцию (ПЦР) проводили в объеме реакционной смеси 25 мкл, содержащей: 67 мМ трис-HCl, рН 8,8; 16,6 мM сульфата аммония; 1,5– 3,0 мM MgCl 2 ; 0,01 % Tween 20; по 0,2 мM каждого dNTP; по 10 пМ праймеров; 10 нг матричной ДНК и 1 единицу Taq-полимеразы (НПО «СибЭнзим», Россия). Амплификацию проводили в термоциклере MiniAmp Plus (Thermo Fisher, США) при следующих температурных режимах: начальная денатурация при 96 °С в течение 2 мин, затем 30–34 цикла при температурно-временном режиме: денатурация при 94 °С – 30 сек, отжиг при 45– 60 °С – 40 сек, элонгация при 70 °С – 1 мин, финальная элонгация при 70 °С – 2 мин. Количество циклов и температура отжига зависели от используемых праймеров. Детекцию продуктов амплификации осуществляли с помощью электрофореза высокого разрешения в 8 %-ном полиакриламидном геле в денатурирующих условиях (параметры силы тока и напряжения: 200 mA, 800 V) с последующей окраской геля азотнокислым серебром. После окрашивания гель фотографировали и анализировали.
Для каждого изучаемого локуса были определены индекс полиморфного информационного содержания (PIC) и эффективное число аллелей (Ne). Вычисления проводили с помощью компьютерного программного обеспечения Gene-Calc [22].
Результаты и обсуждение. Для создания эффективной технологии генотипирования сои необходима система информативных микросателлитных маркеров с достаточно высоким уровнем полиморфизма. Из литературных источников нами были отобраны семь микро- сателлитных локусов ДНК, характеристики которых были получены из библиотечных баз данных Primer-BLAST и Soybase [23; 24]. Их характеристика и нуклеотидные последовательности фланкирующих их праймеров представлены в таблице 1.
Таблица 1
Характеристика микросателлитных локусов ДНК сои, полученная из баз данных Primer-BLAST и Soybase
|
№ |
Локус |
Лока-лиза-ция |
Мотив |
Нуклеотидные последовательности фланкирующих праймеров |
Размер фраг-мента |
|
1 |
SCT 413 |
1 |
(AT)35 |
F:GCGCTCCCTTCTTTTCC ACTGAATTGA R:GCGTTTTCTCTCGGTTT CTCTCTTCTTATTA |
200 |
|
2 |
SАT 420 |
20 |
(AT)19 |
F:GCGGATGGAGCCAACA R:GCGTGTAGCCCTAGAA AGTT |
186 |
|
3 |
SАTT 309 |
18 |
(ATT)1 3 |
F:GCGCCTTCAAATTGGCG TCTT R:GCGCCTTAAATAAAAC CCGAAACT |
147 |
|
4 |
SАTT 149 |
13 |
(ATT)1 7 |
F:TTGCACATTCTTTTTGGT AAACAGTCATAA R:GTTGGAGGCCATAGTC ACATTAATCTTAGA |
274 |
|
5 |
SАTT 307 |
6 |
(ATT)1 2 |
F:GCGCTGGCCTTTAGAAC R:GCGTTGTAGGAAATTTG AGTAGTAAG |
162 |
|
6 |
SАTT 286 |
6 |
(ATT)1 7 |
F:GCGGCGTTAATTTATGC CGGAAA R:GCGTTTGGTCTAGAATA GTTCTCA |
217 |
|
7 |
SАTT 532 |
1 |
(ATT)1 5 |
F:GCGCCAATATTATCATG CTTTATGT R:GCGTGTAAAAATCTTTG AATCTTGA |
168 |
Для каждой из представленных пар праймеров рассчитана температура отжига (в веб-версии программы PrimerBLAST). Однако эти значения являются приблизительными и требуют их оптимизации экспериментальным путем. Для этого с каждой парой праймеров проводили ПЦР. Образцы ДНК пяти сортов сои амплифицировали по установленному протоколу, изменяя температуру отжига в каждом опыте на 3–5 °С.
Выбор оптимального значения температуры отжига основывался на получен- ных спектрах ДНК после электрофоретического разделения амплифицированных фрагментов. Необходимо было добиться получения четких, хорошо различимых фрагментов в характерном для каждого локуса диапазоне длин. А также уменьшить неспецифический отжиг праймеров. На рисунке для примера показаны электрофореграммы продуктов амплификации с праймерами SAT420 и SCT413. Для праймера SAT420 оптимальной выбрана температура отжига 58 °С, так как при снижении температуры появляются дополнительные, неспецифичные фракции ДНК (рис. А). Для праймера SCT413 лучшего качества фореграммы получены также при 58 °С. При слишком высокой температуре отжиг ухудшается и, как следствие, на фореграмме выявляются нечеткие, плохо различимые фрагменты (рис. Б).
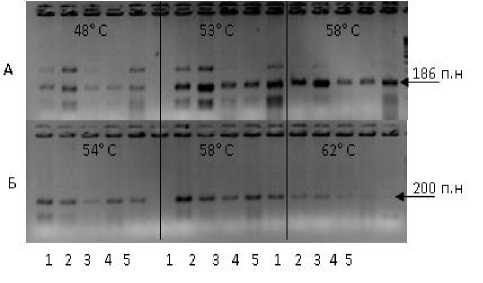
Рисунок – Оптимизация температуры отжига для ПЦР анализа микросателлитных локусов; фореграммы продуктов амплификации А – с праймером SАT420, Б – с праймером SCT413;
дорожки 1–5 – генотипы сои (ориг.)
В результате проведенных экспериментов для каждой пары праймеров были подобраны оптимальные значения температуры отжига, которые представлены в таблице 2. Из таблицы видно, что у всех праймеров расчетная температура отжига отличается от подобранной нами экспериментально.
Таблица 2
Оптимальные температуры отжига для ПЦР анализа микросателлитных локусов ДНК сои
|
№ |
Локус |
Температура отжига (°С) |
|
|
Расчетная |
Экспериментальная (оптимальная) |
||
|
1 |
SCT413 |
64,0 |
58 |
|
2 |
SАT420 |
57,1 |
58 |
|
3 |
SАTT309 |
61,0 |
50 |
|
4 |
SАTT149 |
61,5 |
56 |
|
5 |
SАTT307 |
56,5 |
47 |
|
6 |
SАTT286 |
60,0 |
58 |
|
7 |
SАTT532 |
57,5 |
55 |
По всем изучаемым микросателлит-ным локусам с использованием ПЦР было проведено генотипирование 20 сортов сои коллекции ВИР. Для каждого сорта был получен набор амплифицированных фрагментов ДНК, для которых рассчитали приблизительный размер. Фрагмент наибольшей длины обозначали цифрой 1 и далее цифрами 2, 3, 4 по мере уменьшения их длин. Таким образом, получили характеристики аллельного состояния микросателлитных локусов у образцов сои (табл. 3).
Таблица 3
Полиморфизм микросателлитных локусов ДНК сортов сoи
|
Генотип |
Локус |
||||||
|
SCT 413 |
SAT 420 |
SATT 309 |
SATT 149 |
SATT 307 |
SATT 286 |
SATT 532 |
|
|
Лидия |
2 |
3 |
2 |
1 |
2 |
2 |
2 |
|
Белор |
1 |
3 |
2 |
3 |
1 |
1 |
2 |
|
Даурия |
3 |
3 |
3 |
3 |
2 |
1 |
2 |
|
Соер-3 |
2 |
2 |
2 |
3 |
3 |
3 |
2 |
|
Северная 4 |
2 |
2 |
2 |
3 |
2 |
1 |
1,2 |
|
СибНИИК-315 |
3 |
2 |
2 |
3 |
1 |
1 |
1,2 |
|
Окская |
2 |
2 |
2 |
2 |
2 |
1 |
2 |
|
Ясельда |
3 |
2 |
2 |
3 |
2 |
1 |
1 |
|
Витязь 50 |
2 |
2 |
1 |
2 |
2 |
2 |
1,3 |
|
СибНИИСХОЗ-6 |
1 |
2 |
3 |
3 |
1 |
1 |
2 |
|
Алтом |
1 |
1 |
3 |
3 |
1 |
1 |
3 |
|
ВНИИОЗ-85 |
3 |
2 |
3 |
1 |
2 |
3 |
1,2 |
|
Лучезарная |
2 |
2 |
2 |
1 |
2 |
1 |
1 |
|
Росинка |
2 |
2 |
2 |
3 |
2 |
1 |
1 |
|
Венера (Примоская 915) |
2 |
2 |
3 |
3 |
3 |
1 |
2 |
|
Т-25 |
2 |
2 |
2 |
1 |
2 |
4 |
3 |
|
Альтаир |
3 |
3 |
2 |
3 |
1 |
1 |
1 |
|
Брянская 11 |
1 |
3 |
2 |
1 |
3 |
2 |
3 |
|
Локус |
2 |
3 |
3 |
1 |
3 |
3 |
1 |
|
Грибская 30 |
2 |
2 |
2,3 |
3 |
3 |
2 |
2,3 |
Как видно из таблицы 3, в основном у изученных сортов выявлено по одному аллелю на локус. Однако у сортов Северная 4, СибНИИК-315, Витязь 50, ВНИИ-ОЗ-85 по локусу Satt532, а у сорта Грибская 30 по локусам SATT532 и SATT309 выявлено по два фрагмента разной длины. Это свидетельствует о наличии внутрисортового полиморфизма у этих сортов и требует дальнейшего изучения.
У большинства изучаемых SSR-локусов было выявлено по три аллеля, по локусу SAT286 – четыре, что в среднем составило 3,1 аллеля на локус (табл. 4). При этом эффективное число аллелей для изучаемых генотипов сои варьировало от 1,69 до 2,27 со средним значением 2,01.
Таблица 4
Основные показатели информативности изучаемых микросателлитных локусов
|
№ |
Локус |
Количество аллелей, Na |
Эффективное число аллелей, Ne |
Индекс полиморфного информационного содержания, PIC |
|
1 |
SCT413 |
3 |
2,13 |
0,53 |
|
2 |
SАT420 |
3 |
1,69 |
0,41 |
|
3 |
SАTT309 |
3 |
1,72 |
0,42 |
|
4 |
SАTT149 |
3 |
1,89 |
0,47 |
|
5 |
SАTT307 |
3 |
2,27 |
0,56 |
|
6 |
SАTT286 |
4 |
2,13 |
0,53 |
|
7 |
SАTT532 |
3 |
2,27 |
0,56 |
|
Среднее |
3,1 |
2,01 |
0,50 |
|
Индекс полиморфного информационного содержания (PIC), характеризующий информативность микросателлитных локусов, в изученной группе генотипов сои варьировал от 0,41 до 0,56 со средним значением 0,50. Наибольшее значение PIC было получено по локусам SATT307 и SATT532 – 0,56.
Заключение. В результате исследования были апробированы семь пар праймеров, описанных в литературе как фланкирующие микросателлитные участки ДНК сои. В зависимости от локуса число аллелей варьировало от 3 до 4 (в среднем 3,1 аллеля на локус). Эффективное число аллелей Ne для изученных генотипов сои в среднем составило 2,01, а значение индекса полиморфного информационного содержания (PIC) 0,50. Из двадцати образцов сои пять оказались генетически неоднородными. Проанализированные образцы различались по аллельному состоянию локусов. Таким образом, изученные маркеры пригодны для создания молекулярно-генетических паспортов и определения генетической однородности сортов сои. Можно сделать вывод, что семь апробированных нами локусов могут быть использованы для создания эффективной технологии для идентификации и паспортизации генотипов сои.
Список литературы Поиск новых SSR-локусов ДНК для создания эффективной технологии генотипирования сои
- Давидчук Н.Д., Корабельская Е.М., Еремеева Н.В., Кобыльский Г.И. Полиморфизм запасных белков и использование его в семеноводстве пшеницы и ячменя // Вестник Тамбовского Государственного Университета. -2009. - Т. 14. - С. 116-121.
- Азарин К.В., Маркин Н.В., Лотник B.C., Усатов А.В. ДНК маркеры в селекции растений: учеб. пособие // Ростов-на-Дону: Издательство Южного федерального университета, 2012. - С. 16.
- Сухарева А.С., Кулуев Б.Р. ДНК-маркеры для генетического анализа сортов культурных растений // Биомика. - 2018. - Т. 10. - № 1. - С. 69-84.
- Гучетль С.З., Зайцев Н.И., Фролов С.С., Фролова И.Н., Кузнецова Е.С. Генотипирование инбредных линий и гибридов подсолнечника селекции Армавирской опытной станции ВНИИМК с помощью микросател-литных локусов // Масличные культуры. - 2019. - № 3 (179). - С. 27-34.
- Сиволап Ю.М., Бальвинская М.С., Родер М. SSRP-анализ молекулярно-генетического полиморфизма сортов ярового ячменя южно-украинской селекции // Доклады Росс. акад. сельхоз. наук. - 2001. - № 5. - С. 3-7.
- Колобова О.С., Малюченко О.П. , Шалаева Т.В., Шанина Е.П., Шилов И.А. [и др.]. Генетическая паспортизация картофеля на основе мультиплексного анализа 10 микросателлитных маркеров // Вавиловский журнал генетики и селекции. - 2017. - № 21. - С. 124-127. DOI 10.18699/VJ17.230
- Супрун И.И., Ковалев В.С., Токмаков С.В., Белан К.А. ДНК-паспортизация современных российских сортов риса с применением SSR-маркеров // Науч. журнал КубГАУ - 2017. - Т. 131. - С. 1-11. DOI: 10.21515/19904665-131-065.
- Челюстникова Т.А., Гучетль С.З., Антонова Т.С. Микросателлитные локусы для идентификации сортов льна масличного селекции ВНИИМК: подбор информативных праймеров и оптимальных условий ПЦР ДНК // Масличные культуры. - 2019. - № 2 (178). - С. 41-46.
- Гориславец С.М., Рисованная В.И., Спотарь Г.Ю., Володин В.А. Генотипирование сорта винограда Бессемянный Магарача и анализ его происхождения с использованием SSR-маркеров // Магарач. Виноградарство и виноделие. - 2018. - Т. 20. - С. 19-21.
- Лыжин А.С., Соловченко А.Е. Создание генетических паспортов подвойных форм яблони на основе анализа полиморфизма микросателлитных последовательностей ДНК // Достижения науки и техники АПК. - 2019. - № 33. - C. 11-13. DOI: 10.24411/0235-24512019-10203.
- Sun B., Fu C., Yang C., Ma Q. [et al.] Genetic diversity of wild soybeans from some regions of Southern China based on SSR and SRAP markers // American Journal of Plant Sciences. - 2013. - № 4. - Р. 257-268.
- Ghosh J., Ghosh P.D., Choudhury P.R. An assessment of genetic relatedness between soybean [Glycine max (L.) Merrill] cultivars using SSR markers // American Journal of Plant Sciences. - 2014. - No 5. - Р. 3089-3096.
- Bisen А., Khare D., Nair P., Tripathi N. SSR analysis of 38 genotypes of soybean (Glycine Max (L.) Merr.) genetic diversity in India // Physiol Mol Biol Plants. - 2015. -No 21 (1). - Р. 109-115.
- Абугалиева С.И., Волкова Л.А., Нурланова А.А., Жанпеисова А.С., ТуруспековЕ.К. ДНК-фингерприн-тинг сортов сои Казахстана с использованием SSR-маркеров // Биотехнология. Теория и практика. - 2013. -№ 3. - С. 26-34.
- Кезимана П., Романова Е.В., Трифонова (Кочу-мова) А.А., Шмелькова Е.О. Анализ вариабельности микросателлитных локусов сортов сои (Gtycine max) // Теоретические и прикладные проблемы АПК. - 2016. -№ 4. - С. 3-7.
- Mukuze C., Tucamuhabwa P., Maphosa M., Dari S., Dramadri I.O. [et al.] Genetic diversity analysis among soybean genotypes using SSR markers in Uganda // African Journal of Biotechnology. - 2020. - Vol. 19 (7). - P. 439448.
- Zulj Mihaljevic M., Sarcevic H, Lovric A., Andrijanic Z., Sudaric A. [et al.]. Genetic diversity of European commercial soybean [Glycine max (L.) Merr.] germplasm revealed by SSR markers // Genet Resour Crop Evol. - 2020. -No 67. - Р. 1587-1600.
- Способ идентификации сортов сои на основе микросателлитных (SSR) маркеров: пат. 2388828 РФ : МКП C12Q 1/68 10.05.2010.
- Способ выделения гибридных растений сои с использованием микросателлитных (SSR) локусов ДНК : пат. 2398883 РФ : МКП C12Q 1/68 10.09.2010.
- Рамазанова С.А., Коломыцева А.С. Оптимизация технологии генотипирования сои на основе анализа полиморфизма SSR-локусов ДНК // Масличные культуры. - 2020. - № 1 (181). - С. 42-48.
- Saghai-Maroof M.A., Soliman K.M., Jorgensen R.A., Allard R. W. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics // PNAS USA. - 1984. -81. - P. 8014-8018.
- Binkowski J., Miks S. Gene-Calc [компьютерное программное обеспечение]: [Электронный ресурс]. -Режим доступа: https://gene-calc.pl/pic (дата обращения: 26.10.2021).
- SoyBase. Integrating Genetics and Genomics to Advance Soybean Research: [Электронный ресурс]. -Режим доступа: https://soybase.org/ (дата обращения: 10.09.2021).
- National Center for Biotechnology Information, U.S. National Library of Medicine [Электронный ресурс]. -Режим доступа: https://www.ncbi.nlm.nih.gov/ (дата обращения: 26.10.2021).


