Поиск следов селекции в геномах домашних коз (Capra hircus L.), разводимых в России, с помощью идентификации островков гомозиготности
Автор: Денискова Т.Е., Доцев А.В., Селионова М.И., Айбазов А.М.М., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Статья в выпуске: 4 т.59, 2024 года.
Бесплатный доступ
Геномы современных популяций сельскохозяйственных животных формировались под влиянием длительной селекции, следы которой можно детектировать с помощью биоинформатических подходов. Так, для анализа инбридинга в популяциях сельскохозяйственных животных используют сегменты гомозиготности (runs of homozygosity, ROH). Более того, ROH подходят для выявления признаков отбора через островки ROH. Распределение сегментов гомозиготности в геномах групп коз, разводимых на территории России, было изучено нами ранее, однако информация о признаках отбора в геноме этих групп коз до сих пор отсутствует. В настоящей работе впервые проведен поиск следов селекции в геномах пород и популяций коз, разводимых в России. Идентифицированы гены, локализованные в островках ROH на CHI12, CHI18, CHI25. Среди генов выявлены функциональные кандидаты, ассоциированные с шерстной (IFT88, CUX1) и молочной (LATS2) продуктивностью, регулирующие метаболизм (LCAT, PLA2G15, SMPD3), рост (SLC12A4, SH2B2) , репродуктивные (MRPL57, MICU2, SAP18) и иммунно-адаптивные функции (DDX28, IL17D) . Цель исследования - выявить геномные регионы, находящиеся под давлением селекции в российских популяциях коз, используя поиск островков ROH. Выборка коз (Capra hircus L.) включала следующие породы: алтайскую белую пуховую (n = 20), горноалтайскую пуховую (n = 33), дагестанскую пуховую (n = 34), дагестанскую шерстную (n = 20), оренбургскую (n = 32) и советскую шерстную (n = 29), а также популяцию местных карачаевских коз (n = 36). Геномные исследования популяций зааненской породы, разводимой в России, были проведены нами ранее, поэтому в представляемой работе SNP-профили зааненской породы (n = 33) были использованы как группа сравнения. ДНК выделяли из ушных выщипов с помощью набора ДНК-Экстран-2 (ООО «НПФ Синтол», Россия). SNP-профили были сгенерированы с использованием ДНК-чипа Illumina Goat SNP50 BeadChip («Illumina, Inc.», США) в рамках нашего предыдущего исследования. Работу проводили на базе ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста в 2021-2024 годах. Индивидуальная генетическая сеть была построена по принципу ближайшего соседа (Neighbor Net) в программе SplitsTree 4.14.5. Для поиска ROH в геноме исследуемых коз использовали метод последовательных прогонов, реализованный в пакете R «detectRUNS». В качестве индикаторов островков ROH были отобраны перекрывающиеся сегменты с минимальной длиной ROH в 0,3 Мb, которые встречались у более чем 50 % особей внутри своей группы. Поиск генов-кандидатов, локализованных внутри островков ROH, осуществляли с помощью онлайн-инструмента Genome Data Viewer (NCBI) по сборке генома домашних коз CHIR_1.0 (GCF_000317765.1). Генная онтология была проанализирована с использованием онлайн-ресурса DAVID (Database for Annotation, Visualization and Integrated Discovery). Островки ROH были обнаружены на 12-й хромосоме (CHI12) у горноалтайской, советской шерстной и дагестанской пуховой пород, а также на CHI18 и CHI25 у горноалтайской и алтайской белой пуховой пород. Во всех островках ROH были идентифицированы и аннотированы гены. Островок ROH на CHI12 включал 13 общих для четырех пород генов, среди которых наиболее значимыми были LATS2 (активация лактогенеза и потенциальное влияние на репродукцию), IL17D (функция иммунитета), IFT88 (ассоциация с тониной кашемировых волокон), MRPL57 (регуляция фолликулярной фазы эстрального цикла), MICU2 и SAP18 (развитие яичников и эмбрионов, спермиогенез). Внутри островка ROH на CHI18 были локализованы 16 генов, регулирующих липидный метаболизм (LCAT, PLA2G15 и SMPD3), вовлеченных в формирование реакции на гипоксию (DDX28) и связанных с развитием мышечных волокон (SLC12A4). Геномный регион на CHI25 включал гены PRKRIP, SH2B2, CUX1 и POLR2J, среди которых наиболее перспективными были CUX1, регулирующий развитие волосяных фолликулов, и SH2B2, ассоциированный с ростом и размерами туловища. Таким образом, наше исследование расширяет понимание геномной архитектуры российских локальных пород и популяций коз.
Snp, островки roh, гомозиготность, гены-кандидаты, домашние козы, признаки отбора, локальные породы
Короткий адрес: https://sciup.org/142243758
IDR: 142243758 | УДК: 636.39:575.174 | DOI: 10.15389/agrobiology.2024.4.620rus
Текст научной статьи Поиск следов селекции в геномах домашних коз (Capra hircus L.), разводимых в России, с помощью идентификации островков гомозиготности
В отечественном козоводстве выделяют несколько направлений продуктивного выращивания животных: пуховое, шерстное, молочное и грубошерстное, или мясное (1). По данным на 31 декабря 2022 года, наибольшую долю в российском козоводстве занимает молочное направление (51,5 % от численности коз из идентифицированных пород) с лидерством зааненской породы (42,7 тыс. гол.), тогда как на долю исконно русских направлений козоводства (пуховое и шерстное) приходится соответственно 17,7 и 30,8 % при драматическом сокращении численности пуховых пород (2). Так, за последние 5 лет поголовье горноалтайской породы снизилось с 18,7 до 8,9 тыс. гол., а оренбургской — с 6,0 до 2,9 тыс. гол. Численность советской шерстной породы не такая критическая — 30,5 тыс. гол., однако сокращение поголовья затронуло и эту породу (58,7 тыс. гол. в 2018 году) (2). Грубошерстное направление представлено в основном местными неидентифи-цированными козами (1). Риски исчезновения отечественных пород коз, обладающих ценными свойствами, имеют негативные последствия как для национального, так и для мирового генофонда.
Уникальность отечественных пуховых и шерстных пород заключается прежде всего в том, что эти породы способны производить высококачественную продукцию (например, оренбургский пуховый платок известен и за пределами нашей страны) в условиях резко континентального климата, в котором иностранные породы не могут с ними конкурировать. По сравнению с другими видами сельскохозяйственных животных козы используют скудную растительность для удовлетворения потребности в питательных веществах и энергии (3). Кроме того, локальные породы сельскохозяйственных животных играют важную роль в сохранении культурного наследия человечества (4).
Поиск сегментов гомозиготности (runs of homozygosity, ROH) — это современный метод анализа инбридинга в популяциях сельскохозяйственных животных. Кроме того, ROH подходят для выявления признаков отбора через островки ROH (5), поскольку селекция — это значимый эволюционный фактор, способный повысить гомозиготность (6). Также при положительном отборе для улучшения экономически значимых признаков и поддержания стандарта породы возможно уменьшение изменчивости в целевых участках генома, поэтому ROH могут указывать на отпечатки селекции (6). Примеры, демонстрирующие эффективность поиска следов селекции, сопряженных с островками ROH, описаны у крупного рогатого скота (КРС) (7), свиней (8), овец (9), лошадей (10), кроликов (11) и кур (12).
F. Bertolini с соавт. (6) использовали SNP профили коз из разных стран, сгенерированные в рамках проекта AdaptMap , для выявления общих закономерностей распределения ROH в основных трансграничных и локальных группах. Изучая популяции швейцарских коз, H. Signer-Hasler с соавт. (13) обнаружили в островках ROH гены, связанные с одомашниванием. В частности, вариант гена TSHR мог повлиять на изменения сезонности воспроизводства у современных домашних коз, а ген STC1 подвергался позитивному отбору в отношении увеличения размера тела и/или надоев молока. По сообщению нескольких исследовательских групп, в островках ROH у домашних коз довольно часто локализуются гены, связанные с адаптациями к природно-климатическим условиям разведения (14-16). У китайских локальных пород коз в островках ROH были выявлены гены, связанные с иммунными функциями (14).
Распределение сегментов гомозиготности в геномах групп коз, разводимых на территории России, было изучено в наших предыдущих исследованиях (17). Однако информация о признаках отбора в геноме этих групп коз до сих пор отсутствует.
В настоящей работе впервые проведен поиск следов селекции в геномах российских пород и популяций коз. Идентифицированы гены, локализованные в островках ROH на CHI12, CHI18, CHI25. Среди генов выявлены функциональные кандидаты, ассоциированные с шерстной ( IFT88, CUX1 ) и молочной ( LATS2 ) продуктивностью, регулирующие метаболизм ( LCAT, PLA2G15, SMPD3 ), рост ( SLC12A4, SH2B2 ) , репродуктивные ( MRPL57 , MICU2, SAP18 ) и иммунно-адаптивные функции ( DDX28, IL17D ) .
Цель исследования — выявить геномные регионы, находящиеся под давлением селекции в отечественных популяциях коз, с использованием поиска островков ROH.
Методика. Работу проводили на базе ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста в 2021-2024 годах. Выборка коз ( Capra hircus L.) включала следующие породы: алтайскую белую пуховую (ALTW, n = 20), горноалтайскую пуховую (ALTM, n = 33), дагестанскую пуховую (DAGD, n = 34), дагестанскую шерстную (DAGL, n = 20), оренбургскую (OREN, n = 32) и советскую шерстную (SOVM, n = 29), а также популяцию местных карачаевских коз (KARA, n = 36). Образцы ткани коз ALTW, ALTM, OREN и SOVM были отобраны в племенных хозяйствах и племенных репродукторах, образцы ткани коз DAGD, DAGL и KARA — в частных хозяйствах.
Геномные исследования российских популяций зааненской породы были проведены нами ранее (18), поэтому в представляемой работе SNP-профили зааненских коз (SAAN, n = 33) использовали как группу сравнения.
ДНК выделяли из ушных выщипов с помощью набора ДНК-Экс-тран-2 (ООО «НПФ Синтол», Россия) согласно рекомендациям производителя. SNP-профили были сгенерированы с использованием ДНК-чипа Illumina Goat SNP50 BeadChip («Illumina, Inc.», США) (19, 20) в рамках нашего предыдущего исследования (17).
Индивидуальную генетическую сеть строили по принципу ближайшего соседа (Neighbor Net) в программе SplitsTree 4.14.5 (21).
Для поиска ROH в геноме коз использовали метод последовательных прогонов (22), реализованный в пакете R «detectRUNS» (23). В анализе допускался один SNP с отсутствующим генотипом и до одного возможного гетерозиготного генотипа. Минимальная длина ROH составляла 500000 п.н.
В качестве индикаторов островков ROH в геноме отобрали перекрывающиеся сегменты с минимальной длиной ROH 0,3 Мb, которые встречались у более чем 50 % особей внутри своей группы.
Поиск генов-кандидатов внутри островков ROH осуществляли с помощью онлайн-инструмента Genome Data Viewer (NCBI) по сборке генома домашних коз CHIR_1.0 (GCF_000317765.1) . Генную онтологию анализировали с использованием онлайн-ресурса DAVID (Database for Annotation, Visualization and Integrated Discovery) (24, 25).
Результаты. Анализ индивидуальной генетической сети (рис. 1) продемонстрировал, что SOVM и OREN формировали консолидированные группы, как и KARA. ALTM и ALTW образовали смешанный кластер. Это, вероятно, отражает происхождение ALTW, созданной посредством поглотительного скрещивания козоматок ALTM с козлами советской шерстной породы для формирования группы животных, производящих белый пух (26).
Примечательным выглядело распределение особей DAGD и DAGL.
Большая часть животный DAGD располагалась по обе стороны от SOVM, а большинство коз DAGL — от SAAN. Такая кластеризация, как правило, предполагает, что «центральная» порода оказала влияние на формирование генофонда присоединяющихся группировок. Кроме того, SAAN присоединялась к основной генетической сети через особей DAGL. Это, во-первых, указывает на четкую дифференциацию швейцарской молочной породы от локальных российских групп коз, во-вторых, предположительно может свидетельствовать об использовании этой породы для улучшения DAGL. Документального подтверждения нашего предположения мы не нашли, однако в научной литературе описаны примеры использования производителей зааненской породы для повышения молочной продуктивности аборигенных дагестанских коз в некоторых хозяйствах (27). Кроме того, группа DAGD была более консолидированной по сравнению с DAGL. Касательно DAGD такая кластеризация согласуется с аспектами происхождения этой породы, выведенной при улучшении аборигенных коз производителями советской шерстной породы (28).
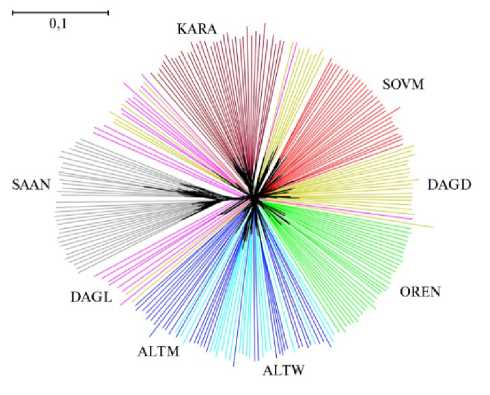
Рис. 1. Индивидуальная генетическая сеть, построенная по принципу ближайшего соседа, для групп коз ( Capra hircus L.) из российских популяций: ALTW — алтайская белая пуховая, ALTM — горноалтайская пуховая, DAGD — дагестанская пуховая, DAGL — дагестанская шерстная, OREN — оренбургская, SOVM — советская шерстная, SAAN — зааненская, KARA — местные карачаевские козы (ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 20212024 годы).
Островки ROH, то есть протяженные сегменты гомозиготности, встречающиеся более чем у 50 % особей внутри своей группы, были обнаружены на CHI12, CHI18 и CHI25 у ALTM, ALTW, DAGD и SOVM (рис. 2). У остальных групп коз островков ROH мы не обнаружили. Это могло объясняться несколькими причинами. Например, местные карачаевские козы до сих пор не выделены как порода, поэтому можно предположить, что давление искусственного отбора (селекции) было выражено слабее по сравнению с заводскими породами.
Возможно, для поисков признаков отбора, связанного с адаптацией к природным особенностям региона разведения, в группе этих животных более информативными будут другие биоинформатические подходы, например расчет значений F ST . Показано (см. рис. 1), что дагестанская шерстная порода испытывает влияние других пород, и это, вероятно, снижает геномный инбридинг и приводит к разделению протяженных сегментов ROH. Относительно заводских пород — оренбургской и зааненской мы полагаем,
что продолжение исследований на больших выборках поможет лучше понять распространения ROH у этих групп.
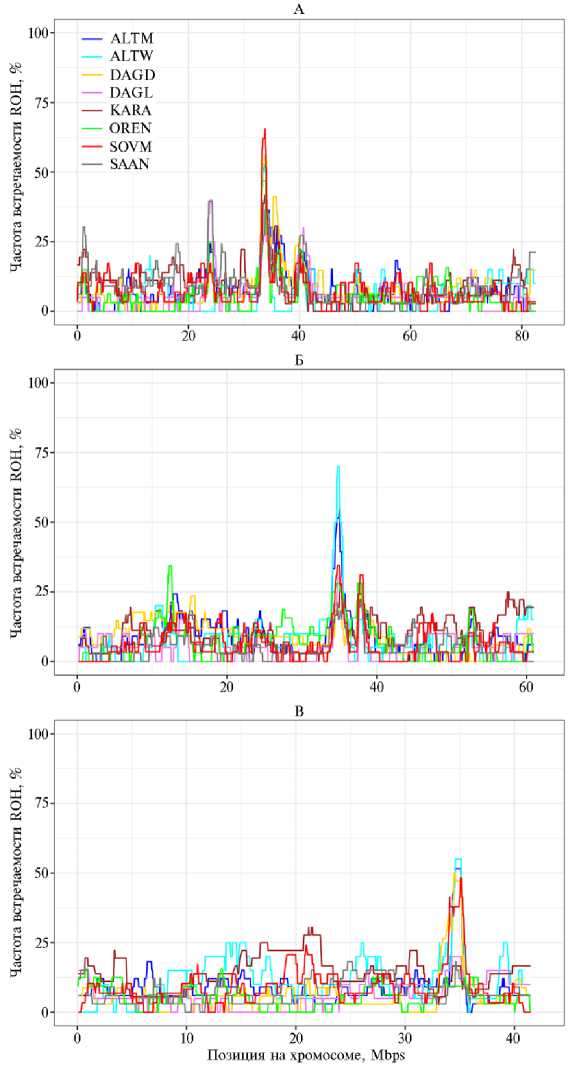
Рис. 2. Локализация островков ROH на CHI12 (А) , CHI18 (Б) и CHI25 (В) в геномах коз ( Capra hircus L.) из российских популяций: ALTW — алтайская белая пуховая, ALTM — горноалтайская пуховая, DAGD — дагестанская пуховая, DAGL — дагестанская шерстная, OREN — оренбургская, SOVM — советская шерстная, SAAN — зааненская, KARA — местные карачаевские козы (ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2021-2024 годы).
В таблице 1 представлены наиболее часто встречающиеся сегменты ROH, которые были классифицированы как островки ROH, с указанием начальной и конечной позиции и числа локализованных SNPs.
-
1. Наиболее распространенные (у ≥ 50 % особей в каждой породе) ROH, обнаруженные на CHI12, CHI18 и CHI25 у коз ( Capra hircus L.) из российских популяций (ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2021-2024 годы)
-
2. Гены, локализованные в идентифицированных островках ROH у коз ( Capra hircus L.) из российских популяций (ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2021-2024 годы)
|
Порода |
CHI |
n SNP |
SNP |
Позиция, bp |
||
|
стартовый |
конечный |
начальная |
конечная |
|||
|
ALTM |
12 |
11 |
rs268262662 |
rs268262652 |
33 412 747 |
33 884 399 |
|
DAGD |
12 |
12 |
rs268262662 |
rs268262650 |
33 412 747 |
33 961 522 |
|
SOVM |
12 |
16 |
rs268262665 |
rs268262649 |
33 230 265 |
33 999 048 |
|
ALTM ALTW |
18 |
8 |
rs268255074 |
rs268240149 |
34 686 451 |
35 017 668 |
|
ALTM ALTW |
25 |
110 |
rs268244509 |
rs268244520 |
34 584 777 |
35 124 678 |
Примечани е. CHI — хромосома коз; n SNP — число SNP, локализованных в островке ROH; ALTW — алтайская белая пуховая, ALTM — горноалтайская пуховая, DAGD — дагестанская пуховая, SOVM — советская шерстная.
Следует отметить, что 6 из 8 SNPs на CHI18 встречались у 60 % особей породы ALTW и 8 из 16 SNPs на CHI12 — у 60 % особей породы SOVM.
В таблице 2 приведены гены, расположенные внутри островков ROH.
|
Порода |
CHI |
Позиция |
Гены |
|
|
начальная |
конечная |
|||
|
ALTM |
12 |
33 412 747 |
33 884 399 |
LATS2 а, XPO4 а, N6AMT2 а, IL17D а, IFT88 а, CRYL1 а, ZDHHC20 а , LOC106502707 а , MICU2 а , SKA3 а, SAP18 а, MRPL57 а, GJA3 а |
|
DAGD |
12 |
33 412 747 |
33 961 522 |
LATS2 а, XPO4 а, N6AMT2 а, IL17D а, IFT88 а, CRYL1 а, ZDHHC20 а, LOC106502707 а, MICU2 а, SKA3 а, SAP1 а, MRPL57 а, GJA3 а, GJB2 , GJB6 , ZMYM2 |
|
SOVM |
12 |
33 230 265 |
33 999 048 |
LATS2 а, XPO4 а, N6AMT2 а , IL17D а, IFT88 а, CRYL1 а, ZDHHC20 а, LOC106502707 а, MICU2 а, SKA3 а, SAP18 а, MRPL57 а, GJA3 а, GJB2 , GJB6 , ZMYM2 , ZMYM5 |
|
ALTM ALTW |
18 |
34 686 451 |
35 017 668 |
NFATC3 , DUS2 , DDX28 , LCAT , PSMB10 , SLC12A4 , PRMT7 , SLC7A6OS , SLC7A6 , PLA2G15 , ESRP2 , LOC102180420 , SMPD3 , LOC102169124 |
|
ALTM ALTW |
25 |
34 584 777 |
35 124 678 |
PRKRIP , SH2B2 , CUX1 , POLR2J |
Примечани е. CHI — хромосома коз; ALTW — алтайская белая пуховая, ALTM — горноалтайская пуховая, DAGD — дагестанская пуховая, SOVM — советская шерстная. а — гены, общие для пород ALTM, DAGD и SOVM.
Таким образом, мы идентифицировали островки ROH на трех хромосомах: CHI12, CHI18 и CHI25. F. Bertolini с соавт. (6) проводили поиск протяженных гомозиготных сегментов в выборках, представленных множеством пород коз из разных стран. Авторы сообщили об общих выявленных закономерностях — островках ROH на CHI12 и CHI18, что по хромосомной локализации совпадает с нашими расчетными данными. Согласно E.S. Kim с соавт. (29), геномный регион, в котором локализовался островок ROH на CHI12, испытывал давление отбора и, вероятно, может быть так или иначе связан с адаптациями к природно-климатическим условиям. Этот же островок ROH ( ∼ 33,9-36,5 Mbp) обнаружен у итальянских пород коз (4). Островок ROH на CHI18 был общим для европейских и азиатских коз и предположительно находился под давлением селекции на улучшение качества шерсти (29). Что касается островка ROH на CHI25, то F. Bertolini с соавт. (30) сообщили о сильном сигнале селекции в этом геномном регионе (34,6936,43 Mbp, содержит ген CUX1 ), данные были подтверждены тремя разными методами. Сигнал выявлен у двух пород, известных качеством шерстной и пуховой продукции, — ангорской и анкара, но не выявлен у кашемировой породы (30).
На CHI12 обнаружен островок ROH, немного различающийся по длине у разных групп животных (33,4-33,8 Mbp у ALTM и ALTW, 33,4-33,9 Mbp у DAGD и 33,2-33,9 Mbp у SOVM) и содержащий 13 общих генов у трех исследуемых пород. Гены GJB2 (gap junction protein beta 2) , GJB6 (gap junction protein beta 6) и ZMYM2 (zinc finger MYM-type protein 2) дополнительно входили в островок у DAGD и SOVM, при этом ген ZMYM5 (zinc finger MYM-type protein 5) был дополнительно идентифицирован у SOVM. В этом островке локализованы гены из общего семейства белков щелевых соединений GJA3 (gap junction protein beta 2), GJB2 и GJB6 . Гены GJB2 и GJB6 связаны с функционированием нервной системы, функциями слуха и эктодермальными процессами (31, 32). Интересно, что гены GJA3, ZMYM5 и ZMYM2 располагались в островках ROH у некоторых пород овец, разводимых в России (33). Кроме того, Y. Li с соавт. (34) предположили, что гены GJB6, SAP18 (Sin3A associated protein 18) и MICU2 (mitochondrial calcium uptake 2) могут быть вовлечены в регуляцию репродуктивной функции у овец, влияя на развитие яичников и эмбрионов, а также на спермиогенез (34).
Известно, что ген CRYL1 (crystallin lambda 1) участвует в развитии нервной системы и почек в эмбриогенезе у овец (35). Основываясь на данных высокопроизводительного секвенирования РНК, полученной из шерстяных волокон у тибетских кашемировых коз, X. Fu с соавт. (36) предположили, что ген IFT88 (intraflagellar transport 88) регулирует тонину кашемировых волокон. Кроме того, этот ген играет важную роль в процессах спермиогенеза (37) и находился под давлением отбора у пород овец, разводимых в России (38). Ген IL17D (interleukin 17D) участвует в формировании иммунного ответа организма против респираторных инфекций (39). Ген LATS2 (large tumor suppressor kinase 2) вовлечен в процессы пролиферации и дифференцировки клеток, изменения размера органов и регенерации тканей, а также играет важную роль в регуляции роста и развития мышц у сельскохозяйственных животных (40). Ген LATS2 влияет на секрецию молочного жира и связан с активацией лактогенеза у коз (40) и коров (41). Имеются сведения, что этот ген также может регулировать репродуктивные качества. Так, экспериментальные данные, полученные при изучении баранов китайской породы Hu, показали, что ген LATS2 влияет на процессы полового созревания самцов и капацитации (43), а у КРС участвует в поддержании нормального развития фолликулов (44). Ген MRPL57 (mitochondrial ribosomal protein L57) может быть вовлечен в регуляцию фолликулярной фазы эстрального цикла и сезонности размножения у овец (45).
На CHI18 был выявлен островок ROH (см. табл. 2) у ALTM и ALTW. Интересно, что L.F.M. Mota с соавт. (46) в результате поиска полногеномных ассоциаций с признаками, ассоциированными с эффективностью кормления у КРС, обнаружили геномный регион, содержащий гены PSMB10 , LCAT , SLC12A4 , DDX28 , DUS2 , NFATC3 , ESRP2 , PLA2G15 , SLC7A6 , SLC7A6OS , PRMT7 и SMPD3 , расположенные на BTA18 (46).
Следует отметить, что некоторые гены-кандидаты идентифицированы и в других исследованиях (46-50). Снижение экспрессии гена LCAT (lecithin-cholesterol acyltransferase) приводит к уменьшению образования липопротеинов высокой плотности, что обусловливает снижение способности транспортировать холестерин из жировой ткани в печень и мышцы (46).
Ген PLA2G15 (phospholipase A2 group XV) регулирует гидролиз фосфолипидов до свободных жирных кислот. Поэтому гены LCAT и PLA2G15 — одни из ключевых кандидатов, связанных с липидным метаболизмом у сельскохозяйственных животных (47, 48). Транскриптомный анализ показал, что ген SMPD3 (sphingomyelin phosphodiesterase 3) регулирует метаболизм липидов (в частности, сфинголипидов) и косвенно влияет на эффективность потребления корма у КРС (49). Согласно результатам анализа в DAVID, у коз этот ген вовлечен в регуляцию различных функций, включая развитие хондроцитов, участвующих в морфогенезе кости, регуляцию формирования хрящей и минерализации костей, метаболизм коллагенов, транспорт полисахаридов и секрецию пептидных гормонов.
Что касается других генов из этого региона, то некоторые из них также были успешно аннотированы. Например, ген NFATC3 (nuclear factor of activated T cells 3) влияет на морфометрию сердца у плода у овец (50). Ген-ортолог у человека вовлечен в регуляцию функционирования иммунной системы (51). Высказываются предположения, что ген DDX28 (DEADbox helicase 28) связан с реакцией организма млекопитающих на гипоксию (52), а ген SLC12A4 (solute carrier family 12 member 4) — с развитием мышечных волокон (53).
Гены PRKRIP , SH2B2 , CUX1 и POLR2J располагались в островке ROH у ALTM и ALTW на CHI25. Согласно результатам функциональной аннотации, ген SH2B2 (SH2B adaptor protein 2) вовлечен в следующие метаболические пути: сигнальный путь рецептора инсулина (GO:0008286), регуляция метаболического процесса (GO:0019222), организация актинового цитоскелета (GO:0030036), дифференцировка бурых жировых клеток (GO:0050873). Ген SH2B2 влияет на энергетический метаболизм и массу тела посредством усиления сигналов лептина — гормона, контролирующего массу тела и аппетит (54). Исследования нескольких научных коллективов показали, что этот ген может влиять на рост и, следовательно, на размеры туловища сельскохозяйственных животных. Так, M. Yang с соавт. (55) идентифицировали индел-му-тацию размером 4 п.н. и три SNPs в гене SH2B2 , ассоциированных с ростом, у пяти китайских пород КРС. Авторы предложили ген SH2B2 в качестве соответствующего кандидата (55). Три SNPs (g.20570G > A, g.20693T > C и g.24070C > A) в гене SH2B2 были достоверно ассоциированы с длиной туловища, длиной крупа и окружностью груди у циньчуаньского (Qinchuan) крупного рогатого скота (54). Ген CUX1 (CUT-like homeobox 1) — это ключевой регулятор развития волосяных фолликулов у овец (56-58). Вероятно, этот же ген может быть вовлечен в формирование шерстного покрова у ALTM и ALTW. Онтология гена POLR2J (RNA polymerase II subunit J) у коз или других сельскохозяйственных животных еще не исследована. Однако имеются сведения, что ген-ортолог у человека связан с функциями иммунной системы и предложен в качестве кандидата, ассоциированного с устойчивостью к респираторным инфекциям (59).
Итак, наше исследование представляет собой одну из первых попыток поиска следов селекции в геномах российских популяций домашних коз. С помощью детекции островков ROH мы выявили целевые регионы на трех хромосомах CHI12, CHI18 и CHI25 у представителей горноалтайской, алтайской белой пуховой, дагестанской пуховой и советской шерстной пород. Наличие у домашних коз этих регионов, испытывающих давление селекции, подтверждалось ранее другими авторами на большом поголовье коз, обитающих в различных географических регионах. Выявление этих геномных участков у исследуемых нами групп соответствует тенденциям, отмеченным другими исследователями, и согласуется с происхождением анализируемых пород коз. Среди выявленных генов были кандидаты, связанные с шерстной продуктивностью (CUX1, IFT88), регулирующие различные метаболические процессы (LCAT, PLA2G15, SMPD3), репродуктивные функции (GJB6, SAP18, MICU2, LATS2, MRPL57), рост и развитие (SLC12A4, SH2B2, GJB2, GJB6), эмбриогенез (CRYL1, NFATC3), а также вовлеченные в формирование ответа на неблагоприятные внешние факторы (на инфекции — IL17D, POLR2J, на гипоксию — DDX28). Полученные нами результаты расширяют понимание геномной архитектуры российских локальных пород и популяций коз. Мы продолжим поиск признаков отбора в геноме коз с использованием других методических подходов.
Список литературы Поиск следов селекции в геномах домашних коз (Capra hircus L.), разводимых в России, с помощью идентификации островков гомозиготности
- Новопашина С.И., Санников М.Ю., Хататаев С.А., Кузьмина Т.Н., Хмелевская Г.Н., Степанова Н.Г., Тихомиров А.И., Маринченко Т.Е. Состояние и перспективные направления улучшения генетического потенциала мелкого рогатого скота. Научный аналитический обзор. М., 2019.
- Ежегодник по племенной работе в овцеводстве и козоводстве в хозяйствах Российской Федерации (2022 год) /Под ред. Г.И. Шичкина, Г.Ф. Сафиной, Х.А. Амерханова, В.В. Чернова, Л.Н. Григорян, Г.Н. Хмелевской, А.В. Равичевой, Н.Г. Степанова. М., 2023.
- Селионова М.И., Трухачев В.И., Айбазов А-М.М., Столповский Ю.А., Зиновьева Н.А. Генетические маркеры в козоводстве (обзор). Сельскохозяйственная биология, 2021, 56(6): 1031-1048 (doi: 10.15389/agrobiology.2021.6.1031rus).
- Dadousis C., Cecchi F., Ablondi M., Fabbri M.C., Stella A., Bozzi R. Keep Garfagnina alive. An integrated study on patterns of homozygosity, genomic inbreeding, admixture and breed traceability of the Italian Garfagnina goat breed. PLoS ONE, 2021, 16(1): e0232436 (doi: 10.1371/journal.pone.0232436).
- Gorssen W., Meyermans R., Janssens S., Buys N. A publicly available repository of ROH islands reveals signatures of selection in different livestock and pet species. Genetics, Selection, Evolution, 2021, 53(1): 2 (doi: 10.1186/s12711-020-00599-7).
- Bertolini F., Cardoso T.F., Marras G., Nicolazzi E.L., Rothschild M.F., Amills M., AdaptMap consortium. Genome-wide patterns of homozygosity provide clues about the population history and adaptation of goats. Genetics, Selection, Evolution, 2018, 50(1): 59 (doi: 10.1186/s12711-018-0424-8).
- Zinovieva N.A., Dotsev A.V., Sermyagin A.A., Deniskova T.E., Abdelmanova A.S., Kharzinova V.R., Sölkner J., Reyer H., Wimmers K., Brem G. Selection signatures in two oldest Russian native cattle breeds revealed using high-density single nucleotide polymorphism analysis. PLoS ONE, 2020, 15(11): e0242200 (doi: 10.1371/journal.pone.0242200).
- Addo S., Jung L. An insight into the runs of homozygosity distribution and breed differentiation in Mangalitsa pigs. Frontiers in Genetics, 2022, 13: 909986 (doi: 10.3389/fgene.2022.909986).
- Mészárosová M., Mészáros G., Moravčíková N., Pavlík I., Margetín M., Kasarda R. Within- and between-breed selection signatures in the original and improved Valachian sheep. Animals, 2022, 12(11): 1346 (doi: 10.3390/ani12111346).
- Szmatoła T., Gurgul A., Jasielczuk I., Oclon E., Ropka-Molik K., Stefaniuk-Szmukier M., Polak G., Tomczyk-Wrona I., Bugno-Poniewierska M. Assessment and distribution of runs of homozygosity in horse breeds representing different utility types. Animals, 2022, 12(23): 3293 (doi: 10.3390/ani12233293).
- Ballan M., Schiavo G., Bovo S., Schiavitto M., Negrini R., Frabetti A., Fornasini D., Fontanesi L. Comparative analysis of genomic inbreeding parameters and runs of homozygosity islands in several fancy and meat rabbit breeds. Animal Genetics, 2022, 53(6): 849-862 (doi: 10.1111/age.13264).
- Fedorova E.S., Dementieva N.V., Shcherbakov Y.S., Stanishevskaya O.I. Identification of key candidate genes in runs of homozygosity of the genome of two chicken breeds, associated with cold adaptation. Biology, 2022, 11(4): 547 (doi: 10.3390/biology11040547).
- Signer-Hasler H., Henkel J., Bangerter E., Bulut Z., VarGoats Consortium, Drögemüller C., Leeb T., Flury C. Runs of homozygosity in Swiss goats reveal genetic changes associated with domestication and modern selection. Genetics, Selection, Evolution, 2022, 54(1): 6 (doi: 10.1186/s12711-022-00695-w).
- Sun X., Guo J., Li L., Zhong T., Wang L., Zhan S., Lu J., Wang D., Dai D., Liu G.E., Zhang H. Genetic diversity and selection signatures in Jianchang Black goats revealed by whole-genome sequencing data. Animals, 2022, 12(18): 2365 (doi: 10.3390/ani12182365).
- Senczuk G., Macrì M., Di Civita M., Mastrangelo S., Del Rosario Fresno M., Capote J., Pilla F., Delgado J.V., Amills M., Martínez A. The demographic history and adaptation of Canarian goat breeds to environmental conditions through the use of genome-wide SNP data. Genetics, Selection, Evolution, 2024, 56(1): 2 (doi: 10.1186/s12711-023-00869-0).
- Ziegler T.E., Molina A., Ramón M., Sanchez M., Muñoz-Mejías E., Antonini A., Demyda-Peyrás S. Analysis of the genomic landscape of inbreeding in two divergent groups of Spanish Florida goats. Journal of Animal Breeding and Genetics = Zeitschrift fur Tierzuchtung und Zuchtungsbiologie, 2023, 140(3): 316-329 (doi: 10.1111/jbg.12759).
- Deniskova T.E., Dotsev A.V., Selionova M.I., Reyer H., Sölkner J., Fornara M.S., Aybazov A.M., Wimmers K., Brem G., Zinovieva N.A. SNP-based genotyping provides insight into the west Asian origin of Russian local goats. Frontiers in Genetics, 2021, 12: 708740 (doi: 10.3389/fgene.2021.708740).
- Денискова Т.Е., Доцев А.В., Форнара М.С., Сермягин А.А., Reyer H., Wimmers К., Brem G., Зиновьева Н.А. Геномная архитектура российской популяции зааненских коз в аспекте генофонда породы из пяти стран мира. Сельскохозяйственная биология, 2020, 55(2): 285-294 (doi: 10.15389/agrobiology.2020.2.285rus).
- International Goat Genome Consortium (IGGC).GoatGenome2013. Режим доступа: http://www.goatgenome.org. Без даты.
- Tosser-Klopp G., Bardou P., Bouchez O., Cabau C., Crooijmans R., Dong Y., Donnadieu-Tonon C., Eggen A., Heuven H.C., Jamli S., Jiken A.J., Klopp C., Lawley C.T., McEwan J., Martin P., Moreno C.R., Mulsant P., Nabihoudine I., Pailhoux E., Palhière I., Rupp R., Sarry J., Sayre B.L., Tircazes A., Jun W., Wang W., Zhang W., International Goat Genome Consortium. Design and characterization of a 52K SNP chip for goats. PLoS ONE, 2014, 9(1): e86227 (doi: 10.1371/journal.pone.0086227).
- Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies. Molecular Biology and Evolution, 2006, 23(2): 254-267 (doi: 10.1093/molbev/msj030).
- Marras G., Gaspa G., Sorbolini S., Dimauro C., Ajmone-Marsan P., Valentini A., Williams J.L., Macciotta N.P. Analysis of runs of homozygosity and their relationship with inbreeding in five cattle breeds farmed in Italy. Animal Genetics, 2015, 46(2): 110-121 (doi: 10.1111/age.12259).
- detectRUNS: Detect Runs of Homozygosity and Runs of Heterozygosity in Diploid Genomes. Rpackageversion 0.9.5. Режим доступа: https://cran.r-project.org/web/packages/detectRUNS/vignet-tes/detectRUNS.vignette.html. Без даты.
- Huang D., Sherman B., Lempicki R. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols, 2009, 4(1): 44-57 (doi: 10.1038/nprot.2008.211).
- Sherman B.T., Hao M., Qiu J., Jiao X., Baseler M.W., Lane H.C., Imamichi T., Chang W. DAVID: a web server for functional enrichment analysis and functional annotation of gene lists (2021 update). Nucleic Acids Research, 2022, 50(W1): W216-W221 (doi: 10.1093/nar/gkac194).
- Каргачакова Т.Б., Чикалев А.И. Алтайские белые пуховые козы. Овцы, козы, шерстяное дело, 2016, 2: 9-10.
- Мусалаев Х.Х., Палаганова Г.А., Абдуллабеков Р.А. Совершенствование продуктивных качеств помесных молочных коз. Овцы, козы, шерстяное дело, 2016, 2: 10-11.
- Мусалаев Х.Х. Результаты скрещивания грубошерстных коз с козлами советской шерстной породы в условиях внутригорного Дагестана. Автореф. канд. дис. Орджоникидзе, 1972.
- Kim E.S., Elbeltagy A.R., Aboul-Naga A.M., Rischkowsky B., Sayre B., Mwacharo J.M., Rothschild M.F. Multiple genomic signatures of selection in goats and sheep indigenous to a hot arid environment. Heredity, 2016, 116(3): 255-264 (doi: 10.1038/hdy.2015.94).
- Bertolini F., Servin B., Talenti A., Rochat E., Kim E.S., Oget C., Palhière I., Crisà A., Catillo G., Steri R., Amills M., Colli L., Marras G., Milanesi M., Nicolazzi E, Rosen B.D., Van Tassell C.P., Guldbrandtsen B., Sonstegard T.S., Tosser-Klopp G., Stella A., Rothschild M.F., Joost S., Crepaldi P., AdaptMap consortium. Signatures of selection and environmental adaptation across the goat genome post-domestication. Genetics, Selection, Evolution, 2018, 50(1): 57 (doi: 10.1186/s12711-018-0421-y).
- Lamartine J., Munhoz Essenfelder G., Kibar Z., Lanneluc I., Callouet E., Laoudj D., Lemaître G., Hand C., Hayflick S.J., Zonana J., Antonarakis S., Radhakrishna U., Kelsell D.P., Christianson A.L., Pitaval A., Der Kaloustian V., Fraser C., Blanchet-Bardon C., Rouleau G.A., Waksman G. Mutations in GJB6 cause hidrotic ectodermal dysplasia. Nature Genetics, 2000, 26(2): 142-144 (doi: 10.1038/79851).
- Ortolano S., Di Pasquale G., Crispino G., Anselmi F., Mammano F., Chiorini J.A. Coordinated control of connexin 26 and connexin 30 at the regulatory and functional level in the inner ear. Proceedings of the National Academy of Sciences, 2008, 105(48): 18776-18781 (doi: 10.1073/pnas.0800831105).
- Денискова Т.Е., Доцев А.В., Зиновьева Н.А. Идентификация генов-кандидатов, ассоциированных с экономически значимыми признаками, на основе анализа островков гомозиготности в геноме пород овец, разводимых в России. ДостижениянаукиитехникиАПК, 2023, 37(9): 80-86.
- Li Y., Chen Z., Fang Y., Cao C., Zhang Z., Pan Y., Wang Q. Runs of homozygosity revealed reproductive traits of Hu sheep. Genes, 2022, 13(10): 1848 (doi: 10.3390/genes13101848).
- Wang W., Chen S., Qiao L., Zhang S., Liu Q., Yang K., Pan Y., Liu J., Liu W. Four markers useful for the distinction of intrauterine growth restriction in sheep. Animals, 2023, 13(21): 3305 (doi: 10.3390/ani13213305).
- Fu X., Zhao B., Tian K., Wu Y., Suo L., Ba G., Ciren D., De J., Awang C., Gun S., Yang B. Integrated analysis of lncRNA and mRNA reveals novel insights into cashmere fineness in Tibetan cashmere goats. PeerJ, 2020, 8: e10217 (doi: 10.7717/peerj.10217).
- San Agustin J.T., Pazour G.J., Witman G.B. Intraflagellar transport is essential for mammalian spermiogenesis but is absent in mature sperm. Molecular Biology of the Cell, 2015, 26(24): 4358-4372 (doi: 10.1091/mbc.E15-08-0578).
- Yurchenko A.A., Deniskova T.E., Yudin N.S., Dotsev A.V., Khamiruev T.N., Selionova M.I., Egorov S.V., Reyer H., Wimmers K., Brem G., Zinovieva N.A., Larkin D.M. High-density genotyping reveals signatures of selection related to acclimation and economically important traits in 15 local sheep breeds from Russia. BMC Genomics, 2019, 20(Suppl 3): 294 (doi: 10.1186/s12864-019-5537-0).
- Elder L.A., Hinnant H.R., Mandella C.M., Claus-Walker R.A., Parrish L.M., Slanzon G.S., McConnel C.S. Differential gene expression in peripheral leukocytes of pre-weaned Holstein heifer calves with respiratory disease. PLoS ONE, 2023, 18(5): e0285876 (doi: 10.1371/journal.pone.0285876).
- Zhang J., Raza S.H.A., Wei D., Yaping S., Chao J., Jin W., Almohaimeed H.M., Batarfi M.A., Assiri R., Aggad W.S., Ghalib S.H., Ageeli A.A. Roles of MEF2A and MyoG in the transcriptional regulation of bovine LATS2 gene. Research in Veterinary Science, 2022, 152: 417-426 (doi: 10.1016/j.rvsc.2022.08.030).
- Chen Z., Luo J., Zhang C., Ma Y., Sun S., Zhang T., Loor J.J. Mechanism of prolactin inhibition of miR-135b via methylation in goat mammary epithelial cells. Journal of Cellular Physiology, 2018, 233(1): 651-662 (doi: 10.1002/jcp.25925).
- Chen Z., Chu S., Liang Y., Xu T., Sun Y., Li M., Zhang H., Wang X., Mao Y., Loor J.J., Wu Y., Yang Z. miR-497 regulates fatty acid synthesis via LATS2 in bovine mammary epithelial cells. Food & Function, 2020, 11(10): 8625-8636 (doi: 10.1039/d0fo00952k).
- Zhang G.-M., Zhang T.-T., An S.-Y., El-Samahy M.-A., Yang H., Wan Y.-J., Meng F.-X., Xiao S.-H., Wang F., Lei Z.-H. Expression of Hippo signaling pathway components in Hu sheep male reproductive tract and spermatozoa. Theriogenology, 2019, 126: 239-248 (doi: 10.1016/j.theriogenology.2018.12.029).
- Plewes M.R., Hou X., Zhang P., Liang A., Hua G., Wood J.R., Cupp A.S., Lv X., Wang C., Davis J.S. Yes-associated protein 1 is required for proliferation and function of bovine granulosa cells in vitro. Biology of Reproduction, 2019, 101(5): 1001-1017 (doi: 10.1093/biolre/ioz139).
- Lakhssassi K., Ureña I., Marín B., Sarto M.P., Lahoz B., Alabart J.L., Calvo J.H., Serrano M. Characterization of the pars tuberalis and hypothalamus transcriptome in female sheep under different reproductive stages. Animal Biotechnology, 2023, 34(8): 3461-3474 (doi: 10.1080/10495398.2022.2155174).
- Mota L.F.M., Santos S.W.B., Júnior G.A.F., Bresolin T., Mercadante M.E.Z., Silva J.A.V., Cyrillo J.N.S.G., Monteiro F.M., Carvalheiro R., Albuquerque L.G. Meta-analysis across Nellore cattle populations identifies common metabolic mechanisms that regulate feed efficiency-related traits. BMC Genomics, 2022, 23(1): 424 (doi: 10.1186/s12864-022-08671-w).
- Pena R.N., Noguera J.L., Casellas J., Díaz I., Fernández A.I., Folch J.M., Ibáñez-Escriche N. Transcriptional analysis of intramuscular fatty acid composition in the longissimus thoracis muscle of Iberian × Landrace back-crossed pigs. Animal Genetics,2013, 44(6): 648-660 (doi: 10.1111/age.12066).
- Yang C., Huang Z., Pan C., Wang S. Characterization of feed efficiency-related key signatures molecular in different cattle breeds. PLoS ONE, 2023, 18(9): e0289939 (doi: 10.1371/journal.pone.0289939).
- Yu H., Raza S.H.A., Pan Y., Cheng G., Mei C., Zan L. Integrative analysis of blood transcriptomics and metabolomics reveals molecular regulation of backfat thickness in Qinchuan cattle. Animals, 2023, 13(6): 1060 (doi: 10.3390/ani13061060).
- Fan X., Turdi S., Ford S.P., Hua Y., Nijland M.J., Zhu M., Nathanielsz P.W., Ren J. Influence of gestational overfeeding on cardiac morphometry and hypertrophic protein markers in fetal sheep. The Journal of Nutritional Biochemistry, 2011, 22(1): 30-37 (doi: 10.1016/j.jnutbio.2009.11.006).
- Song N., Wang X., Gui L., Raza S.H.A., Luoreng Z., Zan L. MicroRNA-214 regulates immunity-related genes in bovine mammary epithelial cells by targeting NFATc3 and TRAF3. Molecular and Cellular Probes, 2017, 35: 27-33 (doi: 10.1016/j.mcp.2017.06.002).
- Evagelou S.L., Bebenek O., Specker E.J., Uniacke J. DEAD box protein family member DDX28 is a negative regulator of hypoxia-inducible factor 2a- and eukaryotic initiation factor 4e2-directed hypoxic translation. Molecular and Cellular Biology, 2020, 40(6): e00610-19 (doi: 10.1128/MCB.00610-19).
- Li Y., Xu Z., Li H., Xiong Y., Zuo B. Differential transcriptional analysis between red and white skeletal muscle of Chinese Meishan pigs. International Journal of Biological Sciences, 2010, 6(4): 350-360 (doi: 10.7150/ijbs.6.350).
- Raza S.H.A., Khan R., Gui L., Schreurs N.M., Wang X., Mei C., Yang X., Gong C., Zan L. Bioinformatics analysis and genetic polymorphisms in genomic region of the bovine SH2B2 gene and their associations with molecular breeding for body size traits in Qinchuan beef cattle. Bioscience Reports, 2020, 40(3): BSR20192113 (doi: 10.1042/BSR20192113).
- Yang M., Fu J., Lan X., Sun Y., Lei C., Zhang C., Chen H. Effect of genetic variations within the SH2B2 gene on the growth of Chinese cattle. Gene, 2013, 528(2): 314-319 (doi: 10.1016/j.gene.2013.06.086).
- Hu T., Huang S., Lv X., Wang S., Getachew T., Mwacharo J.M., Haile A., Sun W. miR-143 targeting CUX1 to regulate proliferation of dermal papilla cells in Hu sheep. Genes, 2021, 12(12): 2017 (doi: 10.3390/genes12122017).
- Lv X., Li Y., Chen W., Wang S., Cao X., Yuan Z., Getachew T., Mwacharo J., Haile A., Li Y., Sun W. Association between DNA methylation in the core promoter region of the CUT-like homeobox 1 (CUX1) gene and lambskin pattern in Hu sheep. Genes, 2023, 14(10): 1873 (doi: 10.3390/genes14101873).
- Zhou H., Huang S., Lv X., Wang S., Cao X., Yuan Z., Getachew T., Mwacharo J.M., Haile A., Quan K., Li Y., Reverter A., Sun W. Effect of CUX1 on the proliferation of Hu sheep dermal papilla cells and on the Wnt/β-Catenin signaling pathway. Genes, 2023, 14(2): 423 (doi: 10.3390/genes14020423).
- Zhu X., Zou Y., Jia L., Ye X., Zou Y., Tu J., Li J., Yu R., Yang S., Huang P. Using multi-tissue transcriptome-wide association study to identify candidate susceptibility genes for respiratory infectious diseases. Frontiers in Genetics, 2023, 14: 1164274 (doi: 10.3389/fgene.2023.1164274).


