Полногеномный анализ ассоциаций с продуктивными и репродуктивными признаками у молочного скота в российской популяции голштинской породы
Автор: Сермягин А.А., Гладырь Елена Александровна, Харитонов Сергей Николаевич, Ермилов А.Н., Стрекозов Н.И., Брем Г., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Геномная селекция
Статья в выпуске: 2 т.51, 2016 года.
Бесплатный доступ
Полногеномный анализ ассоциаций (whole-genome associated study, GWAS) создавался как мощный инструмент для идентификации геномных вариаций, связанных с экономически важными признаками у различных видов сельскохозяйственных животных. Развитие методов геномной селекции открывает новые возможности для улучшения продуктивных и воспроизводительных качеств домашнего скота. Целью настоящих исследований было изучение полногеномных ассоциаций полиморфизмов единичных нуклеотидов (SNP) с племенной ценностью голштинских быков-производителей по признакам молочной продуктивности и воспроизводства. Скрининг SNP проводили с помощью Illumina BovineSNP50K v2 BeadChip у 195 быков-производителей, проверенных по качеству потомства, и 61 молодых быков, оцененных по родословной. Сбор и анализ информации по хозяйственно полезным качествам дочерей быков осуществляли на основе баз данных племенного учета в 77 хозяйствах Московской области. Племенную ценность (EBV) определяли по показателям молочной продуктивности и воспроизводительным качествам: удой за 305 сут лактации (MY), массовая доля жира (FC) и белка (PC), количество молочного жира (FY) и белка (PY), возраст первого отела (CA), трудность отела (CD), кратность осеменений (CR), продолжительность сервис-периода (DO) и стельности (GL), межотельный период (CI). Расчет EBV по каждому из признаков проводили по методологии BLUP Sire Model. Общее число SNP, отобранных по результатам контроля качества, составило 41370. Для расчета прямой геномной племенной ценности (DGV) по методу GBLUP выполняли построение геномной матрицы родства (G). Показатели комбинированной геномной племенной ценности (GEBV) получали на основе значений DGV и EBV. Для повышения достоверности данных GWAS и мощности поиска ассоциаций в целом в анализе использовали геномные оценки племенной ценности как для проверенных ранее, так и для молодых быков-производителей. Тестирование нулевых гипотез значимости ассоциаций осуществляли по критериям Бонферрони и ожидаемой доле ложных отклонений (LocFdr). Значения коэффициентов наследуемости для признаков воспроизводства колебались от 0,035 для CR до 0,221 для CA, тогда как для показателей молочной продуктивности они достигали более высоких значений - от 0,250 для FC до 0,401 для PC. На основании результатов тестов по Бонферрони и LocFdr было обнаружено несколько высокозначимых SNP для признаков молочной продуктивности и воспроизводства. Для MY установлены достоверные ассоциации ARS-BFGL-NGS-50172 на BTA17 и Hapmap54246-rs29017970 на BTA13. Для FC были картированы консервативные локусы на BTA-104917-no-rs и BTB-01604502 (58 Mb, BTA9), ARS-BFGL-NGS-107379 и ARS-BFGL-NGS-4939 (1,8-2,0 Mb, BTA14), для PC - Hapmap 43278-BTA-50082 (BTA20). Полиморфизмы ARS-BFGL-BAC-7205 (BTA1) и Hapmap48395-BTA-58382 (BTA5) оказались ассоциированы с PY. Получены данные по точной локализации мутаций для признаков репродукции: BTB-01622929 на BTA1 для CA, ARS-BFGL-NGS-89711 на BTA27 для CR, ARS-BFGL-NGS-117881 на BTA5 для DO, BTA-31636-no-rs на BTA1 и Hapmap26774-BTA-163037 на BTA27 для CI. Аддитивный эффект по ряду показателей превышал 9,0 %. В геноме отмечали референтные мутации с высокой степенью значимости в генах TRAFD1, LOC786966, DGAT1, SLC16A7, DUSP26 и CCDC58. Показано, что использование полногеномного анализа позволяет с высокой степенью точности картировать QTL признаков продуктивности и воспроизводства с низкой и умеренной наследуемостью.
Полногеномный анализ, наследуемость, молочная продуктивность, воспроизводительные качества
Короткий адрес: https://sciup.org/142213928
IDR: 142213928 | УДК: 636.2:636.082.12:577.21 | DOI: 10.15389/agrobiology.2016.2.182rus
Текст научной статьи Полногеномный анализ ассоциаций с продуктивными и репродуктивными признаками у молочного скота в российской популяции голштинской породы
В традиционных программах селекции изучение продуктивных качеств у сельскохозяйственных животных ведется без идентификации влияния каких-либо функциональных генов. При этом в последнюю четверть
∗ Исследования выполнены при поддержке государства в лице Минобрнауки России (уникальный идентификационный номер проекта RFMEFI60414X0062).
века развитие молекулярно-генетических методов идет опережающими темпами: от определения отдельных генов, контролирующих единичные физиологические процессы, до локусов количественных признаков (quantitative trait loci, QTL) и мононуклеотидных замен (single nucleotide polymorphism, SNP), маркирующих комплекс продуктивных качеств животного (1-3). Установлено, что величина влияния единичного гена, отвечающего за количественные и качественные признаки у животного или группы крови, имеет невысокую долю генетической изменчивости, обусловливающую фенотипическое проявление признака (4, 5). В связи с этим отбор животных не гарантирует точного наследования продуктивного показателя в поколениях. Использование высокополиморфных маркеров — микросателлитов (short tandem repeats, STR) также не дает желаемого эффекта картирования QTL, поскольку, даже несмотря на возможное наличие у животного групп сибсов и полусибсов, анализ сцепления QTL и STR на хромосомах проводится в интервале 20 cM или выше, что делает сложным определение основных мутаций (6).
Полногеномный анализ ассоциаций (genome-wide association study, GWAS) представляет собой развитие метода маркер-зависимой селекции (marker-assisted selection, MAS), когда используемые генетические маркеры распределены по всему геному так, что все QTL находятся в неравновесном сцеплении (linkage disequilibrium, LD) по меньшей мере с одним из них. Исследования MAS сфокусированы на точном картировании некоторых QTL при условии, что ген, входящий в QTL, может быть идентифицирован. В свою очередь, эффективность GWAS зависит от числа SNP. Если SNP расположены на хромосоме слишком далеко друг от друга, то QTL не может быть в достаточном неравновесном сцеплении хотя бы с одним из маркеров и, как следствие, локус не будет найден. Увеличение плотности покрытия генома по SNP позволяет повысить вероятность определения QTL и в значительной степени — точность картирования (7, 8).
Так, у крупного рогатого скота на уровне пород найдены достоверные ассоциации с хозяйственными признаками при использовании 10000 SNP, но по оценке M.E. Goddard (6), для межпородного анализа у вида Bos taurus необходимы 300000 SNP. Число SNP около 50000 может быть вполне достаточным для работы с одной породой, например голштинской, однако для более точного картирования QTL и повышения достоверности геномных оценок племенной ценности преимущество отдается выборкам по нескольким породам и SNP-панелям высокой плотности (9-11).
Число животных, необходимое для изучения ассоциаций, зависит от степени влияния, которая выражается в доле изменчивости признака, раскрываемой точечными мутациями. Этот параметр сочетает в себе частоту аллеля и среднюю разность между SNP генотипами по количественному признаку. На практике некоторые SNP составляют более 4 % генетической дисперсии и выше, что на малой выборке служит достаточной величиной прогноза даже для низконаследуемых признаков (12-14).
Использование метода GWAS позволяет эффективно вести поиск значимых полиморфизмов как среди пород скота, так и внутри разных популяций животных голштинского происхождения (15-17). При этом для хозяйственно ценных признаков проводится картирование и для бычьего, и для маточного поголовья, связанное в первую очередь с поиском достоверных связей по продукции молочного жира и белка (18, 19).
Установлены консервативные регионы генома скота, имеющие значимые ассоциации с признаками молочной продуктивности: казеиновый кластер (CSN), диацилглицерол-О-ацилтрансфераза (DGAT1), рецеп- тор гормона роста (GHR) и другие, а также ряд общепопуляционных мутаций, не описанных ранее (20-22). Изучение сопряженности по признакам с низкой наследуемостью позволяет повысить результативность селекции за счет детекции или элиминации генов, отвечающих за здоровье и воспроизводство животных, и закреплять ценные генотипы в популяции (23-27). Также представляют интерес исследования по поиску локусов, отвечающих за качественные признаки животных (28).
Необходимо отметить, что оценка влияния каждого маркера при расчете прогноза геномной племенной ценности или при ассоциативном анализе происходит одновременно с другими маркерами. Каждая мутация несет определенную долю компоненты генетической изменчивости, при этом допускается, что при влиянии множества SNP сумма всех эффектов в среднем должна быть мала. Это обстоятельство, однако, не препятствует получению ясной картины ассоциаций по изучаемым показателям (29-32).
Поиск и изучение ассоциаций по точечным генетическим мутациям с хозяйственно полезными признаками на голштинской популяции молочного скота в России ранее не проводили. Выполняя GWAS анализ, мы сочетали выборки по ранее проверенным и молодым быкам, чтобы подтвердить эффективность и возможность применения моделей прогноза геномной племенной ценности для селекции молочного скота голштинского происхождения. Эти первые результаты, полученные по полногеномному анализу, станут основой для картирования локусов количественных признаков, ответственных за формирование продуктивных и воспроизводительных качеств животных.
Целью нашей работы было исследование полногеномных ассоциаций единичных нуклеотидов с показателями оценки племенной ценности быков-производителей голштинской породы по признакам молочной продуктивности и воспроизводства.
Методика. Сбор и анализ информации по хозяйственно полезным качествам дочерей быков голштинской породы осуществлялся на основе баз данных племенного учета в 77 хозяйствах Московской области (первичные базы данных были предоставлены Региональным информационноселекционным центром «Мосплеминформ»). С помощью биочипа Bovine SNP50K v2 BeadChip («Illumina Inc.», США) с плотностью 54609 SNP про-генотипировали 256 быков-производителей, принадлежащих ОАО «Головной центр по воспроизводству сельскохозяйственных животных» (пос. Быково, Московская обл.) и ОАО «Московское» по племенной работе (г. Ногинск, Московская обл.). Выборка включала 195 быков-производителей, проверенных по качеству потомства ( n = 47998), со средним числом дочерей-первотелок от каждого, равным 246 гол., и 61 молодого быка, оцененных по родословной (Parent Average, PA). У дочерей быков были изучены следующие фенотипические признаки: удой за 305 сут лактации (milk yield, MY), массовая доля жира (fat content, FC) и белка (protein content, PC), количество молочного жира (fat yield, FY) и белка (protein yield, PY), возраст первого отела (calving age, CA), легкость отела (calving easy, CE), кратность осеменений (conception rate, CR), продолжительность сервис-периода (days open, DO) и стельности (gestation length, GL), межотельный период (calving interval, CI).
Значения племенной ценности (Estimated Breeding Value, EBV) быков-производителей рассчитывались по методологии BLUP Sire Model, после чего полученные оценки дерегрессировались, то есть были скорректированы на влияние систематических средовых эффектов (drpEBV) (33).
Уравнение модели смешанного типа для оценки племенной ценно- сти быков было реализовано в среде языка программирования статистического пакета SAS IML (SAS University Edition) (34):
y i jk = μ + HYS i + b 1 А k + b 2 DO k + sire j + e ijk , [1] где yi j k — показатель признака k -й первотелки; μ — популяционная константа; HYSi — фиксированный эффект i -го «стада-года-сезона» отела; b1 и b2 — коэффициенты линейной регрессии; Аk — возраст первого отела k -й первотелки; DOk — продолжительность сервис-периода k -й первотелки; sire j — рандомизированный эффект j -го быка-производителя, имеющий нормальное распределение со средней, равной 0, и вариансой, равной A σ a2, где A — аддитивная матрица родства; ei j k — эффект неучтенных факторов.
Контроль качества генотипирования и регрессионный анализ по ассоциациям проводили с помощью программы Plink v. 1.07 (35). По его результатам было отобрано 41370 SNP. Ранее мы установили, что точность геномной оценки племенной ценности для молодых быков (Genomic Enhanced Breeding Value, GEBV) на 9-45 % выше по сравнению с оценкой по предкам (36). В связи с этим в анализе ассоциаций использовали значения GEBV как для молодых, так и прошедших проверку быков.
Расчет геномной матрицы родства (G) проводили по алгоритму, разработанному P.M. VanRaden (37), в среде языка программирования R (38). Матрица состояла из элементов — оценок гомозиготных и гетерозиготных локусов: AA = 1, AB = 0, BB = - 1. Коэффициенты наследуемости, генетические и паратипические вариансы были получены на основе двухфакторного иерархического дисперсионного комплекса. Оценки GEBV рассчитывались как комбинация прямого эффекта SNP-маркеров (Direct Genomic Value, DGV) и EBV (PA) согласно подходу GBLUP по следующей линейной модели (37):
DRP = Xb + Zu + e, [2]
где DRP — известный вектор псевдофенотипических данных drpEBV, рас- положенных на диагональной матрице W, с весовыми коэффициентами эффективного вклада информации по дочерям (effective daughter contribution): EDC = k ½ r2/(1 - r2) при вариационном отношении k = (4 - h2)/h2 (здесь r2 — достоверность оценки по признаку, h2 — наследуемость показателя); Xb — матрица, содержащая одну общую константу в модели; u — известный вектор прогнозов для DGV (G — геномная матрица родства), причем Z представляет собой единичную диагональную матрицу.
Система нормальных уравнений имеет следующий вид:
Х ′ WDRP Z ′ WDRP
Х′WX X′WZ b u
Z ′ WX Z ′ WX + kG - 1
Для подтверждения достоверного влияния SNP и определения значимых регионов в геноме скота были использованы несколько тестов для проверки нулевых гипотез: по Бонферрони (пороговое значение P < 1,2½10-6, 0,05/41370), по ожидаемой доле ложных отклонений в модификации B. Efron (Local False discovery rate, LocFdr), оценка которой была проведена методом максимального правдоподобия, реализованного в пакете locfdr на языке R (39), и по показателю Fdr согласно подходу Y. Benjamini и Y. Hochberg (40) с пороговым значением эмпирических оценок P < 0,05.
Для поиска генов, тесно ассоциированных с хозяйственными признаками, использовали базу данных National Center for Biotechnology Information (NCBI). Функциональную идентификацию генов проводили по базе данных Discover EggNOG 4.1 . Визуализацию данных осуществляли в пакете qqman с помощью языка программирования R (41).
Результаты. Полученные коэффициенты наследуемости (h2) по признакам молочной продуктивности и воспроизводства свидетельствовали о возможности проводить селекцию молочного скота в популяции в разных направлениях. Так, для улучшения признаков воспроизводства и фертильности, имеющих низкие h2 в пределах от 0,035±0,005 до 0,068±0,007, требуется сокращение генерационного интервала или(и) повышение интенсивности отбора животных. В тоже время по признакам молочной продуктивности, отличающимся умеренной наследуемостью, возможно прогнозировать более высокий селекционный ответ (табл. 1).
Коэффициенты генетической изменчивости отличались константностью для признаков FC, PC, CA, GL и CI. Вне зависимости от характера наследования хозяйственных качеств, по признакам FY, PY, DO, CR и CE наблюдалось повышение коэффициента вариации, что было связано со значимой долей влияния паратипических факторов.
По результатам анализа признаков молочной продуктивности установлены значимые ассоциативные связи с EBV быков-производителей. Согласно критерию Fdr с пороговым значением P < 0,05, для MY обнаружено 124 SNP, тогда как тесты по LocFdr и Бонферрони показали всего 2 SNP с высокими значениями достоверности (P < 1,2½10-6) — ARS-BFGL-NGS-50172 ( Bos taurus autosome , BTA17) и Hapmap54246-rs29017970 (BTA13) (рис. 1). По FC и PC установлены консервативные локусы (P = 1,2½10-6-4,1½10-8) BTA-104917-no-rs и BTB-01604502 (58 Mb, BTA9), ARS-BFGL-NGS-107379 и ARS-BFGL-NGS-4939 (1,8-2,0 Mb, BTA14), Hapmap 43278-BTA-50082 (BTA20), при этом максимально возможное число значимых точечных мутаций, согласно критерию Fdr, составляло от 3 до 13.
1. Показатели наследуемости и изменчивости по признакам молочной продуктивности и воспроизводства в популяции скота голштинской породы (Московская обл.)
|
Показатель |
\ h2 ±m h2 |
\ σ∩ P |
] Cv P 1 |
σ A |
1 Cv A 1 |
σ U |
1 Cv U |
|
Удой за 305 сут лактации, кг |
0,281±0,014 |
1317 |
21 |
697 |
11 |
986 |
16 |
|
Массовая доля жира, % |
0,250±0,013 |
0,37 |
9 |
0,19 |
5 |
0,27 |
7 |
|
Молочный жир, кг |
0,328±0,015 |
57,8 |
23 |
33,1 |
13 |
39,6 |
16 |
|
Массовая доля белка, % |
0,401±0,016 |
0,19 |
6 |
0,12 |
4 |
0,13 |
4 |
|
Молочный белок, кг |
0,370±0,016 |
43,8 |
22 |
26,6 |
13 |
29,6 |
15 |
|
Возраст первого отела, мес |
0,221±0,012 |
3,8 |
14 |
1,8 |
6 |
2,9 |
11 |
|
Трудность отела, балл |
0,068±0,007 |
1,03 |
242 |
0,27 |
63 |
0,89 |
210 |
|
Кратность осеменений, ед. |
0,035±0,005 |
1,27 |
63 |
0,24 |
12 |
1,23 |
61 |
|
Сервис-период, сут |
0,059±0,006 |
86,5 |
56 |
20,9 |
14 |
83,4 |
54 |
|
Продолжительность стельности, сут |
0,036±0,005 |
12,7 |
5 |
2,4 |
1 |
12,5 |
5 |
|
Межотельный период, сут |
0,063±0,007 |
82,4 |
19 |
20,6 |
5 |
79,2 |
19 |
П р и м еч а ни е. h2 — коэффициент наследуемости, mh2 — ошибка коэффициента наследуемости, σ P, σ A и σ U — соответственно оценки фенотипического, генетического и паратипического стандартных отклонений; Cv — коэффициент изменчивости, %.
По FY не выявлено каких-либо ассоциаций, тогда как для PY получены 48 SNP, расположенных преимущественно на 5-й и 13-й аутосомах (Fdr, P < 0,05). Однако, согласно критериям, рассчитанным по Бонферро-ни и LocFdr, число высокозначимых полиморфизмов снизилось до 4: ARS-BFGL-BAC-7205 (119 Mb, BTA1), Hapmap48395-BTA-58382 (54 Mb, BTA5), Hapmap54246-rs29017970 (30 Mb, BTA13) и ARS-BFGL-NGS-50172 (64 Mb, BTA17). По двум последним SNP имелись характерные ассоциации с удоем за стандартную лактацию, что свидетельствует о тесной генетической взаимосвязи и эффективности селекции по количеству молочного белка.
GWAS по воспроизводительным качествам и фертильности дочерей быков выявил регионы в геноме крупного рогатого скота с локализацией от 9 (по GL) до 66 (по DO) SNP со значением ожидаемой доли 186
ложных отклонений P < 0,05 (рис. 2). Использование более мощных критериев оценки нулевой гипотезы позволило точно локализовать мутации для CA ( BTB-01622929 , 117 Mb, BTA1), CR ( ARS-BFGL-NGS-89711 , 29 Mb BTA27), DO ( ARS-BFGL-NGS-117881 , 82 Mb, BTA5) и CI ( BTA-31636-no-rs , 67 Mb, BTA1; Hapmap26774-BTA-163037 , 42 Mb, BTA27). По признаку GL была определена значимая нуклеотидная замена ( BTA-31636-no-rs ), аналогичная выявленной для CI. По показателю CE не удалось обнаружить достоверных ассоциаций, что, по нашему мнению, связано с высоким влиянием паратипических факторов на признак.
Достоверность оценок ассоциаций по хозяйственно полезным при- знакам приближалась к нормальному распределению клонение от теоретически ожидаемого, как показано тиль-квантиль (q-q график, см. рис. 1, 2).
и имела слабое от-на графиках кван-
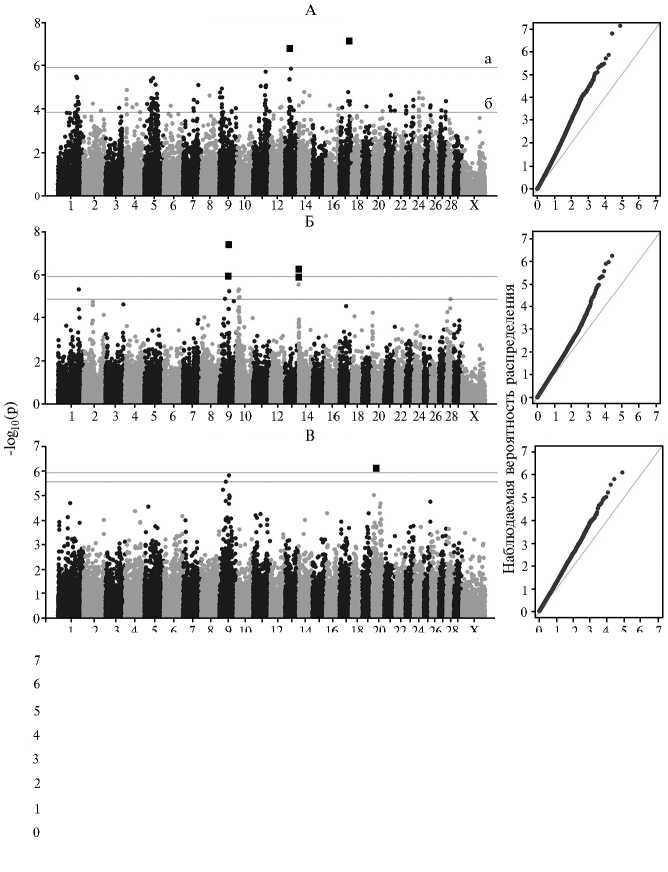
Рис. 1. Результаты GWAS (genome-wide association study, слева) и соответствующие вероятности распределения достоверности оценок (q-q график, справа) по удою (А) , массовой доле жира (Б) , массовой доле белка (В) и количеству молочного белка (Г) в популяции скота голштинской породы (Московская обл.) : 1 и 2 — порог соответственно по Бонферрони и Fdr. Квадраты означают соответствие по достоверности критерию проверки нулевой гипотезы по Бонферрони (высокодостоверные результаты по ассоциациям).
Значимые однонуклеотидные полиморфизмы были локализованы как внутри отдельных генов, так и в ближайших к ним регионах. Аддитивная доля влияния каждого маркера составила от 8,9 до 11,3 % общей изменчивости признаков. Значения регрессионного эффекта маркера для оценок племенной ценности имели как положительную, так и отрицательную величину, что указывает на возможность использования в отборе лучших животных, а также поиска и элиминации вредных мутаций. Достоверность ассоциаций колебалась в пределах от 1,2½10-6 до 7,6½10-8. Частоты минорных аллелей по локусам составляли 0,053-0,445 (табл. 2).
2. Достоверные ассоциации полиморфизмов единичных нуклеотидов (SPN) , сопряженных с оценкой количественных признаков, по точечным мутациям и их геномная локализация (популяция скота голштинской породы, Московская обл.)
|
SNP |
BTA |
Позиция |
Аллель |
Частота аллеля, Х ± m |
Эффект, Х ± m |
P |
R 2 , % |
Ближайший ген |
Расстояние до гена, п.н. |
Удо й з а 3 0 5 сут л акт ац ии, кг
|
ARS-BFGL- |
||||
|
NGS-50172 Hapmap54246- |
17 |
64342186 C 0,275±0,020 - 132,6±23,9 7,6½10-8 10,9 |
TRAFD1 |
10778 |
|
rs29017970 BTA-104917- |
13 |
30878341 G 0,126±0,015 +172,3±32,0 1,7½10-7 10,4 Массовая доля жира, % |
LOC104973750 |
Внутри гена |
|
no-rs ARS-BFGL- |
9 |
58113021 A 0,304±0,020 +0,036±0,0064,1½10-8 11,3 |
EPHA7 |
279992 |
|
NGS-107379 |
14 |
2054457 G 0,387±0,022 +0,030±0,0065,9½10-7 9,5 |
LOC786966 |
298 |
|
BTB-01604502 ARS-BFGL- |
9 |
58145538 A 0,368±0,021 +0,033±0,0071,1½10-6 9,0 |
EPHA7 |
312509 |
|
NGS-4939 Hapmap43278- |
14 |
1801116 G 0,347±0,021 +0,029±0,0061,2½10-6 8,9 Массовая доля белка, % |
DGAT1 |
Внутри гена |
|
BTA-50082 Hapmap48395- |
20 |
25185940 A 0,053±0,010 +0,033±0,0078,0½10-7 9,3 Кол ич е ств о м ол очног о б елка, кг |
ARL15 |
Внутри гена |
|
BTA-58382 ARS-BFGL- |
5 |
54120342 G 0,382±0,022 - 3,7±0,7 4,6½10-7 9,7 |
SLC16A7 |
Внутри гена |
|
NGS-50172 Hapmap54246- |
17 |
64342186 С 0,275±0,020 - 3,9±0,8 6,4½10-7 9,5 |
TRAFD1 |
10778 |
|
rs29017970 ARS-BFGL- |
13 |
30878341 G 0,127±0,015 +5,1±1,0 8,4½10-7 9,3 |
LOC104973750 |
Внутри гена |
|
BAC-7205 |
1 |
119976661 С 0,416±0,022 +3,5±0,7 1,2½10-6 9,1 Возраст первого отела, мес |
HPS3 |
Внутри гена |
|
BTB-01622929 ARS-BFGL- |
1 |
117345968 A 0,445±0,022 - 0,49±0,09 3,7½10-7 9,8 Кр атно сть о с ем енений, ед. |
LOC104970999 |
56 |
|
NGS-89711 ARS-BFGL- |
27 |
29231663 G 0,376±0,022 +0,041±0,0082,1½10-7 10,2 Продолжительность сервис-периода |
DUSP26 , сут |
261502 |
|
NGS-117881 Hapmap26774- |
5 |
82631679 A 0,302±0,020 +2,1±0,4 1,7½10-7 10,4 PPFIBP1 П р о д о лж ит ельн о сть м еж от ельн ог о п ер и од а, сут |
Внутри гена |
|
|
BTA-163037 BTA-31636-no- |
27 |
42578430 A 0,136±0,015 +0,7±0,1 1,1½10-6 9,0 |
LOC101905415 |
6930 |
|
rs BTA-31636-no- |
1 |
67399528 С 0,380±0,022 - 0,5±0,1 1,2½10-6 9,0 CCDC58 П р о д о лж ит ельн о сть ст ель н о ст и, сут |
Внутри гена |
|
|
rs |
1 |
67399528 C 0,380±0,022 - 0,4±0,1 1,3½10-6 9,0 |
CCDC58 |
Внутри гена |
П р и м еч а ни е. ВТА — хромосома крупного рогатого скота, Р — вероятность ошибки теста по нулевой гипотезе, R2 — коэффициент детерминации (доля влияния маркера). Для обозначения генов использованы официально принятые аббревиатуры.
Анализ SNP, сопряженных с оценкой количественных признаков скота голштинской популяции, выявил ряд генов, участвующих в различных биологических процессах (табл. 2): TRAF отвечает за иммунный ответ, биорегуляцию и синтез белков; EPHA7 контролирует механизм передачи клеточных сигналов и регуляцию метаболических процессов; LOC786966 связан с цитоскелетом и белковыми структурами эпителиальных клеток; DGAT1 контролирует транспорт липидов и их метаболизм;
ARL15 отвечает за внутриклеточный обмен, секрецию и везикулярный аппарат; SLC16A7 влияет на метаболизм и транспорт углеводов; HPS3 связан с клеточными процессами и пигментацией; DUSP26 отвечает за защитные механизмы на молекулярном уровне, а также за метаболические процессы фосфат содержащих соединений; CCDC58 контролирует клеточный цикл, деление клеток, фазу мейоза, связанную с разделением хромосом. Из вышеперечисленных генов три ( LOC786966 , DGAT1 , SLC16A7 ) были референтными для скота голштинской породы, что показано в аналогичных исследованиях J.B. Cole (20), проведенных на североамериканской популяции животных. В целом следует отметить, что по большинству признаков ассоциации могут быть коррелированными, т.е. являться частью общей системы взаимосвязи биологических процессов в организме животного.
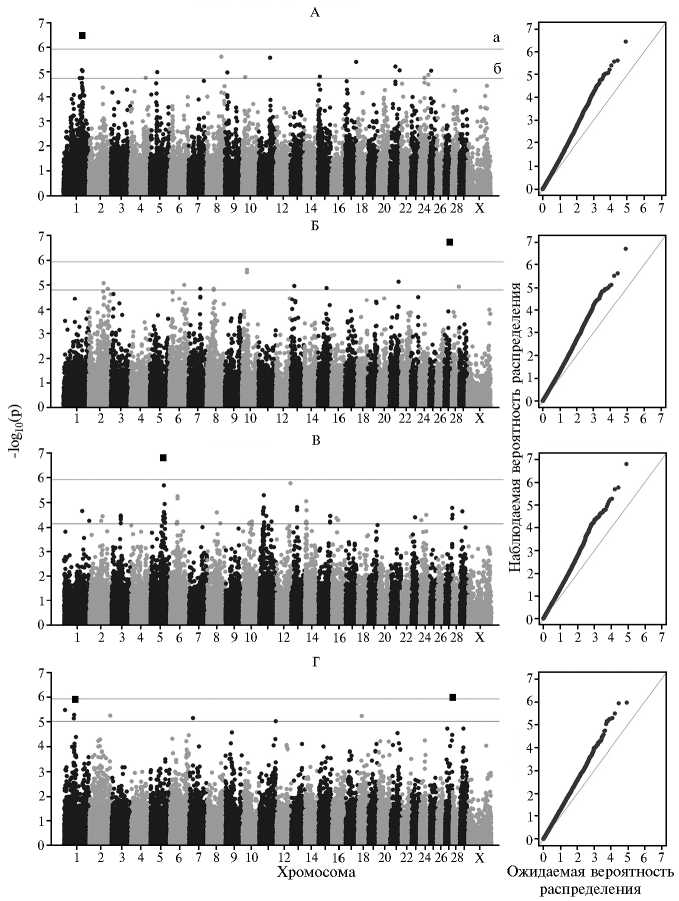
Рис. 2. Результаты GWAS (genome-wide association study, слева) и соответствующие вероятности распределения достоверности оценок (q-q график, справа) по возрасту первого отела (А) , числу осеменений (Б) , продолжительности сервис- (В) и межотельного (Г) периодов в популяции скота голштинской породы (Московская обл.) : 1 и 2 — порог соответственно по Бонферрони и Fdr. Квадраты означают соответствие по достоверности критерию проверки нулевой гипотезы по Бонферрони (высокодостоверные результаты по ассоциациям).
Таким образом, нами получены данные по картированию локусов количественных признаков (молочная продуктивность и воспроизводство) у крупного рогатого скота голштинской породы. Установлены высокодостоверные ассоциации по 14 SNP на хромосомах 1-й, 5-й, 9-й, 13-й, 14-й, 17-й, 20-й и 27-й с аддитивным эффектом более 9 % по ряду показателей. Наибольшей консервативностью в геноме по референтным мутациям обладали гены TRAFD1 , LOC786966 , DGAT1 , SLC16A7 , DUSP26 и CCDC58 . Полногеномные исследования, выполненные для признаков с разной степенью наследуемости, позволяют достаточно точно определять ассоциации по генетическим мутациям. Использование критерия оценки нулевых гипотез по ожидаемой доле ложных отклонений согласно функции максимального правдоподобия позволяет расширить детекцию по SNP. Тест по Бонферрони обладает более высокой мощностью проверки статистических гипотез.
Список литературы Полногеномный анализ ассоциаций с продуктивными и репродуктивными признаками у молочного скота в российской популяции голштинской породы
- Марзанов Н.С., Девришов Д.А., Марзанова С.Н., Комкова Е.А., Озеров М.Ю., Кантанен Ю. Генетическое маркирование, сохранение разнообразия и проблемы разведения животных. Сельскохозяйственная биология, 2011, 2: 3-14.
- Столповский Ю.А. Концепция и принципы генетического мониторинга для сохранения in situ пород доместицированных животных. Сельскохозяйственная биология, 2010, 6: 3-8.
- Зиновьева Н.А., Стрекозов Н.И., Янчуков И.Н., Ермилов А.Н., Ескин Г.В. Система геномной оценки скота: первые результаты. Животноводство России, 2015, 3: 27-29.
- Кузнецов В.М. Племенная оценки животных: прошлое, настоящее, будущее (обзор). Проблемы биологии продуктивных животных, 2012, 4: 18-57.
- Sermyagin A.A., Seltsov V.I., Kostyunina O.V., Gladyr E.A., Kharzinova V.R., Zinovieva N.A. Effect of LALBA polymorphism on the milk production traits of Russian cattle population. Book of Abstract «International scientific genetic conference «XXVI. GENETIC DAYS». Prague, Czech Republic, 2014: 135-138.
- Goddard M.E., Hayes B.J. Mapping genes for complex traits in domestic animals and their use in breeding programmes. Nature Reviews Genetics, 2009, 10: 381-391 ( ) DOI: 10.1038/nrg2575
- Weller J.I. Quantitative trait loci analysis in animals. 2nd ed. CABI, UK, 2009.
- Hayes B.J., Bowman P.J., Chamberlain A.J., Goddard M.E. Invited review: Genomic selection in dairy cattle: Progress and challenges. J. Dairy Sci., 2009, 92: 433-443 ( ) DOI: 10.3168/jds.2008-1646
- Pryce J.E., Gredler B., Bolormaa S., Bowman P.J., Egger-Danner C., Fuerst C., Emmerling R., Sölkner J., Goddard M.E., Hayes B.J. Short communication: Genomic selection using a multi-breed, across-country reference population. J. Dairy Sci., 2011, 94: 2625-2630 ( ) DOI: 10.3168/jds.2010-3719
- Pérez O'Brien A.M., Utsunomiya Y.T., Mészáros G., Bickhart D.M., Liu G.E., Van Tassell C.P., Tad S., Sonstegard T.S., da Silva M.V.B., Garcia J.F., Sölkner J. Assessing signatures of selection through variation in linkage disequilibrium between taurine and indicine cattle. Genetics Selection Evolution, 2014, 46: 19 ( ) DOI: 10.1186/1297-9686-46-19
- Boison S.A., Santos D.J.A., Utsunomiya A.H.T., Carvalheiro R., Neves H.H.R., Perez O'Brien A.M., Garcia J.F., Sölkner J., da Silva M.V.G.B. Strategies for single nucleotide polymorphism (SNP) genotyping to enhance genotype imputation in Gyr (Bos indicus) dairy cattle: Comparison of commercially available SNP chips. J. Dairy Sci., 2015, 98: 4969-4989 ( ) DOI: 10.3168/jds.2014-9213
- Pryce J.E., Bolormaa S., Chamberlain A.J., Bowman P.J., Savin K., Goddard M.E., Hayes B.J. A validated genome-wide association study in 2 dairy cattle breeds for milk production and fertility traits using variable length haplotypes. J. Dairy Sci., 2010, 93: 3331-3345 ( ) DOI: 10.3168/jds.2009-2893
- Bolormaa S., Pryce J.E., Hayes B.J., Goddard M.E. Multivariate analysis of a genome-wide association study in dairy cattle. J. Dairy Sci., 2010, 93: 3818-3833 ( ) DOI: 10.3168/jds.2009-2980
- Chamberlain A.J., Hayes B.J., Savin K., Bolormaa S., McPartlan H.C., Bowman P.J., Van Der Jagt C., MacEachern S., Goddard M.E. Validation of single nucleotide polymorphisms associated with milk production traits in dairy cattle. J. Dairy Sci., 2012, 95: 864-875 ( ) DOI: 10.3168/jds.2010-3786
- Qanbari S., Pimentel E.C.G., Tetens J., Thaller G., Lichtner P., Sharifi A.R., Simianer H. A genome-wide scan for signatures of recent selection in Holstein cattle. Animal Genetics, 2010, 41(4): 377-389 ( ) DOI: 10.1111/j.1365-2052.2009.02016.x
- Maxa J., Neuditschko M., Russ I., Förster M., Medugorac I. Genome-wide association mapping of milk production traits in Braunvieh cattle. J. Dairy Sci., 2012, 95: 5357-5364 ( ) DOI: 10.3168/jds.2011-4673
- Fang M., Fu W., Jiang D., Zhang Q., Sun D., Ding X., Liu J. A multiple-SNP approach for genome-wide association study of milk production traits in Chinese Holstein cattle. PLoS ONE, 2014, 9(8): e99544 ( ) DOI: 10.1371/journal.pone.0099544
- Schopen G.C.B., Visker M.H.P.W., Koks P.D., Mullaart E., van Arendonk J.A.M., Bovenhuis H. Whole-genome association study for milk protein composition in dairy cattle. J. Dairy Sci., 2011, 94: 3148-3158 ( ) DOI: 10.3168/jds.2010-4030
- Maurice-Van Eijndhoven M.H.T., Bovenhuis H., Veerkamp R.F., Calus M.P.L. Overlap in genomic variation associated with milk fat composition in Holstein Friesian and Dutch native dual-purpose breeds. J. Dairy Sci., 2015, 98: 6510-6521 ( ) DOI: 10.3168/jds.2014-9196
- Cole J.B., Wiggans G.R., Ma L., Sonstegard T.S., Lawlor T.J. Jr., Crooker B.A., Van Tassell C.P., Yang J., Wang S., Matukumalli L.K., Da Y. Genome-wide association analysis of thirty one production, health, reproduction and body conformation traits in contemporary U.S. Holstein cows. BMC Genomics, 2011, 12: 408 ( ) DOI: 10.1186/1471-2164-12-408
- Wang X., Wurmser C., Pausch H., Jung S., Reinhardt F., Tetens J., Thaller G., Fries R. Identification and dissection of four major QTL affecting milk fat content in the German Holstein-Friesian population. PLoS ONE, 2012, 7(7): e40711 ( ) DOI: 10.1371/journal.pone.0040711
- Cole J.B., Waurich B., Wensch-Dorendorf M., Bickhart D.M., Swalve H.H. A genome-wide association study of calf birth weight in Holstein cattle using single nucleotide polymorphisms and phenotypes predicted from auxiliary trait. J. Dairy Sci., 2014, 97: 3156-3172 ( ) DOI: 10.3168/jds.2013-7409
- Sahana G., Guldbrandtsen B., Lund M.S. Genome-wide association study for calving traits in Danish and Swedish Holstein cattle. J. Dairy Sci., 2011, 94: 479-486 ( ) DOI: 10.3168/jds.2010-3381
- Fritz S., Capitan A., Djari A., Rodriguez S.C., Barbat A., Baur A., Grohs C., Weiss B., Boussaha M., Esquerre D., Klopp C., Rocha D., Boichard D. Detection of haplotypes associated with prenatal death in dairy cattle and identification of deleterious mutations in GART, SHBG and SLC37A2. PLoS ONE, 2013, 8(6): e65550 ( ) DOI: 10.1371/journal.pone.0065550
- Mészáros G., Eaglen S., Waldmann P., Sölkner J. A genome wide association study for longevity in cattle. Open Journal of Genetics, 2014, 4: 46-55 ( ) DOI: 10.4236/ojgen.2014.41007
- Sahana G., Guldbrandtsen B., Thomsen B., Holm L-E., Panitz F., Brøndum R.F., Bendixen C., Lund M.S. Genome-wide association study using high-density single nucleotide polymorphism arrays and whole-genome sequences for clinical mastitis traits in dairy cattle. J. Dairy Sci., 2014, 97: 7258-7275 ( ) DOI: 10.3168/jds.2014-8141
- Suchocki T., Szyda J. Genome-wide association study for semen production traits in Holstein-Friesian bulls. J. Dairy Sci., 2015, 98: 5774-5780 ( ) DOI: 10.3168/jds.2014-8951
- Mészáros G., Petautschnig E., Schwarzenbacher H., Sölkner, J. Genomic regions influencing coat color saturation and facial markings in Fleckvieh cattle. Animal Genetics, 2015, 46(1): 65-68 ( ) DOI: 10.1111/age.12249
- Misztal I., Legarra A., Aguilar I. Computing procedures for genetic evaluation including phenotypic, full pedigree, and genomic information. J. Dairy Sci., 2009, 92: 4648-4655 ( ) DOI: 10.3168/jds.2009-2064
- Aguilar I., Misztal I., Johnson D.L., Legarra A., Tsuruta S., Lawlor T.J. Hot topic: A unified approach to utilize phenotypic, full pedigree, and genomic information for genetic evaluation of Holstein final score. J. Dairy Sci., 2010, 93: 743-752 ( ) DOI: 10.3168/jds.2009-2730
- Legarra A., Ducrocq V. Computational strategies for national integration of phenotypic, genomic, and pedigree data in a single-step best linear unbiased prediction. J. Dairy Sci., 2012, 95: 4629-4645 ( ) DOI: 10.3168/jds.2011-4982
- Přibyl J., Madsen P., Bauer J., Přibylová J., Šimečková M., Vostrý L., Zavadilová L. Contribution of domestic production records, Interbull estimated breeding values, And single nucleotide polymorphism genetic markers to the single-step genomic evaluation of milk production. J. Dairy Sci., 2013, 96: 1865-1873 ( ) DOI: 10.3168/jds.2012-6157
- Garrick D.J., Taylor J.F., Fernando R.L. Deregressing estimated breeding values and weighting information for genomic regression analyses. Genetics Selection Evolution, 2009, 41: 55 ( ) DOI: 10.1186/1297-9686-41-55
- SAS/STAT 9.1 User's Guide. SAS Institute Inc., Cary, NC, 2005.
- Purcell S., Neale B., Todd-Brown K., Thomas L., Ferreira M., Bender D., Maller J., Sklar P., de Bakker P., Daly M.J., Sham P.C. PLINK: a toolset for whole-genome association and population-based linkage analysis. American Journal of Human Genetics. 2007, 81(3): 559-575 ( ) DOI: 10.1086/519795
- Сермягин А.А., Гладырь Е.А., Харитонов С.Н., Ермилов А.Н., Янчуков И.Н., Стрекозов Н.И., Зиновьева Н.А. Использование геномной оценки для совершенствования племенных качеств скота голштинской и черно-пестрой пород России. Мат. Межд. науч.-практ. конф. «Пути продления продуктивной жизни коров на основе оптимизации разведения, технологий содержания и кормления животных». Дубровицы, 2015: 115-120.
- VanRaden P.M. Efficient methods to compute genomic predictions. J. Dairy Sci., 2008, 91: 4414-4423 ( ) DOI: 10.3168/jds.2007-0980
- The R Project for Statistical Computing: Free software environment for statistical computing and graphics (http://www.r-project.org).
- Efron B., Turnbull B.B., Narasimhan B. Locfdr: Computes local false discovery rates. R package version 1.1-7. 201 (http://CRAN.R-project.org/package=locfdr).
- Benjamini Y., Hochberg Y. Controlling the false discovery rate: a practical and powerful approach to multiple testing. Journal of the Royal Statistical Society, Series B, 1995, 57: 289-300.
- Turner S.D. qqman: an R package for visualizing GWAS results using Q-Q and Manhattan plots. bioRxiv, 2014. Preprint first posted online May 14, 2014 ( ) DOI: 10.1101/005165


