Полногеномный поиск ассоциаций однонуклеотидных замен с показателями качества молока у овец (Ovis aries L.) породы лакон
Автор: Селионова М.И., Трухачев В.И., Зиновьева Н.А., Белоус А.А., Айбазов А.М.М.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Полногеномный анализ
Статья в выпуске: 4 т.59, 2024 года.
Бесплатный доступ
Молочное овцеводство в последние годы демонстрирует динамичное развитие в России, что обусловлено растущим потребительским спросом на молочную продукцию премиум класса. В селекции молочных овец наиболее перспективными признаны маркер-ассоциированный (marker-associated selection, MAS) и полногеномный ассоциативный анализ (genome-wide association study, GWAS) благодаря секвенированию генома овец в рамках проекта Sheep HapMap и разработке ДНК-чипы различной плотности. Для ряда молочных пород овец GWAS позволил выявить локусы, находящиеся под давлением селекции и влияющие на признаки продуктивности и технологические свойства овечьего молока. В то же время полногеномных исследований для молочных овец выполнено значительно меньше, чем для молочного скота и молочных коз, что определило цель настоящего исследования. Впервые выполнен полногеномный поиск ассоциаций однонуклеотидных замен с показателями качества молока у овец породы лакон и определены геномные регионы, связанные с их молочной продуктивностью. SNP-профили исследованных овец были сгенерированы с использованием чипа высокой плотности Ovine HD BeadChip 600K («Illumina Inc.», CШA). Контроль качества, фильтрацию данных, поиск ассоциаций для каждого однонуклеотидного полиморфизма с содержанием компонентов в молоке, определенным на автоматическом анализаторе CombiFoss 7 DC («FOSS», Дания), проводили множественным линейным регрессионным анализом в программе plink 1.90 (https://www.cog-genomics.org/plink/), визуализацию данных - в пакете qqman с использованием языка программирования R (https://github.com/stephenturner/qqman), идентификацию генов по позициям SNP, заданным в соответствии со сборкой генома овец OAR версии 3.1, с помощью веб-ресурса Ensembl Genes release 103, структурную аннотацию геномных регионов, покрывающих окно ±0,20 Mb от идентифицированных SNPs, в программе DAVID v6.8 (https://david.ncifcrf.gov/). Выявлено 64 полногеномных (p -6) и 270 предположительных (p -5) SNP, при этом наибольшее число полногеномных SNPs - 14, 6 и 13 идентифицировали соответственно на 1-й, 3-й и 13-й хромосомах. С показателями массовая доля белка и содержание β-казеина ассоциации демонстрировали 22 и 19 полногеномных SNPs, идентифицированных на пяти (OAR1, OAR3, OAR12, OAR13 и OAR26) и четырех (OAR1, OAR3, OAR13 и OAR26) хромосомах, с массовой долей жира - 6 SNPs на трех хромосомах (OAR1, OAR3, OAR26), с количеством полиненасыщенных, насыщенных и мононенасыщенных жирных кислот - 7, 5 и 4 SNPs соответственно на четырех (OAR1, OAR3, OAR5 и OAR17) и трех (OAR1, OAR24, OAR26 и OAR1, OAR6, OAR17) хромосомах. Два полногеномных SNPs (oar3_OAR1_24401030 и oar3_OAR2_241055039) были общими для трех признаков - массовой доли белка, содержания β-казеина и массовой доли жира. Структурная аннотация геномных регионов выявила 61 ген с описанными биологическими функциями, из которых 11 демонстрировали связь с фенотипическими признаками овец, определенными в других GWAS исследованиях. Выявленные гены в соответствии с терминами генной онтологии были вовлечены в формирование и регуляцию функций иммунной системы (GPX7, JAK1, CD8B), клеточный метаболизм и транспорт веществ (UBE2U, ACO1, FABP1, ACBD5), формирование и развитие внутренних органов и скелетно-хрящевой системы (BMPR1B, OXSM), процессы оогенеза, оплодотворения и раннего развития эмбрионов (ZPBP, ZP2) .
Ovis aries, овцы, компоненты молока, белок, жир, насыщенные жирные кислоты, мононенасыщенные жирные кислоты, полиненасыщенные жирные кислоты, гены-кандидаты, gwas, snp, qtl
Короткий адрес: https://sciup.org/142243766
IDR: 142243766 | УДК: 636.32/.38:575.2 | DOI: 10.15389/agrobiology.2024.4.735rus
Текст научной статьи Полногеномный поиск ассоциаций однонуклеотидных замен с показателями качества молока у овец (Ovis aries L.) породы лакон
Мировое производство молока овец характеризуется динамичным ростом. К 2022 году его объем составил 11,0 млн т, и, исходя из динамики последних лет, прогнозируется, что к 2030 году повышение составит 26,0 % — до 14,0 млн т (1). Лидер по объему овечьего молока — Китай, который в 2022 году произвел около 1,3 млн т, с небольшим отставанием следуют Турция и Греция с 1,16 и 1,10 млн т. Сирия, Испания, Алжир и Италия производят примерно 0,5-0,6 млн т (FAOSTAT, 2022).
Для нашей страны специализированное молочное овцеводство — это
∗ Исследования выполнены при финансовой поддержке РНФ в рамках проекта ¹ 21-76-20008.
новое направление. Стимулом послужила необходимость сформировать собственную сырьевую базу для производства более широкой линейки молочной продукции, особенно сыров премиум класса. В 2015-2018 годах в Краснодарском крае, Республике Марий Эл, Тверской области были созданы первые фермы по разведению молочных овец, в 2021-2022 годах ареал расширился и такие фермы появились в Московской области.
К отличительным особенностям овечьего молоко относятся высокое содержание белка и жира и ненасыщенных короткоцепочечных жирных кислот. Казеин овечьего молока более устойчив к нагреванию, содержит больше неорганического фосфора и кальция, поэтому при производстве сыра не требуется дополнительного внесения хлорида кальция, что дает технологическое преимущество. Кроме того, из овечьего молока получается значительно больший выход сыра, который составляет 1:5, тогда как из молока коров 1:10 (2).
В селекции молочных овец, как и других видов сельскохозяйственных животных, в последнее время наиболее перспективными признаны генетические методы (3, 4). Прогресс в их развитии способствует расширению научного поиска и применяемых новых инструментов геномного анализа. Благодаря проекту Sheep HapMap, выполненному Международным консорциумом по геномике овец (ISGC, , проведено полное секвенирование и представлена первая версия генома овец (OAR_v1.0), с последующим усовершенствованием (OAR_v2.0 и OAR_v3.1) , разработаны ДНК-чипы различной плотности. Это стало чрезвычайно важным ресурсом для анализа полногеномных ассоциаций (genome-wide association study, GWAS) и поиска генов, формирующих ценные фенотипические признаки молочных овец (5). При этом следует признать, что ассоциативных геномных исследований в молочном овцеводстве выполнено значительно меньше в сравнении с молочным скотоводством и козоводством (6-8), хотя имеются и определенные достижения (9-11).
В исследовании H. Li с соавт. (12) с использованием GWAS у крос-сбредных молочных овец идентифицировано 288 высокозначимых SNPs для удоя, 115 и 74 — для выхода соответственно жира и белка. На хромосомах 1-й, 3-й, 4-й, 5-й, 7-й и 11-й выявлено наибольшее число SNPs с высокой значимостью. Это позволило авторам предположить, что именно указанные хромосомы лидируют по числу генов, контролирующих молочную продуктивность овец. Аннотация геномных регионов выявила ряд генов — TTC7B , SLC27A6 , LRP1B , GALNT14 , CNTN4 , CDH13 , SLC39A12 , которые были ассоциированы с изученными признаками, а также связаны с липидным и белковым обменом у других видов сельскохозяйственных животных. Гены ITPR2 и SLC27A6 были также определены как гены-кандидаты продуктивности у молочного скота, что делает их перспективными для использования в селекции молочных овец (12).
Гены GAL3ST3 и SPATA6 были определены как высокозначимые для селекции по признакам удоя и продолжительности лактации для овец породы авасси (13). У молочных овец породы валле-дель-белис выявлено 23 геномно значимых SNPs: 3 ассоциированы с удоем, 9 — с выходом жира и 11 — с его массовой долей. Несколько SNPs совпали с известными генами-кандидатами или ранее зарегистрированными QTL (quantitative trait loci) для признаков молочной продуктивности у разных видов домашнего скота. Кроме того, на 3-й хромосоме обнаружены новые SNPs для изученных признаков (14). На овцах пород ассаф и чурра, помимо традиционных показателей молочной продуктивности (удой, массовая доля и выход белка и жира, количество соматических клеток) в GWAS были включены технологические параметры, связанные с производством сыра (время свертывания и образованием творожного сгустка, время его затвердевания, упругость через 30 и 60 мин после добавления сычужного фермента). В итоге идентифицировали соответственно 84 и 139 значимых ассоциаций SNPs, функциональная аннотация которых выявила 71 потенциальный ген-кандидат для определения наиболее перспективных в селекции (15).
Известно, что селекция на повышение продуктивности зачастую сопровождается снижением устойчивости к заболеваниям и приспособительных реакций к неблагоприятным факторам окружающей среды. Учитывая это, целью исследования A. Argyriadou с соавт. (16) было проведение полногеномного ассоциативного анализа косвенных признаков здоровья (степень упитанности животных, продолжительность лактации, число соматических клеток в молоке) у молочных овец пород хиос и фризарта. Было установлено 19 генов-кандидатов, в том числе 2 ассоциированных с упитанностью ( POT1 , TMEM229A) , 13 — с числом соматических клеток ( NTAQ1 , ZHX1 , ZHX2 , LOC101109545 , HAS2 , DERL1 , FAM83A , ATAD2 , RBP7 , FSTL1 , CD80 , HCLS1 , GSK3B ) и 4 — с продолжительностью лактации ( GRID2, FAIM, CEP70 , GRIP1 ). В результате аннотации генов и поиска онтологий ген TMEM229A, регулирующий синтез белка клеточных мембран, и ген POT1, контролирующий длину теломер и служащий индикатором клеточного старения, были определены как маркеры параметров тела у крупного рогатого скота, а для генов GRID2 и CEP70 , регулирующих секрецию гонадотропина и пролактина, выявлена связь с состоянием вымени и молочной продуктивности у овец и крупного рогатого скота (16).
Указывается на важность изучения SLC2A2 , CSN2 , SCD и SOCS2 в качестве генов-кандидатов молочной продуктивности у овец, поскольку эти гены демонстрировали высокозначимую связь с продуктивностью молочного скота. Так, ген SLC2A2 ассоциирован с транспортировкой растворимых молекул из крови в молочную железу, ген СSN2 — с синтезом казеина A2, ген SCD — с выработкой ненасыщенных жирные кислоты и процентом молочного жира, ген SOCS2 — с числом соматических клеток в молоке и предрасположенностью к маститу (17).
Таким образом, метод GWAS, основанный на генотипировании однонуклеотидных полиморфизмов по всему геному, открывает большие возможности для выявления генов-кандидатов и понимания генетической архитектуры признаков молочной продуктивности у овец. Несмотря на то, что GWAS выполнен для ряда молочных пород овец, локусы, находящиеся под давлением селекции и влияющие на признаки молочной продуктивности и технологические свойства овечьего молока, остаются в значительной степени неизвестными.
В настоящем исследовании впервые выполнен полногеномный поиск ассоциаций однонуклеотидных замен с показателями качества молока у овец породы лакон и определены геномные регионы, связанные с их молочной продуктивностью. На 1-й, 3-й и 13-й хромосомах для всех исследованных показателей идентифицировано наибольшее число полногеномных SNPs, из которых большая часть была связана с массовой долей белка и β-казеина. Структурная аннотация геномных регионов выявила 61 ген с описанными биологическими функциями, из которых 11 демонстрировали связь с другими фенотипическим признаками у овец. Выявленные гены в соответствии с терминами генной онтологии были вовлечены в процессы оогенеза, оплодотворения и раннего развития эмбрионов, в формирование и развитие внутренних органов и скелетно-хрящевой системы, в регуляцию функций иммунной системы, клеточного метаболизма, в транспорт веществ.
Целью настоящего исследования был поиск генов-кандидатов, связанных с показателями молочной продуктивности у овец породы лакон, на основе полногеномного анализа ассоциаций.
Методика . Объектом исследований были овцы породы лакон первой ( п = 61) и второй ( п = 35) лактаций (всего п = 96, КФХ Николаев М.И., Краснодарский край, 2022 год). Средние суточные пробы молока получали при 2-кратном доении (доильная установка (тип «параллель» Mdisplacement 2½16, доильный аппарат TOP FLOW, «Gea Farm Technologies», Германия) в течение трех контрольных доений через 14 сут. Компонентный анализ молока проводили в ФИЦ животноводства — ВИЖ им. академика Л.К. Эрнста на базе ОНИС БиоТехЖ с использованием метода инфракрасной спектрометрии (автоматический анализатор CombiFoss 7 DC, «FOSS», Дания). Определяли массовую долю истинного белка (МДБ), содержание β-казе-ина, массовую долю жира (МДЖ), содержание моно- и полиненасыщен-ных (МНЖК, ПНЖК) и насыщенных жирных кислот (НЖК), трансизомеры жирных кислот (ТЖК).
SNP-профили были сгенерированы с использованием чипа высокой плотности Ovine HD BeadChip 600K («Illumina, Inc.», США). Фильтрацию результатов генотипирования проводили с использованием программного продукта plink 1.9 .
Контроль качества и фильтрацию данных генотипирования для каждого SNP и каждого образца выполняли с помощью программного пакета plink 1.9 и применением следующих фильтров: сall-rate по всем исследуемым SNP для индивидуального образца не ниже 90 % (--mind); сall-rate для каждого из исследованных SNPs по всем генотипированным образцам не ниже 90 % (--geno); частота встречаемости минорных аллелей (MAF) более 0,01 или 0,05 (--maf 0.01); отклонение числа SNP генотипов от распределения Харди-Вайнберга в совокупности протестированных образцов с уровнем значимости p-value < 10-6 (--hwe). Кроме того, проводили оценку неравновесного сцепления исследуемых SNPs (LD оценка) с r2 < 0,2 с шагом 50 kb (--indep-pairwaise). Для GWAS было использовано 47269 SNPs, прошедших контроль качества данных.
Полногеномный порог значимости был установлен на уровне p < 0,05, предположительный (пограничный) — в расчете на одну ложноположительную ассоциацию (18). Полногеномный порог (высокая значимость) после коррекции с использованием поправки Бонферрони для множественных сравнений составил p < 1,06½10-6, или 0,05/47269, что соответствует отрицательному десятичному логарифму -log105,97, предположительный (пограничный) — p < 2,11½10-5, что соответствует -log104,67. Для определения допустимого для модели уровня значимости SNP полученные значения р (р < 0,005) для всех выбранных полиморфных сайтов проверяли с помощью построения графиков квантиль-квантиль Quantatile-Quantatile (Q-Q) plot. Результат определялся с помощью коэффициента λ, значение которого выше 1 свидетельствует о доверии к модели, использующей заданные значения р выбранных полиморфных сайтов. Анализ проводили с помощью программного обеспечения GEMMA версии 0.98.1 (19). Геномные графики -log10 (p-values), иллюстрирующие уровень ассоциации SNPs с изучаемыми признаками и соответствующие квантиль-квантиль графики (Q-Q plot), были созданы в RStudio с использованием R версии 4.1.2 (RRID:SCR_001905; и статистического пакета R qqman (20).
Анализ нормального распределения изучаемых признаков прово- дился с помощью описательной статистики в программе STATISTICA 10 . Определяли минимальное и максимальное значение признака (min и max), среднее значение по выборке (M), его ошибку (±SEM), коэффициент вариации (Cv). Средовые и постоянные эффекты исключали методом обобщенной линейной модели (generalizes linear regression, GLM):
у = HYt + Ь1Аде + е, где у — соответствующая GLM оценка животного; НYi — фиксированный эффект «год рождения животного»; Age — регрессионный эффект в сутках лактации на момент оценки животного; b1 — регрессионный коэффициент модели; e — нераспределенный остаток модели.
Идентификация генов по позициям SNP, заданным в соответствии со сборкой генома овец OAR версии 3.1, выполнялась с помощью веб-ресурса Ensembl Genes release 103. Структурная аннотация геномных регионов, покрывающих окно ±0,20 Mb от идентифицированных SNPs, проводилась в программе DAVID v6.8 .
Результаты . Описательная статистика по показателям молока, изученным методом инфракрасной спектрометрии, представлена в таблице 1.
1. Описательная статистика по изученным показателям качества молока у овец ( Ovis aries L.) породы лакон (КФХ Николаев М.И., Краснодарский край, 2022 год)
|
Группа показателей |
Наименование |
Min |
Max |
M |
±SEM |
Cv , % |
|
Белок |
Массовая доля белка (истинная) |
3,80 |
10,60 |
5,93 |
0,07 |
16,8 |
|
и β-казеин, % |
β-Казеин |
3,27 |
9,43 |
5,13 |
0,06 |
17,7 |
|
Жир, % |
Массовая доля жира |
3,03 |
8,64 |
7,65 |
0,15 |
30,1 |
|
и жирные кислоты |
Насыщенные жирные кислоты |
1,10 |
7,26 |
5,26 |
0,11 |
29,9 |
|
(ЖК), трансизомеры |
Мононенасыщенные жирные кислоты |
0,59 |
4,48 |
1,97 |
0,04 |
25,4 |
|
ЖК, г/100 г |
Полиненасыщенные жирные кислоты |
0,08 |
1,47 |
1,32 |
0,03 |
28,7 |
|
Трансизомеры жирных кислот |
0,05 |
0,64 |
0,22 |
0,01 |
52,7 |
Анализ полученных данных показывает, что наименьшая изменчивость была характерна для массовой доли белка и β-казеина, которая не превышала 18,0 %. Далее следовали показатели содержания жирных кислот и массовой доли жира — 25,4-30,1 %. Наибольшую изменчивость (52,7 %) демонстрировал уровень трансизомеров жирных кислот.
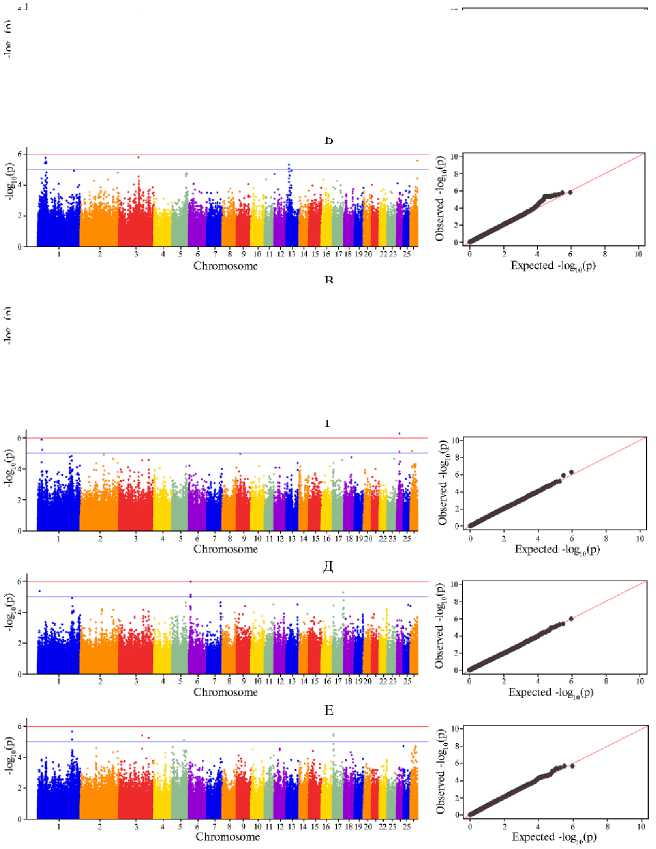
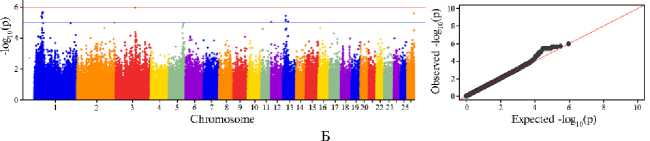
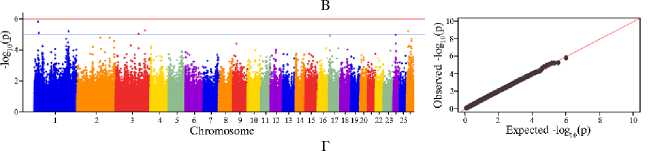
Визуализация расположения статистически значимых SNPs на 25 аутосомах овец породы лакон представлены на рисунке 1 (А-Ж, слева). Для всех исследованных признаков квантили вероятностного распределения ожидаемого и наблюдаемого отклонений от нормального распределения свидетельствовали о достаточном уровне значимости (см. рис. 1, А-Ж, справа).
В результате выполненного GWAS для изученных показателей молока овец мы выявили 64 полногеномных и 270 предположительных (с допороговой значимостью) SNPs. При этом наибольшее число полногеномных SNPs идентифицировали на 1-й, 3-й и 13-й хромосомах — соответственно 14, 6 и 13. На 5-й, 15-й, 17-й, 24-й и 26-й хромосомах было выявлено по одному полногеномному SNP. Наибольшее число нуклеотидных полиморфизмов допороговой значимости, как и в случае с SNPs высокой значимости, мы обнаружили на 1-й, 3-й и 13-й хромосомах, соответственно 61, 19 и 13. Большее в сравнении с другими хромосомами число SNPs допороговой значимости, равное 9, было выявлено на 26-й хромосоме. На других хромосомах (номера 2, 5, 6, 9, 11, 15, 16, 17, 23, 24 и 25) обнаружили или один, или два SNP. На 19-й, 20-й, 21-й и 22-й хромосомах мы не выявили ни одного однонуклеотидного полиморфизма, ассоциированного с показателями качества молока у овец (табл. 2).
А
Ж
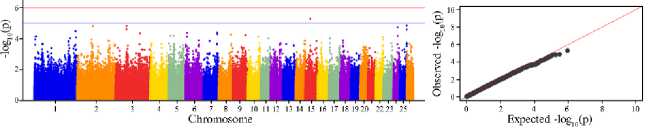
Распределение SNP, ассоциированных с признаками качества молока у овец ( Ovis aries L.) породы лакон (КФХ Николаев М.И., Краснодарский край, 2022 год) по хромосомам (ось X) в зависимости от достоверности ассоциаций ( - log 10 p-value, ось Y, нижняя линия) и критерию Бонфер-рони (ось Y, верхняя линия) (слева) и квантиль вероятностного распределения ожидаемого и наблюдаемого отклонений от нормального распределения для значений достоверности ассоциаций (Quantatile-Quantatile Q-Q plot, справа): А — массовая доля белка, Б — содержание β-казеина, В — массовая доля жира, Г — насыщенные жирные кислоты, Д — мононенасыщенные жирные кислоты, Е — полиненасыщенные жирные кислоты, Ж — трансизомеры жирных кислот.
С показателем массовая доля белка и β-казеина ассоциации демонстрировали соответственно 73 и 66 SNP, в том числе 22 и 19 полногеномных, которые были распределены соответственно между пятью (OAR 1, 3, 12, 13 26) и четырьмя (OAR 1, 3, 13 и 26) хромосомами. При этом 3 полногеномных SNP на 1-й хромосоме (oar3_OAR1_45935458, _53855439, _55048881, _50079454), 12 SNP на 13-й хромосоме (oar3_OAR13_17148993, _17153638,
_17155105, _17156335, _19523580.1, _17168795, _17171855, _17176409, _17180926, _17207481, _17218518, _17220761), а также 1 SNP на 26-й хромосоме (oar3_OAR26_43767472) совпадали для обоих признаков. Таким образом, для массовой доли белка из 22 SNP высокой значимости 16 SNP были идентичны выявленным для β-казеина (72,7 %), тогда как для последнего таких было соответственно 16 SNP из 19 SNP (84,2 %). Также следует отметить, что из 51 SNPs допороговой значимости для массовой доли белка и из 47 SNPs для β-казеина 43 SNPs были идентичны, то есть совпадение для указанных признаков составляло соответственно 84,3 и 91,5 %.
2. Число и распределение по хромосомам SNPs с разным уровнем значимости ассоциаций с показателями качества молока у овец ( Ovis aries L.) породы лакон (КФХ Николаев М.И., Краснодарский край, 2022 год)
|
Показатель |
Полногеномные |
Предположительные |
||
|
число |
номера хромосом |
число |
номера хромосом |
|
|
МДБ |
22 |
1, 3, 12, 13, 26 |
51 |
1, 2, 3, 5, 6, 9, 11, 13, 16, 17, 26 |
|
β-Казеин |
19 |
1, 3, 13, 26 |
47 |
1, 2, 3, 5, 6, 9, 11, 12, 13, 16, 17, 26 |
|
МДЖ |
6 |
1, 3, 26 |
46 |
1, 2, 3, 4, 9, 12, 16, 17, 18, 24, 26 |
|
НЖК |
5 |
1, 24, 26 |
34 |
1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 13, 14, 16, 17, 18, 23, 24, 26 |
|
МНЖК) |
4 |
1, 6, 17 |
32 |
1, 2, 3, 5, 6, 7, 9, 11, 13, 17, 25 |
|
ПНЖК) |
7 |
1, 3, 5, 17 |
42 |
1, 2, 3, 4, 5, 9, 10, 11, 12, 15, 17, 25, 26 |
|
ТЖК Всего |
1 |
15 64 SNP на 10 хромосомах |
18 |
1, 2, 3, 5, 6, 7, 8, 9, 10, 11, 15, 24, 25 270 SNP на 22 хромосомах |
|
Примечан |
и е. МДБ — массовая доля белка в молоке, МДЖ — |
массовая доля жира, НЖК — насыщен- |
||
|
ные жирные кислоты, МНЖК — мононенасыщенные жирные кислоты, ПНЖК — полиненасыщенные жирные кислоты, ТЖК — трансизомеры жирных кислот |
||||
Для показателя массовая доля жира в общей сложности было идентифицировано 52 SNPs, из которых 6, расположенных на хромосомах 1-й, 3-й и 26-й, имели полногеномный характер значимости.
Для изученных классов жирных кислот наибольшее число ассоциаций было выявлено для полиненасыщенных жирных кислот — 49 SNPs, для насыщенных и мононенасыщенных было установлено близкое их число — соответственно 39 и 36 SNPs. При этом SNPs, имеющих полногеномный характер ассоциаций, было 7, 5 и 4, они располагались соответственно на OAR1, OAR3, OAR5 и OAR17 (ПНЖК), OAR1, OAR24, OAR26 (НЖК), OAR1, OAR6 и OAR17 (МНЖК).
С трансизомерами жирных кислот были связаны 19 SNPs, при этом лишь один SNP на 15-й хромосоме имел полногеномную значимость.
Для массовой доли жира, жирных кислот и их трансизомеров выявлен ряд общих SNPs. Так, для трех признаков — МДЖ, НЖК, ПНЖК идентифицировали четыре SNPs (oar3_OAR1_24401030, oar3_OAR1_220774480, oar3_OAR3_149398814 и oar3_OAR3_191472136) с полногеномным уровнем значимости и шесть SNPs (oar3_OAR1_220784114, oar3_OAR1_220798463, oar3_OAR2_210881102, oar3_OAR2_210881505, oar3_OAR2_240690950, oar3_OAR17_60807141), которые для одних признаков демонстрировали полногеномный уровень значимости, для других — предположительный. Для показателей МДЖ и НЖК выявлен один общий SNP полногеномной значимости — oar3_OAR26_9949123, для МНЖК и ТЖК — три общих SNPs: oar3_OAR1_10731857, oar3_OAR6_11914049 и oar3_OAR6_11894952, которые были полногеномными для первого показателя и предположительной значимости для второго. Следует отметить, что наибольшее число совпадающих SNPs допороговой значимости (по 10) выявили для признаков МДЖ и НЖК, МДЖ и ПНЖК, тогда как для МНЖК и ПНЖК, МНЖК и ТЖК таких было по 3 SNP.
Следует выделить два полногеномных SNPs высокой значимости — oar3_OAR1_24401030 и oar3_OAR2_241055039, которые были общими для трех признаков: для массовой доли белка, содержания β-казеина и массовая доли жира.
Анализ в базе данных для аннотаций, визуализации и интегрированного обнаружения (DAVID) по биологической библиотеке (GO) позволил установить, что из 328 структурно обнаруженных генов у 61 имеются описанные биологические функции (табл. 3). При этом из 61 гена всего два имеют регистрационные номера в базе количественных признаков овец (SheepQTLdb, : это гены CD8B (16 номеров) и BMPR1B (5 номеров).
3. Гены-кандидаты, ассоциированные с показателями качества молока у овец ( Ovis aries L.) породы лакон (КФХ Николаев М.И., Краснодарский край, 2022 год)
|
Признак |
Хромосома SNP |
Ген |
|
|
МНЖК, ТЖК |
1-я |
oar3_OAR1_10731857 |
PSMB2 |
|
МДБ, β-казеин |
oar3_OAR1_26966899 |
GPX7 |
|
|
МДБ |
oar3_OAR1_39850348 |
UBE2U |
|
|
oar3_OAR1_40315643 |
JAK1 |
||
|
МДБ, β-казеин |
oar3_OAR1_55047459 |
PTGFR |
|
|
МДБ |
oar3_OAR1_58721514 |
ADGRL2 |
|
|
НЖК |
oar3_OAR1_214967961 |
SPATA16 |
|
|
ПНЖК |
oar3_OAR1_222968891 |
SI |
|
|
МДЖ |
oar3_OAR1_222567308 |
SI |
|
|
МДБ, β-казеин |
oar3_OAR1_234111845 |
GPR149 |
|
|
НЖК |
oar3_OAR1_252980839 |
NCK1 |
|
|
ТЖК |
s39199.1 |
TDRP a |
|
|
ПНЖК, ТЖК |
2-я |
oar3_OAR2_100941856 |
ACO1 |
|
МДЖ, ПНЖК, НЖК |
oar3_OAR2_101399457 |
||
|
МНЖК |
oar3_OAR2_137345624 |
PDK1 |
|
|
МДЖ |
OAR2_166814077.1 |
STAM2 |
|
|
МДЖ |
oar3_OAR2_240690950 |
CATSPER4 , PAFAH2 , PAQR7 |
|
|
oar3_OAR2_240701010 oar3_OAR2_241044500 oar3_OAR2_241055039 |
|||
|
ПНЖК НЖК |
oar3_OAR2_240690950 |
||
|
МДБ, β-казеин |
oar3_OAR2_241478119 |
MACO1 |
|
|
oar3_OAR2_241480853 |
|||
|
НЖК |
3-я |
oar3_OAR3_58715245 |
CD8B , CD8A , SMYD1 , FABP1 |
|
МДБ, β-казеин |
oar3_OAR3_94997846 |
SFXN5 a, EMX1 , RAB11 , FIP5 |
|
|
s48071.1 oar3_OAR3_95001099 |
|||
|
МДБ, β-казеин |
oar3_OAR3_127246410 oar3_OAR3_127246743 OAR3_135788851.1 oar3_OAR3_127881773 oar3_OAR3_127885301 oar3_OAR3_128271531 |
EPYC |
|
|
МДЖ |
4-я |
oar3_OAR4_6603407 |
ZPBP a |
|
ПНЖК |
5-я |
oar3_OAR5_404672 |
FLT4 |
|
ТЖК |
oar3_OAR5_12170540 |
PIN1 |
|
|
МДБ, β-казеин |
oar3_OAR5_92684368 |
GPR150 |
|
|
МДБ, β-казеин |
6-я |
OAR6_34500087_X.1 oar3_OAR6_30821158 |
BMPR1B a, PDLIM5 |
|
ТЖК |
8-я |
oar3_OAR8_78424209 |
OPRM1 |
|
НЖК |
oar3_OAR8_88857890 |
TBXT, SFT2D1 |
|
|
МНЖК |
9-я |
oar3_OAR9_56200041 |
HEY1 |
|
ПНЖК |
11-я |
oar3_OAR11_60396583 |
ABCA5 , ABCA10 |
|
МДБ, β-казеин |
12-я |
oar3_OAR12_1463246 |
LAX1 |
|
МДБ, β-казеин |
13-я |
oar3_OAR13_17148993 oar3_OAR13_17153638 oar3_OAR13_17155105 oar3_OAR13_17156335 OAR13_19523580.1 |
ACBD5 |
|
oar3_OAR13_17168795 |
|||
|
oar3_OAR13_17171855 oar3_OAR13_17176409 |
|||
|
oar3_OAR13_17180926 |
|||
|
oar3_OAR13_17207481 oar3_OAR13_17218518 |
|||
Продолжение таблицы 3
|
oar3_OAR13_17220761 |
|||
|
МДБ НЖК ТЖК |
15-я |
oar3_OAR13_17267663 oar3_OAR13_17280407 OAR13_19692036.1 oar3_OAR13_30141706 oar3_OAR13_73566109 oar3_OAR15_33907633 |
PTER PI3, WFDC12 , SLPI GRAMD1B a, SCN3B |
|
МДБ, β-казеин |
16-я |
oar3_OAR16_11651869 |
PIK3R1 |
|
МДЖ, НЖК МНЖК |
17-я |
oar3_OAR16_53250752 oar3_OAR17_69645464 |
CDH18 a OSBP2 a, PLA2G3 |
|
НЖК |
21-я |
oar3_OAR17_69649241 oar3_OAR21_23194924 |
NAV2 , CSRP3 |
|
β-Казеин |
24-я |
oar3_OAR24_5898966 |
RBFOX1 a |
|
НЖК ТЖК МДЖ, ПНЖК, НЖК |
26-я |
oar3_OAR24_19579007 oar3_OAR24_28998229 oar3_OAR26_32527902 |
ZP2, CRYM SBDS STAR , DDHD2 , LSM1 , PLPP5 , LETM2 , |
|
ПНЖК |
oar3_OAR26_32571082 oar3_OAR26_32618174 oar3_OAR26_39386944 |
FGFR1 OXSM |
|
Примечани е. Гены, содержащие в своем составе обнаруженные SNP, помечены верхним индексом a; МДБ — массовая доля белка в молоке, МДЖ — массовая доля жира, НЖК — насыщенные жирные кислоты, МНЖК — мононенасыщенные жирные кислоты, ПНЖК — полиненасыщенные жирные кислоты, ТЖК — трансизомеры жирных кислот
Так, ассоциация гена CD8B с обхватом груди у коммерческих пород зарегистрирована под четырьмя номерами (QTL:55219-QTL:255222), с длиной тела — под пятью (QTL:255214-QTL:255218), с массой тела — под шестью (QTL:255208-QTL:255213) и с обхватом голени — под одним номером (QTL:255223) (21). Ассоциация гена BMPR1B с репродуктивной функцией, а именно с многоплодием, у короткохвостых ханьских овец и у овец пород исландская, финская, романовская и тексель зарегистрирована под четырьмя номерами (QTL:13975 и QTL:154661-QTL:154663), с живой массой у овец пород кира блэк и мериноландшафт — под одним (QTL:264134) (22).
Наибольшее число выявленных SNPs — 15, ассоциированных с массовой долей белка и содержанием β-казеина в молоке овец породы лакон, попадает в область гена ACBD5 (OAR13). Ацил-КоА-связывающий домен 5 (ген ACBD 5) представляет собой пероксисомальный белок с ацил-КоА-свя-зывающим доменом в N-концевой области, биологическая функция которого заключается в регуляции метаболизма жирных кислот (GO: 0006631). Мы выявили ассоциацию этого гена с массовой долей молочного белка и количеством β-казеина, что, по-видимому, обусловлено тесной взаимосвязью между содержанием белка и жира в молоке, обнаруженной у многих видов животных (23, 24). Кроме того, показано, что у коз ген ACBD5 также вовлечен в иммунный ответ: его продукт функционирует как рецептор, регулирующий процессы деградации поврежденных пероксисом и белков. Мутация в гене ACBD5 приводила к возникновению тромбоцитопении — заболевания, при котором число тромбоцитов опускалось ниже допустимого уровня (25).
Еще одним геном, который в нашем исследовании демонстрировал связь с массовой долей белка и содержанием β-казеина, а в других работах — с показателем, связанным с синтезом жира, был ген GPX7 (глутатионпероксидаза 7) . J. Yang с соавт. (26) на основе полногеномного секвенирования и анализа ассоциаций выявили высокодостоверную связь гена GPX7 с метаболизмом арахидоновой кислоты у китайских локальных пород овец.
Некоторые гены, демонстрировавшие связь с белковыми компонентами молока у овец породы лакон, в других исследованиях показывали ассоциации с экстерьерными признаками овец и коз. Так, в работе M. Yang с соавт. (27) ген JAK1 (янус-киназа 1) был значимо связан с косой длиной тела, глубиной и шириной груди у овец китайской породы ху (р < 0,05), а также с косой длиной тела, высотой креста и шириной груди у тощехвостых овец породы хань (р < 0,05). При этом описанная биологическая функция JAK1 заключается в передаче сигналов альфа-, бета-, гамма-интерферонам и цитокинам, что служит основным элементом иммунного и воспалительного ответа интерлейкина-6 (28). Для остальных генов, функции которых связаны с иммунными реакциями — LAX1, PI3, CD8B и SLPI, имеются сведения лишь о связи гена CD8B (кластер дифференцировки 8 бета) с фенотипическими показателями у овец, а именно с массой туши и экстерьерными признаками, отвечающих за размер тела (21). Ген UBE2U, показавший связь с массовой долей молочного белка у овец породы лакон, ранее был обнаружен у карачаевских коз как связанный с широтными промерами тела (29).
Следующая рассматриваемая группа — гены, которые показали ассоциации с синтезом молочного жира и жирных кислот у овец. При этом в указанную группу мы включили те гены, функции которых описаны и имеются сведения об их влиянии на другие признаки продуктивности животных. Это гены ACO1 , FABP1 , ACBD5 , OXSM и ZP2.
В работе J. Wang с соавт. (30) при изучении генетического разнообразия локальных пород овец северо-восточной части Таримского бассейна на основе определения индекса фиксации ( F ST ) в группе генов цитратного цикла (цикл Кребса, или цикл лимонной кислоты) был идентифицирован ген ACO1 (аконитаза 1) . Это один из основных генов в регуляции дыхательного метаболизма клеток, что напрямую определяет активность синтеза аминокислот, углеводов, жирных кислот (30). Поэтому связь гена ACO1 с массовой долей молочного жира и количеством жирных кислот (ПНЖК, ТЖК, НЖК) представляется вполне логичной и подтверждает непосредственное влияние ACO1 на формирование этих показателей.
Функциональная роль гена FABP1 (белок, связывающий жирные кислоты 1) хорошо известна и впервые была подробно описана для фенотипических признаков, связанных с жиронакоплением в организме человека (31). Установлена ключевая роль гена FABP1 в синтезе, отложении и деградации жира и различных классов жирных кислот. В дальнейшем обнаружили гомологию с такими же функциями у свиней (32), что позволило предложить ген FABP1 в качестве генетического маркера качественных характеристик свинины — с высоким или низким количеством межмышечных и межпучковых жировых включений. У овец были идентифицированы гены FABP5 и FABP7 , которые, как и ген FABP1 , относятся к одному семейству генетических детерминант белков, связывающих жирные кислоты (FABP). Установлено, что ген FABP5 контролирует метаболизм липидов, а ген FABP7 участвует во внутриклеточном транспорте полиненасыщенных жирных кислот (33, 34). Показано, что в семействе FABP существует значительная структурная гомология генов не только между изоформами белков у человека, но и между их изоформами у разных видов животных, что указывает на дупликацию и дивергенцию одного предкового гена с липид-связывающими свойствами кодируемого им белкового продукта (35).
Ген OXSM (3-оксоацил-АСР-синтаза митохондриальная) связан с метаболизмом белков через регуляцию процессов удлинения цепей жирных кислот в митохондриях. Этой биологической функцией L. Zhang с соавт. (36) обосновывают выявленную методом GWAS у овец породы сунит ассоциацию с массой ягнят в раннем онтогенезе.
Гены семейства ZP контролируют синтез четырех гликопротеинов зоны пеллюцида (zona pellucida, ZP) — ZP1, ZP2, ZP3, ZP4, которые играют 744
решающую роль во время оогенеза, оплодотворения и преимплантационного развития эмбрионов (37). За последние десятилетия проведены обширные исследования по оценке роли гликопротеинов ZP в опосредованном распознавании гамет во время оплодотворения. Известно, что гены ZP3 и ZP2 кодируют первичные и вторичные рецепторы сперматозоидов у мышей (38). S. Chen с соавт. (39) идентифицировали генетические вариации гена ZP2 на внутри- и межвидовом уровнях у крупного рогатого скота и доказали определяющую роль ZP2 в репродуктивной изоляции между видами. По-види-мому, вовлеченность выявленных нами генов ZP2 и ZPBP в функции, связанные с лактацией и синтезом полиненасыщенных жирных кислот у молочных овец обусловлена связью генов семейства ZP с функциями воспроизводства.
Остальные выявленные нами гены в соответствии биологическими функциями распределились в следующие группы: 1-я — формирование и регуляция функций иммунной системы (гены GPX7 , PTGFR , LAX1 , PI3 , WFDC12 , SLPI ); 2-я — формирование и развитие нервной системы, включая центральную нервную систему (гены GPR149 , MACO1 , EMX1 , NAV2 , RBFOX1 ); 3-я — клеточный метаболизм и транспорт веществ (гены PSMB2 , UBE2U , SI , NCK1 , PDK1 , PAQR7 , CD8A , SFXN5 , RAB11FIP5 , PIN1 , GPR150 , SFT2D1 , ABCA5 , ABCA10 , PTER , GRAMD1B , SCN3B , PIK3R1 , OSBP2 , PLA2G3 , STAR , DDHD2 , PLPP5 ); 4-я — формирование и развитие органов и скелетно-хрящевой структуры (гены ADGRL2 , MACO1 , EMX1 , EPYC , FLT4 , BMPR1B , PDLIM5 , TBXT , HEY1 , CDH18 , CSRP3 , SBDS , FGFR1 ); 5-я — процессы оогенеза, оплодотворения и раннего развития эмбрионов (гены SPATA16 , TDRP , CATSPER4 ).
Мы планируем продолжить подобные исследования, в том числе на других породах овец, чтобы выявить общие однонуклеотидные замены и гены-кандидаты, перспективные для селекционных программ в молочном овцеводстве.
Итак, при полногеномном ассоциативном исследовании идентифицированы 64 полногеномных (p < 1,06½10-6) и 270 предположительных (p < 2,11½10-5) SNP, связанных с качественными характеристиками молока у овец породы лакон. Наибольшее число полногеномных SNPs — 14, 6 и 13 идентифицировано соответственно на 1-й, 3-й и 13-й хромосомах. Ассоциации с массовой долей молочного белка и β-казеина демонстрировали 22 и 19 полногеномных SNPs, которые распределились между пятью (OAR1, OAR3, OAR12, OAR13 и OAR26) и четырьмя (OAR1, OAR3, OAR13 и OAR26) хромосомами, с массовой долей жира — 6 SNP на трех хромосомах (OAR1, OAR3, OAR26), с содержанием полиненасыщенных, насыщенных и мононенасыщенных жирных кислот — 7, 5 и 4 SNPs соответственно на четырех (OAR1, OAR3, OAR5 и OAR17) и трех (OAR1, OAR24, OAR26 и OAR1, OAR6, OAR17) хромосомах. Два SNPs высокой значимости (oar3_OAR1_24401030 и oar3_OAR2_241055039) были общими для трех признаков — массовой доли молочного белка, β-казеина и массовой доли молочного жира. Структурная аннотация геномных регионов выявила 61 ген с описанными биологическими функциями, из которых 11 демонстрировали связь с другими фенотипическим признаками овец. Выявленные гены в соответствии с терминами генной онтологии были вовлечены в формирование и регуляцию функций иммунной системы (GPX7, JAK1, CD8B), в клеточный метаболизм и транспорт веществ (UBE2U, ACO1, FABP1, ACBD5), в формирование и развитие внутренних органов и скелетно-хрящевой системы (BMPR1B, OXSM), в процессы оогенеза, оплодотворения и раннего развития эмбрионов (ZPBP, ZP2). Таким образом, ассоциативные иссле- дования на овцах породы лакон, выполненные в нашей стране впервые, позволили получить новые данные для понимания генетических механизмов формирования признаков продуктивности у молочных овец. Эти данные при успешной валидации на разных породах молочных овец могут в перспективе использоваться в программы селекции.
Список литературы Полногеномный поиск ассоциаций однонуклеотидных замен с показателями качества молока у овец (Ovis aries L.) породы лакон
- Popescu A., Stanciu M., Antonie I. Livestock and milk and meat production in the top five EU countries rearing sheep and goats, 2012-2021. Management. Economic Engineering in Agriculture and rural development, 2022, 22(3): 2284-7995.
- Шувариков А.С, Канина К.А., Робкова Т.О., Юрова Е.А. К вопросу оценки состава и свойств овечьего, козьего и коровьего молока. Овцы, козы, шерстяноедело, 2018, 1: 20-22.
- Dettori M.L., Pazzola M., Noce A., Landi V., Vacca G.M. Variations in casein genes are associated with milk protein and fat contents in Sarda goats (Capra hircus), with an important role of CSN1S2 for milk yield. Animals, 2023, 14(1): 56 (doi: 10.3390/ani14010056)
- Кручинин А.Г., Бигаева А.В., Туровская С.Н., Илларионова Е.Е. Сравнение полиморфизма генов молочных белков козьего и овечьего молока: мировой опыт. Пищевая промышленность, 2020, 8: 36-40 (doi: 10.24411/0235-2486-2020-10083).
- Arranz J.J., Gutiérrez-Gil B. Detection of QTL underlying milk traits in sheep: An update. In: Milk production. Advanced genetic traits. Сellular mechanism. Animal management health. InTech, 2012: 97-126 (doi: 10.5772/50762).
- Wang P., Li X., Zhu Y., Wei J., Zhang C., Kong Q., Nie X., Zhang Q., Wang Z. Genome-wide association analysis of milk production, somatic cell score, and body conformation traits in Holstein cows. Frontiers in Veterinary Science, 2022, 9: 932034 (doi: 10.3389/fvets.2022.932034).
- Getaneh M., Wondimagegn K. Candidate genes associated with economically important traits in dairy goats. Cogent Food & Agriculture, 2022, 8(1): 2149131 (doi: 10.1080/23311932.2022.2149131).
- Zhang T.Y., Zhu C.Y., Du L.X., Zhao F.P. Advances in genome-wide association studies for important traits in sheep and goats. Yi Chuan, 2017, 39(6): 491-500 (doi: 10.16288/j.yczz.17-021).
- Marina H., Gutiérrez-Gil B., Esteban-Blanco C., Suárez-Vega A., Pelayo R., Arranz J.J. Analysis of whole genome resequencing datasets from a worldwide sample of sheep breeds to identify potential causal mutations influencing milk composition traits. Animals, 2020, 10(9): 1542 (doi: 10.3390/ani10091542).
- Pulina G., Milan M.J., Lavin M.P., Theodoridis A., Morin E., Capote J. Thomas D.L., Francesconi A.H.D., Caja G. Invited review: Current production trends, farm structures, and economics of the dairy sheep and goat sectors. Journal Dairy Science, 2018, 101: 6715-6729 (doi: 10.3168/jds.2017-14015).
- Mucha S., Tortereau F., Doeschl-Wilson A., Rupp R., Conington J. Animal Board invited review: meta-analysis of genetic parameters for resilience and efficiency traits in goats and sheep. Animal, 2022, 16(3): 100456 (doi: 10.1016/j.animal.2022.100456).
- Li H., Wu X.-L., Tait R.G., Jr, Bauck S., Thomas D.L., Murphy T.W., Rosa G.J.M. Genome-wide association study of milk production traits in a crossbred dairy sheep population using three statistical models. Animal Genetics, 2020, 51: 624-628 (doi: 10.1111/age.12956).
- Jawasreh K., Haddad N., Abulubbad A., Al-Amareen A. Genome-wide association study for milk production of Awassi sheep in Jordan. Journal of the Saudi Society of Agricultural Sciences, 2022, 21(5): 302-309 (doi: 10.1016/j.jssas.2021.09.006).
- Sutera A.M., Tolone M., Mastrangelo S., Di Gerlando R., Sardina M. T., Portolano B., Pong-Wong R., Riggio V. Detection of genomic regions underlying milk production traits in Valle del Belice dairy sheep using regional heritability mapping. Journal of Animal Breeding and Genetics, 2021, 138(5): 552-561 (doi: 10.1111/jbg.12552).
- Marina H., Pelayo R., Suárez-Vega A., Gutiérrez-Gil B., Esteban-Blanco C., Arranz J.J. Genome-wide association studies (GWAS) and post-GWAS analyses for technological traits in Assaf and Churra dairy breeds. Journal of Dairy Science, 2021, 104(11): 11850-11866 (doi: 10.3168/jds.2021-20510).
- Argyriadou A., Michailidou S., Vouraki S., Tsartsianidou V., Triantafyllidis A., Gelasakis A., Banos G., Arsenos G. A genome-wide association study reveals novel SNP markers associated with resilience traits in two Mediterranean dairy sheep breeds. Frontiers in Genetics, 2023, 14: 1294573 (doi: 10.3389/fgene.2023.1294573).
- Li R., Ma Y., Jiang L. Review: Research progress of dairy sheep milk genes. Agriculture, 2022, 12: 169 (doi: 10.3390/agriculture12020169).
- Duggal P., Gillanders E.M., Holmes T.N., Bailey-Wilson J.E. Establishing an adjusted P-value threshold to control the family-wide type 1 error in genome wide association studies. BMC Genomics, 2008, 9(1): 516 (doi: 10.1186/1471-2164-9-516).
- Zhou X., Stephens M. Genome-wide efficient mixed-model analysis for association studies. Nature Genetics, 2012, 44(7): 821-824 (doi: 10.1038/ng.2310).
- Turner S. qqman: an R package for visualizing GWAS results using Q-Q and manhattan plots. Journal of Open Source Software, 2018, 3: 731 (doi: 10.21105/joss.00731).
- Lin C., Li F., Zhang X., Zhang D., Li X., Zhang Y., Zhao Y., Song Q., Wang J., Zhou B., Cheng J., Xu D., Li W., Zhao L., Wang W. Expression and polymorphisms of CD8B gene and its associations with body weight and size traits in sheep. Animal Biotechnology, 2023, 34(4): 1214-1222 (doi: 10.1080/10495398.2021.2016432).
- Akhatayeva Z., Bi Y., He Y., Khan R., Li J., Li H., Pan C., Lan X. Survey of the relationship between polymorphisms within the BMPR1B gene and sheep reproductive traits. Animal Biotechnology, 2023, 34(3): 718-727 (doi: 10.1080/10495398.2021.1979023).
- Saranjam N., Farhoodi M., Ghasem A., Mohammadsadegh M., Farzaneh N. Associations between milk fat, protein and fat-to-protein ratio with some reproductive indices in dairy cows. The Indian Journal of Animal Sciences, 2020, 90: 764-767 (doi: 10.56093/ijans.v90i5.104622).
- Guliński P., Socha S. The relationship between milk fat to protein ratio and selected production traits of Polish Holstein-Friesian cows. Animal Science Papers and Reports, 2021, 39: 213-223.
- Khanduri A., Sahu A.R., Wani S.A., Khan R.I.N., Pandey A., Saxena S., Malla W.A., Mondal P., Rajak K.K., Muthuchelvan D., Mishra B., Sahoo A.P., Singh Y.P., Singh R.K., Gandham R.K., Mishra B.P. Dysregulated miRNAome and proteome of PPRV infected goat PBMCs reveal a coordinated immune response. Frontiers in Immunology, 2018, 9: 2631 (doi: 10.3389/fimmu.2018.02631).
- Yang J., Li W.R., Lv F.H., He S.G., Tian S.L., Peng W.F., Sun Y.W., Zhao Y.X., Tu X.L., Zhang M., Xie X.L., Wang Y.T., Li J.Q., Liu Y.G., Shen Z.Q., Wang F., Liu G.J., Lu H.F., Kantanen J., Han J.L., Liu M.J. Whole-genome sequencing of native sheep provides insights into rapid adaptations to extreme environments. Molecular Biology and Evolution, 2016, 33(10): 2576-2592 (doi: 10.1093/molbev/msw129).
- Yang M., Zhao W., Wang Z., Liu J., Sun X., Wang S. Detection of key gene InDels in JAK/STAT pathway and their associations with growth traits in four Chinese sheep breeds. Gene, 2023, 888: 147750 (doi: 10.1016/j.gene.2023.147750).
- Chaimowitz N.S., Forbes L.R. Human diseases caused by impaired signal transducer and activator of transcription and Janus kinase signaling. Current Opinion Pediatrics, 2019, 31(6): 843-850 (doi: 10.1097/MOP.0000000000000841).
- Easa A., Selionova M., Aibazov M., Mamontova T., Sermyagin A., Belous A., Abdelmanova A., Deniskova T., Zinovieva N. Identification of genomic regions and candidate genes associated with body weight and body conformation traits in Karachai goats. Genes, 2022, 13(10): 1773 (doi: 10.3390/genes13101773).
- Wang J., Suo J., Yang R., Zhang C.L., Li X., Han Z., Zhou W., Liu S., Gao Q. Genetic diversity, population structure, and selective signature of sheep in the northeastern Tarim Basin. Frontiers in Genetics, 2023, 14: 1281601 (doi: 10.3389/fgene.2023.1281601).
- Smathers R.L., Petersen D.R. The human fatty acid-binding protein family: evolutionary divergences and functions. Human Genomics, 2011, 5(3): 170-191 (doi: 10.1186/1479-7364-5-3-170).
- Yang R., Zhou D., Yan Z., Zhao Z., Wang Y., Li J., Ren L., Xie L., Wang X. Fatty acid binding protein 1 and fatty acid synthetase over-expression have differential effects on collagen III synthesis and cross-linking in Zongdihua pig primary adipocytes. PLoS ONE, 2023, 8(5): e0270614 (doi: 10.1371/journal.pone.0270614).
- Listyarini K., Sumantri C., Rahayu S., Islam M.A., Akter S.H., Uddin M.J., Gunawan A. Hepatic transcriptome analysis reveals genes, polymorphisms, and molecules related to lamb tenderness. Animals, 2023, 13(4): 674 (doi: 10.3390/ani13040674).
- Gunawan A., Listyarini K., Harahap R.S., Jakaria Roosita K., Sumantri C., Inounu I., Akter S.H., Islam M.A., Uddin, M.J. Hepatic transcriptome analysis identifies genes, polymorphisms and pathways involved in the fatty acids metabolism in sheep. PLoS ONE, 2021, 16(12): e0260514 (doi: 10.1371/journal.pone.0260514).
- Ishimura S., Furuhashi M., Watanabe Y., Hoshina K., Fuseya T., Mita T., Okazaki Y., Koyama M., Tanaka M., Akasaka H., Ohnishi H., Yoshida H., Saitoh S., Miura T. Circulating levels of fatty acid-binding protein family and metabolic phenotype in the general population. PLoS ONE, 2013, 8(11): e81318 (doi: 10.1371/journal.pone.0081318).
- Zhang L., Liu J., Zhao F., Ren H., Xu L., Lu J., Zhang S., Zhang X., Wei C., Lu G., Zheng Y., Du L. Genome-wide association studies for growth and meat production traits in sheep. PLoS ONE, 2013, 8(6): e66569 (doi: 10.1371/journal.pone.0066569).
- Wassarman P.M., Litscher E.S. Mammalian fertilization: the egg’s multifunctional zona pellucida. The International Journal of Developmental Biology, 2008, 52(5-6): 665-676 (doi: 10.1387/ijdb.072524pw).
- Bleil J.D., Greve J.M., Wassarman P.M. Identification of a secondary sperm receptor in the mouse egg zona pellucida: role in maintenance of binding of acrosome-reacted sperm to eggs. Developmental Biology, 1988, 128(2): 376-385 (doi: 10.1016/0012-1606(88)90299-0).
- Chen S., Costa V., Beja-Pereira A. Evolutionary patterns of two major reproduction candidate genes (Zp2 and Zp3) reveal no contribution to reproductive isolation between bovine species. BMC Evolutionary Biology, 2011, 11: 24 (doi: 10.1186/1471-2148-11-24).


