Получение нового продуцента рекомбинантного афлатоксин-деградирующего фермента с помощью гетерологичной экспрессии в Pichia pastoris
Автор: Синельников И.Г., Зоров И.Н., Денисенко Ю.А., Микитюк О.Д., Синицын А.П., Щербакова Л.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика, геномика, генетическая инженерия
Статья в выпуске: 6 т.57, 2022 года.
Бесплатный доступ
Загрязнение микотоксинами наносит значительный экономический ущерб пищевой и кормовой промышленности и серьезно угрожает здоровью человека и животных из-за мутагенности, онкогенности и других опасных свойств этих вторичных метаболитов грибов. Метод ферментативной деградации является эффективной и экологически приемлемой альтернативой химическим методам деконтаминации сельскохозяйственного сырья и пищевой продукции. В проведенном нами исследовании система экспрессии рекомбинантных белков, которую мы адаптировали для повышения копийности дрожжевых гетерологичных генов в хромосоме дрожжей Pichia pastoris, была впервые применена для получения фермента ADTZ-оксидазы из Armillaria tabescens , разлагающей афлатоксин В1. Cинтетический ген adtz указанного фермента был интегрирован в геном штамма P. pastoris GS115 под контролем промотора глицеральдегид-3-фосфатдегидрогеназы . Для амплификации гена adtz сконструировали олигонуклеотидные последовательности, к 5´-концу которых добавили специфические сайты рестрикции HindIII и NotI. После получения на основе вектора pPIG-1 плазмиды pPIG-ADTZ, содержащей ген adtz , ее линеаризовали посредством расщепления эндонуклеазой рестрикции ApaI и методом электропорации трансформировали клетки реципиентного штамма P. pastoris GS115. Полученные трансформанты дрожжевых клеток отбирали на среде Yeast Extract-Peptone-Dextrose (YPD) с антибиотиком. Вставку целевого гена подтверждали с помощью ПЦР-амплификации, рестрикционным анализом и секвенированием по Сэнгеру. В результате получили 54 трансформанта штамма P. pastoris GS115, содержавшие вставку целевого гена adtz , и среди них отобрали наиболее активный продуцент - клон ADTZ-14 (выход общего внеклеточного белка 2,1 мг/мл). Секретируемый этим клоном рекомбинантный фермент ADTZ представлял собой мономерный белок с молекулярной массой 78±3 кДа, обладающий высокой аффинностью к афлатоксину В1 (АФВ1). Сохранение функциональных свойств полученного белка было подтверждено экспериментами по оценке его способности деградировать АФВ1 при кратковременной и длительной инкубации. Так, под воздействием ADTZ концентрация АФВ1, добавленного в бесклеточную культуральную жидкость (КЖ) клона ADTZ-14, снижалась на 14 % уже через 2 ч инкубации при 40 °С. После более длительной инкубации при 30 °С содержание добавленного АФВ1 (5 мг/мл) в бесклеточной КЖ было на 50 % ниже, чем в контроле (КЖ нетрансформированного штамма P. pastoris GS115), через 3 сут, а через 5 сут инкубации в тех же условиях деградация токсина достигала 80 %. Полученные данные свидетельствуют о достаточно высоком биотехнологическом потенциале нового продуцента рекомбинантного белка ADTZ и целесообразности дальнейших исследований по созданию на его основе ферментного препарата для деконтаминации растениеводческой продукции, загрязненной АФВ1.
Афлатоксин в1, микотоксины, энзиматическая деградация, adtz из armillaria tabescens, синтетический ген adtz, рекомбинантные белки, гетерологичная экспрессия, pichia pastoris
Короткий адрес: https://sciup.org/142237381
IDR: 142237381 | УДК: 636.085.19:573.6.086.83:577.21 | DOI: 10.15389/agrobiology.2022.6.1166rus
Текст научной статьи Получение нового продуцента рекомбинантного афлатоксин-деградирующего фермента с помощью гетерологичной экспрессии в Pichia pastoris
Афлатоксины (группа сходных по структуре вторичных метаболитов грибов рода Aspergillius , широко распространенных в природе) известны как опасные микотоксины, загрязняющие корма и другую сельскохозяйственную продукцию (1-4). В настоящее время идентифицировано более 20 афлатоксинов (АФ), их производных и близкородственных соединений (5). Среди них наиболее серьезную озабоченность в связи с контаминацией кормов для животноводства и птицеводства вызывают АФ В- и G-типов (6, 7). Эти микотоксины представляют собой производные дифуранокумарина, имеющие в своем составе связанную с кумариновым ядром бифурановую структуру и циклопентановое (у АФ В-типа) или лактонное кольцо (у АФ G-типа) (8, 9). Из-за токсичности, карценогенности и мутагенности этих соединений, а также из-за их устойчивости к термическим обработкам (10, 11)
корма и другая продукция растениеводства, загрязненная АФ выше допу-
* Исследования поддержаны Российским научным фондом (проект РНФ № 22-16-00153).
стимых гигиеническими регламентами концентраций, не годится для непосредственного использования или дальнейшей переработки в пищевые продукты. Во всем мире загрязнение этими микотоксинами, особенно АФB 1 , который по своей гепатотоксичности и опасности для теплокровных превосходит все остальные АФ (6, 7), наносит серьезный экономический ущерб как сельскому хозяйству, так и пищевой промышленности, а также создает проблемы для здоровья людей (1, 3).
Для деконтаминации используются физические, химические и микробиологические методы, которые, однако, имеют ряд известных ограничений (1, 12), поэтому постоянно ведется поиск иных эффективных, экологически безопасных и не влияющих на качество агропродукции средств и способов деградации АФ и детоксикации. С этой точки зрения весьма многообещающим представляется подход, основанный на способности ряда грибов (13-15) и бактерий (16-19) синтезировать ферменты, трансформирующие АФ до нетоксичных или менее токсичных соединений (20, 22). Использование бесклеточных препаратов, содержащих такие ферменты, позволяет избежать проблем, которые могут возникать при применении самих продуцентов (например, ухудшения органолептических свойств обрабатываемых продуктов, снижения их пищевой ценности). Кроме того, ферментные препараты технологически более удобны для обработки кормов (23) и, в отличие от препаратов для пищевой промышленности, не требуют дорогостоящей многоступенчатой очистки целевого продукта.
Известно, что источниками ферментов, деградирующих и детоксицирующих АФ, могут быть некоторые ксилотрофные базидиомицеты родов Ple-urotus (24, 25), Phanerochaete и Armillaria (26-28). Так, из мицелия одного из представителей этих грибов — опенка дубового ( Armillaria tabescens ) с помощью гидрофобной и металл-хелатной хроматографии был выделен фермент с оксидазной активностью (28), названный авторами афлатоксин-детоксифи-зим (aflatoxin-detoxifizyme, ADTZ). Оказалось, что он способен катализировать раскрытие и последующий гидролиз дифуранового кольца (29) — структуры, связанной с токсичностью АФ B-типа. Дальнейшие исследования показали, что ADTZ представляет собой мономерный белок с молекулярной массой 76 кДа, обладающий высокой аффинностью к АФВ 1 (29). При контакте с ADTZ токсичность и мутагенность АФВ 1 значительно снижались (28, 30).
Эти данные свидетельствуют о перспективности разработки детоксицирующих препаратов, содержащих ADTZ. Однако их созданию прежде всего препятствует отсутствие доступной технологии получения внутриклеточного ADTZ из мицелия A. tabescens и отчасти тот факт, что для глубинного культивирования A. tabescens необходимы жидкие среды сложного состава, включающие весьма специфические и дорогостоящие компоненты (31), или требуется многостадийная процедура ферментации (28). Указанные препятствия могли бы быть преодолены при использовании гетерологичной системы экспрессии и создании доступного продуцента рекомбинантного белка ADTZ. Тем не менее до сих пор нет подходящей системы, позволяющей получить внеклеточный гетерологичный ADTZ в количестве, достаточном для его применения с целью деконтаминации растениеводческой продукции. В то же время в ряде современных работ (32, 33) продемонстрировано успешное использование клеток дрожжей Pichia pastoris в качестве реципиентов для гетерологичной экспрессии.
Ранее мы провели адаптацию системы экспрессии в P. pastoris, заключавшуюся в модификации интеграционного вектора для повышения ко-пийности гетерологичных генов в хромосоме дрожжей (интеграционный вектор и его получение запатентованы) (34). В настоящем исследовании этот подход впервые использован для создания нового продуцента афлаток- син-деградирующего фермента.
Нашей целью была оптимизация и применение указанной системы для гетерологичной экспрессии ADTZ в Pichia pastoris GS115, а также оценка способности препарата бесклеточной культуральной жидкости (КЖ) полученного штамма P. pastoris ADTZ-14, содержащего внеклеточный рекомбинантный фермент ADTZ, деградировать АФВ 1 .
Методика . Для экспрессии гена adtz , кодирующего афлатоксин-де-токсифизим , использовали штамм дрожжей Pichia pastoris GS115 (syn. Koma-gataellaphaffii ) («Thermo Fisher Scientific», США). Дрожжевые клетки культивировали в течение 3 сут при 30 ° С на жидкой среде YPD (г/л: глюкоза — 20,0; дрожжевого экстракт — 10,0; мясной пептон — 20,0). Для получения плазмидной ДНК штамм Escherichia сoli XL1-Blue («Agilent», США) выращивали при 37 ° С на среде Лурия-Бертрани (г/л: триптон — 10, дрожжевой экстракт — 5, NaCl — 5; pH 7,2-7,5). Для экспрессии ADTZ использовали плазмиду pPIG-1 (34). Ген adtz , кодирующий фермент деградации афлатоксина у A. tabescens (GenBank AY941095.1), был синтезирован в ЗАО «Евроген» (Россия) с учетом кодонового состава у P. pastoris .
Амплификацию гена adtz осуществляли методом ПЦР. Смесь для ПЦР (50 мкл) содержала 1½ буфер с 3 мМ MgCl 2 и 5 ед. Taq полимеразы («NEB», Великобритания), 0,2 мкМ олигонуклеотидов ADTZ-fwd (5´-gaagcttctATGG-CTACTACAACTG-3´) и ADTZ-rev (5´-cgcggccgcTTAC-AATCTTCTCTC-3´) и 0,1 нг ДНК в качестве матрицы. Реакцию проводили на амплификаторе T-100 («Bio-RAD», США) при следующих условиях: 95 ° С в течение 15 с, 62 ° С в течение 15 с, 72 ° С в течение 120 с (25 циклов). Результаты амплификации оценивали методом электрофореза в 1 % агарозном геле в камере Sub-Cell GT Cell («Bio-RAD», США).
Продукт амплификациии — вектор pPIG-1 расщепляли с помощью эндонуклеаз рестрикции HindIII и NotI согласно рекомендациям производителя («NEB», Великобритания).
Обработанные фрагменты лигировали Т4 ДНК-лигазой (ЗАО «Евроген», Россия), и смесью (2 мкл) трансформировали клетки Escherichia coli XL1-blue («Agilent», США) методом теплового шока. Трансформанты отбирали на агаризованной среде Лурия-Бертрани, содержащей селективный антибиотик ампициллин (100 мкг/мл). Из ампициллин-резистентных трансформантов выделяли плазмиду pPIG-ADTZ, используя набор Plasmid Miniprep (ЗАО «Евроген», Россия). Наличие вставки целевого гена в плазмиде pPIG-ADTZ подтверждали с помощью ПЦР-амплификации, рестрикционным анализом, как описано выше, а также секвенированием по Сэнгеру. Секвенирование осуществляли в обоих направлениях с праймеров, для амплификации гена. Секвенирование генов и синтез праймеров, использованных для амплификации, были выполнены в ООО «Синтол» (Россия).
Плазмиду pPIG-ADTZ линеаризовали расщеплением эндонуклеазой рестрикции ApaI («NEB», Великобритания) в соответствии с протоколом производителя и интегрировали в P. pastoris GS115 методом электропорации (35). Селекцию трансформантов осуществляли на агаризованой среде YPD, в которую добавляли антибиотик зеоцин («Thermo Fisher Scientific», США) в концентрации 200 мкг/мл. Из колоний, устойчивых к антибиотику, выделяли ДНК (36) и проверяли наличие вставки ADTZ с помощью ПЦР.
Рекомбинантный белок ADTZ получали при культивировании штамма-продуцента P. pastoris ADTZ-14 в 24-луночных планшетах (3 мл жидкой среды YNB, 30 ° С, аэрация 200 об/мин, 3 сут). Каждые 24 ч в лунки добавляли 40 % раствор глюкозы в 20 мМ калий-фосфатном буфере (рН 6,0) до конечной концентрации 2 %.
Анализ бесклеточной КЖ на наличие рекомбинантного ADTZ осу- ществляли методом электрофореза в полиакриламидном геле с додецилсульфатом натрия (ДНС-ПААГ, Mini-PROTEAN® Tetra, «Bio-RAD», США). Концентрацию общего белка измеряли по Лоури (37).
Кинетику деградации АФВ 1 и АФG 1 изучали в экспериментах с кратковременной инкубацией бесклеточной КЖ P. pastoris ADTZ-14 . Коммерческие препараты АФB 1 и АФG 1 (ВНИИВСГЭ, Россия) растворяли в 20 мМ Na-фосфатном буфере (рН 6,7) до конечной концентрации каждого 2,5 мкг/мл. Контроль концентрации осуществляли, используя значения молярных коэффициентов экстинкции s = 21800 и s = 17700 (при X = 362 нм) соответственно для АФB 1 и АФG 1 . КЖ трансформанта P. pastoris ADTZ-14 инкубировали с растворами токсинов в ячейках планшета автосамплера, термостатированного при 30 или 40 ° С. С помощью автосамплера из реакционной смеси каждые 30 мин в течение 2,5 ч отбирали аликвоты по 5 мкл и определяли содержание АФ методом обращенно-фазовой хроматографии на термостатированной при 30 ° С колонке Kromasil Ethernity 5-C18 (4,6x250 мм) («Akzo Nobel», Швеция), снабженной соответствующей предколонкой, используя хроматографическую систему Agilent 1200 («Agilent Technologies», США) c диодно-матричным детектированием. Хроматографическое разделение проводили в градиенте вода/ацетонитрил (от 40 % до 68 % ацетонитрила за 20 мин, детектирование при 360, 235 и 225 нм, ширина щели 8 нм). Степень деградации АФB 1 и АФG 1 оценивали по изменению площади соответствующего хроматографического пика. В качестве контроля использовали КЖ нетрансформированного штамма P. pastoris GS115.
Для оценки способности рекомбинантного фермента ADTZ деградировать АФВ 1 при длительной инкубации к 1 мл образцов КЖ P. pastoris ADTZ-14 (2,1 мг общего белка/мл) после предварительной стерилизации фильтрованием (мембраны с размером пор 0,22 мкм, «Millipore», США) добавляли 1,0 мкг АФВ 1 («Sigma-Aldrich», США), растворенного в минимальном объеме метанола. В качестве контроля использовали образцы (1 мл) КЖ нетрансформированного штамма P. pastoris GS115, в которые вносили то же количество АФВ 1 . Образцы инкубировали в течение 3 и 5 сут при рН 7,0 и 30 ° С. Содержание АФВ 1 после инкубации определяли с помощью высокоэффективной жидкостной хроматографии (ВЭЖХ) на термостатированной (27 ° С) колонке Symmetry C18 (5мкм, 150x4,6 мм) в изократическом режиме элюции (подвижная фаза метанол:вода 60:40, объем введенной пробы 10 мкл, λ = 362 нм), используя систему Waters 1525 Breeze c детектором Waters UV 2487 («Waters Corp.», США) (12, 38). Перед ВЭЖХ-анализом пробы КЖ разводили подвижной фазой в 100 раз. Измерения концентраций токсина вели в линейном диапазоне детектирования, рассчитывая концентрацию по калибровочной кривой зависимости площади пика стандарта АФВ 1 («Sigma-Aldrich», США) от его концентрации в опыте и контроле, и определяли процент деградации относительно количества токсина, обнаруженного к соответствующей контрольной пробе.
Статистическую обработку данных количественного определения АФB 1 проводили, используя программу STATISTIСA 6.1 («StatSoft, Inc.», США). Достоверность различий при р < 0,05 подтверждали с помощью t -теста Стьюдента для независимых переменных. В таблице и на рисунках указаны средние значения ( М ) двух измерений для каждого из трех биологических повторов со стандартными отклонениями (±SD).
Результаты . Ген разлагающего афлатоксин фермента из A. tabescens , был клонирован методом ПЦР с использованием разработанных олигонуклеотидов. Размер продукта амплификации, соответствовавшего синтезированному гену adtz , составил 2088 п.н. Секвенирование полученного продукта подтвердило его идентичность с последовательностью A. tabescens
(GenBank AY941095.1) (рис. 1).
|
ADTZ_Sint |
1 |
ATGGCTAC7ACAACTGTTCACAGAGAGAGATTCTTGGCTGACAAGTC7GCTCCATTGTGT Hill II llllllll III 1 III Hill ПН II lllllllllll llllll |
50 |
|
ADTZ_A.tabescens |
92 |
ATGGCCACCACAACTGTCCACCGGGAGC6ATTCCTGGCAGATAAGTC7GCTCCTT7GTG7 |
151 |
|
ADTZ_sint |
61 |
GGTATG6ACATCAGAAAGTCTTTCGATCAACTGTCTTCCAAAGAGAAGCTGTACACTCAC |
120 |
|
ADTZ_A.tabescens |
152 |
llllllll II llllllll II Hill II II II II Hill Hill II GGTATGGATATTAGAAAGICATTTGATCAGCTCAGCTCTAAGGAAAAGCTCTACACGCAT |
211 |
|
ADTz"sint |
" 2 1 |
TATG7CACAGAAGCCTCA7GGGCTGGTGCTCGTATCATTCAAGCTCAATGGACTCCACAA |
180 |
|
ADTZ_A.tabescens |
212 |
II II II Hill II Hill II II 1 Hill II Hill Hill II II TACGTGACCGAAGCTTCTTGGGCGGGCGCAAGAATCATCCAGGCTCAGTGGACCCCGCAG |
271 |
|
ADTZ_sint |
181 |
GCTACTGACTTGTATGATCTGTTGATTCTGACCTTCTCTGTCAATGGTAAGCTGGCAGAT |
240 |
|
ADTZ_A.tabescens |
272 |
II II II 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 II II III II Hill Hill II II GCGACAGATCTATATGATCTGTTGATCCTTACGTTCAGCGTAAATGGAAAGCTCGCCGAC |
331 |
|
ADTZ~sint |
241 |
TTGAATGCTCTCAAGACTTCTTCTGG--TTTGAGTGAAGATGACTGGGAAGCCTTGATTC |
293 |
|
ADTZ_Autabescens |
332 |
HHHI II Hill II II II HI II 1 II II Hill llllllll 1 CTGAATGCCCTTAAGACGTCGTCAGGCCTTTCAGAG--GACGATTGGGAGGCCTTGATAC |
389 |
|
ADTZ_sint |
299 |
AGTACACTGTTCAAGTTTTGTCCAACTTGGTGAACTACAAGACCTTCGGATTCACTAAGA |
358 |
|
ADTZ_A.tabescens |
390 |
HHHI II II II HI III 1 II lllllllllll llllllll II Illi AGTACACGGTCCAGGTATIGAGCMTCTTGTCAACTACAAGACGTTCGGATTTACGAAGA |
449 |
|
ADTz"sint |
3 59 |
TCATTCCTAGAGTTGATGCTGAGAAATTCGAATCTGTTGTCAAAGCATCTTCCAATGCTG |
418 |
|
ADTZ_Autabescens |
450 |
HHHI 1 II II II II II II II II II llllllll III HI II 1 TCATTCCCCGCGTCGACGCAGAAAAGTTIGAGTCAGTGGTCAAAGCCICTAGCAACGCAG |
509 |
|
ADTZ_sint |
419 |
ATCAAGGTTCTGCCTTGTTCACCAAATTGAAACAACACATCTATGCCTTGTCTCCAGAGT |
478 |
|
AD tz_a. tabescens |
510 |
1 II II II II 1 llllllll lllllllIlli III Hill 1 Hill Illi ACCAGGGC7CGGCACTAT7CACCAAGTTGAAACAACACATATATGCGCTTTCTCCTGAGT |
569 |
|
ADTZ_sint |
479 |
CTGCCTTGTTCATTQGTAAGCGTAAAGATtiGTCATCTTTCCAACTACTACTTGGGTGAAC |
538 |
|
AD7Z_A.tabescens |
570 |
1 II 1 llllllll II 1 II II Hill II II II Hill 1 lllllll CAGCGCTA7TCATTGGCAAAAGGAAGGACGGTCACG7ATCAAATTAC7ATCTTGGTGAAC |
629 |
|
ADIZ_sint |
539 |
CAGTTGGTGATGCAGAAGITGATGCCATTCAGAATGTTGCTGAGAAAITGGGTGTTGACA |
598 |
|
AD7Z_A.tabescens |
630 |
1 Hill Hill II II Hill II llllllll llllllll II II Hill 1 CTGTTGGAGATGCTGAGG7CGATGCTATCCAGAATGTCGCTGAGAAG7TAGGCGTTGATA |
689 |
|
AD7Z_sint |
599 |
TTTTGAACACTAGAGTTAAGAAGAATGGIGCTGGAGATTACACTCTGITGGTTGCTTCTG |
658 |
|
AD7Z_A.tabescens |
690 |
1 1 II III 1 II lllllllllll II II llllllll II II Hill Illi TCCTCAATACTCGCGTGAAGAAGAATGGAGCGGGTGATTACACGCTC7TAGTTGCCTCTG |
749 |
|
AD7Z_sint |
659 |
CCAAAACA7CTCCTCCATCTGTTCATGACTTCCAGATTGATTCTACTCCTGCCAAGTTGA |
718 |
|
AD7Z_A.tabescens |
750 |
1 Hill III II II II lllllllllll II II II Hill II II Illi CTAAAACCAGTCCACCCTCCGTGCATGACrTCCAAATCGACTCAACTCCGGCTAAATTGA |
899 |
|
ADTZ^Sint |
719 |
CCATTGAGTATGGTGACTATGCTTCTTCCTTGACTAAAGTTG7TGCAGCTTTGCAAGAAG |
778 |
|
AD7Z_A.tabescens |
810 |
1 lllllllllll Hill II II II 1 II II Hill II II 1 II II 1 CGATTGAGTATGGCGACTACGCGTCATCTCTAACGAAGGTTGTCGCCGCCCTTCAGGAGG |
869 |
|
ADTZ_sint |
779 |
CTAAACAGTATACTGCTAATGATCATCAATCTGCTATGATTGAAGGCTATGTCAAGTCCT |
838 |
|
AD7Z_A.tabescens |
879 |
1 lllllllllll II II lllllllllll II Hill lllllllllllllllll 1 CCAAACAGTATACCGCGAACGATCATCAATCAGCGATGATCGAAGGCTATGTCAAGTCGT |
929 |
|
AD7z"sint |
8 39 |
TCAACTCTGGTTCCATTCCAGAACACAAAGCTGCCTCAACTGAGTGGGTTAAGGACATTG |
898 |
|
AD7Z_A.tabescens |
930 |
lllllll II II Hill llllllllllllll Hill II Hill II II Illi TCAACTCAGGATCAATTCCGGAACACAAAGCTGCGTCAACAGAA7GGG7GAAAGATATTG |
989 |
|
AD7Z_sint |
899 |
GTCCAGTTGTTGAGTCCTACATTGGTTTCGTTGAGACCTATGTTGATCCATATGGTGGAA |
958 |
|
adtz_a. tabescens |
990 |
1 II Hill lllllllllll II Hill II llllllll II llllllll III GACCGGTTG7AGAGTCCTACATCGGGTTCGTCGAAACCTATGTCGACCCATATGGCGGAC |
1049 |
|
ADTZ_sint |
959 |
GAGCTGAGTGGGAAGGTTTCACAGCCATTGTTGACAAACAACTGTCTGCCAAGTATGAAG |
1018 |
|
ADTZAutabescens |
1050 |
1 II II Hill llllllll Hill II Hill II III HI Hill Illi GCGCGGAATGGGAGGGTTICACTGCCATCGTCGACAAGCAGCTGAGTGCGAAGTACGAAG |
1109 |
|
ADTZ_sint |
1019 |
CTCTGGTGAATGGAGCACCAAAGTTGATCAAGTCATTGCCATGGGGTACTGACTTCGAAG |
1078 |
|
ADTZ_A.tabescens |
1110 |
1 Illi II II II II llllllllllll 1 II Hill II llllllll 1 CATTGGTTAACGGTGCTCCTAAGTTGATCAAGAGTC7TCCGTGGGGAACGGACTTCGAGG |
1169 |
|
ADTZ_sint |
1079 |
TTGATGTGTTCAGAAAACCAGACTTCACTGCTTTGGMGTTGTTTCCITCGCTACTGGTG |
1138 |
|
adtz_a. tabescens |
1170 |
Illi II Hill II II Hill Hill llllllll II II II II II II 1 7TGACGTC7TCAGGAAGCCGGACTTTACTGCGTTGGAAGTCGTATCA7TTGCAACAGGAG |
1229 |
|
ADTZ_Siilt |
1139 |
GTAT7CCTGCTGGTATTAACATTCCAAACTATTATGAAGTCAGAGAAAGTACTGGTTTCA |
1198 |
|
ADTZ_A.tabescens |
1230 |
HIHIIHI II II II II 111111111111111111 1 Hill II II II 1 GTAT7CCTGCCGGAATCAATATACCAAACTATTATGAAGTCCGGGAAAGCACAGGGTTTA |
1289 |
|
ADTZ_sint |
1199 |
AAAATGTTTCATTGGCTAACATTCTGGCTGCTAAAGTTCCAAACGAAGAGTTGACTTTCA |
1258 |
|
ADTZ_A.tabescens |
1290 |
1 llllllll 1 II II III Illi II II II llllllll Hill lllllll AGAA7GTT7CGCTAGCGAATATTTTGGCGGCCAAGGTACCAAACGAGGAGTTAACTTTCA |
1349 |
|
ADTZ_Siilt |
1259 |
7TCA7CCAGACGATGTTGAGTTGTACAATGCTTGGGATTCTAGAGCT7TCGAGCTGCAAG |
1318 |
|
ADTZ_A.tabescens |
1350 |
1 Hill II II II II 1 II II lllllllll 1 1 II II II II II 1 TCCATCCTGATGACGTAGAACTATATAACGCTTGGGATAGTCGCGCGTTTGAACTTCAGG |
1409 |
|
ADTZ_sint |
1319 |
7TGC7AATCATGAGTTGT7GGGACATGGTAGTGGCAAGTTGTTCCAAGAAGGTGCAGATG |
1378 |
|
ADTZ_A.tabescens |
1410 |
1 II II II II 1 Hill Hill llllll 1 llllllllllllll Illi TGGCCAACCACGAACTmGGGTCATGGCrCCGGCAAGCTTTTCCAAGAAGGTGCTGATG |
1469 |
|
AD7Z_sint |
1379 |
GTAAACTGAACTTCGATCC TGAGAAAGTCATCAATCCATTGACTGGCAAACC TATCAC TT |
1438 |
|
AD7Z_A.tabescens |
1470 |
1 lllllllllllllllll II II Hill II II lllllll II II II Illi GGAAACTGAACTTCGATCCCGAAAAGGTCATAAACCCTCTGACTGGAAAGCCGATAACTT |
1529 |
|
AD7Z_sint |
1439 |
CTTGGTACAAACCAGGTCAAACACCAGA7TCTGTGTTGGGTGAAGTTAGTTCTTCCATGG |
1498 |
|
ADTZ_A.tabescens |
1530 |
1 Hill II Hill Hill II llllllll II II Hill II II Illi CATGGTATAAGCCAGGGCAAACGCCGGAITCTGTTTTAGGCGAAGTGTCGTCGTCAATGG |
1589 |
|
AD7Z_sint |
1499 |
AAGAGTGCAGAGCTGAGACTGTTGCCTTGTAC7TGGTTTCCAATTTGGACATCTTGAAAA |
1558 |
|
AD7Z_A.tabescens |
1590 |
Illi II 1 II Hill II II 1 lllllllll III 1 II II 1 Illi AAGAA7GTCGGGCGGAGACCGTAGCGCTCTAC7TGGTTAGCAACCTCGATATTCTTAAAA |
1649 |
|
AD7Z_sint |
1559 |
TCTTCAACTACGTTGACAAACAAGATAT7GAAGACATTCAGTACATCACTTTCTTGTTGA |
1618 |
|
adtz_a, tabescens |
1650 |
1 Hill Hill Hill Hill llllllll II lllllllllll llllll 1 1 TTTTCAATTACGTCGACAAGCAAGACATTGAAGATATCCAGTACATCACGTTCTTGCTTA |
1709 |
|
AD7Z~sint |
1619 |
TGGCTAGAGCTGGTTTGAGAGCTTTGGAATTC7ATGATCCTGCTACCAAGAAACATGGTC |
1678 |
|
AD7Z_A.tabescens |
1710 |
Illi 1 llllll II 1 II 1 II II llllllll II llllllll II II 1 TGGCCCGCGCTGG7CTGCGGGCACTAGAGTTT7ATGATCCAGCCACCAAGAAGCACGGAC |
1769 |
|
AD7Z_sint |
1679 |
AAGCTCACA7GCAAGCTCGTATGGGTATCACTCAGTACTTGA7TCAAGCTGGTATTGCTA |
1738 |
|
AD7Z_A.tabescens |
1770 |
1 II II Hill II 1 Hill II II llllll 1111111111111 Hill 1 AGGCACATATGCAGGCCAGAATGGGCATAACCCAGTACCTGATTCAAGCTGGGATTGCGA |
1829 |
|
AD7Z_sint |
1739 |
GATTGGAAT7GДТ7СAAGATGCTAATGG7GAA7TGGAGAACT7G7ATG7TAGAGTTGATA |
1798 |
|
AD7Z_A.tabescens |
1830 |
II 1 llllllll II Hill II II HI 1 II Hill II III 1 Hill GACTTGAAT7GATCCAGGA7GCCAACGGCGAACTCGAAAACT7A7ACG7TCGGGTTGACC |
1889 |
|
ADIZ_sint |
1799 |
GAGAGAAAGTCTTGTCCAAAGGCMAGAAGTTGTTGGTCAACTGTTGA7TGAATTGCAAG |
1858 |
|
ADIZ_A.tabescens |
1890 |
1 llllllll lllllllllll II II llllllllllll II Illi III 1 Illi GGGAGAAAGTGTTGICCAAAGGAAAGGAGGTTG TT6GTCAATT6C TGA7 CGAACTCCAAG |
1949 |
|
AD7Z_sint |
1859 |
TTAG--AAAGTCAACTGCTGATGGTACAGGATCTAGAGACTTCTACAC7ACCTTGACTGA |
1916 |
|
AD7Z_A.tabescens |
1950 |
1 1 Hill II II II II II II II Illi llllllll II Illi II TCCGGAAAAGT--ACCGCAGACGGCACCGGCTCCCGAGATTTCTACACAACGCTGACCGA |
2007 |
|
ADTZ_sint |
1917 |
ACCAATCTCTGGTTGGGAAGGTAAGATCAGAGACATTGTGCTGAAGAAGAAGTTGCCTCG |
1976 |
|
ADTZ_A.tabescens |
2008 |
llllllllllll Hill II llllll lllllll II lllllllllll 1 Hill ACCAATCTCTGGATGGGAGGGCAAGATCCGAGACATCGTTTTGAAGAAGAAGCTTCCTCG |
2067 |
|
ADTZ_sint |
1977 |
TAAGATCTTCGTTCAACC7AACACCTTCGTTGTCAATGGTGAAGTTCAGTTGAAAGAGTA |
2036 |
|
ADTZ_A.tabescens |
2068 |
II Hill II Hill II II II II Hill II Hill HI 1 llllllll AAAAATCT7TGTCCAACCCAATACAT7TGTCGTCAACGGCGAAGTCCAGCTCAAAGAGTA |
2127 |
|
ADTZ_sint |
2037 |
TCCATTGACTGCAGCTGGAGTGATTGAATCCTTCATTGAGAGAAGATTGT |
2086 |
|
adtz_a. tabescens |
2128 |
HI Hill II II II II llllll llllllllllll llllll 7CCT7TGACGGCTGCCGGGGTAATTGAAAGTTTCATTGAGAGACGAT7GT |
2177 |
Рис. 1. Визуализация выравнивания последовательности гена adtz с оптимизированными кодонами и природного гена adt из Armillaria tabescens (GenBank AY941095.1, . Оптимизация кодонового состава осуществлялась ЗАО «Евроген» (Россия) с использованием таблицы частоты встречаемости кодонов для Pichia pastoris .
Полученный рекомбинантный ген был интегрирован в вектор pPIG-1 рестриктазно-лигазным методом. Рестрикционный анализ полученной в результате этого новой плазмиды после ее двойного расщепления рестрик-тазами HindIII и NotI приводил к получению продуктов размерами 5500 и 2100 п.н., что подтверждало правильность интеграции целевой последовательности в вектор pPIG-1. Полученная рекомбинантная плазмида (рис. 2) была названа pPIG-ADTZ.
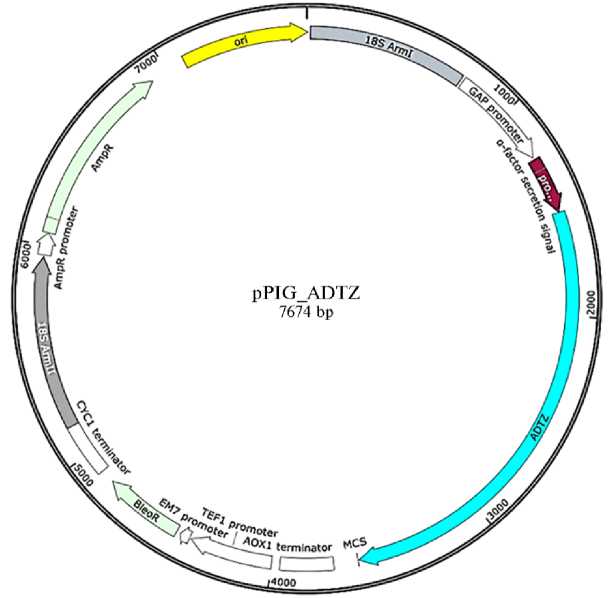
Рис. 2. Карта плазмиды pPIG-ADTZ, полученной при клонировании последовательности синтезированного гена афлатоксин-детоксифизима (ADTZ) adtz ( synthetic_ADTZ) в вектор pPIG-1.
Плазмиду pPIG-ADTZ, линеаризованную эндонуклеазой рестрикции ApaI, электропорировали в компетентные клетки P. рastoris GS115 и отбирали трансформанты на среде YPD с зеоцином. При клонировании было получено 154 клональных колонии. Геномная ДНК, выделенная из 70 случайно выбранных трансформированных клонов, была проанализирована методом ПЦР для идентификации вставки гена adtz. ПЦР-анализ ДНК этих трансформантов, выращенных на селективной среде, показал, что по крайней мере 54 клона содержали целевую вставку adtz. Среди них наиболее продуктивным оказался клон ADTZ-14, который отобрали для дальнейшей работы. Экспрессия ADTZ в клетках этого клона уже после 72 ч культивирования приводила к накоплению в КЖ внеклеточного рекомбинантного белка, наблюдаемый размер которого согласно анализу с помощью ДСН-ПААГ составлял 78±3 кДа (рис. 3), тогда как в КЖ нетрансформированного реципиентного P. pastoris GS115 белков с сопоставимой массой мы не обнаружили. В КЖ трансформированного клона ADTZ-14 концентрация об- щего белка составила 2,1 мг/мл.
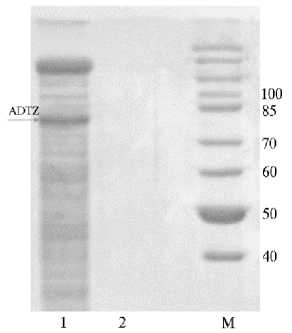
Рис. 3. Электрофореграмма белков культуральной жидкости штамма Pichia pastoris ADTZ-14 (1) , трансформированного рекомбинантным вектором pPIG-ADTZ с синтезированным геном афлатоксин-детоксифизима (ADTZ) adtz , и реципиента P. pastoris GS115 (2); М — маркер молекулярной массы PageRuler™ 26614 («Thermo Fisher Scientific», США).
Сравнение кинетики деградации АФВ 1 и АФG 1 в КЖ P. pastoris ADTZ-14 показало, что рекомбинантный фермент способен разрушать оба токсина, однако его эффективность в отношении АФВ 1 оказалась значительно выше, чем для АФG 1 . Так, под действием КЖ продуцента рекомбинантного внеклеточного ADTZ уже через 2 ч инкубации концентрация токсина АФВ 1 снижалась примерно на 14 % по сравнению с исходной, в то время как для АФG 1 снижение составило лишь 4 % (рис. 4).
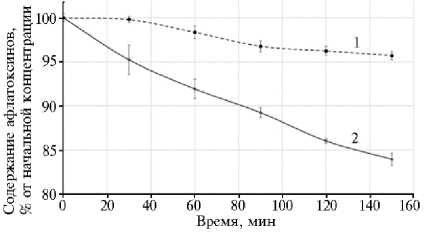
Рис. 4. Кинетика деградации афлатоксинов G 1 (1) и B 1 (2) в бесклеточной культуральной жидкости штамма Pichia pastoris ADTZ-14, трансформированного рекомбинантным вектором pPIG-ADTZ с синтезированным геном афлатоксин-детокси-физима (ADTZ) adtz , при 40 ° С и рН 6,7 ( n = 3, М ±SD).
Полученные результаты соответствовали данным других авторов, отмечавших высокую специфичность внутриклеточного ADTZ из A. tabescens в отношении АФВ1 (29). В связи с этим мы исследовали деградационную активность рекомбинантного ADTZ в отношении указанного токсина при его более длительной инкубации с бесклеточной КЖ штамма P. pastoris ADTZ-14.
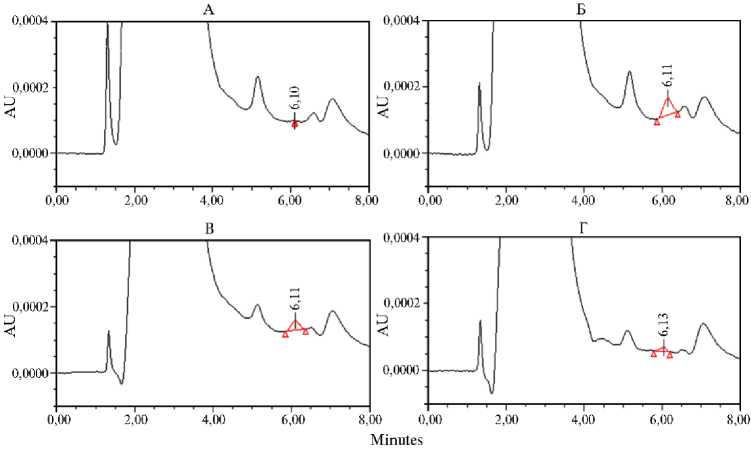
Рис. 5. Хроматограммы образцов культуральной жидкости (КЖ) Pichia pastoris (инкубация при
30 С и рН 7,0).
А: КЖ нетрансформированного реципиентного штамма GS115 (контроль).
Б: КЖ GS115 + афлатоксин В 1 (АФВ 1 , 1 мкг/мл) после инкубации (контрольный образец). Пик на хроматограмме соответствует 10 нг АФВ 1 во введенной пробе.
В и Г: КЖ штамма P. pastoris ADTZ-14, трансформированного рекомбинантным вектором pPIG-ADTZ с синтезированным геном афлатоксин-детоксифизима (ADTZ) adtz . + АФВ 1 (1 мкг/мл) соответственно через 3 и 5 сут инкубации.
В этих экспериментах было обнаружено, что через 3 сут содержание добавленного в КЖ токсина снижалось почти в 2 раза, а через 5 сут эффективность его деградации достигала 80 % (рис. 5, табл.).
Ферментативная деградация афлатоксина В 1 (АФВ 1 ) в бесклеточной культуральной жидкости (КЖ) штамма-трансформанта Pichia pastoris ADTZ-14, секретирующего рекомбинантный афлатоксин-детоксифизим (ADTZ), в зависимости от времени инкубации ( n = 6, M ±SD)
|
Штамм |
Продолжительность инкубации |
||
|
0 ч |
72 ч |
120 ч |
|
|
АФВ 1 , мкг/мл |
АФВ 1 , мкг/мл деградация, % |
АФВ 1 , мкг/мл деградация, % |
|
ADTZ-14 0,97±0,01 0,57±0,06 41,2а 0,19±0,04 80,4b
GS115 (контроль) 0,98±0,01 1,01±0,01 0,0с 0,90±0,05 0,1с
Примечани е. Штамм ADTZ-14 получен трансформацией реципиента P. рastoris GS115 рекомбинантным вектором pPIG-ADTZ с синтезированным геном афлатоксин-детоксифизима (ADTZ) adtz . Перед инкубацией в КЖ вносили АФВ 1 до концентрации 1 мкг/мл; для 0 ч указаны концентрации, обнаруженные в пробах КЖ перед инкубацией (открываемость от 96 до 99 %).
abc Различия между процентами деградации, отмеченными разными буквами, статистически значимы при р < 0,05.
Представленные нами данные свидетельствуют о достаточно высоком биотехнологическом потенциале нового продуцента рекомбинантного ADTZ и расширяют пока что ограниченный спектр полученных с помощью системы гетерологичной экспрессии в P. pastoris рекомбинантных ферментов других ксилоторофных грибов, разлагающих АФВ 1 (39).
Следует также отметить, что продуцент ADTZ-14 характеризовался достаточно высоким для P. pastoris уровнем экспрессии внеклеточных белков. Вероятно, использование синтетического гена с оптимизированными кодонами способствовало увеличению продуктивности дрожжевых клеток. Подобный подход был успешно применен ранее для экспрессии бактериальной а -амилазы в P. pastoris (40). Однако по последним данным, использование синтетических генов способно приводить к неправильному фолдингу белка, деградации и снижению его активности и стабильности, что может быть причиной частичной деградации секретируемых рекомбинантных белков (41), которую, как отмечено выше, мы наблюдали и в наших экспериментах при электрофоретическом анализе КЖ трансформированного клона ADTZ-14. Поэтому необходимо продолжить исследования по повышению эффективности деградации АФВ 1 рекомбинантным ADTZ, дополнительной проверке воздействия этого рекомбинантного фермента на другие афлатоксины, а также эксперименты по обработке ферментным препаратом растениеводческой продукции, загрязненной АФВ 1 . Не исключено также, что гетерологичная экспрессия с использованием других эукариот, например штаммов мицелиальных грибов, которые применяются для биопереработки корма с целью повышения его питательной ценности, позволит получить новые продуценты высокоактивного экстрацеллюлярного ADTZ. Подобные продуценты могли бы оказаться перспективным для одновременной деконтаминации растительных кормов, загрязненных афлатоксином, и повышения доступности их питательных компонентов.
Таким образом, разработанная нами ранее система экспрессии для повышения копийности гетерологичных генов в Pichia pastoris впервые применена для получения рекомбинантного белка, способного разлагать АФВ1. При этом была осуществлена трансформация дрожжевых клеток плазмидой pPIG-ADTZ и получены 154 рекомбинантных клона P. pastoris, 77 % которых содержали целевую последовательность синтетического гена афлаток-син-детоксифизима ADTZ — adtz. Выход белка у наиболее продуктивного трансформанта ADTZ-14 составил 2,1 мг/мл бесклеточной культуральной жидкости, причем в этом случае около половины пула всех внеклеточных белков приходилось на рекомбинантный ADTZ, инкубация АФВ1 с которым приводила к деградации 80 % добавленного токсина. Полученный штамм-трансформант P. pastoris ADTZ-14, секретирующий функциональный ADTZ, можно рассматривать в качестве продуцента доступного и достаточно активного в отношении АФВ1 рекомбинантного фермента, на основе которого в будущем могут разрабатываться препараты для энзиматической деградации этого микотоксина. Подтверждение деконтаминационного потенциала рекомбинантного фермента будет свидетельствовать о целесообразности оптимизации биотехнологии для увеличения выхода целевого продукта и разработки его препаративной формы.
Список литературы Получение нового продуцента рекомбинантного афлатоксин-деградирующего фермента с помощью гетерологичной экспрессии в Pichia pastoris
- Джавахия В.Г., Стацюк Н.В., Щербакова Л.А., Поплетаева С.Б. Афлатоксины: ингибирование биосинтеза, профилактика загрязнения и деконтаминация агропродукции. М., 2017.
- Coppock R.W., Christian R.G., Jacobsen B.J. Aflatoxins. In: Veterinary toxicology /R.C. Gupta (ed.). Academic Press, 2018: 983-994 (doi: 10.1016/B978-0-12-811410-0.00069-6).
- Кононенко Г.П., Зотова Е.В., Буркин А.А. Опыт микотоксикологического обследования зернофуражных культур. Сельскохозяйственная биология, 2021, 56(5): 958-967 (doi: 10.15389/agrobiology.2021.5.958rus).
- Кононенко Г.П., Буркин А.А. Токсины микромицетов в генеративных органах растений семейства Fabacea. Сельскохозяйственная биология, 2021, 56(5): 968-978 (doi: 10.15389/agrobiology.2021.5.968rus).
- Kumar P., Mahato D.K., Kamle M., Mohanta T.K., Kang S.G. Aflatoxins: A global concern for food safety, human health and their management. Front. Microbiol., 2017, 7: 2170 (doi: 10.3389/fmicb.2016.02170).
- Romani L. Immunity to fungal infections. Nat. Rev. Immunol., 2004, 4(11): 1-23 (doi: 10.1038/nri1255).
- El-Sayed R.A., Jebur A.B., Kang W., El-Demerdash F.M. An overview on the major mycotoxins in food products: characteristics, toxicity, and analysis. Journal of Future Foods, 2022, 2(2): 91-102 (doi: 10.1016/j.jfutfo.2022.03.002).
- Schuda P.F. Aflatoxin chemistry and syntheses. In: Syntheses of natural products. Topics in current chemistry, V. 91. Springer, Berlin, Heidelberg, 1980: 79-81 (doi: 10.1007/3-540-09827-5_3).
- Mahato D.K., Lee K.E., Kamle M., Devi S., Dewangan K.N., Kumar P., Kang S.G. Aflatoxins in food and feed: an overview on prevalence, detection and control strategies. Front. Microbiol., 2019, 10: 2266 (doi: 10.3389/fmicb.2019.02266).
- Probst C., Njapau H., Cotty P.J. Outbreak of an acute aflatoxicosis in Kenya in 2004: identification of the causal agent. Appl. Environ. Microbiol., 2007, 73(8): 2762-2764 (doi: 10.1128/aem.02370-06).
- Medina A., Gilbert M.K., Mack B.M., O’Brian G.R., Rodriguez A., Bhatnagar D., Payne G., MaganN. Interactions between water activity and temperature on the Aspergillus flavus transcriptome and aflatoxin B1 production. Int. J. Food Microbiol., 2017, 256: 36-44 (doi: 10.1016/j.ijfoodmicro.2017.05.020).
- Shcherbakova L.A., Statsyuk N.V., Mikityuk O.D., Nazarova N.A., Dzhavakhiya V.G. Aflatoxin B1 degradation by metabolites of Phoma glomerata PG41 isolated from natural substrate colonized by aflatoxigenic Aspergillus flavus. Jundishapur J. Microbiol., 2015, 8(1): e24324 (doi: 10.5812/jjm.24324).
- Ji C., Fan Y., Zhao L. Review on biological degradation of mycotoxins. Anim. Nutr., 2016, 2(3): 127-133 (doi: 10.1016/j.aninu.2016.07.003).
- Verheecke C., Liboz T., Mathieu F. Microbial degradation of aflatoxin B1: current status and future advances. Int. J. Food Microbiol., 2016, 237: 1-9 (doi: 10.1016/j.ijfoodmicro.2016.07.028).
- Alberts J.F, Gelderblom W.C.A., Botha A., van Zyl W.H. Degradation of aflatoxin B1 by fungal laccase enzymes. Int. J. Food Microbiol., 2009, 135: 47-52 (doi:10.1016/j.ijfoodmicro.2009.07.022).
- Taylor M.C., Jackson C.J., Tattersall D.B., French N., Peat T.S., Newman J., Briggs L.J., Lapal-ikar G.V., Campbell P.M., Scott C., Russell R.J., Oakeshott J.G. Identification and characteri-zation of two families of F420H2-dependent reductases from Mycobacteria that catalyse aflatoxin degradation. Mol. Microbiol., 2010, 78(3): 561-575 (doi: 10.1111/j.1365-2958.2010.07356.x).
- Zhao L.H., Guan S., Gao X., Ma Q.G., Lei Y.P., Bai X.M., Ji C. Preparation, purification and characteristics of an aflatoxin degradation enzyme from Myxococcus fulvus ANSM068. J. Appl. Microbiol., 2011, 110(1): 147-155 (doi: 10.1111/j.1365-2672.2010.04867.x).
- Wang Y., Zhao C., Zhang D., Zhao M., Zheng D., Lyu Y., Cheng W., Guo P., Cui Z. Effective degradation of aflatoxin B1 using a novel thermophilic microbial consortium TADC7. Bioresource Technology, 2017, 224: 166-173 (doi: 10.1016/j.biortech.2016.11.033).
- Guo Y.P., Qin X.J., Tang Y., Ma Q.G., Zhang J.Y., Zhao L.H. CotA laccase, a novel aflatoxin oxidase from Bacillus licheniformis, transforms aflatoxin B-1 to aflatoxin Q(1) and epi-aflatoxin Q(1). Food Chem., 2020, 325: 126877 (doi: 10.1016/j.foodchem.2020.126877).
- Adebo O.A., Njobeh P.B., Gbashi S., Nwinyi O.C., Mavumengwana V. Review on microbial degradation of aflatoxins. Crit. Rev. Food Sci. Nutr., 2017, 57(15): 3208-3217 (doi: 10.1080/10408398.2015.1106440).
- Xu H.W., Wang L.Z., Sun J.D., Wang L.P., Guo H.Y., Ye Y.L., Sun X.L. Microbial detoxifica-tion of mycotoxins in food and feed.Crit. Rev. Food Sci. Nutr., 2022, 62(18): 4951-4969 (doi: 10.1080/10408398.2021.1879730).
- Li C.H., Li W.Y., Hsu I.N., Liao Y.Y., Yang C.Y., Taylor M.C., Liu Y.F., Huang W.H., Chang H.H., Huang H.L., Lo S.C., Lin T.Y., Sun W.C., Chuang Y.Y., Yang Y.C., Fu R.H., Tsai R.T. Recombinant aflatoxin-degrading F420H2-dependent reductase from Mycobacterium smegmatis protects mammalian cells from aflatoxin toxicity. Toxins, 2019, 11(5): 259 (doi: 10.3390/toxins11050259).
- Kolosova A., Stroka J. Substances for reduction of the contamination of feed by mycotoxins: a review. World Mycotoxin Journal, 2011, 4(3): 225-256 (doi: 10.3920/WMJ2011.1288).
- Loi M., Fanelli F., Zucca P., Liuzzi V.C., Quintieri L., Cimmarusti M.T., Monaci L., Haidu-kowski M., Logrieco A.F., Sanjust E., Mulè G. Aflatoxin B1 and M1 degradation by Lac2 from Pleurotus pulmonarius and redox mediators. Toxins, 2016, 8(9): 245 (doi: 10.3390/toxins8090245).
- Brana M.T., Sergio L., Haidukowski M., Logrieco A.F., Altomare C. Degradation of aflatoxin B-1 by a sustainable enzymatic extract from spent mushroom substrate of Pleurotus eryngii. Toxins, 2020, 12(1): 49 (doi: 10.3390/toxins12010049).
- Wang J., Ogata M., Hirai H., Kawagishi H. Detoxification of aflatoxin B1 by manganese peroxi-dase from the white-rot fungus Phanerochaete sordid YK-624. FEMS Microbiol. Lett., 2011, 314(2): 164-169 (doi: 10.1111/j.1574-6968.2010.02158.x).
- Yao D.S., Liang R., Liu, D.L., Gu L.Q., Ma L., Chen W.Q. Screening of the fungus whose multienzyme system has catalytic detoxification activity towards aflatoxin B1 (Part I). Ann. N.Y. Acad. Sci., 1998, 864: 579-585 (doi: 10.1111/j.1749-6632.1998.tb10385.x).
- Liu D.-L., Yao D.-S., Liang Y.Q., Zhou T.-H., Song Y.-P., Zhao L., Ma L. Production, purifi-cation, and characterization of an intracellular aflatoxin-detoxifizyme from Armillariella tabescens (E-20). Food Chem. Toxicol., 2001, 39(5): 461-466 (doi: 10.1016/s0278-6915(00)00161-7).
- Cao H., Liu D., Mo X., Xie C., Yao D. A fungal enzyme with the ability of aflatoxin B1 conver-sion: purification and ESI-MS/MS identification. Microbiol. Res., 2011, 166(6): 475-483 (doi: 10.1016/j.micres. 2010.09.002).
- Wu Y.Z., Lu F.P., Jiang H.L., Tan C.P., Yao D.S., Xie C.F., Liu D.L. The furofuran-ring selectivity, hydrogen peroxide-production and low Km value are the three elements for highly effective detoxification of aflatoxin oxidase. Food Chem. Toxicol., 2015, 76: 125-131 (doi: 10.1016/j.fct.2014.12.004).
- Xingming Y. Oral liquid medicine from ferments of Armilarilla tabescens. China PAT # CN1679642. Priority date 11. 05.2004. Publication date 01.08.2007.
- Chahardooli M., Niazi A., Aram F., Sohrabi S.M. Expression of recombinant Arabian camel lactoferricin-related peptide in Pichia pastoris and its antimicrobial identification. J. Sci. Food Agric., 2016, 96(2): 569-575 (doi: 10.1002/jsfa.7125).
- Wang Y., Wang Y., Jiang J., Zhao Y., Xing F., Zhou L. High expression of zearalenone de-grading enzyme in Pichia pastoris. Chinese Journal of Biotechnology, 2020, 36(2): 372-380 (doi: 10.13345/j.cjb.190150).
- Синельников И.Г., Зоров И.Н., Синицына О.А., Синицын А.П., Рожкова А.М. Интегра-ционный вектор для многокопийной интеграции генов в 18Sр РНК дрожжей Pichia pastoris. № RU 2752904C1, Моск. ФГУ Исследовательский Центр «Основы Биотехнологии» РАН (РФ). Заявл. 02.09.20. Опубл. 11.08.21. Бюл. № 23.
- Lin-Cereghino J., Wong W.W., Xiong S., Giang W., Luong L.T., Vu J., Johnson S.D., Lin-Cereghino G.P. Condensed protocol for competent cell preparation and transformation of the methylotrophic yeast Pichia pastoris. Biotechniques, 2005, 38(1): 44-48 (doi: 10.2144/05381BM04).
- Lõoke M., Kristjuhan K., Kristjuhan A. Extraction of genomic DNA from yeasts for PCR-based applications. Biotechniques, 2011, 50(5): 325-328 (doi: 10.2144/000113672).
- Waterborg J.H., Matthews H.R. The Lowry method for protein quantitation. In: Proteins. Methods in molecular biology™, vol 1 /J.M. Walker (ed.). Humana Press, 1984: 1-3 (doi: 10.1385/0-89603-062-8:1).
- Dzhavakhiya V.G., Voinova T.M., Popletaeva S.B., Statsyuk N.V., Limantseva L.A., Shcherbakova L.A. Effect of various compounds blocking the colony pigmentation on the aflatoxin B1 production by Aspergillus flavus. Toxins, 2016, 8(11): 313 (doi: 10.3390/toxins8110313).
- Yang P., Xiao W., Lu S., Jiang S., Zheng Z., Zhang D., Zhang M., Jiang S., Jiang S. Recombinant expression of Trametes versicolor aflatoxin B1-degrading enzyme (TV-AFB1D) in engineering Pichia pastoris GS115 and application in AFB1 degradation in AFB1-contaminated peanuts. Toxins, 2021, 13(5): 349 (doi: 10.3390/toxins13050349).
- Wang J.R., Li Y.Y., Liu D.N., Liu J.S., Li P., Chen L.Z., Xu S.D. Codon optimization significantly improves the expression level of -amylase gene from Bacillus licheniformis in Pichia pastoris. BioMed Research International, 2015, 2015: 248680 (doi: 10.1155/2015/248680).
- Liu K., Ouyang Y., Lin R., Ge C., Zhou M. Strong negative correlation between codon usage bias and protein structural disorder impedes protein expression after codon optimization. J. Biotechnol., 2022, 343: 15-24 (doi: org/10.1016/j.jbiotec.2021.11.001).


