Применение анализа кривых плавления нуклеиновых кислот высокого разрешения и МАЛДИ-минисеквенирования для генотипирования по полиморфизмам гена параоксоназы-1 человека
Автор: Курдюков Иван Дмитриевич, Мурашко Е.А., Дубровский Я.А., Бабаков В.Н., Радилов А.С.
Журнал: Научное приборостроение @nauchnoe-priborostroenie
Рубрика: Хроматография, ПЦР-, ДНК-анализ
Статья в выпуске: 1 т.23, 2013 года.
Бесплатный доступ
Для генотипирования по однонуклеотидным полиморфизмам (rs854560 и rs662) гена параоксоназы-1 человека были проведены анализ кривых плавления нуклеиновых кислот высокого разрешения и МАЛДИ-минисеквенирование. Аллельные частоты, полученные в результате генотипирования 94 жителей Кировской области (66.0 % для L-аллеля и 71.8 % для Q-аллеля), оказались сходными с таковыми для европейцев и белого населения США. Экспериментальные данные подтвердили применимость HRM-анализа и МАЛДИ-минисеквенирования для генотипирования по однонуклеотидным полиморфизмам L55M и Q192R гена параксоназы-1 человека.
Параоксоназа-1, малди минисеквенирование
Короткий адрес: https://sciup.org/14264837
IDR: 14264837 | УДК: 575.174.015.3:
Текст научной статьи Применение анализа кривых плавления нуклеиновых кислот высокого разрешения и МАЛДИ-минисеквенирования для генотипирования по полиморфизмам гена параоксоназы-1 человека
Параоксоназа-1 (PON1) является сывороточной эстеразой, заякоренной на поверхности частиц липопротеинов высокой плотности (ЛПВП). PON1 защищает липиды от окисления и предотвращает развитие атеросклероза. Данный фермент обладает широким спектром субстратной специфичности и способен гидролизовать фосфорорганические соединения, хотя его основными физиологическими субстратами являются лактоны [1]. В кодирующей области гена PON1 обнаружено два клинически значимых полиморфизма, которые определяют активность фермента в сыворотке [2, 3]: аминокислотная замена Leu / Met (L/M) в позиции 55 (rs854560), а также Gln / Arg (Q/R) в позиции 192 (rs662). В настоящее время МАЛДИ-масс-спектро-метрия (matrix-assisted laser desorption / ionization,
MALDI, матрично-активированная лазерная десорбция / ионизация) широко применяется в различных областях биологии и медицины, в частности для идентификации патогенных микроорганизмов и определения однонуклеотидных полиморфизмов генов (single nucleotide polymorphism, SNP) у человека [4, 5]. В то же время генотипирование по анализу кривых плавления нуклеиновых кислот высокого разрешения (high-resolution melting analysis, HRMA, HRM-анализ) является простым и достаточно дешевым методом [6], который в настоящее время заметно популярен из-за своих преимуществ. Таким образом, проблема экспериментальной оценки применимости HRM-анализа и МАЛДИ-минисеквенирования для определения генетических полиморфизмов параоксоназы-1 достаточно актуальна в прикладном плане.
Табл. 1. Условия амплификации при проведении HRM-анализа генетических полиморфизмов параоксоназы-1
|
Полиморфизм |
Праймер |
Последовательность |
T a , °C |
|
L55M, rs854560 |
Прямой (первая пара) |
5' GAA GAG TGA TGT ATA GCC CCA G 3' |
64.5 |
|
Обратный (первая пара) |
5' TTT AAT CCA GAG CTA ATG AAA GCC 3' |
||
|
Прямой (вторая пара) |
5' AAG AGG ATT CAG TCT TTG AGG 3' |
60 |
|
|
Обратный (вторая пара) |
5' TTG AAA TGA CCT GTG AAG TAG 3' |
||
|
Q192R, rs662 |
Прямой |
5' TAT TGT TGC TGT GGG ACC TGA G 3' |
63 |
|
Обратный |
5' CAC GCT AAA CCC AAA TAC ATC TC 3' |
PON1
T, Leu
PON1 Q192R rs662
A, Glu
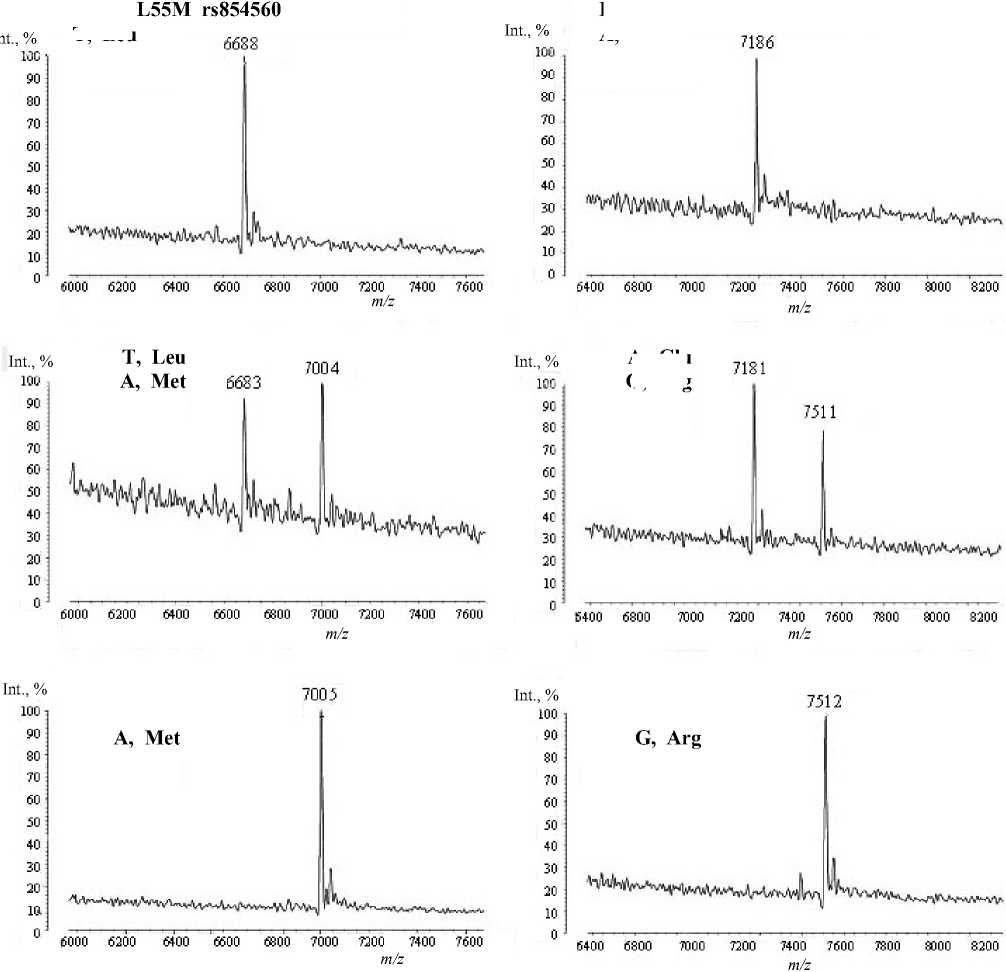
Рис. 1. Масс-спектры продуктов реакций минисеквенирования по полиморфизмам rs854560 и rs662. Указан аллельный вариант нуклеотида и соответствующая аминокислота в белковом продукте
A, Glu
G, Arg
МАТЕРИАЛЫ И МЕТОДЫ
Отбор биологического материала и выделение геномной ДНК проводили согласно условиям и методам, описанным нами ранее [7]. Всего проанализировали образцы ДНК девяноста четырех здоровых доноров г. Кирова, пгт. Оричи и п. Мирный Кировской области: сорока восьми мужчин и сорока шести женщин.
Амплификацию и HRM-анализ полиморфизм-содержащих фрагментов ДНК проводили на ам- плификаторе CFX96 (Bio-Rad, США) c использованием реагента SsoFast EvaGreen Supermix (BioRad, США). Последовательности использованных в работе праймеров указаны в табл. 1. Реакции проводили в объеме 25 мкл, количество геномной ДНК составляло 30–40 нг на реакцию, количество каждого праймера — 10 пмоль. Амплификацию проводили по следующей программе: 96 °С — 5 мин; далее 40 циклов (96 °С — 15 с, Ta — 30 с); затем 72 °С — 5 мин. Плавление продуктов амплификации проводили в диапазоне
65–95 °C с увеличением температуры на 0.2 °С каждые 10 с. Обработку полученных данных проводили в программной среде Precision Melt Analysis Software (Bio-Rad).
Анализ образцов геномной ДНК проводили также методом минисеквенирования и детектировали в линейном режиме с помощью МАЛДИ-масс-спектрометра Axima Perfomance. В ходе исследования использовали наборы для определения полиморфизма rs854560 и rs662 производства OOO НПФ "Литех". В случае набора MS-045 (rs854560) анализ проводили полностью согласно протоколу производителя. В случае набора MS-046 (rs662) стадию амплификации фрагмента исследуемого гена проводили так же, как и при рестрикционном анализе; последующие стадии анализа проводили согласно протоколу производителя с использованием прилагаемых к набору реагентов. К образцам, содержащим продукты минисеквенирования, добавляли по 15 мкл катион-обменной смолы, суспендированной в воде. Образцы помещали на подвижную платформу и оставляли перемешиваться на 1 ч при комнатной температуре. Затем пробу оставляли на 10 мин для выпадения смолы в осадок. Супернатант использовали для масс-спектрометрического анализа. На мишень для МАЛДИ-масс-спектрометрии наносили 0.5 мкл матрицы (3-гидроксипиколиновой кислоты). Аликвоту образца, полученного после процедуры очистки, в объеме 0.5 мкл смешивали с каплей раствора матрицы на мишени и оставляли при комнатной температуре до полного высыхания и кристаллизации. Интерпретацию генотипов проводили на основе масс-спектров продуктов реакции минисеквенирования.
Для верификации полученных результатов HRM-анализа продукты амплификации подвергали рестрикционному анализу. В случае рестрикционного анализа полиморфизма rs854560 использовали рестриктазу Hin1II (NlaIII) (Fermentas), в случае полиморфизма rs662 — рестриктазу BspPI (AlwI) (Fermentas). Реакции рестрикции проводили согласно протоколам производителя с использованием буфера Tango (Fermentas). Фрагменты ДНК разделяли электрофоретически в 1 % либо 3 % агарозном геле, содержащем 0.1 мкг/мл бромистого этидия, при напряжении 5 В/см в течение 60 мин. Использовали буфер TAE. Визуализацию фрагментов проводили на трансиллюминаторе при длине волны 312 нм. Ампликон на полиморфизм rs854560 размером 170 пар нуклеотидов (первая пара праймеров) в случае генотипа ММ полностью гидролизовался на фрагменты размером 126 и 44 нуклеотидных пар [8]. В случае использования второй пары праймеров формировался ампликон размером 712 пар нуклеотидов (п. н.), в котором имелось три конститутивных сайта рестрикции. В случае аллеля М появлялся еще один, в результате чего наблюдали гидролиз фрагмента размером 421 п. н. на фрагменты 295 и 126 п. н. Ампликон на полиморфизм rs662 размером 99 п. н. в случае генотипа RR полностью гидролизовался на фрагменты размером 66 и 33 п. н. [8].
Равновесность популяции и принадлежность выборок одной генеральной совокупности проверяли методом χ2 ( ν = 2; α = 0.05), теоретические частоты фенотипов получали в соответствии с уравнением Харди—Вайнберга.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
На рис. 1 представлены масс-спектры для всех проанализированных генотипов (указан аллельный вариант нуклеотида и соответствующая аминокислота в белковом продукте). В табл. 2 указаны массы продуктов минисеквенирования и указано соответствие аллельным вариантам. Незначительные различия в массах на рис. 1 и в табл. 2 можно объяснить погрешностью прибора в данном диапазоне масс, но эти различия не влияли на однозначную идентификацию полиморфизмов.
HRM-анализ по полиморфизму rs854560 был затруднен заменой A → T. Такой тип замен (как и G → C) является слабым местом данного метода из-за сходных термодинамических свойств ампликона [9]. При использовании первой пары праймеров (табл. 1) не удавалось различить между собой гомозиготы LL и MM, которые образовывали единый кластер (рис. 2).
Табл. 2. Соответствие масс-спектров продуктов минисеквенирования аллельным вариантам параоксоназы-1
|
Полиморфизм |
Кат. номер набора |
Зонд |
Аллель А (нуклеотид, аминокислота) |
Аллель В (нуклеотид, аминокислота) |
|
L55M, rs854560 |
MS-045-100 |
6382 Да |
6679 Да (T, Leu) |
6999 Да (A, Met) |
|
Q192R, rs662 |
MS-046-100 |
6876 Да |
7173 Да (A, Glu) |
7502 Да (G, Arg) |
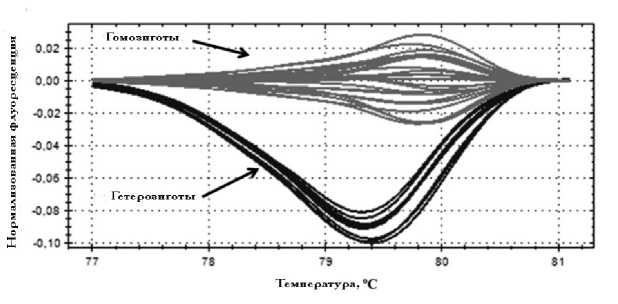
Рис. 2. Кривые плавления ампликонов (первая пара праймеров), содержащих полиморфизм rs854560 (нормализация по гомозиготам LL и MM).
Кривые плавления гомозигот LL и MM не различаются между собой, образуя единый кластер, но заметно отличаются от кривой плавления гетерозигот LM
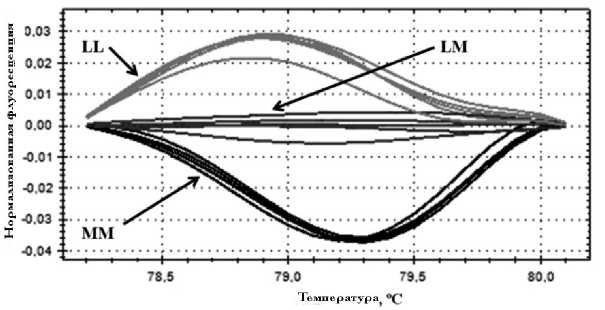
Рис. 3. Кривые плавления ампликонов (вторая пара праймеров), содержащих полиморфизм rs854560 (нормализация по гетерозиготам LM).
Видно отчетливое различие кривых плавления всех трех генотипов (LL, LM, MM) между собой
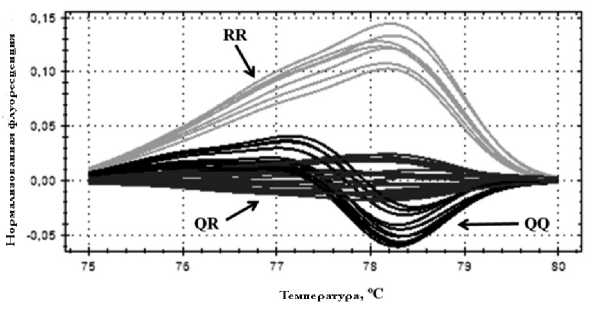
Рис. 4. Кривые плавления ампликонов, содержащих полиморфизм rs662 (нормализация по гетерозиготам QR).
Видно отчетливое различие кривых плавления всех трех генотипов (QQ, QR, RR) между собой
Использование второй пары праймеров оказалось более успешным и позволяло уверенно различать все три генотипа (рис. 3). Анализируемый регион кривой плавления устанавливали в диапазоне 78.2–80.1 °С. Ограничивающие (pre-melt, postmelt) регионы устанавливали в диапазонах 78.1– 78.2 °С и 80.1–80.2 °С соответственно. Успешность процедуры может быть связана с более выгодными для HRM-анализа термодинамическими характеристиками ампликона, нежели в случае с ампликоном размером 170 п. н. Другим объяснением может быть возможная сцепленность полиморфизма rs854560 с каким-то другим, попадающим в тот же ампликон. В этом случае можно было бы говорить об определении гаплотипов по двум SNP. Данное предположение предстоит проверить секвенированием. В случае же полиморфизма rs662 простота генотипирования определялась заметным влиянием замены (A или G) на Tm ампликона (порядка 78 °С для QQ и 78.5 °С для RR); кластеризация кривых плавления в соответствии с генотипами не вызывала затруднений (рис. 4). Анализируемый регион кривой плавления устанавливали в диапазоне 75–80 °С. Ограничивающие (pre-melt, post-melt) регионы устанавливали в диапазонах 74–75 °С и 80–81 °С соответственно.
Результаты минисеквенирования на полиморфизм rs854560 и rs662 полностью совпадали с результатами рестрикционного анализа.
Табл. 3. Наблюдаемое соотношение генотипов по полиморфизмам параоксоназы-1 и аллельные частоты
|
Полиморфизм |
Соотношение генотипов (индивидуумы) |
Аллельные частоты (%) |
|
L55M (rs854560) |
LL:LM:MM 43 : 38 : 13 |
L:M 66.0 : 34.0 |
|
Q192R (rs662) |
QQ:QR:RR 50 : 35 : 9 |
Q:R 71.8 : 28.2 |
При генотипировании населения Кировской области нами было получено распределение генотипов (табл. 3), которое соответствовало таковому в равновесной популяции с полученными аллельными частотами.
ЗАКЛЮЧЕНИЕ
Использование относительно простого и дешевого HRM-анализа применимо для генотипирования полиморфизмов rs854560 и rs662 параоксоназы-1. Данные были верифицированы рестрикционным анализом и МАЛДИ-минисеквенирова-нием. Использование данного подхода позволило вычислить частоты аллелей L (rs854560) и Q (rs662) среди населения Кировской области, которые оказались равными 0.660 и 0.718 соответственно. Полученные значения оказались сходны с таковыми для Санкт-Петербурга [10]. В целом же частоты аллельных вариантов по полиморфизмам параоксоназы-1 rs854560 и rs662 у населения Европейской части России сопоставимы с таковыми у населения Западной Европы [11] и белого населения США [12].


