Разнообразие бактерий, выделенных из района разработок месторождения калийных солей Верхнекамья
Автор: Ястребова О.В., Ананьина Л.Н., Пастухова Е.С., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 10, 2009 года.
Бесплатный доступ
Из почвы, воды и галитовых отходов района солеразработок (г. Соликамск, Пермский край) методом накопительных культур выделено 12 различных по грампринадлежности штаммов бактерий. По результатам генотипирования методами REP-ПЦР и ARDRA выделенные штаммы были объединены в пять различных геномогрупп. У представителей геномогрупп определены нуклеотидные последовательности гена 16S рРНК, сравнительный анализ которых показал принадлежность штаммов к родам Bacillus, Virgibacillus, Pseudomonas, Chromohalobacter и Idiomarina. Из образца породы калийно-магниевых солей выделены два штамма, идентифицированные как представители родов Bacillus и Streptomyces. В накопительной культуре из данного образца на полноценной среде c 20% NaCl обнаружены бактерии, близкородственные бактериям родов Ralstonia и Stenotrophomonas (сходство 98%). Большинство выделенных штаммов растет при 15-20% NaCl в среде культивирования, штаммы С2 и К52 способны к росту в щелочных условиях (pH 9.0). Штамм Pseudomonas sp. К513 использует в качестве субстрата бензойную кислоту в высокой концентрации (5 г/л) при содержании в среде культивирования 3% хлорида натрия.
Короткий адрес: https://sciup.org/147204449
IDR: 147204449 | УДК: 579.26:579.222.2
Текст научной статьи Разнообразие бактерий, выделенных из района разработок месторождения калийных солей Верхнекамья
-
aИнститут экологии и генетики микроорганизмов УрО РАН, 614081, Пермь, ул. Голева, 13
-
bПермский государственный университет, 614990, Пермь, ул. Букирева, 15
Из почвы, воды и галитовых отходов района солеразработок (г. Соликамск, Пермский край) методом накопительных культур выделено 12 различных по грампринадлежности штаммов бактерий. По результатам генотипирования методами REP-ПЦР и ARDRA выделенные штаммы были объединены в пять различных геномогрупп. У представителей геномогрупп определены нуклеотидные последовательности гена 16S рРНК, сравнительный анализ которых показал принадлежность штаммов к родам Bacillus, Virgibacillus , Pseudomonas, Chromohalobacter и Idiomarina . Из образца породы калийно-магниевых солей выделены два штамма, идентифицированные как представители родов Bacillus и Streptomyces. В накопительной культуре из данного образца на полноценной среде c 20% NaCl обнаружены бактерии, близкородственные бактериям родов Ralstonia и Stenotrophomonas (сходство 98%) . Большинство выделенных штаммов растет при 15-20% NaCl в среде культивирования, штаммы С2 и К52 способны к росту в щелочных условиях (pH 9.0). Штамм Pseudomonas sp. К513 использует в качестве субстрата бензойную кислоту в высокой концентрации (5 г/л) при содержании в среде культивирования 3% хлорида натрия.
В последние десятилетия интенсивное исследование микрофлоры экосистем с высоким засолением среды позволило выделить и охарактеризовать умеренно и экстремально галофильные бактерии, различные по систематическому положению и отличающиеся разнообразием ферментных систем (Ventosa et al., 1998). Данные бактерии представляют интерес в связи с высоким биотехнологическим потенциалом, в частности они могут быть использованы для получения осмопротектерных соединений, ферментов, активных при высоком осмотическом давлении среды, а также для создания биопрепаратов очистки почв и стоков со сложным, полихимическим характером загрязнения (Margesin, Schinner, 2001).
Цель работы – исследование разнообразия микроорганизмов почвы, воды, образца породы и галитовых отходов района солеразработок.
Методы исследования
Образцы почвы, воды, галитовых отходов, а также образец породы для постановки накопительных культур были отобраны на территории Верхнекамского месторождения калийно-магниевых солей (ОАО ”Сильвинит” г. Соликамск). Отбор образца почвы производился на глубине 5 см на расстоянии 2 м от солеотвала в осеннее время года. Проба воды была взята из прилегающего к солеотвалу водоема с глубины 5 см от поверхности и на расстоянии 70 см от береговой линии. Образец галитовых отходов отобран с глубины 5 см непосредственно с солеотвала.
Метод накопительного культивирования
Для выделения штаммов микроорганизмов использовали метод накопительного культивирования. Для постановки накопительных культур (НК) использовалась жидкая минеральная среда Раймонда (Розанова, Назина, 1982) с разными концентрациями NaCl (5, 10, 15, 20, 25%). В качестве субстратов использовались: бензоат (1 г/л) или триптон (0.5 г/л) и дрожжевой экстракт (0.25 г). Образцы почвы, воды или галитовых отходов (1 г) были помещены в 250 мл колбы с 100 мл ростовой среды. Инкубация проводилась в течение 2 недель на термокачалке (100 об/мин.) при температуре 28оС. Из накопительных культур путем высева на селективные агаризованные среды выделяли чистые культуры микроорганизмов. Чистота культур контролировалась путем высева на агаризованную полноценную среду Раймонда, содержащую 3% хлорида натрия.
Изучение физиологических свойств
Устойчивость бактерий к NaCl (концентрация соли от 3 до 25%) определяли на полноценной агаризованной среде Раймонда. Рост бактерий учитывали на седьмой день.
Рост бактерий при разных значениях рН определяли при концентрации Na+ 0.8-0.85 М в буферных системах (для рН 5 - ацетатный буфер, для рН 6-8 - фосфатный буфер, для рН 810 - трис-HCl буфер (“Методы общей бактериологии”, 1983), приготовленных на основе минеральной среды Раймонда, содержащей глюкозу в качестве источника углерода и энергии. Штаммы культивировались на агаризованной среде при рН 5, 7, 8, 9, 10. Рост учитывали на седьмой день.
Идентификация бактерий
Идентификацию бактерий проводили на основе изучения их культуральных, морфологических и биохимических свойств (Определитель бактерий Берджи, 1997; Методы общей бактериологии, 1983). Морфологию и жизненный цикл бактерий изучали у выращенных на агаризованной среде 12-72часовых культур с использованием световой микроскопии (фазовый контраст).
Амплификация гена 16S рРНК. Для амплификации использовали бактериальные праймеры 27F и 1492R. Амплификацию проводили так, как описано у Гавриш и др. (2004).
REP-ПЦР (полимеразная цепная реакция повторяющихся экстрагенетических палиндром-ных последовательностей ДНК) проводили по методу Versalovic (1994). Продукты реакции разделяли электрофорезом в 1.5% агарозном геле, приготовленном на 1 х ТВЕ буфере (Маниатис и др., 1984).
Расщепление ДНК эндонуклеазами рестрикции. 5 мкл ПЦР продукта были обработаны 1 ед. рестрикционных ферментов Hha I и Mse I (Fermentas, Литва), используя для каждой эндонуклеазы рекомендованный буфер и соответствующий температурный режим. Фрагменты рестрикции разделяли электрофорезом в 1.2% агарозном геле (Scortichini et al., 2002).
Денатурирующий градиентный гель-электрофорез ампликонов 16S рРНК был выполнен в 6% (в/об.) полиакриламидном геле, содержащем линейный денатурирующий химический градиент от 35 до 50%, где 100% составляет 7М мочевина и 40% - формамид, согласно протоколу Muyzer et al. (1993).
Определение нуклеотидной последовательности гена 16S рРНК. Секвенирование гена 16S рРНК, амплифицированного с использованием универсальных бактериальных праймеров (Гавриш и др., 2004), проводили с применением набора реактивов CEQ Dye Terminator Cycle Sequencing Kit на автоматическом секвенаторе MegaBASE 1000 (JSC GE “Healthcare”, США).
Филогенетический анализ .
Нуклеотидную последовательность 16S рДНК изучаемого изолята сравнивали с нуклеотидными последовательностями типовых штаммов близкородственных видов из базы данных GenBank с помощью программы CLUSTAL W и корректировали вручную (Thompson et al., 1994). Эволюционное расстояние, выраженное как число замен на 100 нуклеотидов, рассчитывали согласно методу Jukes, Cantor (1969). Построение филогенетического древа производили с помощью пакета программ TREECON c использованием метода “neighbor-joining” (NEIGHBOR) (Van de Peer, DeWachter, 1994). Оценку статистической достоверности ветвления (“bootstrap-анализ”) проводили с использованием соответствующей функции программы TREECON на основе 1000 альтернативных деревьев.
Результаты и обсуждение
Из образцов почвы, воды и галитовых отходов района солеразработок ОАО “Сильвинит” (г. Соликамск, Пермский край) методом накопительного культивирования (НК) на полноценной и минеральной среде Раймонда с разным содержанием NaCl были выделены 14 штаммов бактерий, различных по грампринадлежности, морфологии клеток и колоний.
Из НК на полноценной среде с содержанием NaCl 5 %, 10 и 15% выделено два грамположительных (С32, С41) и шесть грамотрицательных (В151, В152, В153, В154, В201, В202) штаммов бактерий. Выделенные штаммы обладают каталазной активностью, оксидазоотрицательны. Из НК на минеральной среде с бензоатом в качестве субстрата и содержанием NaCl 5% выделено четыре бактериальных штамма (К51, К52, К511 и К513), различных по грампринадлежности, каталазной и оксидазной активностям.
Проведено сравнение выделенных штаммов с использованием методов REP-ПЦР и ARDRA. Анализ полученных REP-ПЦР профилей фрагментов геномной ДНК исследуемых штаммов показал, что два штамма - К511 и К513 входят в одну геномогруппу (I тип) (рис. 1). Штаммы В201 и В202 также составляют одну геномогруппу (II тип). Штамм В153 отличается от представителей данных групп и выделен в отдельную геномогруппу (III тип).
м 1 2 3 4 5
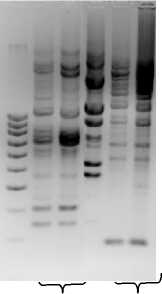
м 1 2 3 4 5 6 7 8 1 2 3 4 5 6 7 8
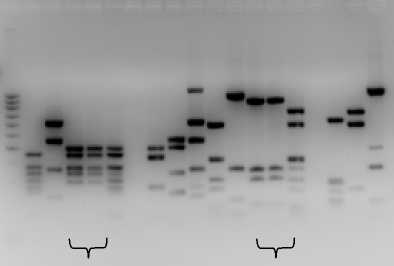
IV V I II III IV V I II III
I III II
А
Mbo I Hha I
B
Рис. 1. Электрофореграмма продуктов амплификации REP-ПЦР штаммов-деструкторов (A) и 16S рДНК, обработанных рестриктазами Mbo I и Hha I (B). На электрофореграмма А: м - маркер 1 kb (“Силекс”, Россия), 1 - К511, 2 - К513, 3 - В153, 4 - В201, 5 - В202. На электрофореграмма В: м - маркер 1 kb (“Силекс”, Россия), 1 - В151, 2 - В152, 3 - К513, 4 - К511, 5 - В201, 6 - Paenibacillus sp. SN501, 7 - Bacillus sp. 2508, 8 - В153.
0.02
I----------------------------------1
I B. sabtilis subsp. suhriiis DSM101 (AJ2 76351)
B. mojavensis IFO157181 (AB021191)
B. subtilis subsp. spiziaeui NRRLB-23049T(AFD749 7D)i
50 I B. vaiiismortis DSM110311
B. omrZatiy!Me/aciezisATCC23350T(X60605)
B. atrophaeus JCM90701 (ABO21181)
B. soHorensis NRRL B-231541 (EU138473)
В. lichMformis DSM131 (X68416)
63 -C2
B. as^us 24 KT fAJ831843^
B. tequileKsis 10b1 (AY197613)
98J B. pumiius DSMZ271 (AY456263)
1 B. safensis FO-OMb1 (AF234854)
B. uhtotdims 41KF2bT (AJS31842)
B. aerophihisl'SK1 (AJ831844)
B. stratosphericiis 41KF2aT (AJ831841)
B. idrienss S MC 4352-2T (AY904033)
100 I B. in&cus Sdtt1 (AJ583158)
B. cibi JG-3O1 (AY55O276)
----Б. taeanensis BH0300171 (AY6039^)
В. «igicola KMM3 7371 (AY22S462)
— В. hwajinpoensis SW-721 (AF541966) B. decoleradouis LMG 19507T(AJ315075)
AlicydobacUlus addocaMarius NBRC 15б52т (AB271754)
Рис. 2. Филогенетическое древо, построенное с использованием метода “neighbor-joining”, отображающее положение штамма С2 в системе рода Bacillus . Масштаб соответствует 2 нуклеотидным заменам на каждые 100 нуклеотидов. Цифрами показана статистическая достоверность порядка ветвления, определенная с помощью “bootstrap”-анализа 1000 альтернативных деревьев
Результаты рестрикционного анализа амплифицированных 16S рДНК (ARDRA) с использованием рестриктаз Mbo I и Hha I подтвердили принадлежность штаммов К511 и К513 к одной геномогруппе (I тип). Штаммы В201, В153, В151 и В152 отличаются от представителей данной группы и различаются между собой. Они выделены в отдельные геномогруппы II, III, IV и V типа, соответственно (рис. 2).
У представителей геномогрупп были определены нуклеотидные последовательности гена 16S рРНК размером около 1400 п.н.
Сравнительный анализ нуклеотидной последовательности гена 16S рРНК показал 99.5% сходство штамма К51 с типовым штаммом вида Bacillus alсalophilus . Штамм К513 имеет наибольший уровень гомологии с типовым штаммом вида Pseudomonas putida (99%), штамм К52 - с типовым штаммом вида Virgibacillus picturae (99,5%), штаммы В201 и В151 – c Chromohalobacter canadensis (99.4%), штаммы B152 и В153 – с типовым штаммом вида Idiomarina loihensis (99.5%).
Из образца породы калийно-магниевых солей методом НК на полноценной среде Раймонда, содержащей 5% NaCl, выделено два штамма: С31 и С2. Филогенетический анализ нуклеотидной последовательности гена 16S рРНК штамма С2 выявил его принадлежность к роду Bacillus . Наибольшее сходство (100%) изолят С2 имеет с B. licheniformis DSM13T
(X68416) (рис. 3). Штамм С31 имеет наибольший уровень гомологии с типовым штаммом вида Streptomyces ambofaciens (99.4%).
Грамположительные штаммы способны к росту как в отсутствие соли, так и при концентрации NaCl до 3%. Они являются галотолерантными микроорганизмами по классификации Кашнера (Кашнер, 1981). Грамотрицательные штаммы не растут в отсутствие соли в среде, способны расти при концентрации NaCl 10%–15%, а штамм В201 – до 20%. Данные штаммы относятся к галофильным микроорганизмам (таблица 1).
Все выделенные бактерии растут при значении рН 7.0, штаммы Bacillus sp. C2 и Virgibacillus sp. К52 являются алкалофильными и способны к росту в щелочных условиях среды - рН 9 (табл. 1). Способность спорообразующих бактерий к росту в условиях повышенной солености и высоких значений pH среды описана для ряда галоалкалофильнх штаммов рода Bacillus, выделенных из содовых озер и солончаков (Vargas et al., 2005; Sorokin et al., 2008).
Два штамма - Bacillus sp. К51 и Virgibacillus sp. К52 - способны к слабому росту на бензойной кислоте в среде, содержащей NaCl в концентрации до 4 %.
Грамотрицательный штамм Pseudomonas sp. К513 эффективно растет на бензоате (1 г/л) в среде без добавления соли и в среде, содержащей до 6% хлорида натрия.
Таблица 1
Рост штаммов бактерий в полноценной среде Раймонда при различных значениях рН среды и в присутствии различных концентраций хлорида натрия
|
Штамм |
Концентрация NaCl (%) |
Значение pH |
||||||
|
Без NaCl |
3 |
15 |
20 |
6.0 |
7.0 |
8.0 |
9.0 |
|
|
Bacillus sp. C2 |
+ |
+ |
+ |
+ |
+ |
+ |
± |
|
|
Streptomyces sp. C31 |
+ |
+ |
+ |
+ |
+ |
|||
|
Chromohalobacter sp. B151 |
+ |
+ |
||||||
|
Idiomarina sp. B152 |
+ |
+ |
||||||
|
Idiomarina sp. B153 |
+ |
+ |
+ |
|||||
|
Chromohalobacter sp. B201 |
+ |
+ |
+ |
+ |
||||
|
Chromohalobacter sp. B202 |
+ |
+ |
+ |
|||||
|
Pseudomonas sp. К511 |
+ |
+ |
+ |
|||||
|
Pseudomonas sp. К513 |
+ |
+ |
+ |
|||||
|
Bacillus sp. К51 |
+ |
+ |
+ |
|||||
|
Virgibacillus sp. К52 |
+ |
+ |
± |
+ |
+ |
+ |
||
Примечание: “+” – хороший рост (колонии размером более 3 мм); “±” - средний рост (колонии размером 1-2 мм); “–“ - отсутствие роста бактерий.
Известна способность ряда грамотрицательных галофильных штаммов, в частности Halomonas campisalis и Chromohalobacter sp. HS-2, к утилизации бензоата в концентрации 1.6 г/л и 0.6 г/л соответственно (Kim et al., 2008; Oie et al., 2007). Выделенный нами штамм К513 растет на данном субстрате в высокой концентрации - до 5 г/л (рис. 3).
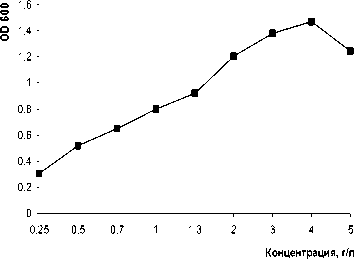
Рис. 3. Максимальное значение оптической плотности штамма К513 в минеральной среде Раймонда (3% NaCl) при разных концентрациях бензойной кислоты
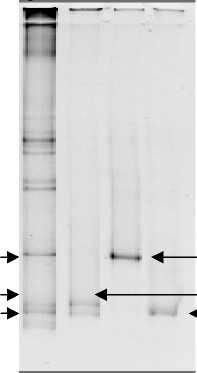
I Uncultured bacterium
II « Ralstonia pickettii »
III « Stenotrophomonas maltophilia »
Рис. 4. ДГГЭ продуктов амплификации фрагмента гена 16S рРНК, полученных с матрицы суммарной бактериальной ДНК накопительной культуры, культивируемой на 20% NaCl ( 1 ); 2, 3, 4 – реамплифицированные элюаты
1 2 3 4
Из образца породы калийно-магниевых солей получена накопительная культура на полноценной среде Раймонда, содержащей 20% NaCl. В НК преобладали палочковидные прямые и искривленные клетки, одиночные, в парах и собранные в цепочки. Размер клеток варьировал от 0.4 х 0.9 мкм до 0.5 х 2.8 мкм. Однако при высеве на агаризованные среды с разным содержанием хлорида натрия (от 3% до 20%) бактериального роста не наблюдалось. Для дальнейшего исследования НК был использован метод денатурирующего градиентного гель-электрофореза (рис. 4).
У фрагментов 16S рДНК, отличающихся электрофоретической подвижностью и элюированных из агарозного геля, определены нуклеотидные последовательности. Показано, что в составе НК присутствуют бактерии разных классов, близкородственные бактериям рода Ralstonia (сходство с типовым штаммом Ralstonia pickettii HPC578T 95%) и рода Stenotrophomonas (сходство с типовым штаммом Stenotrophomonas maltophilia PSM-1T 99%).
Заключение
В исследуемых образцах почвы, воды и галитовых отходов района солеразработок обнаружены бактерии, отличающиеся по физиологическим характеристикам и таксономическому положению. Выделены бактерии класса Gammaproteobacteria (родов Pseudomonas, Chromohalobacter, Idiomarina, Stenotrophomonas), класса Betaproteobacteria (рода Ralstonia), класса Bacilli (родов Bacillus, Virgibacillus) и класса Actinobacteria (рода Streptomyces). Исследуемые штаммы представлены галофильными, галотолерантными и галоалкалофильными бактериями, ряд штаммов способен к деструкции бензоата при высокой солености среды. Изолированные штаммы бактерий являются перспективными для использования при биоремедиации почв и стоков с повышенным содержанием солей и решения ряда других биотехнологических задач.
Работа поддержана грантом РФФИ-Урал №07-04-96078_а., ФЦП “Научные и научнопедагогические кадры инновационной России”, тема ”Разработка биокаталитических технологий синтеза органических кислот и энантиомерно-чистых соединений для полимерной химии, медицины и экологической биотехнологии на основе микроорганизмов-продуцентов, селекционированных из природных и антропогенно-измененных почв” ГК№02.740.11.0078.
Список литературы Разнообразие бактерий, выделенных из района разработок месторождения калийных солей Верхнекамья
- Гавриш, Е.Ю. Три новых вида бревибактерий -Brevibacterium antiguum sp. nov., Brevibacterium aurantiacum sp. nov. и Brevibacterium permense sp. nov./Е.Ю. Гавриш, В.И. Краузова, Н.В. Потехина и др.//Микробиология. 2004. Т. 73., №2. С. 218-225.
- Кашнер Д. Жизнь микробов в экстремальных условиях. М.: Мир, 1981. 365 с.
- Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии//Молекулярное клонирование. М.: Мир, 1984. 390 с.
- Методы общей бактериологии//пер. с англ.; под ред. Ф. Герхардт и др. М.: Мир, 1983. Т. 1, 2, 3.
- Определитель бактерий Берджи/пер. с англ.; под ред. Дж. Хоулта и др. М.: Мир, 1997. Т. 1, 2.
- Розанова Е.П. Углеводородокисляющие бактерии и их активность в нефтяных пластах/Е.П. Розанова, Т.Н. Назина//Микробиология. 1982. Т. 51. С. 324-348.
- Jukes T.H.//Mamallian protein metabolism/T.H. Jukes, C.R. Cantor. New York: Acad. Press. 1969. P. 21-132.
- Kim D. Molecular cloning and functional characterization of the genes encoding benzoate andp -hydroxybenzoate degradation by the halophilic Chromohalobacter sp. strain HS-2/D. Kim, S.W. Kim, K.Y. Choi//FEMS Microbiol. Lett. 2008. Vol. 280. P. 235-241.
- Margesin R. Potential of halotolerant and halophilic microorganisms for biotecnilogy/R. Margesin, Schinner F.//Extremophiles. 2001. Vol. 5. P. 73-83.
- Muyzer G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA/G. Muyzer, E.C. de Waal, A.G. Uitterlinden//Appl. Environ. Microbiol. 1993. Vol. 59. P. 695-700.
- Oie C.S.I. Benzoate and salicylate degradation by Halomonas campisalis, an alkaliphilic and moderately halophilic microorganism/C.S.I. Oie, C.E. Albaugh, B.M. Peyton//Water research. 2007. Vol. 41. № 6. P. 1235-1242.
- Scortichini М. Bacteria associated with hazelnut (Corylus avellana L.) decline are of two groups: Pseudomonas avellanae and strains resembling P. syringae pv. syringae/М. Scortichini, U. Marchesi, M.-P. Rossi//Appl. Environ. Microbiol. 2002. Vol. 68. P. 476-484.
- Sorokin I.D., Haloalkaliphilic diazotrophs in soda solonchak soils/I.D. Sorokin, I.K. Kravchenko, E.V. Doroshenko//FEMS Microbiol Ecol. 2008. V. 65. P. 425-433.
- Thompson J.D. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice/J.D. Thompson, D.G. Higgins, T.J. Gibson//Nucleic. Acids. Res. 1994. Vol. 22. P. 4673-4680.
- Van de Peer Y. TREECON for Windows a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment/Y. Van de Peer, R. DeWachter//Comput. Appl. Biosci. 1994. Vol. 10. P. 569-570.
- Vargas A.V. Bacillus bogoriensis sp. nov., a novel alkalophilic, halotolerant bacterium isolated from Kenyan soda lake/A.V. Vargas, O.D. Delgado, R. Hatti-Kaul, B. Mattiasson//International Journal of Systematic and Evolutionar Microbiology. 2005. Vol. 55. P. 899-902.
- Ventosa A. Biology of halophilic aerobic bacteria/A. Ventosa, J.N. Joaquhn, A. Oren//Microbiol. and Molec.Biol. Rev. 1998. Vol.2. P. 504-544.
- Versalovic J. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction/J. Versalovic, M. Schneider, F.J. de Bruijn//Meth. Cell. Mol. Biol. 1994. Vol. 5. P. 25-40.


