Разработка ДНК-маркеров признака устойчивости подсолнечника к расе G заразихи (Orobanche cumana Wallr.)
Автор: Савиченко Д.Л., Гучетль С.З., Логинова Е.Д.
Рубрика: Селекция, семеноводство и биотехнология сельскохозяйственных растений
Статья в выпуске: 1 (193), 2023 года.
Бесплатный доступ
Заразиха кумская ( Orobanche cumana Wallr.) - облигатный паразит, способный вызвать значительное снижение урожая подсолнечника. Наиболее распространенной и вирулентной расой заразихи в Российской Федерации является раса G. Экологичным и экономически выгодным способом борьбы с ней является выращивание генетически устойчивых сортов и гибридов подсолнечника. Применение ДНК-маркеров при отборе устойчивых растений значительно повышает эффективность селекционного процесса. Для маркирования устойчивости подсолнечника к расе G заразихи с помощью биоинформатических подходов были разработаны новые ДНК-маркеры - четыре SCAR (sequence characterized amplified region) и 10 SSR (simple sequence repeat). Маркеры были проверены на 20 линиях и гибридах подсолнечника. По результатам проверки были поэтапно исключены из исследования все SSR- и два SCAR-маркера. SCAR-маркер RORS1 характеризовался наличием амплифицированного фрагмента ДНК длиной ≈ 168 п.н. и показал ассоциацию с признаком устойчивости, а SORS1 с длиной фрагмента ≈ 322 п.н. - связь с восприимчивым фенотипом. Из них была составлена маркерная система для мультиплексной ПЦР, которая позволила различать устойчивые и восприимчивые линии и гибриды, кроме линии ВК678 и гибридов F1, полученных с использованием ее в качестве родительской. Для устранения недостатков маркерной системы были сконструированы семь SCAR-маркеров, аналогичных SORS1. По результатам оценки был отобран маркер SORS9, характеризующийся наличием амплифицированного фрагмента ДНК длиной ≈ 217 п.н. у восприимчивых генотипов. Новая система маркеров RORS1/SORS9 для мультиплексной ПЦР была валидирована на 70 линиях и гибридах подсолнечника. Восприимчивые линии и гибриды характеризовались амплификацией только маркера SORS9, устойчивые линии - только маркера RORS1, а устойчивые гибриды - обоих маркеров. Разработанная маркерная система позволила отличить все восприимчивые линии и гибриды от устойчивых генотипов подсолнечника.
Подсолнечник, заразиха кумская, днк-маркер, маркер-вспомогательная селекция, устойчивость
Короткий адрес: https://sciup.org/142237981
IDR: 142237981 | УДК: 633.854.78:575:632.9 | DOI: 10.25230/2412-608X-2023-1-193-3-13
Текст научной статьи Разработка ДНК-маркеров признака устойчивости подсолнечника к расе G заразихи (Orobanche cumana Wallr.)
Введение : Подсолнечник является важной масличной и кондитерской культурой, четвертой по популярности в мире после пальмы, сои и рапса. Его масло содержит ненасыщенные жирные кислоты, фитостеролы и витамин Е, который, как известно, снижает уровень липидов низкой плотности, повышает иммунитет и защищает от сердечно-сосудистых заболеваний [1]. Существуют биотические факторы, ограничивающие выращивание подсолнечника, и одним из них является облигатный паразит – заразиха кумская ( Orobanche cumana Wallr.). Сильное поражение этим паразитом способно полностью уничтожить урожай. Популяции заразихи обычно подразделяют на расы в зависимости от их вирулентности. К настоящему времени в мире известно о восьми расах (A, B, С, D, E, F, G и H) [2]. Исследования расового состава заразихи с 25 полей шести регионов Российской Федерации (Самарская, Оренбургская, Воронежская, Белгородская области, Ставропольский и Краснодарский края) показали, что на большинстве полей доминирует раса G заразихи, на некоторых преобладают менее вирулентные – Е и F. A наиболее вирулентный биотип (Н) выявлен на полях практически всех иссле-4
дованных регионов [3]. Для снижения негативных последствий, вызываемых паразитом, применяются различные дорогостоящие агротехнологические приемы, в особенности обработка посевов гербицидами [4]. Возделывание же устойчивых сортов и гибридов подсолнечника отечественного происхождения позволит более экономно и экологически безопасно противодействовать заразихе. На данный момент отбор генотипов подсолнечника по признаку устойчивости к заразихе проводится только по результатам фенотипической оценки на инфекционном фоне [5]. Этот процесс характеризуется высокой трудозатратностью и недостаточно высокой пропускной способностью. Использование молекулярных маркеров позволит осуществлять отбор генотипов, содержащих ген устойчивости, в том числе при комбинировании разных генов и интрогрессии в другой селекционный материал, без обязательной массовой фитопатологической оценки. Разработка и дальнейшее использование маркерных систем для ускоренной селекции подсолнечника активно применяется во многих странах [6; 7; 8; 9]. Для разработки молекулярных маркеров устойчивости к заразихе зарубежными учеными выполнен ряд исследований по поиску местоположения генов устойчивости подсолнечника к разным ее расам. Расоспецифичный ген, контролирующий устойчивость к расе E – Or5, картирован в теломерной области хромосомы 3 на 7,5 сМ выше маркера ORS1036 [10]. В той же хромосоме позже был локализован ген устойчивости к расе выше F – orab-vl-8 [11]. Несмотря на близкое расположение к гену Or5, доказано, что эти гены различны. Позже I. Imerovski et al. картировали до 23 значимых QTL (Quantitative trait loci) устойчивости в геноме подсолнечника. Два основных QTL – or3.1 и or3.2 – располагались в хромосоме 3. QTL or3.1 был расположен в геномной области, где был картирован предшествующий ген устойчивости к за- разихе Or5, в то время как QTL or3.2 впервые был идентифицирован в нижней области той же хромосомы [12]. Также один ген устойчивости к расе F HaOr7 картирован на хромосоме 7 [13]. По некоторым данным, этот ген также может контролировать устойчивость к расам выше F [14]. Ген устойчивости к расе G OrDeb2, интрогрессированный в культурный подсолнечник от H. debilis subsp. tardiflorus, картирован в верхней половине хромосомы 4 [15]. На данный момент неизвестна локализация и не маркировано большинство генов устойчивости к расам заразихи у подсолнечника, возделываемого на территории РФ. Aвторами данной работы было исключено расположение гена, контролирующего устойчивость к расе G заразихи подсолнечника у устойчивых линий ВНИИМК, в непосредственной близости к гену orab-vl-8 на хромосоме 3 [16]. Поскольку на полях РФ все еще преобладает раса G заразихи, актуальна разработка системы маркеров признака устойчивости подсолнечника к этой расе для осуществления маркер-вспомогательной селекции.
Мaтериaлы и методы. Материал исследования – 66 известных устойчивых и восприимчивых к расе G заразихи линий и гибридов селекции ВНИИМК, две линии-дифференциатора к расе F заразихи LC1093 и P96 и два устойчивых коммерческих гибрида иностранной селекции СИ 'естер (Syngenta, Швейцария) и P64LC108 (Pioneer Hi-Bred International, Inc., СШA) (рис. 1).
Для разработки ДНК-маркеров использовались нуклеотидные последовательности из баз генетических данных RefSeq (Reference Sequence) и GenBank® [17]. Поиск микросателлитных локусов был проведен с помощью программного обеспечения GMATA [18]. Дизайн пар праймеров проводился с помощью онлайн-ресурса Primer-BLAST [19; 20] с in silico проверкой специфичности отжига на репрезентативном геноме подсолнечника HanXRQr2.0-SUNRISE [21; 22]. Для анализа биоинформатических данных использовалось программное обеспечение
UGENE (Унипро, РФ) [23]. Визуализация результатов исследования проводилась с помощью пакета R ggplot2 [24] с использованием программного обеспечения R версии 4.2.2 [25].
Выделение и очистка ДНК из растительного материала проводилась методом СТAВ с некоторыми модификациями [26], Diamond DNA Plant kit (Aлтайбио-тех, РФ) а также набором МагноПрайм® ФИТО (НекстБио, РФ) при помощи автоматической станции экстракции и очистки нуклеиновых кислот Auto-pure 96 (Allsheng, КНР). Качество выделенной ДНК определяли с помощью микроспектрофотометра Nano-300 (Allsheng, КНР).
Для проведения полимеразной цепной реакции (ПЦР) использовали 25 мкл реакционной смеси следующего состава: 2,5 мкл 10xПЦР-Буфер-Б для Taq ДНК-полимеразы; 2,5 мM MgCl 2 ; 0,2 мM каждого дезоксирибонуклеозидфосфата; по 10 пМ каждого праймера; 10–30 нг матричной ДНК и 1 ед. SynTaq ДНК-полимеразы (Синтол, РФ). ПЦР проводилась в амплификаторе нуклеиновых кислот MiniAmp™ (Thermo Fisher Scientific, СШA). Условия амплификации: начальная денатурация – 3 мин 94 °С, затем 35 циклов: денатурация 94 °С – 30 сек, отжиг при 60 °С в течение 40 сек, элонгация – 40 сек при 72 °С, финальная элонгация – 5 мин. Для ПЦР анализа использовали два типа праймеров: SCAR ( s equence c haracterized a mplified r egion) и SSR ( s imple s equence r epeat).
Электрофорез продуктов амплификации проводили в агарозном геле (1,5 % агароза, 1хSB-буфер) с использованием камеры для горизонтального электрофореза SE.2 (Хеликон, РФ) в течение 1 ч при силе тока 50–58 mA и напряжении 80– 100 V. Окрашивание продуктов ПЦР осуществляли бромистым этидием. Результаты электрофореза документировали при помощи гель-документирующей видеосистемы GenoSens 2200 (Clinx, КНР). Размер фрагментов ДНК определяли с использованием программного обеспечения Image Lab Software (Bio-Rad, СШA) относительно маркера молекулярного веса 100–1000 п.н. (Синтол, РФ).
Разнообразие генотипов подсолнечника

Сурус Спринт СИ Честер
Натали Кубанский 93 Кубанский 86 Клип
Горфилд Горстар ВК989 ВК680А ВК678А ВК585 ВК580 ВК551 ВК508 ВК305 ВК304 ВКЗОЗ ВК302 ВК301
ВК23ИМИ ВК21сур ВК2ІКЛП
ВК1сур ВК1клл ВК1ими ВК1О1Б ВК101А
ВК101 ВД342
ВД22 ВА760 ВА568 ВАЗ 8 9 Аурус Ар неб
Амели RGP2 RGP1
RGN RGM RGL2 RGL1
RGB RGA RG
Р96 P64LC108 LC1093 2RGS 2RGNV 2RGN 2RGB 2RGA
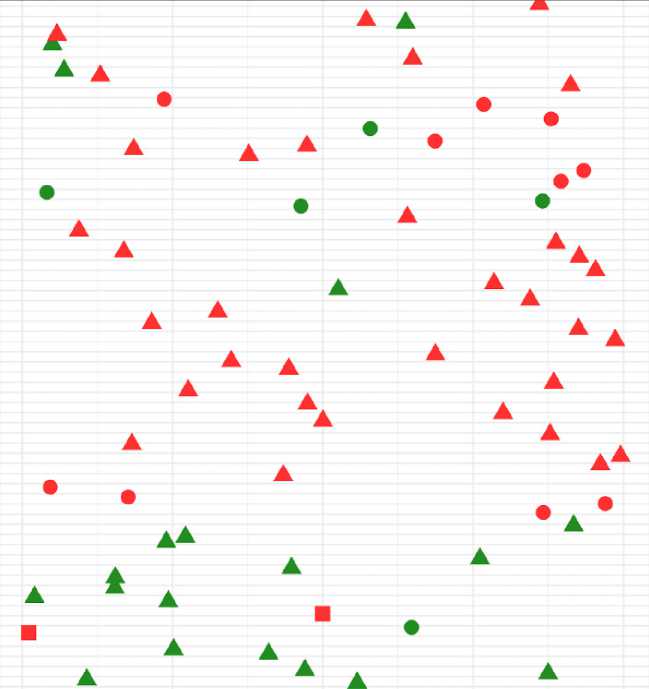
Гибрид
^^ Линия
U Ли ния-дифференциатор
Восприимчив
Устойчив
Случайное рассеивание
Рисунок 1 – Разнообразие генотипов подсолнечника, используемых в исследовании
Результaты и обсуждение . В наших предыдущих исследованиях по маркированию гена устойчивости подсолнечника к расе G заразихи было сделано предположение о расположении этого гена в нижней части третьей группы сцепления [16]. Для проверки данного предположения были разработаны новые ДНК-маркеры микросателлитных локусов этого региона генома подсолнечника. Также для проверки связи гена HaOr7 [13] с устойчивостью к расе G (РФ) заразихи нами были разработаны SCAR-маркеры на основе последовательностей Genbank®: MN219479, MN219480, MF374791, MF374792 [17]. В общей сложности сконструировано 10 SSR- и четыре SCAR-маркера.
Для анализа ассоциации маркеров с признаком устойчивости использовали 20 генотипов подсолнечника: один устойчивый коммерческий гибрид, 10 устойчивых и девять восприимчивых линий селекции ВНИИМК (табл. 1).
Таблица 1
ДНК-маркерами
|
Фенотип |
Название образца |
Маркер |
||||
|
SSR11 |
SSR27 |
RORS1 |
SORS1 |
SORS2 |
||
|
Устойчивый |
СИ 'естер |
247* |
179/191 |
+** |
+ |
+ |
|
RGP1 |
253 |
191 |
+ |
*** - |
- |
|
|
RGB |
247 |
191 |
+ |
- |
- |
|
|
RGP2 |
253 |
179 |
+ |
- |
- |
|
|
2RGNV |
253 |
179 |
+ |
- |
- |
|
|
RGA |
247 |
179 |
+ |
- |
- |
|
|
RGL1 |
253 |
197 |
+ |
- |
- |
|
|
RG |
253 |
197 |
+ |
- |
- |
|
|
RGN |
247 |
191 |
+ |
- |
- |
|
|
RGM |
247 |
191 |
+ |
- |
- |
|
|
RGL2 |
253 |
179 |
+ |
- |
- |
|
|
Восприимчивый |
ВК680 |
247 |
191 |
- |
+ |
+ |
|
Куб86 |
247 |
191 |
- |
+ |
+ |
|
|
ВA760 |
253 |
191 |
- |
+ |
+ |
|
|
Куб93 |
253 |
179 |
- |
+ |
+ |
|
|
ВК585 |
247 |
197 |
- |
+ |
+ |
|
|
ВК508 |
253 |
191 |
- |
+ |
+ |
|
|
ВК551 |
253 |
191 |
- |
+ |
+ |
|
|
ВК580 |
247 |
179 |
- |
+ |
+ |
|
|
ВК678 |
253 |
179 |
- |
+ |
+ |
|
*длина амплифицированного фрагмента ДНК
**наличие специфичного ПЦР-продукта
***отсутствие ПЦР-продукта
В ходе анализа маркеры оценивали на способность к стабильной амплификации, отсутствие неспецифичной гибридизации с ДНК-матрицей и амплификации нецелевого продукта ПЦР, и ассоциацию с признаком. По результатам анализа десяти SSR-маркеров шесть были исключены из исследования по причине отсутствия целевого ПЦР продукта, два – из-за отсутствия полиморфизма длин фрагментов. Таким образом, из 10 SSR-маркеров для дальнейших исследований было отобрано два – SSR11 и SSR27. Из четырех SCAR маркеров один показал отсутствие амплификации и был забракован.
Маркер SSR11 показал наличие двух аллелей микросателлитного локуса, показавших случайное распределение на выборке из 20 различающихся по устойчивости генотипов подсолнечника. Маркер SSR27 характеризовался наличием трех аллелей, также показавших случайное распределение. Таким образом, оба маркера не показали ассоциации с признаком устойчивости к расе G заразихи.
SCAR-маркер SORS2 демонстрировал ассоциацию с признаком на изучаемых генотипах, однако оказался непригоден для дальнейшей работы из-за амплификации неспецифичного ПЦР-продукта при анализе устойчивых линий. Маркер SORS1 показал отсутствие продуктов амплификации у всех устойчивых генотипов, кроме гибрида F 1 , у которого наблюдалось наличие амплифицирован-ного фрагмента ДНК, как и у всех восприимчивых линий. Маркер RORS1 демонстрировал наличие ПЦР-продукта у всех устойчивых и его отсутствие у восприимчивых генотипов подсолнечника (табл. 1). Длины амплифицированных фрагментов для маркера SORS1 соответствовали ожидаемым и составили ≈ 322 п.н., а для RORS1 ≈ 168 п.н., что позволяет эффективно разделять их в агарозном геле.
Пары праймеров были проверены in silico [27] и показали совместимость по температурному режиму, а также отсут- ствию вторичных структур, что позволило создать из них маркерную систему для мультиплексной ПЦР. Данная система была проверена на 29 генотипах подсолнечника (рис. 2).
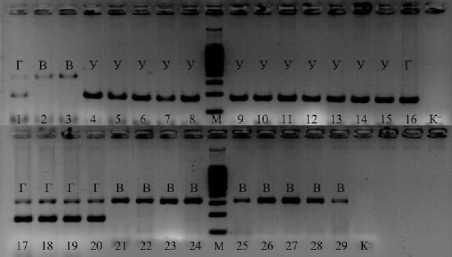
рисGнок 2 – Электрофоретические спектры амплифицированных фрагментов ДНК мультиплексной ПЦР с маркерами RORS1/SORS1.
Дорожки: 1 – СИ 'естер; 2 – ВК680; 3 – Куб86;
4 – RGP1; 5 – RGB; 6 – RGP2; 7 – 2RGNV;
8 – RGA; 9 – RGL1; 10 – RG; 11 – RGN;
12 – RGM; 13 – RGL2; 14 – 2RGB; 15 – 2RGA;
16 – F 1 ВК678 × RGM; 17 – F 1 ВК680 × RGP1;
18 – F 1 ВК680 × RGM; 19 – F 1 ВК680 × RGL2;
20 – F 1 ВК680 × RGL1; 21 – ВA760; 22 – Куб93; 23 – ВК585; 24 – ВК508; 25 – ВК551; 26 – ВК304; 27 – ВК580; 28 – ВA389; 29 – ВA568; М – маркер молекулярного веса; К- – отрицательный контроль;
Г – гибридное растение; У – устойчивая линия;
В – восприимчивая линия
Разработанная система маркеров RORS1 и SORS1 для мультиплексной ПЦР продемонстрировала кодоминантный характер наследования и полную ассоциацию с целевым признаком. Исключение составил гибрид материнской восприимчивой линии ВК678 и устойчивой линии RGM (рис. 2, № 16). У этого гибрида на электрофореграмме отсутствовал фрагмент ДНК, характерный для восприимчивого фенотипа. Для последующего анализа были взяты следующие генотипы: линия ВК678 и два гибрида – F1 (ВК678 × RGB) и F1 (ВК678 × RGP2). В качестве положительного контроля использовали гибридную комбинацию F1 (ВК680 × RGP1). Проведены две ПЦР: отдельно с маркером SORS1 и мультиплексом RORS1/SORS1 (рис. 3).
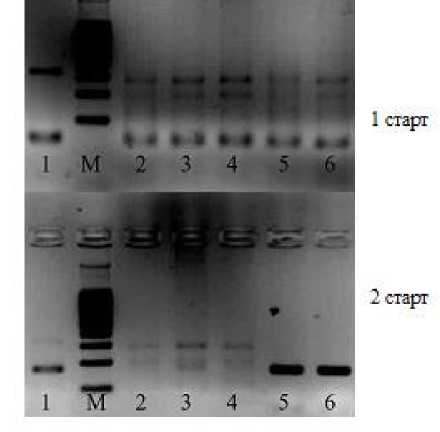
рисGнок 3 – Электрофоретические спектры амплифицированных фрагментов ДНК:
1 старт – с маркером SORS1; 2 старт – c маркерами RORS1/SORS1.
Дорожки: 1 – F 1 (ВК680 × RGP1);
М – маркер молекулярного веса; 2–4 – ВК678;
5 – F 1 (ВК678 × RGB); 6 – F 1 (ВК678 × RGP1)
В ходе обеих реакций контрольная гибридная комбинация F 1 (ВК678 × RGP1) повторно показала ожидаемые результаты: с маркером SORS1 получен один ПЦР продукт длиной ≈ 322 п.н., а в мультиплексной реакции RORS1/SORS1 получены два фрагмента ДНК размером ≈ 168 и 322 п.н. соответственно. У линии ВК678 использование маркера SORS1 привело к образованию двух неспецифичных продуктов длиной ≈ 300 и 200 п.н. Данный эффект наблюдался в обеих ПЦР с маркером SORS1 только у линии ВК678 и гибридов F1 (ВК678 × RGB) и F 1 (ВК678 × RGP1). В мультиплексной ПЦР у этих гибридов амплифицировались фрагменты ДНК длиной ≈ 168 п.н., соответствующие специфичному ПЦР-продукту маркера RORS1 и характеризующие родительские линии RGB и RGP2, а также отсутствовали неспецифичные продукты ПЦР. Таким образом, появление неспецифичных продуктов ПЦР наблюдалось у линии ВК678 и у гибридов, где она являлась родительской формой, только при отсутствии специфичной гибридизации праймеров маркера RORS1 с ДНК-матрицей. Это ограничило использование разработанной мультиплексной системы RORS1/SORS1 при анализе гибридов с линией ВК678.
Для устранения данного недостатка были in silico разработаны семь SCAR-маркеров аллеля дикого типа, альтернативных маркеру SORS1, с учетом совместимости с маркером RORS1 для постановки мультиплексной ПЦР. Новые маркеры были проверены на 30 линиях и гибридах подсолнечника селекции ВНИИМК, включая ВК678. В результате был отобран маркер SORS9. Он продемонстрировал стабильную амплификацию фрагмента ДНК с ожидаемым размером ≈ 217 п.н. у восприимчивых генотипов и отсутствие неспецифичного ПЦР-продукта в реакциях с устойчивыми линиями. Возможность постановки мультиплексной ПЦР с маркерами RORS1/SORS9 была проверена на 13 линиях и гибридах селекции ВНИИМК (рис. 4).
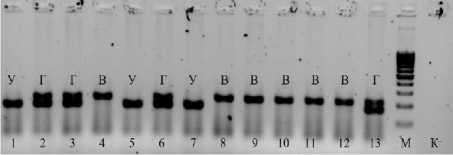
рисGнок 4 – Электрофоретические спектры амплифицированных фрагментов ДНК мультиплексной ПЦР с маркерами RORS1/SORS9. Дорожки: 1 – RGM;
2 – F 1 (ВК680 × RGM); 3 – F 1 (ВК680 × RGP1);
4 – ВК678; 5 – RGB; 6 – F 1 (ВК678 × RGP1);
7 – RG; 8 – ВК304; 9 – ВК551; 10 – ВA389;
11 – ВA568; 12 – ВК508; 13 – F 1 (ВК678 × RGM); М – маркер молекулярного веса;
К- – отрицательный контроль
Результаты анализа 13 генотипов подсолнечника с помощью мультиплексной
ПЦР с маркерами RORS1/SORS9 показали полное соответствие ожидаемых in silico и полученных ПЦР-продуктов – RORS1 ≈ 168 п.н. и SORS9 ≈ 217 п.н. Гибридные растения подсолнечника, полученные от скрещивания восприимчивого и устойчивого растений, характеризовались наличием обоих фрагментов.
Для последующей валидации разработанной системы маркеров был проведен анализ по наличию/отсутствию ПЦР-про-дуктов каждого маркера мультиплексной системы на 70 генотипах подсолнечника: 57 устойчивых и восприимчивых к расе G заразихи линий, в том числе двух линиях-дифференциаторах к расе F заразихи, пяти устойчивых и 10 восприимчивых гибридов (рис. 5).
Все устойчивые к расе G заразихи линии подсолнечника характеризовались наличием ПЦР-продукта длиной ≈ 168 п.н., специфичного маркеру RORS1, и отсутствием фрагмента ДНК длиной ≈ 217 п.н., специфичного маркеру SORS9. Все восприимчивые линии, в том числе две линии-дифференциатора к расе F, а также восприимчивые гибриды показали наличие специфичного ПЦР-продукта только маркера SORS9. Устойчивые гибриды характеризовались наличием обоих фрагментов ДНК маркеров RORS1 и SORS9.
Характеристика разработанных маркеров RORS1 и SORS9 представлена в таблице 2.
Таблица 2
%арактеристика ра7работанных маркеров RORS1 и SORS9
|
Название маркера |
Название праймера |
Нуклеотидная последовательность 5'-3' |
Длина амплифи-цируемого фрагмента ДНК, п.н. |
|
RORS1 |
RORS1 F |
ACCATCTTTGCT TCCTATGGCA |
168 |
|
RORS1 R |
CGTCACCCTTCT ATGTCGCT |
||
|
SORS9 |
SORS9 F |
AGTGCATTCGC AATCTTCGC |
217 |
|
SORS9 R |
TGCCATCCTCAT CATTAAGGG |
Анализ ассоциации маркерной системы с признаком устойчивости подсолнечника к расе G заразихи
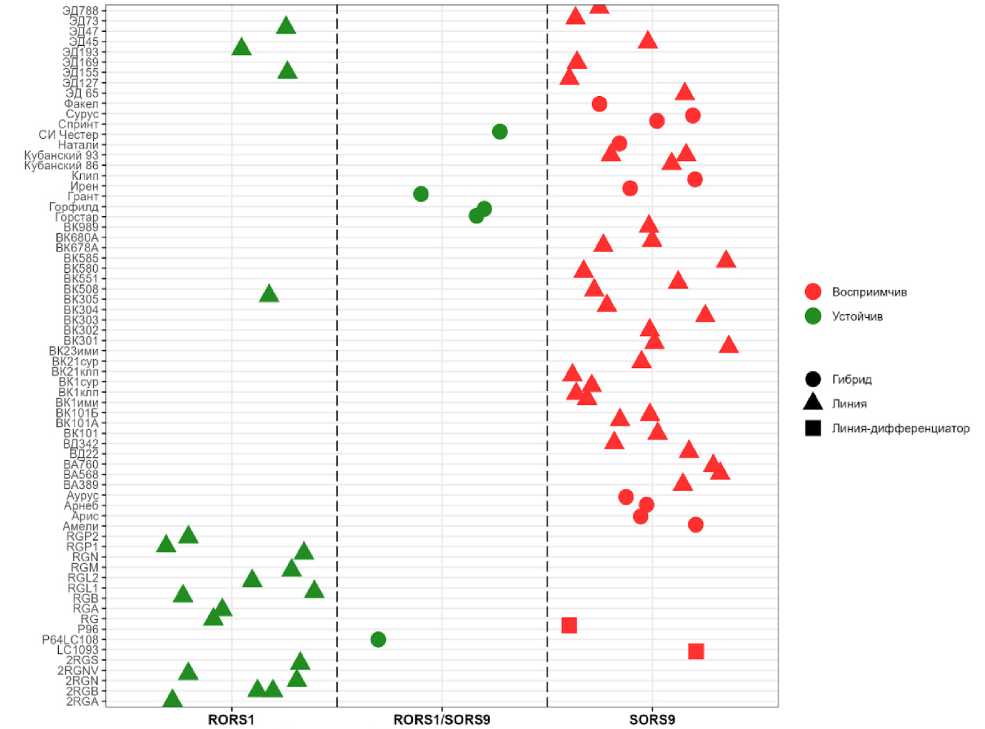
Наличие ПЦР-продукта маркеров
Рисунок 5 – Результаты анализа ассоциации маркерной системы RORS1/SORS9 с признаком устойчивости подсолнечника к расе G заразихи
Результаты наших исследований позволяют сделать вывод, что ген устойчивости к расе G заразихи у подсолнечника, возделываемого на территории РФ, локализуется на хромосоме 7 и является идентичным гену устойчивости к расе F HaOr7 , описанному Duriez et al. [13]. Однако на данный момент не были опубликованы ДНК-маркеры данного гена, что впервые в мире было сделано в нашей работе . В исследовании Martín‐Sanz et al. сообщалось, что этот ген также может контролировать устойчивость к расам выше F [14], что и было подтверждено с помощью разработанных нами маркеров. Для дальнейшей валидации маркерной системы получены семена самоопылен-ных гибридов, которые участвовали в данном исследовании. Будет проведен анализ сцепления маркерных фрагментов ДНК с целевым признаком в расщепляющихся популяциях F 2 .
Зaключение. Разработаны надежные SCAR маркеры RORS1 и SORS9 и создана маркерная система для мультиплексной ПЦР на наличие/отсутствие признака устойчивости к расе G заразихи в растениях подсолнечника. По результатам анализа 70 генотипов подсолнечника, отличающихся по устойчивости к расе G заразихи, установлено, что разработанная маркерная система позволяет различать устойчивые и восприимчивые линии подсолнечника, а также гибриды F 1 . Для дальнейшей валидации маркерной системы будет проведен анализ расщепляющихся популяций F 2 .
Список литературы Разработка ДНК-маркеров признака устойчивости подсолнечника к расе G заразихи (Orobanche cumana Wallr.)
- Staughton J. The amazing benefits of sunflower oil // Oilseeds Focus. - 2019. - V. 5. - I. 2. -P. 40-41.
- Cvejic S., Radanovic A., Dedic B., Jockovic M., Jocic S., Miladinovic D. Genetic and genomic tools in sunflower breeding for broomrape resistance // Genes. - 2020. - Vol. 11. - No. 2. -P. 152.
- Антонова Т.С., Арасланова Н.М., Сауко-ва С.Л., Ивебор М.В. К вопросу о засоренности полей в регионах РФ семенами заразихи (Orobanche cumana Wallr.) - облигатного паразита подсолнечника // Вестник российской сельскохозяйственной науки. - 2022. - № 4. -С. 29-32. DOI: 10.31857/2500-2082/2022/4/29-32.
- Лукомец В.М., Трунова М.В., Демурин Я.Н. Современные тренды селекционно-генетического улучшения сортов и гибридов подсолнечника во ВНИИМК // Вавиловский журнал генетики и селекции. - 2021. - Т. 25. -№ 4. - С. 388-393.
- Антонова Т.С., Стрельников Е.А., Арасланова Н.М., Гучетль С.З., Челюстнико-ва Т.А. Отбор на устойчивость к расе G заразихи из расщепляющихся популяций подсолнечника в искусственных условиях выращивания // Масличные культуры. Науч.-тех. бюл. ВНИИМК. - 2017. - Вып. 3 (171). - C.18-22.
- Davar R., Darvishzadeh R., Ahmad M. [et al.]. QTL mapping of partial resistance to basal stem rot in sunflower using recombinant inbred lines // Phytopathol. Mediterr. - 2010. - V. 49. -I. 3. - P. 330-341.
- Dimitrijevic A., Horn R. Sunflower hybrid breeding: from markers to genomic selection // Front Plant Sci. - 2018. - V. 8. - P. 2238. DOI: 10.3389/fpls.2017.02238.
- Dimitrijevic A., Imerovski I., Miladinovic D. [et al.]. Oleic acid variation and marker-assisted detection of Pervenets mutation in high-and low-oleic sunflower cross // Crop Breed. Appl. Bio-technol. - 2017. - V. 17. - I. 3. - P. 235-241. DOI: 10.1590/1984-70332017v17n3a36.
- Rauf S., Warburton M., Naeem A., Kainat W. Validated markers for sunflower (Helianthus an-nuus L.) breeding // OCL. - 2020. - V. 27. -P. 47.
- Tang S., Heesacker A., Kishore V.K., Fernandez A., Sadik E.S., Cole G., Knapp S.J. Genetic mapping of the Or¡ gene for resistance to Orobanche race E in sunflower // Crop Science. - 2003. - Vol. 43. - No. 3. - P. 1021-1028.
- Imerovski I., Dimitrijevic A., Miladinovic D., Dedic B., Jocic S., Tubic N.K., Cvejic S. Mapping of a new gene for resistance to broom-rape races higher than F // Euphytica. - 2016. -Vol. 209. - P. 281-289.
- Imerovski I. Dedic B., Cvejic S., Miladinovic D., Jocic S., Owens G.L., Rieseberg L.H. BSA-seq mapping reveals major QTL for broomrape resistance in four sunflower lines // Molecular Breeding. - 2019. - Vol. 39. - P. 1-15.
- Duriez P., Vautrin S., Auriac M.C., Ba-zerque J., Boniface M.C., Callot C. [et al.]. A receptor-like kinase enhances sunflower resistance to Orobanche cumana // Nature Plants. -2019. - Vol. 5. - No. 12. - P. 1211-1215.
- Martín-Sanz A. Pérez-Vich B., Rueda S., Fernández-Martínez J.M., Velasco L. Characterization of post-haustorial resistance to sunflower broomrape // Crop Science. - 2020. - Vol. 60. -No. 3. - P. 1188-1198. DOI: 10.1002/ csc2.20002.
- Fernández-Aparicio M., Del Moral L., Muños S., Velasco L., Pérez-Vich B. Genetic and physiological characterization of sunflower resistance provided by the wild-derived OrDeb2 gene against highly virulent races of Orobanche cumana Wallr. // Theoretical and Applied Genetics. - 2022. - Vol. 135. - No. 2. - P. 501-525.
- Гучетль С.З., Савиченко Д.Л. Анализ сцепления гена устойчивости к расе G заразихи с микросателлитными локусами у линии-донора подсолнечника селекции ВНИИМК RGP1 // Масличные культуры. - 2021. - Вып. 2 (186). - С. 3-9.
- База данных открытого доступа GenBank: [Электронный ресурс]. - Режим доступа: https://www.ncbi.nlm.nih.gov.
- WangX., Wang L. GMATA: an integrated software package for genome-scale SSR mining, marker development and viewing // Frontiers in Plant Science. - 2016. - Vol. 7. - P. 1350. DOI: 10.3389/ fpls.2016.01350.
- Онлайн-ресурс Primer-BLAST: [Электронный ресурс]. - Режим доступа: https ://www.ncbi.nlm.nih. gov/tools/primer-blast.
- Ye J., Coulouris G., Zaretskaya I. [et al.]. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction // BMC Bio-informatics. - 2012. -Vol. 13. - No. 134. DOI: 10.1186/1471-2105-13-134.
- Badouin H., Gouzy J., Grassa C.J. [et al.]. The sunflower genome provides insights into oil metabolism, flowering and Asterid evolution // Nature. - 2017. - Vol. 546. - No. 7656. - P. 148-152. DOI: 10.1038/ nature22380.
- Референсный геном подсолнечника: [Электронный ресурс]. - Режим доступа: https ://www.ncbi.nlm.nih.gov/ assembly/GCF_ 002127325.2.
- Okonechnikov K., Golosova O., Fursov M. Unipro UGENE: a unified bioinformatics toolkit // Bioinformatics. - 2012. - Vol. 28. - No. 8. - P. 1166-1167. DOI: 10.1093/bioinformatics/ bts091.
- Wickham H. Elegant graphics for data analysis (ggplot2) // Applied Spatial Data Analysis R. - 2009. - P. 65-90.
- Программная среда R для статистических вычислений и графики: [Электронный ресурс]. - Режим доступа: https://www.r-project.org/
- Boom R., Salmans M.M., Jansen C.L., Wertheim-van Dillen P.M. [et al.]. Rapid and simple method for purification of nucleic acids // Journal of clinical microbiology. - 1990. - Vol. 28. - No. 3. - P. 495-503.
- Онлайн-ресурс Multiple Primer Analyzer: [Электронный ресурс]. - Режим доступа: https://www.thermofisher.com/nl/en/home/brands /thermo-scien-tific/molecular-biology/molecular-biology-learning-cen-ter/molecular-biology-resour-ce-library/thermo-scientific-web-tools/multiple-primer-analyzer.html.


