Реконструкция генной сети шизофрении для поиска генов-мишеней
Автор: Дохоян А.Ю., Глущенко М.В., Орлов Ю.Л.
Журнал: Ульяновский медико-биологический журнал @medbio-ulsu
Рубрика: Клиническая медицина
Статья в выпуске: 3, 2022 года.
Бесплатный доступ
На данный момент шизофрения является плохо изученным заболеванием с многообразием симптомов, характерных для иных патологических состояний, и сложной диагностикой без однозначного лечения. Для поиска мишеней терапии необходима реконструкция генной сети заболевания, кластеризация генов в сети, выявление ключевых генов, обладающих наибольшим числом контактов в сети, нахождение категорий генных онтологий. Цель исследования - анализ генов, связанных с шизофренией, определение их положения в генной сети, установление их взаимосвязи, распознавание ключевых при протекании болезни, оценка их перспективности в качестве генов-мишеней для лекарственных воздействий. Материалы и методы. С помощью онлайн-инструментов биоинформатики OMIM, PANTHER и DAVID, GeneMANIA и STRING-DB, GeneCards был проанализирован актуальный на данный момент массив данных, связанных с шизофренией, рассчитаны категории генных онтологий для списка из 200 генов, такие как биологические процессы, молекулярные функции и клеточные ком-партменты, которые отражают влияние шизофрении на передачу нейронных импульсов, визуализированы и построены генные сети, содержащие выявленные ключевые объекты и их взаимосвязи, выделен сильно связанный кластер, включающий гены BDNF, SLC6A4, HTR2A, HTR2C, CHRM1, SRC, AKT, YWHAE, DISC1, DRD2, COMT, NDEL1, NOS1, CAMK28 и др., определены наиболее релевантные гены шизофрении: COMT, DISC1, HTR2A, NRXN1. Результаты. Биологическая интерпретация полученных результатов все еще остается сложной задачей, так как шизофрения является генетически сложной болезнью с большой расходимостью причин и условий возникновения. Анализ генов, связанных с шизофренией, определение их положения в генной сети (связанности) позволяет выявить их взаимосвязь, установить, какие из них являются ключевыми при протекании болезни, оценить их перспективность в качестве генов-мишеней для лекарственных воздействий.
Биоинформатика, шизофрения, реконструкция генных сетей, генные онтологии
Короткий адрес: https://sciup.org/14125357
IDR: 14125357 | УДК: 616.895.8, | DOI: 10.34014/2227-1848-2022-3-6-22
Текст научной статьи Реконструкция генной сети шизофрении для поиска генов-мишеней
Введение. Шизофрения – тяжелое полиморфное расстройство психики (или группа психических расстройств), для которого характерен распад процессов мышления и эмоциональных реакций [1]. По данным Global Health Data Exchange (GHDx) за 2019 г., шизофренией страдают примерно 0,32 % людей во всем мире, среди взрослых этот показатель составляет 0,45 % [2]. У мужчин шизофрения диагностируется немного чаще, чем у женщин (1,4:1) [3], причем пик заболеваемости прихо- дится на 20–28 лет, в то время как у женщин имеется тенденция к более позднему началу болезни (26–32 года) [4]. В детском возрасте (до 13 лет) шизофрения проявляется гораздо реже (1:10 000 детей [5] или 1:30 000 детей [6]), также редки случаи поздней (40–60 лет) и очень поздней (после 60 лет) шизофрении, составляющие 15 % и 4 % соответственно [7].
Единой причины развития шизофрении пока не установлено. Считается, что заболевание может развиваться в результате сложного комплексного взаимодействия генов и факторов окружающей среды [8–10]. Генетический вклад в развитие шизофрении составляет примерно 70–80 % [11], при этом разделить влияние генов и факторов окружающей среды достаточно сложно [12]. Отягощенная наследственность является одной из причин заболевания. Если болен один из родителей, риск развития заболевания у ребенка составляет около 13 %, если больны оба – почти 50 % [13]. Тем не менее гетерогенность фенотипических характеристик заболевания даже у родственников и слабое влияние каждого конкретного генетического варианта на риск развития болезни указывают на сложные механизмы наследования [14–16]. Также предполагается возможная генетическая корреляция между шизофренией и наличием других психических расстройств: расстройств аутистического спектра, биполярного расстройства, большого депрессивного расстройства, синдрома дефицита внимания и гиперактивности [17].
Таким образом, шизофрения – актуальная медицинская проблема. Активный исследовательский интерес к данной патологии обусловлен неуклонным ростом частоты ее встречаемости (распространение заболевания в популяции выросло с 13,1 млн в 1990 г. до 20,9 млн в 2016 г. [18]); инвалидизирующим влиянием на здоровье, психоэмоциональную, профессиональную сферы жизни пациентов; сложностью диагностики и отсутствием однозначного лечения. Реконструкция и анализ структуры генных сетей с помощью биоинформати-ческих методов позволяет выявить ключевые гены заболевания. Это необходимо для определения эффективности диагностики и терапии, а также дальнейшего подбора и исследования потенциальных вариантов фармакологических веществ, способных взаимодействовать с генами-мишенями, что в перспективе приведет к созданию лекарственных средств на их основе. Решение этих задач должно начаться со сбора списка генов, ассоциированных с развитием шизофрении, анализа категорий генных онтологий для такого списка и реконструкции генной сети. Для ключевых генов заболевания, полученных с помощью анализа структуры генной сети, будут рассмотрены варианты поиска лекарственных средств (веществ, взаимодействующих с данным белком).
Цель исследования. Анализ генов, связанных с шизофренией, определение их положения в генной сети (связанности), установление их взаимосвязи, распознавание ключевых при протекании болезни, оценка их перспективности в качестве генов-мишеней для лекарственных воздействий.
Материалы и методы. Составление списка генов, связанных с наследственной предрасположенностью к шизофрении, осуществлялось с использованием интернет-ресурса OMIM (Online Mendelian Inheritance in Man, . Поиск проводился по ключевому слову “schizophrenia” (шизофрения). Полученный список уточнялся и отфильтровывался вручную. Использовались только белок-кодирующие гены. Дополнительно применялся онлайн-ресурс для уточнения генов, ассоциированных с заболеванием.
С помощью ресурсов PANTHER (Protein ANalysis THrough Evolutionary Relationships, и DAVID (The Database for Annotation, Visualization and Integrated Discovery, был выполнен анализ обогащения категорий генных онтологий для заданного списка генов. Использовались стандартные статистические критерии, параметры описаны в тексте. Применялась корректировка на множественность гипотез (Бонферрони).
Реконструкция сети взаимодействий генов шизофрении проводилась с помощью двух ресурсов для оценки оптимальных возможностей обоих ресурсов для одного и того же списка генов: GeneMANIA и STRING-DB . Рисунки были построены в графическом формате.
Для определения наиболее релевантных генов шизофрении использовался интернет-ресурс GeneCards . Поиск проводился по ключевому слову “schizophrenia” (шизофрения).
Таким образом, была построена сеть для исследования функциональной и молекулярной связи генов в контексте заболевания.
Результаты. Используя ресурс OMIM, по запросу “schizophrenia” был получен актуаль- ный список из 256 наименований генов. Из набора исходных генов часть идентификаторов была не распознана системой или не могла быть определена в геноме человека однозначно. В итоге был получен список из 200 наименований генов. Использование информации о мутациях – наследственной информации в связи с заболеванием из базы OMIM – служит только основой для составления списка генов. Получившийся список можно использовать для поиска и анализа определенных генов, мутации которых часто встречаются при шизофрении, в целях нахождения более релевантных генов, передающих сигнал в сети.
Описание общих категорий генных онтологий позволяет выявить общие сущности в списке генов, например для молекулярных функций или типов структур белка, чего нельзя ожидать только от упоминания генов в связи с заболеванием. Выполнен анализ генных онтологий для того же списка генов с помощью ресурса PANTHER. Для получения наиболее информативных результатов значения p ограничили до 1,00E-07 (коррекция Бонферрони). Таким образом, с помощью PANTHER построена таблица онтологий для категорий биологических процессов (табл. 1).
Таблица 1
Table 1
Категории генных онтологий для генов шизофрении по PANTHER
(биологические процессы), порог значимости 1,00E-07
Gene ontology categories for schizophrenia genes by PANTHER (biological processes), significance threshold 1.00E-07
|
Категории генных онтологий для биологических процессов Gene ontology categories for biological processes |
Класс онтологий GO Ontology class |
Число генов Number of genes |
FC* |
p (Бонферрони) (Bonferroni) |
|
Регуляция процессов в многоклеточном организме Regulation of multicellular organismal process |
GO:0051239 |
54 |
3,27 |
3,21E-12 |
|
Регулирование транспортировки Regulation of transport |
GO:0051049 |
43 |
3,95 |
1,88E-11 |
|
Регулирование локализации Regulation of localization |
GO:0032879 |
53 |
3,09 |
7,00E-11 |
|
Позитивная регуляция биологического процесса Positive regulation of biological process |
GO:0048518 |
82 |
2,11 |
8,78E-11 |
|
Модуляция химической синаптической передачи Modulation of chemical synaptic transmission |
GO:0050804 |
22 |
8,30 |
4,90E-10 |
|
Регуляция транссинаптической сигнализации Regulation of trans-synaptic signaling |
GO:0099177 |
22 |
8,28 |
5,13E-10 |
|
Положительная регуляция метаболического процесса Positive regulation of metabolic process |
GO:0009893 |
61 |
2,58 |
7,29E-10 |
|
Регулирование каталитической активности Regulation of catalytic activity |
GO:0050790 |
47 |
3,18 |
1,72E-09 |
|
Категории генных онтологий для биологических процессов Gene ontology categories for biological processes |
Класс онтологий GO Ontology class |
Число генов Number of genes |
FC* |
p (Бонферрони) (Bonferroni) |
|
Позитивная регуляция клеточного процесса Positive regulation of cellular process |
GO:0048522 |
75 |
2,10 |
6,88E-09 |
|
Регуляция уровня нейротрансмиттеров Regulation of neurotransmitter levels |
GO:0001505 |
16 |
11,68 |
1,39E-08 |
|
Синаптический сигналинг Synaptic signaling |
GO:0099536 |
21 |
7,43 |
1,49E-08 |
|
Сигналинг Signaling |
GO:0023052 |
70 |
2,16 |
2,40E-08 |
|
Регуляция молекулярной функции Regulation of molecular function |
GO:0065009 |
52 |
2,70 |
2,80E-08 |
|
Клеточная коммуникация Cell communication |
GO:0007154 |
70 |
2,12 |
7,93E-08 |
Примечание. Таблица ограничена до значений ненормированного p-value<1,00E-07; * – диапазон изменения.
Note. The table was restricted to non-normalized p-value<1.0E-07; * – fold change.
Приведенные в табл. 1 данные показывают, что при шизофрении одними из значимых категорий для биологических процессов являются модуляция химической синаптической передачи (гены HTR2A , NRXN1 , DISC1 , DRD3 и др.), регуляция транссинаптической сигнализации ( HTR2A , NRXN1 , DISC1 , DRD3
и др.), регуляция уровня нейротрансмиттеров ( DBH , HTR2A , COMT , NRXN1 и др.), которые указывают на влияние шизофрении на нейронную передачу сигнала.
Далее с помощью PANTHER была построена таблица онтологий для молекулярных функций (табл. 2).
Таблица 2
Table 2
Категории генных онтологий для генов шизофрении по PANTHER (молекулярные функции), порог значимости 1,00E-02
Gene ontology categories for schizophrenia genes by PANTHER (molecular functions), significance threshold 1.00E-02
|
Категории генных онтологий для молекулярных функций Gene ontology categories for molecular functions |
Класс онтологий GO Ontology class |
Число генов Number of genes |
FC* |
p (Бонферрони) (Bonferroni) |
|
Активность регулятора ионных каналов Ion channel regulator activity |
GO:0099106 |
9 |
10,11 |
1,32E-03 |
|
Активность регулятора канала Channel regulator activity |
GO:0016247 |
9 |
9,77 |
1,74E-03 |
|
Категории генных онтологий для молекулярных функций Gene ontology categories for molecular functions |
Класс онтологий GO Ontology class |
Число генов Number of genes |
FC* |
p (Бонферрони) (Bonferroni) |
|
Связывание кальмодулина Calmodulin binding |
GO:0005516 |
10 |
7,92 |
2,51E-03 |
|
Активность рецепторов нейротрансмиттеров Neurotransmitter receptor activity |
GO:0030594 |
8 |
11,22 |
2,82E-03 |
|
Активность трансмембранных сигнальных рецепторов Transmembrane signaling receptor activity |
GO:0004888 |
25 |
2,98 |
2,84E-03 |
|
Активность молекулярного трансдуктора Molecular transducer activity |
GO:0060089 |
27 |
2,76 |
4,15E-03 |
|
Активность сигнальных рецепторов Signaling receptor activity |
GO:0038023 |
27 |
2,76 |
4,15E-03 |
|
Связывание сигнальных рецепторов Signaling receptor binding |
GO:0005102 |
27 |
2,69 |
6,69E-03 |
|
Связывание серотонина Serotonin binding |
GO:0051378 |
4 |
52,81 |
7,46E-03 |
|
Связывание аминов Amine binding |
GO:0043176 |
4 |
48,75 |
9,70E-03 |
Примечание. Таблица ограничена до значений ненормированного p-value<1,00E-02; * – диапазон изменения.
Note. The table was restricted to non-normalized p-value<1.00E-02; * – fold change.
Данные табл. 2 позволяют выявить наиболее значимые категории молекулярных функций в проявлении шизофрении: активность регулятора ионных каналов (гены NOS1 , NRXN1 , AKT1 , YWHAE и др.), связывание кальмодулина ( NOS1 , AKT1 , CAMK2B , PLCB и др.), активность рецепторов нейротрансмиттеров ( HTR2A , DRD3 , HTR2C и др.) и активность трансмембранных сигнальных рецепторов ( HTR2A , NRXN1 , DRD3 , HTR2C и др.), что свидетельствует о влиянии шизофрении на нейромедиаторы.
Далее с помощью PANTHER была построена таблица онтологий для клеточных компартментов (табл. 3).
Согласно табл. 3 наиболее значимыми категориями клеточных компартментов явля-
ются синапс (гены DBH , HTR2A , NRXN1 , DISC1 и др.), клеточный стык ( DBH , HTR2A , NRXN1 , DISC1 и др.), проекция нейронов ( HTR2A , COMT , NRXN1 , DISC1 и др.). Следует отметить также такую категорию с относительно невысоким уровнем значимости, как сомато-дендритный компартмент ( HTR2A , COMT , NRXN1 , APOE и др.). Эти данные подтверждают, что шизофрения влияет на синапс и, следовательно, передачу нервного импульса.
Далее список из 200 генов человека был загружен через интерфейс DAVID для поиска значимых категорий генных онтологий для этой группы генов. Было распознано 129 идентификаторов (табл. 4).
|
Таблица 3 Table 3 |
Категории генных онтологий для генов шизофрении по PANTHER (клеточные компартменты), порог значимости 1.00E-04
Gene ontology categories for schizophrenia genes by PANTHER (cellular compartments), significance threshold 1,00E-04
|
Категории генных онтологий для клеточных Класс Число генов компартментов онтологий GO Number FC* Gene ontology categories for cellular compartments Ontology class of genes |
p (Бонферрони) (Bonferroni) |
|
Синапс GO:0045202 41 4,92 Synapse |
1,11E-14 |
|
Клеточный стык GO:0030054 47 3,56 Cell junction |
4,51E-12 |
|
Глутаматергический синапс GO:0098978 19 9,18 Glutamatergic synapse |
9,45E-10 |
|
Постсинапс GO:0098794 22 5,69 Postsynapse |
1,06E-07 |
|
Проекция нейронов , Neuron projection |
1,23E-07 |
|
Проекция клетки, ограниченная плазматической мембраной GO:0120025 41 2,93 Plasma membrane bounded cell projection |
2,48E-07 |
|
Проекция клеток GO:0042995 42 2,86 Cell projection |
2,64E-07 |
|
Периферия клетки GO:0071944 72 1,78 Cell periphery |
1,67E-05 |
|
Интегральный компонент плазматической мембраны GO:0005887 32 3,05 Integral component of plasma membrane |
1,70E-05 |
|
Дендрит GO:0030425 19 4,88 Dendrite |
2,66E-05 |
|
Дендритное дерево GO:0097447 19 4,86 Dendritic tree |
2,79E-05 |
|
Сомато-дендритный компартмент GO:0036477 22 4 15 Somatodendritic compartment , |
3,02E-05 |
|
Пресинапс GO:0098793 17 5,39 Presynapse |
4,04E-05 |
|
Интегральный компонент пресинаптической мембраны GO:0099056 8 18,11 Integral component of presynaptic membrane |
4,83E-05 |
|
Категории генных онтологий для клеточных компартментов Gene ontology categories for cellular compartments |
Класс онтологий GO Ontology class |
Число генов Number of genes |
FC* |
p (Бонферрони) (Bonferroni) |
|
Внутренний компонент плазматической мембраны Intrinsic component of plasma membrane |
GO:0031226 |
32 |
2,91 |
5,21E-05 |
|
Плазматическая мембрана plasma membrane |
GO:0005886 |
67 |
1,80 |
7,81E-05 |
|
Внутренний компонент пресинаптической мембраны Intrinsic component of presynaptic membrane |
GO:0098889 |
8 |
16,46 |
9,57E-05 |
Примечание. Таблица ограничена до значений ненормированного p-value<1,00E-04; * – диапазон изменения.
Note. The table was restricted to non-normalized p-value<1.00E-04; * – fold change.
Таблица 4
Table 4
Категории генных онтологий для генов шизофрении по DAVID, порог значимости 1,0E-04Gene ontology categories for schizophrenia genes by DAVID, significance threshold 1.0E-04
|
Группа онтологий Ontology group |
Категория онтологий Ontology category |
Число генов Number of genes |
p-value (значимость) p-value (significance) |
Нормированное p по Бенджамини Normalized p (Benjamini) |
|
UP_KEYWORDS |
Synapse |
14 |
5,4E-8 |
6,3E-6 |
|
GOTERM_CC_DIRECT |
Cell junction |
14 |
3,2E-6 |
1,4E-4 |
|
GOTERM_CC_DIRECT |
Postsynaptic membrane |
10 |
4,6E-6 |
1,6E-4 |
|
UP_KEYWORDS |
Cell junction |
15 |
1,3E-5 |
6,1E-4 |
|
UP_KEYWORDS |
Postsynaptic cell membrane |
8 |
4,7E-5 |
1,8E-3 |
|
GOTERM_BP_DIRECT |
Lipoprotein metabolic process |
8 |
3,3E-9 |
2,0E-6 |
|
GOTERM_MF_DIRECT |
Lipid binding |
8 |
4,4E-5 |
4,7E-3 |
|
GOTERM_BP_DIRECT |
Visual learning |
8 |
1,1E-8 |
4,7E-6 |
|
GOTERM_BP_DIRECT |
Cellular calcium ion homeostasis |
9 |
1,2E-7 |
1,8E-5 |
|
KEGG_PATHWAY |
Neuroactive ligandreceptor interaction |
14 |
9,5E-7 |
5,1E-5 |
|
SMART |
SM01381 |
9 |
1,2E-6 |
1,0E-4 |
|
Группа онтологий Ontology group |
Категория онтологий Ontology category |
Число генов Number of genes |
p-value (значимость) p-value (significance) |
Нормированное p по Бенджамини Normalized p (Benjamini) |
|
KEGG_PATHWAY |
cAMP signaling pathway |
11 |
1,1E-5 |
3,4E-4 |
|
GOTERM_CC_DIRECT |
Plasma membrane |
44 |
4,4E-5 |
1,0E-3 |
|
UP_KEYWORDS |
Cell membrane |
34 |
6,7E-5 |
2,2E-3 |
|
GOTERM_BP_DIRECT |
Synapse assembly |
8 |
1,0E-7 |
1,8E-5 |
|
KEGG_PATHWAY |
Serotonergic synapse |
8 |
6,2E-5 |
1,7E-3 |
Примечание. Таблица ограничена до значений ненормированного p-value<1,0E-04; удалены категории (строки таблицы) для групп меньше 8 генов.
Note. The table was restricted to non-normalized p-value<1.0E-04; categories (table rows) for groups of less than 8 genes are removed.
Приведенные в табл. 4 данные показывают, что наиболее значимыми категориями для генов шизофрении являются синапс (Synapse) (гены HTR2A , DISC1 , NRXN1 , NOS1 и др.), клеточный стык (cell junction) ( HTR2A , DISC1 , NOS1 , NRXN1 и др.) и нейроактивное лиганд-рецепторное взаимодействие (Neuroactive ligand-receptor interaction) ( HTR2A , HTR2C , DRD3 , CHRM1 и др.).
Таким образом, анализ категорий генных онтологий по PANTHER и DAVID для генов шизофрении подтверждает наличие категорий генных онтологий, связанных с синаптической сигнализацией и передачей межклеточных сигналов при помощи нейромедиаторов, что соответствует довольно распространенной гипотезе развития данного заболевания – катехоламиновой. Действительно, различные исследования показывают, что у многих больных шизофренией повышен уровень дофамина и серотонина в некоторых отделах мозга [19]. Были найдены часто встречаемые гены: HTR2A , NRXN1 , DISC1 , DRD3 , DBH , COMT , NOS1 , AKT1 , HTR2C .
Для нахождения генов-мишеней использовались все результаты поиска в OMIM с запасом, при этом все гены не обязательно должны были взаимодействовать друг с другом – часть генов не входит в сеть, не образует связей (редкие проявления заболевания, обусловленные наследственностью). На рис. 1 представлена генная сеть из 138 генов шизофрении, реконструированная с помощью GeneMANIA. Сеть включает 1767 взаимодействий, автоматически построенных по литературным данным. Гены в реконструированной сети имеют много взаимодействий разных типов, установленных как экспериментально, так и по косвенным данным. Автоматически сгенерированное изображение достаточно громоздко для восприятия, поэтому оно было отредактировано вручную (рис. 1). Сеть получилась достаточно связной, хотя связи выставлялись только по параметрам «коэкспрессия» и «белок-белковые взаимодействия». Снизу построенной сети находятся гены, которые являются статистически значимыми для шизофрении по данным GeneCards, – COMT, DISC1, HTR2A, NRXN1. Чуть выше – гены, имеющие наибольшее число связей с другими элементами, – POMC, PLCB1, APP, YWHAE. Справа расположены гены, которые являются основными генами-мишенями для лекарственных воздействий при терапии шизофрении, – DRD2, BDNF, COMT, SRR.
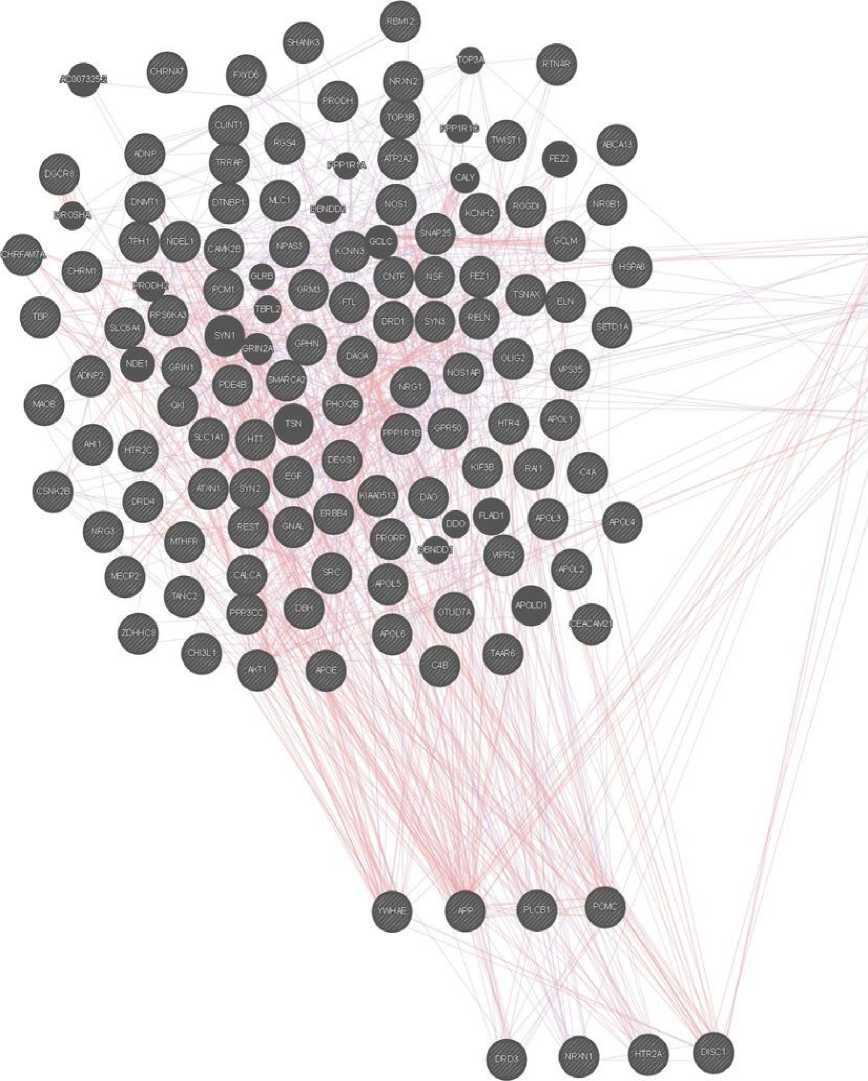
ООО®
Рис. 1. Генная сеть генов шизофрении, реконструированная с помощью GeneMANIA
Fig. 1. Schizophrenia gene network reconstructed with GeneMANIA
На рис. 2 представлена реконструкция генной сети с помощью ресурса STRING-DB. STRING-DB распознал 136 генов из списка и построил сетевые структуры, функционально связанные с известными генами риска развития шизофрении. Первоначальный вариант реконструированной с помощью ресурса STRING-DB сети оказался достаточно разреженным, некоторые объекты сети не контактировали с другими, что говорит об отсутствии взаимодействий между генами. Тем не менее в первичной реконструкции выделялся центральный плотно связанный кластер генов. Также можно увидеть более разреженную сеть второго порядка и одиночные гены. Статистика по STRING-DB показала, что сеть имеет неслучайно большое число связей (со значимостью <1,0E-16), средняя степень связности узла сети составляет 9,43, коэффициент кластеризации – 0,487. Ри- сунок из STRING-DB был перестроен с удалением несвязанных генов и с учетом анализа научных текстов, курируемых баз данных, экспериментально доказанных взаимодействий с максимальным уровнем достоверности (рис. 2).
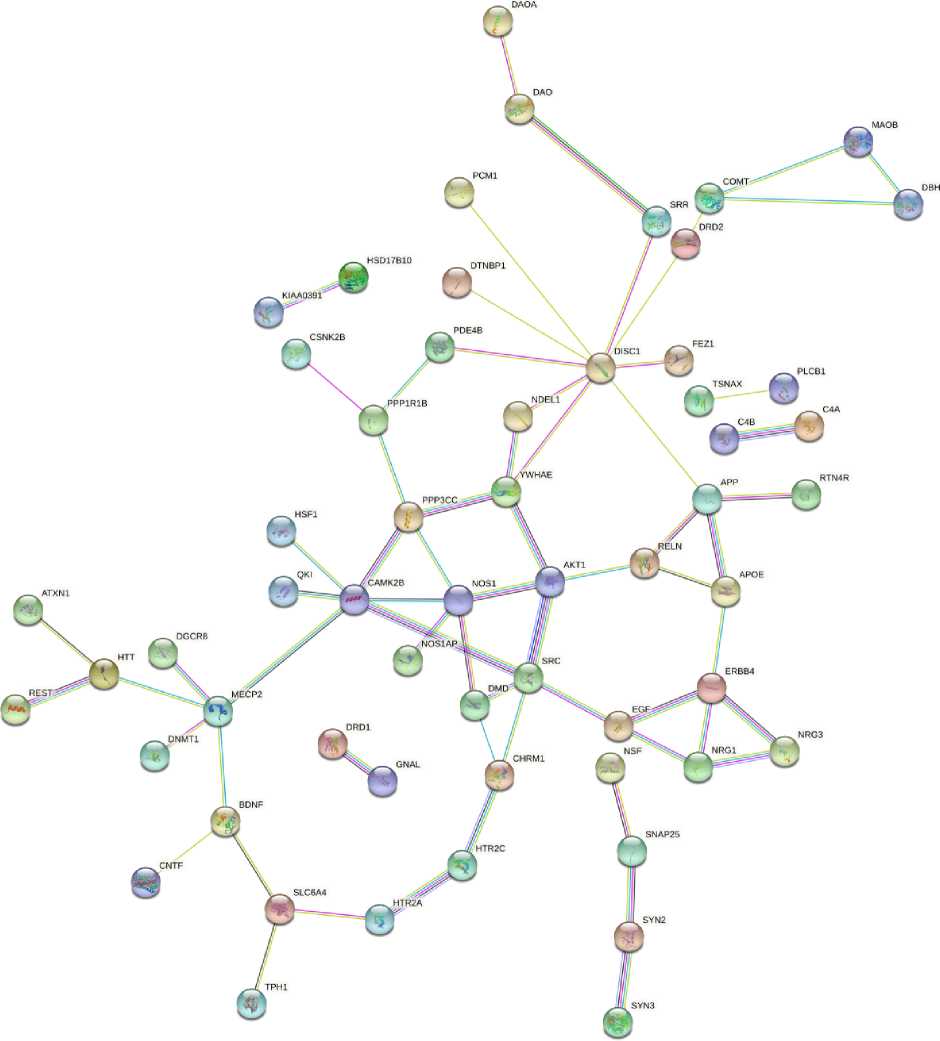
Рис. 2. Реконструкция сети взаимодействий генов шизофрении с помощью STRING-DB с учетом анализа научных текстов, курируемых баз данных, экспериментально доказанных взаимодействий. Связи соответствуют только надежно определенным взаимодействиям (оценка взаимодействия (interaction score) >0,9)
Fig. 2. Reconstruction of the schizophrenia gene interaction network using STRING-DB. Analysis of scientific texts, curated databases, and experimentally proven interactions was taken into account. Links correspond only to reliably defined interactions (interaction score >0.9)
Анализ полученной генной сети позволяет сделать вывод, что наибольшее количество связей имеет ген DISC1 . Мутации, возникающие в данном гене, связаны как с шизофренией, так и с другими психическими расстройствами, например с биполярным расстройством. Также на рис. 2 можно выделить несколько кластеров, наибольший из которых включает гены BDNF , SLC6A4 , HTR2A , HTR2C . Представленные гены участвуют в регуляции передачи синаптических сигналов и в передаче межклеточных сигналов при помощи нейромедиаторов.
Гены узлового, центрального расположения в сети ( NOS1 , APOE , DISC1 , SRC ) можно использовать как гены-мишени для терапии.
Многие гены в функциональном списке имеют молекулярные взаимодействия, обра- зуют связный кластер в сети, чего нельзя было ожидать по случайным причинам. Часть генов не имела связей в генной сети шизофрении, и они не показаны в сети (на рис. 2 число узлов в сети гораздо меньше, чем общее число в списке). Выявление молекулярных взаимодействий белковых продуктов, новых ассоциаций и является новым результатом. Таким образом, построение сети на основе максимально широкого исходного списка дает качественно новую информацию.
Для определения наиболее релевантных генов шизофрении рассмотрим структуру белков, кодируемых генами шизофрении. Поиск по ключевому слову “schizophrenia” (шизофрения) в базе данных GeneCards дал 10 118 генов. По оценке релевантности поиск вывел 10 наиболее значимых генов (табл. 5).
Таблица 5
Table 5
|
Ген Gene |
Описание Description |
Счет GIFtS* Score (GIFtS*) |
Оценка релевантности** Relevance score** |
|
COMT |
Катехол-О-метилтрансфераза Catechol-O-Methyltransferase |
50 |
47,12 |
|
DISC1 |
Нарушенный при шизофрении 1 белок Disrupted under Schizophrenia 1 Protein |
44 |
40,58 |
|
HTR2A |
5-гидрокситриптаминовый рецептор 2A 5-Hydroxytryptamine Receptor 2A |
47 |
39,86 |
|
NRXN1 |
Нейрексин 1 Neurexin 1 |
47 |
38,01 |
|
DISC2 |
Нарушенный при шизофрении 2 белок Disrupted under Schizophrenia 2 Protein |
19 |
37,70 |
|
DRD3 |
Дофаминовый рецептор D3 Dopamine Receptor D3 |
44 |
37,50 |
|
DAOA |
Активатор оксидазы D-аминокислот D-Amino Acid Oxidase Activator |
32 |
36,47 |
|
SCZD1 |
Расстройство шизофрении 1 Schizophrenia Disorder 1 |
3 |
36,26 |
|
SCZD6 |
Расстройство шизофрении 6 Schizophrenia Disorder 6 |
3 |
35,51 |
|
SCZD3 |
Расстройство шизофрении 3 Schizophrenia Disorder 3 |
3 |
35,50 |
Примечание. * – счет (оценка) функциональной аннотации гена GIFtS (GeneCards Inferred Functionality Scores) по представленности в базах данных, следуя [15]; ** – счет релевантности оценивает соответствие поисковому запросу в базе данных для шизофрении.
Наиболее значимые гены шизофрении по данным GeneCards
Most significant schizophrenia genes according to GeneCards
Note . * – GeneCards Inferred Functionality Scores (GIFtS) is taken from the databases according to [15];
** – relevance score evaluates relevance to a search query in the database for schizophrenia.
Согласно табл. 5 наибольшей оценкой релевантности для шизофрении обладают гены COMT, DISCI, HTR2A, NRXN1, следовательно, они являются ключевыми.
COMT катализирует перенос метильной группы с S-аденозилметионина на катехоламины, включая нейротрансмиттеры допамин, эпинефрин и норэпинефрин. Это O-метилирование приводит к одному из основных путей деградации катехоламиновых трансмиттеров. COMT содержится в тканях в двух формах -растворимой (S-COMT) и мембраносвязанной (MB-COMT). Различия между S-COMT и MB-COMT заключаются в N-концевых участках. Несколько вариантов образуются благодаря использованию альтернативных сайтов инициации трансляции и промоторов (предоставлено RefSeq, сентябрь 2008 г.).
DISC1 участвует в регуляции многочисленных аспектов эмбрионального и взрослого нейрогенеза [20, 21]. Он необходим для пролиферации нейронных предшественников в вентрикулярной/субвентрикулярной зоне во время эмбрионального развития мозга и в зубчатой извилине гиппокампа у взрослых. Участвует в Wnt-опосредованной пролиферации нейронных предшественников в качестве положительного регулятора, модулируя активность GSK3B и количество CTNNB1 [20]. Играет роль модулятора сигнального пути AKT-mTOR, контролирующего темп процесса интеграции новорожденных нейронов в ходе взрослого нейрогенеза, включая позиционирование нейронов, развитие дендритов и формирование синапсов. Ингибирует активацию AKT-mTOR-сигнального пути при взаимодействии с CCDC88A. Регулирует миграцию рано родившихся предшественников гранулезных клеток в зубчатую извилину во время развития гиппокампа [21]. Ингибирует активность транскрипционного фактора ATF4 в нейронах, нарушая димеризацию ATF4 и связывание с ДНК.
HTR2A кодирует один из рецепторов серотонина. Мутации в этом гене связаны с предрасположенностью к шизофрении и обсессивно-компульсивному расстройству, а также с ответом на антидепрессант циталопрам у пациентов с большим депрессивным расстройством (БДР). У пациентов с БДР, имеющих мутацию в интроне 2 этого гена, значительно снижен ответ на циталопрам, поскольку этот антидепрессант снижает экспрессию гена. Для этого гена было обнаружено несколько вариантов транскриптов, кодирующих различные изоформы (предоставлено RefSeq, сентябрь 2009 г.).
NRXN1 кодирует однопроходный мембранный белок типа I, принадлежащий к семейству нейрексинов. Нейрексины - это рецепторы клеточной поверхности, которые связывают нейролигины, образуя Ca(2+)-за-висимые комплексы «нейрексин/нейролигин» в синапсах центральной нервной системы. Этот комплекс необходим для эффективной нейротрансмиссии и участвует в формировании синаптических контактов. Три члена этого семейства генов были подробно изучены и, по оценкам, генерируют более 3000 видов за счет использования двух альтернативных промоторов (альфа и бета) и обширного альтернативного сплайсинга в каждом члене семейства. Недавно для этого гена был идентифицирован третий промотор (гамма) в 3'-области. Мутации в гене связаны с синдромом Питта -Хопкинса-2 и могут способствовать предрасположенности к шизофрении (предоставлено RefSeq, август 2016 г.).
Обсуждение. Таким образом, шизофрения - это генетически сложная болезнь, этиология которой представляет огромный набор возможных взаимодействий генетических рисков и окружающих факторов. На данном этапе развития медицины это заболевание является неизлечимым, однако длительное и последовательное проведение комплексной терапии позволяет достичь стойкой продолжительной ремиссии. Существующие методы терапии носят скорее заместительный характер (позволяют продлить время жизни пациента и улучшить ее качество) и не направлены на устранение непосредственно причины заболевания, что свидетельствует о необходимости проводить дальнейшие исследования генетических основ болезни. В настоящее время происходит активное накопление генетических данных в области изучения шизофрении. Анализ литературы (PubMed) показал продолжающийся рост публикаций по данной теме, всего с 1921 г. по настоящее время (2022 г.)
опубликовано 154 159 исследований, что свидетельствует о сохраняющемся интересе к проблемам, связанным с заболеванием.
Составление списка ключевых генов, ассоциированных с развитием шизофрении, анализ категории генных онтологий для этого списка и реконструкция генных сетей позволяют лучше понять генез болезни, выбрать гены-мишени для лекарственных воздействий, модифицирующих степень выраженности, а также предложить новые варианты лечения. Применение полного спектра экспериментальных и компьютерных методов способно уже в ближайшем будущем приблизить создание лекарственных средств, облегчающих состояние больных шизофренией или даже, возможно, полностью предотвращающих появление этой болезни.
Заключение. Установлено, что шизофрения связана с множеством генных онтологий, таких как биологические процессы (модуляция химической синаптической передачи, регуляция транссинаптической сигнализации, регуляция уровня нейротрансмиттеров), молекулярные функции (активность регулятора ионных каналов, связывание кальмодулина, активность рецепторов нейротрансмиттеров, активность трансмембранных сигнальных рецепторов), клеточные компартменты (синапс, клеточный стык, проекция нейронов, сомато-дендритный компартмент).
Построены и проанализированы генные сети, отобраны входящие в кластеры гены, которые можно использовать как гены-мишени: NOS1 , APOE , DISC1 , SRC .
Определены гены-мишени для шизофрении с наибольшей релевантностью: COMT (47,12), DISC1 (40,58), HTR2A (39,86), NRXN1 (38,01).
Список литературы Реконструкция генной сети шизофрении для поиска генов-мишеней
- Schizophrenia: Concise Medical Dictionary. Oxford University Press. 2010. URL: https://www.oxford-reference.com/view/10.1093/acref/9780199557141.001.0001/acref-9780199557141-e-9060 (дата обращения: 12.02.2022). DOI: 10.1093/acref/9780199557141.001.0001.
- Institute of health Metrics and Evaluation (IHME). Global Health Data Exchange (GHDx). URL: http://ghdx.healthdata.org/gbd-results-tool?params=gbd-api-2019-permalink/27a7644e8ad28e739382d31e77589dd7 (дата обращения: 12.02.2022).
- PicchioniM.M., MurrayR.M. Schizophrenia. BMJ. 2007; 335 (7610): 91-95. URL: https://www.bmj.com/ content/335/7610/91 (дата обращения: 12.02.2022). DOI: 10.1136/bmj.39227.616447.BE.
- Castle D., Wesseley S., Der G., Murray R.M. The incidence of operationally defined schizophrenia in Camberwell 1965-84. British Journal of Psychiatry: journal Royal College of Psychiatrists. 1991; 159: 790-794. URL: https://www.cambridge.org/core/journals/the-british-journal-of-psychiatry/article/abs/ incidence-of-operationally-defined-schizophrenia-in-camberwell-196584/6FD5AA8394856650FE3004 A71B49A564 (дата обращения: 12.02.2022). DOI: 10.1192/bjp.159.6.790.
- GonthierM., Lyon M.A. Childhood-onset schizophrenia: An overview. Psychology in the Schools. 2004; 41 (7): 803-811. URL: https://onlinelibrary.wiley.com/doi/10.1002/pits.20013 (дата обращения: 13.02.2022). DOI: 10.1002/pits.20013.
- Mattai A.K., Hill J.L., LenrootR.K. Treatment of early-onset schizophrenia. Curr Opin Psychiatry. 2010; 23 (4): 304-310. URL: https://pubmed.ncbi.nlm.nih.gov/20502331 (дата обращения: 13.02.2022). DOI: 10.1097/YTO.0b013e32833b027e.
- Howard R., Rabins P.V., Seeman M.V., Jeste D.V. Late-onset schizophrenia and very-late-onset schizophrenia-like psychosis: an international consensus. The International Late-Onset Schizophrenia Group. Am. J. Psychiatry. 2000; 157 (2): 172-178. URL: https://pubmed.ncbi.nlm.nih.gov/10671383 (дата обращения: 13.02.2022). DOI: 10.1176/appi.ajp.157.2.172.
- Owen M.J., Sawa A., Mortensen P.B. Schizophrenia. Lancet. 2016; 388 (10039): 86-97. URL: https://pubmed.ncbi.nlm.nih.gov/26777917/ (дата обращения: 14.02.2022). DOI: 10.1016/S0140-6736(15)01121-6.
- Mullin A.P., Gokhale A., Moreno-De-Luca A., Sanyal S., Waddington J.L., Faundez V. Neurodevelop-mental disorders: mechanisms and boundary definitions from genomes, interactomes and proteomes. Transl. Psychiatry. 2013; 3 (12): e329. URL: https://www.nature.com/articles/tp2013108 (дата обращения: 14.02.2022). DOI: 10.1038/tp.2013.108.
- Davis J., Eyre H., Jacka F.N., Dodd S., Dean O., McEwen S., Debnath M., McGrath J., Maes M., Amminger P., McGorry P.D., Pantelis C., Berk M. A review of vulnerability and risks for schizophrenia: Beyond the two hit hypothesis. Neurosci Biobehav Rev. 2016; 65: 185-194. URL: https://pub-med.ncbi.nlm.nih.gov/27073049 (дата обращения: 14.02.2022). DOI: 10.1016/j.neubiorev.2016.03.017.
- Van de Leemput J., Hess J.L., Glatt S.J., TsuangM.T. Genetics of Schizophrenia: Historical Insights and Prevailing Evidence. Adv Genet. 2016; 96: 99-141. URL: https://pubmed.ncbi.nlm.nih.gov/27968732 (дата обращения: 16.02.2022). DOI: 10.1016/bs.adgen.2016.08.001.
- Torrey E.F., Yolken R.H. Schizophrenia as a pseudogenetic disease: A call for more gene-environmental studies. Psychiatry Res. 2019; 278: 146-150. URL: https://pubmed.ncbi.nlm.nih.gov/31200193 (дата обращения: 16.02.2022). DOI: 10.1016/j.psychres.2019.06.006.
- Combs D.R., Mueser K.T., Gutierrez M.M. Chapter 8: Schizophrenia: Etiological considerations. In: Hersen M., Beidel D.C. Adult psychopathology and diagnosis. 6th ed. John Wiley & Sons; 2011.
- HassettA., Ames D., Chiu E. Psychosis in the Elderly. London: Taylor and Francis; 2005.
- Sullivan P.F., Kendler K.S., NealeM.C. Schizophrenia as a complex trait: evidence from a meta-analysis of twin studies. Archives of General Psychiatry. 2003; 60: 1187-1192. URL: https://jamanetwork.com/jour-nals/jamapsychiatry/fullarticle/208134 (дата обращения: 17.02.2022). DOI: 10.1001/archpsyc.60.12.1187.
- Thibaut F. Schizophrenia: An example of complex genetic disease. World Journal of Biological Psychiatry. 2006; 7: 194-197. URL: https://www.tandfonline.com/doi/full/10.1080/15622970600994313 (дата обращения: 17.02.2022). DOI:10.1080/15622970600994313.
- Cross-Disorder Group of the Psychiatric Genomics Consortium. Identification of risk loci with shared effects on five major psychiatric disorders: a genome-wide analysis. Lancet. 2013; 381 (9875): 13711379. URL: https://pubmed.ncbi.nlm.nih.gov/23453885 (дата обращения: 17.02.2022). DOI: 10.1016/S0140-6736(12)62129-1.
- Charlson F.J., Ferrari A.J., Santomauro D.F., Diminic S., Stockings E., Scott J.G., McGrath J.J., White-ford H.A. Global Epidemiology and Burden of Schizophrenia: Findings From the Global Burden of Disease Study 2016. Schizophr Bull. 2018; 44 (6): 1195-1203. URL: https://www.ncbi.nlm.nih.gov/ pmc/arti-cles/PMC6192504 (дата обращения: 10.03.2022). DOI: 10.1093/schbul/sby058.
- Howes O.D., Kapur S. The dopamine hypothesis of schizophrenia: version III - the final common pathway. Schizophr Bull. 2009; 35 (3): 549-562. URL: https://pubmed.ncbi.nlm.nih.gov/19325164 (дата обращения: 10.03.2022). DOI: 10.1093/schbul/sbp006.
- Mao Y., Ge X., Frank C.L., Madison J.M., Koehler A.N., Doud M.K., Tassa C., Berry E.M., Soda T., Singh K.K., Biechele T., Petryshen T.L., Moon R.T., Haggarty S.J., Tsai L.H. Disrupted in schizophrenia 1 regulates neuronal progenitor proliferation via modulation of GSK3beta/beta-catenin signaling. Cell. 2009; 136 (6): 1017-1031. URL: https://pubmed.ncbi.nlm.nih.gov/19303846 (дата обращения: 11.03.2022). DOI: 10.1016/j.cell.2008.12.044.
- Meyer K.D., Morris J.A. Disc1 regulates granule cell migration in the developing hippocampus. Hum Mol Genet. 2009; 18 (17): 3286-3297. URL: https://academic.oup.com/hmg/article/18/17/3286/2527378 (дата обращения: 14.03.2022). DOI:10.1093/hmg/ddp266.


