Сцепленное наследование микросателлитного локуса ORS 822 с геном PLARG, контролирующим устойчивость к ложной мучнистой росе, у подсолнечника
Автор: Рамазанова С.А., Бадьянов Е.В., Иванов С.В., Савиченко В.Г., Гучетль С.З.
Рубрика: Селекция, семеноводство и биотехнология сельскохозяйственных растений
Статья в выпуске: 4 (192), 2022 года.
Бесплатный доступ
Устойчивость к возбудителю ложной мучнистой росы Plasmopara halstedii (Farl.) Berlese et de Toni у подсолнечника контролируется генами устойчивости Pl. Ген Plarg в настоящее является перспективным в селекции на устойчивость, так как эффективен против всех известных рас патогена. Этот ген интрогрессирован из дикорастущего вида Helianthus argophyllus. Молекулярные маркеры и, в частности, простые микросателлитные повторы (SSR) позволяют контролировать перенос и пирамидирование генов устойчивости к болезням. Однако необходима валидация молекулярного маркера для доказательства его надежности. Для идентификации гена Plarg был апробирован микросателлитный маркер ORS 822. Исследование проводили на гибридной комбинации восприимчивой линии подсолнечника селекции ВНИИМК ВК 925 и устойчивой линии RHA 419, являющейся донором гена Plarg. Установлено, что эти линии отличаются друг от друга аллельным состоянием этого локуса. Молекулярный анализ поколения F1 показал, что микросателлитный локус наследуется кодоминантно. Путем самоопыления было получено поколение F2 и проведена фитопатологическая оценка устойчивости к P. halstedii. Анализ расщепления по фено-20 типу показал, что фактически наблюдаемое расщепление соответствовало теоретически ожидаемой модели 3 : 1 при моногенном наследовании признака. На основании полученных данных было определено, что ген Plarg и микросателлитный локус ORS 822 сцеплены с частотой рекомбинации 0,26. В результате проведенного исследования сделан вывод, что для применения в маркер-ассоциированной селекции (МАС) подсолнечника на устойчивость к возбудителю ложной мучнистой росы этот маркер можно использовать для отбора гомозиготных устойчивых растений.
Подсолнечник, plasmopara halstedii, гены устойчивости, микросателлитные маркеры, мас, локус
Короткий адрес: https://sciup.org/142236129
IDR: 142236129 | УДК: 631.523:633.854.78 | DOI: 10.25230/2412-608X-2022-4-192-20-28
Текст научной статьи Сцепленное наследование микросателлитного локуса ORS 822 с геном PLARG, контролирующим устойчивость к ложной мучнистой росе, у подсолнечника
Acknowledgements. The work was conducted with the financial support of the Kuban Science Foundation as part of the scientific project № N-21.1/6.
The authors express gratitude to Iwebor Maria Vyacheslavovna, leading researcher of the immunity laboratory of V.S. Pustovoit All-Russian Research Institute of Oil Crops, for her assistance in phytopathologi-cal evaluation for resistance to the downy mildew pathogen.
Введение. Ложная мучнистая роса (Plasmopara halstedii (Farl.) Berlese et de Toni) является одной из наиболее опасных болезней культурного подсолнечника (Helianthus annuus), так как вызывает серьезные экономические потери [1; 2]. При благоприятных для развития патогена погодных условиях потери урожая могут доходить до 70 %. Многочисленные исследования показывают, что поражение патогеном нарушает процессы жизнедеятельности растений (фотосинтез, дыхание, транспирацию, обмен веществ), это приводит к снижению продуктивности, ухудшению товарных и посевных качеств семян [3; 4].
Первые генетические исследования устойчивости к ложной мучнистой росе показали, что контролируется она доминантными генами, обозначенными Pl. На сегодняшний день у подсолнечника идентифицировано 36 основных генов Pl и три QTL [5; 6].
Один или несколько основных генов Pl 1 , Pl 2 , Pl 5 , Pl 6 , Pl 7 и Pl 8 обеспечивали устойчивость ко всем расам P. halstedii и до начала 2000-х годов широко использовались в селекции на устойчивость. Причем часть этих генов Pl 6 , Pl 7 и Pl 8 интрогресси-рована в культурный подсолнечник из дикорастущих видов Helianthus и контролирует устойчивость почти ко всем расам P. halstedii [7; 8]. Например, ген Pl 6 был получен из дикорастущегого H. annuus , тогда как Pl 7 произошел из H. praecox, а Pl 8 – от H. argophyllus [9]. Дальнейшие исследования показали, что локус Pl 6 представляет собой по крайней мере две генетически отличные области, каждая из которых придает устойчивость различным расам ложной мучнистой росы [10]. Кроме того, M.F. Bouzidi с соавторами предположили, что в локусе Pl 6 находится 11 функциональных генов Pl , поскольку линии подсолнечника, которые содержали этот локус, были устойчивы к 11 расам мучнистой росы [8].
На сегодняшний день перспективным в селекции подсолнечника на устойчивость к ложной мучнистой росе является использование гена Plarg, так как он обеспечивает устойчивость ко всем известным расам па- тогена. В 1989 г. американскими исследователями этот ген был интрогрессирован в культурный подсолнечник из дикорастущего вида H. argophyllus [11]. G.J. Seiler путем скрещивания линии культурного подсолнечника HA 89 и H. argophyilus получил линию ARG-1575-2, несущую ген Plarg [11]. Далее были получены две инбредные линии RHA 419 и RHA 420, которые использовались в селекции подсолнечника на устойчивость к ложной мучнистой росе [14; 15]. Хотя гены Plarg и Pl8 и были интро-грессированы от H. argophyilus, Pl8 был получен из H. argophyllus 415 [11], тогда как Plаrg происходит от H. argophyllus 1575 [15]. Ген Plarg был картирован в группе сцепления LG1, а Pl8 – в LG13 [9; 17]. Классический генетический анализ, основанный на фенотипировании сегрегирующих популяций, показал, что ген Plarg не сцеплен с генами Pl13, Pl14 и Pl16, находящимися в этой же группе сцепления. Однако изучение молекулярной структуры области ДНК, содержащей ген Plarg, не исключает того, что он является кластером тесно сцепленных генов устойчивости [9; 17; 18]. Тем не менее он является основным локусом Pl и по-прежнему эффективен против всех известных рас P. halstedii, включая и расы, преодолевшие действие металаксила [19]. Введение этого гена в селекционный материал позволит получить устойчивые линии и гибриды подсолнечника. Молекулярное маркирование данного гена значительно ускорит их создание, так как позволит проводить раннюю и точную диагностику носителей гена Plarg в гибридной популяции. В литературе опубликован ряд молекулярных маркеров разных типов, сцепленных с локусом Plarg [16; 20; 21; 22], но перед практическим использованием маркера в селекции всегда необходима валидация молекулярных маркеров на своем наборе генотипов.
Данная работа является продолжением комплекса наших работ по оценке диагно-22
стической ценности молекулярных маркеров для идентификации гена Pl arg [23]. В предыдущих исследованиях нами была изучена диагностическая ценность маркера ORS 822 на гибридной комбинации RHA 419 × ВК 776. Было установлено сцепление пары локусов Pl arg – ORS 822 с частотой рекомбинации 0,31 ± 0,05 [28]. Однако часто при разработке молекулярного маркера используется ограниченное число генотипов, а степень сцепления с маркируемым геном определяется, как правило, на основе анализа одной конкретной расщепляющейся популяции. Поэтому перед практическим использованием маркера в селекции необходимо не только апробировать маркер на широком наборе генотипов, но и провести его валидацию на других гибридных комбинациях. В связи с этим целью данного исследования являлся анализ сцепления пары генов Pl arg – ORS 822 у гибридной комбинации RHA 419 × ВК 925 и определение возможности применения микросателлитного локуса в маркер-ассоциированной селекции на устойчивость к возбудителю ложной мучнистой росы
Материалы и методы. Материалом для исследования являлись растения F 1 и F 2 гибридной комбинации линий подсолнечника RHA 419 × ВК 925.
Скрещивания проводили в условиях теплицы по стандартной методике [24]. Гибриды F 1 выращивали в поле. Затем было проведено самоопыление растений F 1 и получено потомство F 2 . Растения поколения F 2 тестировали в лабораторных условиях методом искусственного заражения на устойчивость и восприимчивость к ложной мучнистой росе. Пораженными считали все растения со спороношением как на семядолях, так и на настоящих листьях [25].
Для выделения ДНК использовали фрагменты зеленых листьев подсолнечника. Экстракцию ДНК проводили с использованием набора для выделения Diamond
DNA Plant kit (РФ). Концентрацию ДНК в полученных препаратах определяли на микроспектрофотометре Nano-300 (Allsheng, Китай). Для ПЦР-анализа применили пару праймеров, разработанных для маркирования локуса Pl arg ORS 822 (F: CAATGCCATC TGTCATCAGCTAC, R: AAACAAACCTTTGGACGA AACTC) [26].
Полимеразную цепную реакцию выполняли в реакционной смеси объемом 25 μл следующего состава: 67 mM трис-HCl, рН 8,8; 16,6 mM сульфата аммония; 1,5– 3,0 mM MgCl 2 ; 0,01 % Tween 20; 200 μM dNTP; по 0,5 μM каждого праймера; 10 нг геномной ДНК и 1 ед. рекомбинантной термостабильной ДНК полимеразы (НПО «Сибэнзим», РФ). Амплификацию ДНК проводили в термоциклере MiniAmp Plus (Thermo Fisher Scientific, США). Температурно-временные режимы амплификации были описаны ранее [23].
Электрофорез продуктов амплификации проводили в 2%-ном агарозном и 8%-ном полиакриламидном гелях. Использовали камеры SE-20 и VE-20 для горизонтального и вертикального электрофореза соответственно (Хеликон, Россия). Результаты электрофореза фиксировали при помощи гель-документирующей видеосистемы BIO-PRINT (Vilber Lourmat, Франция). Размер фрагментов ДНК определяли с использованием программного обеспечения BioCapture (Vilber Lourmat, Франция) относительно маркера длины фрагментов ДНК GeneRuler 100 bp DNA Ladder Thermo Scientific (Сибэнзим, Россия).
Математическую обработку результатов расщепления проводили с использованием χ2-критерия соответствия фактических расщеплений теоретически ожидаемым в моно- и дигибридных скрещиваниях. Расчет частоты рекомбинации r и ошибку рекомбинации s p по результатам расщепления в F 2 рассчитывали по методу максимального правдоподобия [27].
Результаты и обсуждение. В данном исследовании оценку диагностической ценности микросателлитного маркера ORS 822 проводили на устойчивой ко всем расам P. halstedii линии подсолнечника RHA 419 и восприимчивой линии ВК 925. Было установлено, что линии отличаются по микросателлитным профилям локуса ORS 822 друг от друга (рис. 1).
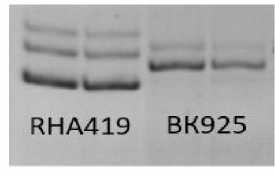
Рисунок 1 – Электрофореграмма продуктов амплификации ДНК линий подсолнечника RHA 419 и ВК 925 с праймером ORS 822 (ориг.)
Анализ наследования микросателлитно-го локуса ORS 822 в поколении F 1 показал, что он наследуется кодоминантно. У полученных растений поколения F 1 по изучаемому локусу были выявлены фрагменты обеих родительских линий (рис. 2).
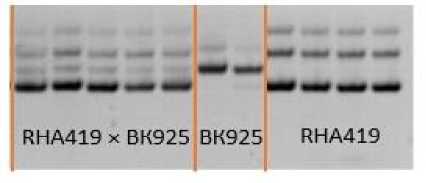
Рисунок 2 – Электрофореграмма продуктов амплификации ДНК линий подсолнечника RHA 419, ВК 925 и растений поколения F 1 гибридной комбинации RHA 419 × ВК 925 с праймером ORS 822 (ориг.)
На основании полученных результатов из поколения F 1 было отобрано пять растений для получения поколения F 2 . Семена одной из полученных семей F 2 прорастили 23
в лабораторных условия и провели фитопатологическую оценку устойчивости к ложной мучнистой росе. Из 102 проанализированных растений пораженных было 18. Анализ расщепления, выполненный по полученным результатам, показал, что фактически наблюдаемое расщепление соответствовало теоретической модели 3 : 1 для моногенного наследования. Критерий χ2, характеризующий соответствие фактических расщеплений теоретически ожидаемым, подтвердил доминантное моногенное наследование устойчивости в данной комбинации (табл. 1).
Таблица 1
Наследование признака устойчивости к ложной мучнистой росе в поколении F 2 при скрещивании линий подсолнечника
RHA 419 и ВК 925
|
Комбинация скрещивания |
Теоретически ожидаемое соотношение |
Практическое соотношение |
χ2 |
df |
P |
|
RHA 419 × ВК 925 |
3 : 1 |
84 : 18 |
3,794 |
1 |
>0,05 |
Эти результаты согласуются с данными S. Wieckhorst с соавторами [17] и С.А. Рамазановой с соавторами [28], которые определили, что локус Pl arg контролируется одним геном и наследуется доминантно.
Результаты проведенного гибридологического анализа поколения F 2 по локусу ORS 822 представлены в таблице 2.
Таблица 2
Наследование SSR-локуса ORS 822 в потомстве F 2 при скрещивании линий RHA 419 и ВК 925
|
Локус |
Всего растений, шт. |
Теоретически ожидаемое соотношение |
Практическое соотношение |
χ2 |
df |
P |
|
ORS 822 |
86 |
1 : 2 : 1 |
28 : 33 : 25 |
4,86 |
2 |
> 0,05 |
В проведенных нами ранее исследованиях данной комбинации скрещивания по локусам ORS 509 и ORS 662 критерий χ2 24
составил 4,6 и 3,9 соответственно [28]. Вероятно, это связано с искажением расщепления всей геномной области при скрещивании культурных линий с линиями, происходящими из дикорастущих видов подсолнечника.
Следующим этапом работы было проведение теста на совместное наследование гена Pl arg с изучаемым SSR-локусом. На рисунке 3 показаны электрофореграммы продуктов амплификации ДНК образцов подсолнечника поколения F 2. Все изученные образцы распределились в пять классов. Это устойчивые растения, имеющие фракцию размером приблизительно 134 п.н., такую же, как у линии RHA 419 (рис. 3, дорожки 4, 6, 9, 10, 14, и 17), восприимчивые растения с фракцией 162 п.н., как у линии ВК 925 (рис. 3, дорожки 7, 20, 21, 25 и 27). Растения, имеющие фракции обоих родителей, были как среди устойчивых, так и восприимчивых (рис. 3, дорожки 3, 5, 8, 11–13, 15, 16, 18, 19, 22–24, и 26) и устойчивые растения с фрагментом 162 п.н. Пораженных растений с генотипом, как у линии RHA 419 не было обнаружено. Полученные результаты показывают, что этот молекулярный маркер можно использовать для отбора гомозиготных устойчивых растений из расщепляющейся популяции.
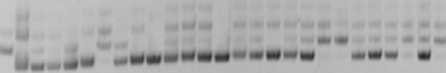
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 В 26 21 22 23 24 25 26 27
Рисунок 3 – Электрофореграмма продуктов амплификации ДНК образцов подсолнечника поколения F 2 (RHA 419 × ВК 925) в полиакриламидном геле по локусу ORS 822, дорожки: 1 – ВК 925;
2 – RHA 419; 3 –27 – образцы подсолнечника поколения F 2 (ориг.)
В таблице 3 представлены данные по оценке совместного наследования гена Plarg с локусом ORS 822. Тест на совместное наследование показал их сцепление.
Таблица 3
Значения χ2 между геном Pl arg и SSR-локусом ORS 822 в потомстве F 2 (RHA 419 × ВК 925)
|
Локус |
Всего растений, шт. |
Фактически наблюдаемое соотношение, шт. |
Теоретически ожидаемое соотношение |
χ2 |
df |
P |
|
Pl arg – ORS 822 |
86 |
28 : 30 : 16 :0: 3 : 9 |
3 : 6 : 3 : 1 : 2 : 1 |
22,31 |
5 |
< 0,01 |
Оценка частоты рекомбинации между этой парой локусов, составившая 0,26 ±
0,05, показана в таблице 4
Таблица 4
Значения частоты рекомбинации r между геном Pl arg и локусом ORS 822 в потомстве F 2
(RHA 419 x ВК 925)
|
Локус |
r |
s p |
|
Pl arg – ORS 822 |
0,26 |
0,05 |
В предыдущих исследованиях нами было установлено сцепление пары локусов Pl arg – ORS 822 у гибридной комбинации RHA 419 × ВК 776 с частотой рекомбинации 0,31 ± 0,05 [28]. На оценку расстояния между локусами могли оказать влияние ошибки фенотипирования, поскольку, как отмечают некоторые авторы, идентификация восприимчивого фенотипа как устойчивого при классификации приводит к увеличению класса рекомбинантных генотипов и искажает дистанции на генетической карте [9; 22]. А также на оценку расстояния между генами могло повлиять искажение расщепления в поколении F 2 у SSR-локуса ORS 822. Такие данные показывают, что диагностическую ценность молекулярных маркеров необходимо проводить на большем количестве гибридных комбинаций.
Заключение. Таким образом, апробация молекулярного маркера ORS 822 гена Plarg с использованием гибридной комбинации линий подсолнечника RHA 419 × ВК 925 показала, что маркерные аллели в реципи-ентной линии ВК 925 отличались от аллелей линии RHA 419. Изученный микросателлитный локус наследуется кодоминантно. Ген устойчивости и SSR-локус наследуются сцеплено с частотой рекомбинации 0,26 ± 0,05. Для практического использования в маркерной селекции локус ORS 822 можно применять для отбора гомозиготных растений из расщепляющейся гибридной популяции.
Список литературы Сцепленное наследование микросателлитного локуса ORS 822 с геном PLARG, контролирующим устойчивость к ложной мучнистой росе, у подсолнечника
- Markell S.G., Humann R.H., Gilley M., Gulya T.J. [et al.]. Downy mildew pathogen // Compendium of sunflower diseases and pests. - American Phytopathology Press, 2016. - P. 115-117.
- Molinero-Ruiz M.L., Melero-Vara J.M., Dominguez J. Inheritance of resistance to two races of sunflower downy mildew (Plasmopara halstedii) in two Helianthus annuus L. lines // Eu-phytica. - 2003. - Vol. 131. - P. 47-51. DOI: 10.1023/A: 1023063726185.
- Пересыпкин В.Ф. Болезни сельскохозяйственных культур. Том 2.: Болезни технических культур и картофеля. - Киев: Урожай, 1990. -248 с.
- Голощапова Н.Н., Гончаров С.В. Селекция подсолнечника на долговременную устойчивость к ложной мучнистой росе // Современное экологическое состояние природной среды и научно-практические аспекты рационального природопользования. II Международная научно-практическая Интернет-конференция. -ФГБНУ «Прикаспийский научно-исследовательский институт аридного земледелия», 2017. -С. 1383-1386.
- Pecrix Y., Penouilh-Suzette C., Munos S., Vear F., Godiard L. Ten broad spectrum resistances to downy mildew physically mapped on the sunflower genome // Front. Plant Sci. - 2018. -Vol. 9. - P. 1780. DOI: 10.3389/fpls.2018.01780.
- Vincourt P., Assadi F., Bordat A., Langlade N.B., Gouzy J., Pouilly N., Vear F. Consensus mapping of major resistance genes and independent QTL for quantitative resistance to sunflower downy mildew // Theoretical and Applied Genetics. - 2012. - No 125 (5). - P. 909-920. DOI: 10.1007/s00122-012-1882-y.
- Bert P.F., Tourvieille de Labrouhe D., Philippon J., Mouzeyar S., Jouan I., Nicolas P., Vear F. Identification of a second linkage group carrying genes controlling resistance to downy mildew (Plasmopara halstedii) in sunflower (Helianthus annuus L.) // Theor. Appl. Genet. -2001. - No 103. - P. 992-997.
- Bouzidi M.F., Badaoui S., Cambon F., Vear F., Tourvieille de Labrouhe D., Nicolas P., Mouzeyar S. Molecular analysis of a major locus for resistance to downy mildew in sunflower with specific PCR-based markers // Theor. Appl. Genet. - 2002. - No 104 - P. 592-600.
- Dufile C.M., Hahn V., Knapp S.J., Bauer E. Plarg from Helianthus argophyllus is unlinked to other known downy mildew resistance genes in sunflower // Theor. Appl. Genet. - 2004. - Vol. 109. - P. 1083-1086. DOI: 10.1007/s00122-004-1722-9.
- Vear F., Gentzbittel L., Philippon J., Mouzeyar S., Mestries E., Roeckel-Drevet P., Tourvieille de Labrouhe D., Nicolas P. The genetics of resistance to five races of downy mildew (Plasmopara halstedii) in sunflower (Helianthus annuus L.) // Theor. Appl. Genet. -1997. - No 95. - P. 584-589.
- Seiler G.J. Registration of 13 downy mildew tolerant interspecific sunflower germplasm lines derived from wild annual species // Crop Science. - 1991. - No 31 (6). - P. 1714-1716. DOI: 10.2135/cropsci1991.0011183x003100060 093x.
- Miller J.F., Gulya T.J., Seiler G.J. Registration of five fertility restorer sunflower germplasms // Crop Science. - 2002. - No 42 (3). -P. 983-989. DOI: 10.2135/cropsci2002.9890.
- Hulke B.S., Miller J.F., Gulya T.J, Vick B.A. Registration of the oilseed sunflower genetic stocks HA 458, HA 459, and HA 460 possessing genes for resistance to downy mildew // Journal of Plant Registrations. - 2010. - No 4. -P. 93-97. F.
- Viranyi F., Gulya T.J., Tourvieille de Labrouhe D. Recent changes in the pathogenic variability of Plasmopara halstedii (sunflower downy mildew) populations from different continents // Helia. - 2015. - Vol. 38. - P. 149-162. DOI: 10.1515/helia-2015-0009.
- Miller J.F., Gulya T.J. Inheritance of resistance to race 4 of downy mildew derived from interspecific crosses in sunflower // Crop Sci. -1991. - No 31. - P. 40-43.
- Tang S., Yu J.-K., Slabaugh M.B., Shintani D.K., Knapp S.J. Simple sequence repeat map of the sunflower genome // Theor. Appl. Genet. - 2002. - No 105. - Р. 1124-1136.
- Wieckhorst S., Bachlava E., Duble C., Tang S., Gao W., Saski C., Knapp S., Schön C.C., Hahn V., Bauer E. Fine mapping of the sunflower resistance locus PIarg introduced from the wild species Helianthus argophyllus // Theor. Appl. Genet. -2010. - Vol. 121. - P. 1633-1644. DOI: 10.1007/s00122-010-1416-4.
- Livaja M., Wang Y., Wieckhorst S., Haseneyer G., Seidel M., Hahn V., Bauer E. BSTA: a targeted approach combines bulked seg-regant analysis with next-generation sequencing and de novo transcriptome assembly for SNP discovery in sunflower // BMC Genomics. - 2013. - No 14. - Р. 628. DOI: 10.1186/1471-2164-14-628.
- Brahm L., Hahn V., Röcher T., Friedt W. Molecular markers as a tool in breeding for resistance against sunflower downy mildew // In: Proceedings of the 15th International Sunflower Conference, Toulouse, France, June 12-15, 2000. - Vol. 3. - Р. J43- J48.
- Imerovski I., Dimitrijevic D., Miladinovic A., Jocic S., Dedic B. [et al.]. Identification and validation of breeder-friendly DNA markers for Plarg gene in sunflower // Molecular Breeding. -2014. - Vol. 34 (3). - P. 779-788. DOI: 10.1007/s11032-014-0074-7.
- Solodenko A. Validation of microsatellite markers of Pl resistance genes to downy mildew of sunflower // Helia. - 2018. - Vol. 41 (68). - P. 7382. DOI: 10.1515/helia-2017-0026.
- Qi L.L., Ma G., Talukder Z.I., Seiler G.J., Hulke B.S., Jan C.C. [et al.]. Molecular mapping of the disease resistance gene and its impact on sunflower breeding // In: Proceedings of the 19th Intetnational Sunflower Conference, Edirne, Turkey. - 2016(b). - P. 20-30.
- Рамазанова С.А., Бадьянов Е.В., Гучетль С.З. Молекулярные маркеры генов Ph, Pli3 и Plarg для использования в селекции подсолнечника на устойчивость к ложной мучнистой росе // Масличные культуры. - 2020. - Вып. 3 (183). -С.20-26.
- Волгин В.В., Обыдало А.Д. Сравнительная эффективность способов стерилизации пыльцы цветков подсолнечника // Масличные культуры. Науч.-тех. бюл. ВНИИМК. - 2014. -Вып. 1 (157-158). - С. 10-15.
- Iwebor M., Antonova T., Saukova S. Occurrence and distribution of races 713, 733 and 734 of sunflower downy mildew pathogen in the Russian Federation // Helia. - 2018. - Vol. 41 (69). - P. 141-151. DOI: 10.1515/helia-2018-0015.
- Tang S., Yu J.K., Slabaugh M.B., Shintani K., Knapp J. Simple sequence repeat map of the sunflower genome // Theoretical and Applied Genetics. - 2002. - Vol. 105. - P. 1124-1136. DOI: 10.1007/s00122-002-0989-y.
- Гершензон С.М. Основы современной генетики. - Киев: Наукова думка, 1979. - 508 с.
- Рамазанова С.А., Бадьянов Е.В., Сави-ченко В.Г., Гучетль С.З., Стрельников Е.А. Оценка сцепления гена Plarg, контролирующего устойчивость к ложной мучнистой росе у подсолнечника, и микросателлитных локусов ДНК // Масличные культуры. - 2022. - Вып. 3 (191). -С. 14-23.


