Секвенирование фрагментов V3-V4 гена 16S rRNA для определения состава и взаимосвязи микробиоты при воспалении молочной железы и репродуктивного тракта у коров (Bos taurus)
Автор: Соколова О.В., Безбородова Н.А., Бытов М.В., Зубарева В.Д., Шкуратова И.А., Зайцева О.С., Мартынов Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Ветеринарная микробиология, микология
Статья в выпуске: 2 т.59, 2024 года.
Бесплатный доступ
Воспалительные заболевания молочной железы и репродуктивного тракта крупного рогатого скота наносят наибольший экономический ущерб молочному животноводству. Секвенирование нуклеотидных последовательностей гена 16S rRNA значительно расширило знание о бактериальных сообществах (микробиомах) и позволило выявить бактерии, о существовании которых ранее не было известно, поскольку их не удается культивировать in vitro. В представленной работе впервые показаны взаимосвязи между микробиотой репродуктивного тракта и молочной железы коров (Bos taurus) при развитии воспалительных заболеваний и выявлены новые этиологически значимые патогены. Нашей целью было определение состава микробиоты репродуктивного тракта и молочной железы коров при воспалении и сравнение полученных бактериальных профилей для обнаружения общей этиологии таких патологий. Для исследования на каждом из сельскохозяйственных предприятий, расположенных в пяти районах Свердловской области, сформировали 4 экспериментальных группы: 1-я - животные без признаков воспаления молочной железы и репродуктивного тракта (З); 2-я - животные с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М); 3-я - животные с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э); 4-я - животные с признаками воспаления молочной железы и репродуктивного тракта (ЭМ). От каждой коровы в группах получали образцы биологического материала (секрет молочной железы, цервикальные смывы), который использовали для метагеномного анализа состава бактериальных сообществ. Анализ последовательностей вариабельных участков гена 16S rRNA показал, что подавляющее большинство идентифицированных операционных таксономических единиц (operational taxonomic units, ОТU) принадлежат домену Bacteria, остальные - филуму Euryarchaeota домена Archaea (0,38 % в пробах секрета молочной железы, 0,44 % - в цервикальных смывах). В образцах секрета молочной железы идентифицированы представители 19 бактериальных филумов, в том числе 43 класса, 85 порядков, 165 семейств и 484 рода. Половину бактериальных ОТU составлял филум Firmicutes (680 ОТU, 51,7 %), второй и третий по численности филумы - Actinobacteria и Bacteroidetes (соответственно 14,5 % и 11,3 %). В цервикальных смывах обнаружены представители 22 бактериальных филумов, в их числе 50 классов, 93 порядка, 172 семейства и 365 родов. Преобладающим бактериальным филумом оказался Firmicutes (876 ОТU, 55,3 %), вторым и третьим по численности - филумы Bacteroidetes и Actinobacteria (соответственно 13,3 % и 11,6 %). Секвенирование вариабельных участков гена 16S rRNA позволило установить наличие бактерий класса Clostridia и рода Facklamia, которые мы не обнаружили ранее, исследуя образцы с помощью культуральных методов. Этим подтверждается клиническое значение метагеномного секвенирования для уточнения состава этиологических агентов в случае некультивируемых или трудно культивируемых бактерий. Определена взаимосвязь микробиоты молочной железы и репродуктивного тракта при воспалительном процессе по относительному содержанию в секрете молочной железы и цервикальных смывах при воспалении, Turicibacter sanguinis, Staphylococcus aureus, Peptostreptococcus anaerobius, Peptoniphilus indolicus и Helcococcus ovis описаны как инфекционные агенты, провоцирующие воспалительный процесс. Полученные данные важны для углубленного понимание связи бактериальной этиологии и патогенеза при воспалительных заболеваниях молочной железы и репродуктивного тракта животных.
Голштинская порода, 16s rrna, микробиота, молочная железа, репродуктивный тракт
Короткий адрес: https://sciup.org/142242449
IDR: 142242449 | УДК: 636.2:619:575:579 | DOI: 10.15389/agrobiology.2024.2.328rus
Текст научной статьи Секвенирование фрагментов V3-V4 гена 16S rRNA для определения состава и взаимосвязи микробиоты при воспалении молочной железы и репродуктивного тракта у коров (Bos taurus)
Микробиота может проявлять как синергические, так и коммен-сальные свойства, а также оказывать патогенное воздействие в зависимости от местоположения в организме хозяина. Наличие некоторых бактерий не приводит к возникновению воспалительной реакции в одной системе
∗ Работа выполнена при финансовой поддержке РФФИ и Свердловской области в рамках научного проекта ¹ 20-416-660004 «Молекулярно-генетическая и фенотипическая характеристика микробиоты репродуктивной системы крупного рогатого скота».
органов, но те же виды могут стать причиной развития патологического процесса в других органах (1). Известно, что условно-патогенные бактерии, в норме находящиеся в желудочно-кишечном тракте крупного рогатого скота, например Escherichia coli , в молочной железе или матке могут вызывать воспаление и развитие инфекционного процесса (1). Здоровая молочная железа считается стерильной средой, особенно у животных в период полового созревания до начала секреции молока (2). Во время первых родов и после начала доения молочная железа становится функционально открытой системой, имеющей прямую связь с окружающей средой. Существует предположение о наличии естественного сообщества микробов (микробиоты) вымени (3). Воспаление молочной железы (мастит) чаще всего вызывает интрамаммарная инфекция (ИМИ) бактериального происхождения. С помощью классических методов культивирования в образцах молока, взятых из пораженных долей молочной железы коров, описаны различные виды бактерий, которые могут участвовать в развитии воспалительного процесса (2, 4). Чаще всего в образцах маститного молока выявляют стафилококки, энтеробактерии и стрептококки, вызывающие подавляющее большинство ИМИ (2, 5). Однако возможности культуральных методов исследований для изучения всего спектра возможных патогенов ограничены.
Воспалительные заболевания репродуктивной системы непосредственно связаны развитием инфекционного процесса. Вероятность бактериального заражения и развития воспалительных заболеваний органов репродуктивной системы у коров возрастает в послеродовый период, так как иммунные клетки эндометрия обладают слабой способностью нейтрализовать патогены, а ткани подвержены влиянию экзогенных факторов (6). Предыдущие метагеномные исследования позволили обнаружить резкие различия в составе микробиоты у здоровых коров и коров с репродуктивными заболеваниями (7, 8). Раннее выявление микробиоты, вызывающей воспалительные заболевания репродуктивного тракта, важно для проведения своевременных лечебно-профилактических мероприятий и предотвращения инфекционного процесса (9-11).
Развитие современных методов секвенирования позволило идентифицировать микробные сообщества, которые населяют различные органы и системы организма или биологические жидкости, в том числе ранее считавшиеся стерильными (12), изучить физиологическую роль, а также вирулентные и патогенные свойства микроорганизмов для определения их значения в развитии инфекционного процесса (13). Так, в литературе имеются сведения о том, что всего 15 % бактерий, входящих в состав микробиоты рубца, можно культивировать в лабораторных условиях. Проекты по мета-геномному анализу расширили понимание о микробной экосистеме рубца и ее роли в переваривании клетчатки (14, 15). В ранних исследованиях с использованием культуральных методов диагностики при изучении микробиоты матки было выделено ограниченное число бактериальных видов, участвующих в развитии воспаления. Предполагалось, что основными возбудителями послеродовой патологии эндометрия являются Escherichia coli , Fusobacterium spp., Prevotella spp. и Trueperella pyogenes . Однако в последние годы при помощи метагеномных подходов были открыты ранее недооцененные представители микробного сообщества, способные играть роль бактериальных патогенов (16).
Секвенирование последовательности гена 16S субъединицы рибосомы наиболее часто применяется для идентификации и сравнения состава бактериальных сообществ, присутствующих в образцах (17). В большинстве литературных источников приводятся раздельные результаты исследований микробиоты молочной железы и репродуктивного тракта животных с использованием секвенирования гена 16S rRNA, касающиеся изучения состава нормофлоры, ее локальных и региональных различий, изменений при развитии маститов и воспалительных заболеваний репродуктивных органов (18, 19). Однако данные о применении метагеномного анализа для поиска взаимосвязи между микробиотой молочной железы и репродуктивного тракта коров и изучения общей бактериальной этиологии заболеваний отсутствуют.
В настоящем исследовании мы впервые показали, что состав микробиоты молочной железы и репродуктивного тракта коров при воспалении характеризуется наличием общих бактерий, не определявшихся ранее культуральными методами, что дает основание предполагать их связь в этиологии воспалительных процессов.
Цель работы — сравнить состав бактериальных сообществ в секрете молочной железы и в репродуктивном тракте коров для выявления общих агентов воспалительного процесса.
Методика . Исследования выполняли в пяти хозяйствах Свердловской области в 2021 году на коровах ( Bos taurus ) голштинской породы 2-3-й лактации. В каждом хозяйстве сформировали 4 группы коров: 1-я группа — животные без признаков воспаления молочной железы и репродуктивного тракта (З, n = 22); 2-я — с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М, n = 24); 3-я — с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э, n = 18); 4-я — с признаками воспаления молочной железы и репродуктивного тракта (ЭМ, n = 17), всего 81 животное.
С соблюдением правил асептики от каждой коровы получали образцы биологического материала (секрет молочной железы, цервикальные смывы). В каждом хозяйстве пробы по каждой экспериментальной группе объединяли, в итоге получили 39 образцов, включая 19 образцов секрета молочной железы и 20 — цервикальных смывов.
Тотальную ДНК из образцов биологического материала выделяли стандартным фенольным методом (20). Концентрацию выделенной ДНК определяли на флуориметре Qubit 4 («ThermoFisher Scientific», США).
Библиотеку ДНК для секвенирования готовили с использованием коммерческого набора реактивов 16S V3-V4 Library Preparation Kit («Illumina, Inc.», США) согласно рекомендациям производителя . Амплификацию (CFX-96, «Bio-Rad», США) проводили с универсальными бактериальными праймерами 16S-8-f-B (5′-AGRGTTTGATCCTGGCTCA-3′) и 16S-1350-r-B (5′-GACG-GGCGGTGTGTACAAG-3′). Режим амплификации: начальная денатурация — 98 °С, 30 с; 25 циклов: денатурация 98 °С, 10 с, 55 °С,15 с, 72 °С, 30 с; завершающая элонгация — 72 °С, 10 мин.
Секвенирование полученной библиотеки проводили на платформе Illumina MiSeq («Illumina, Inc.», США). последовательности выравнивали в программе ClustalW .
Полученные последовательности анализировали при помощи пай-плайна UPARSE в Usearch v.10.0 (21). Пайплайн UPARSE включает соотношение парных считанных последовательностей, фильтрацию последовательностей по качеству, их обрезку по необходимой длине, слияние идентичных последовательностей, исключение одиночных последовательностей, исключение химер. Операционные таксономические единицы (operational taxonomic unit, ОТU) объединяли в кластеры с помощью алгоритма UPARSE-OTU, 330
таксоны аттрибутировали при совместном использовании SINTAX (22), тренажера 16S RDP v.16 и BLAST+ 2.12.0 (23). Разреженность данных по OTU оценивали индивидуально для каждой пробы при помощи программы Usearch.
Относительную численность бактериальных таксонов на уровне фи-лумов, семейств и ОТU сравнивали в парном тесте Манна-Уитни для непараметрических данных по несвязанным измерениям в программе STATIS-TICA v.12 («StatSoft, Inc.», США) для образцов молока и цервикальных смывов в двух вариантах от животных во всех экспериментальных группах и в каждом хозяйстве.
Индексы β-биоразнообразия (сравнение таксономического разнообразия между исследованными образцами) (24) вычисляли с применением коэффициента Жаккарда KJ с помощью программы Usearch . Результаты представлены как медианы (Мe), минимальные и максимальные значения.
Результаты. Данные высокопроизводительного секвенирования доступны в NCBI GenBank по ссылкам: OK048814-OK050076 (микробиота секрета молочной железы коров) и OK037659-OK039186 (микробиота цервикальных смывов коров).
Таксономические профили микробиоты репродуктивного тракта и молочной железы. После проверки на качество и удаления химер для секрета молочной железы и цервикальных смывов получили соответственно 1315 и 1584 ОТU. Анализ последовательностей генов 16S rRNA показал, что подавляющее большинство ОТU принадлежат домену Bacteria , остальные — филуму Euryarchaeota домена Archaea (0,38 % в пробах секрета молочной железы, 0,44 % — в цервикальных смывах).
В секретах молочной железы идентифицировали представителей 19 бактериальных филумов, в том числе 43 класса, 85 порядков, 165 семейств и 484 рода. Половину бактериальных операционных таксономических единиц составлял филум Firmicutes (680 ОТU, 51,7 %), вторым и третьим филу-мами по числу ОТU были Actinobacteria и Bacteroidetes (соответственно 14,5 % и 11,3 %). В некоторых образцах секрета молочной железы обнаружили ОТU филумов, чей вклад также значителен. В пробах ¹ 11 и ¹ 18 мы обнаружили ОТU филума Fusobacteria (соответственно 49 % и 17 %) (рис. 1). Самым многочисленным (481 ОТU, 36,6 %) оказался класс Clostridia , в котором доминировало семейство Ruminococcaceae (192 ОТU, 14,6 % от общего числа ОТU), второе и третье место по численности заняли соответственно семейства Lachnospiraceae (113 ОТU, 8,6 %) и unc _Clostridiales (89 ОТU, 6,8 %) (рис. 2). По всем образцам секрета молочной железы, в которых изучили бактериальный состав, только 25 ОТU (или 1,9 % от всех ОТU) имели вклад ≥ 5 % в относительное содержание. Эти ОТU соответствуют представителям 6 филумов, 8 классов, 11 порядков, 16 семейств и 22 родов, что численно намного меньше по таксономическому разнообразию, чем весь список ОТU (рис. 3).
В цервикальных смывах обнаружены представители 22 бактериальных филумов, в их числе 50 классов, 93 порядка, 172 семейства и 365 родов. Преобладающим бактериальным филумом был Firmicutes (876 ОТU, 55,3 %), вторым и третьим по численности — соответственно Bacteroidetes (13,3 %) и Actinobacteria (11,6%) (рис. 4). В классе Clostridia (самая большая группа по числу ОТU — 637 ОТU, 40,2 %) доминирующим оказалось семейство Pep-toniphilaceae (256 ОТU, 16,2 %), на втором и третьем месте были соответственно семейства Lachnospiraceae (149 ОТU, 9,4 %) и unc_ Clostridiales (130 ОТU, 8,2 %) (рис. 5).
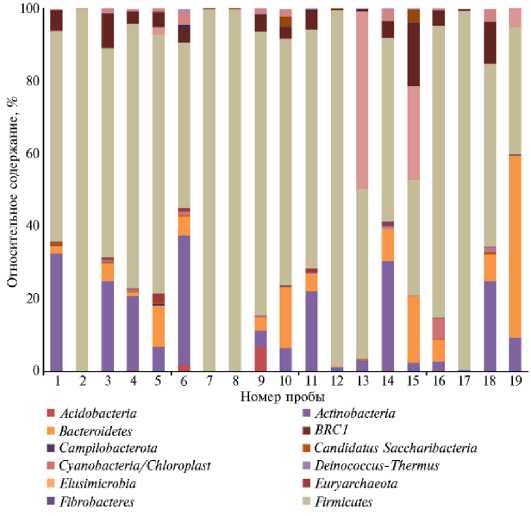
Рис. 1. Относительное содержание OTU на уровне филумов бактерий в образцах секрета молочной железы у коров ( Bos taurus ) голштинской породы (Свердловская обл., 2021 год). Группы по оси абсцисс: животные без признаков воспаления молочной железы и репродуктивного тракта (З) (4-19), с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М) (1-16), с признаками воспаления молочной железы и репродуктивного тракта (ЭМ) (3-18), с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э) (2-17).
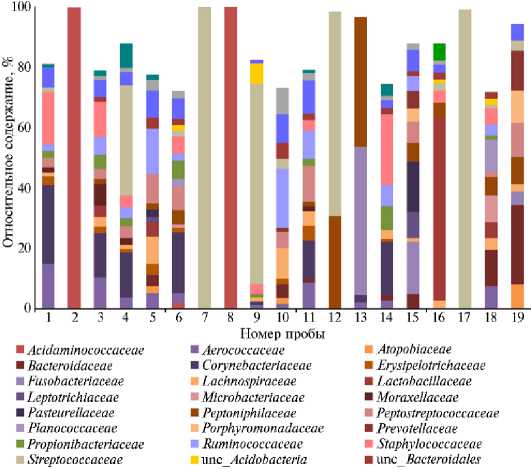
Рис. 2. Относительное содержание ОТU по преобладающим семействам бактерий в образцах секрета молочной железы у коров ( Bos taurus ) голштинской породы (Свердловская обл., 2021 год). Группы по оси абсцисс: животные без признаков воспаления молочной железы и репродуктивного тракта (З) (4-19), с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М) (1-16), с признаками воспаления молочной железы и репродуктивного тракта (ЭМ) (3-18), с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э) (2-17). Общее содержание ОТU для некоторых образцов не достигает 100 %, поскольку использованы данные только для ОТU, чей вклад составлял > 5 %; unc — unclassified.
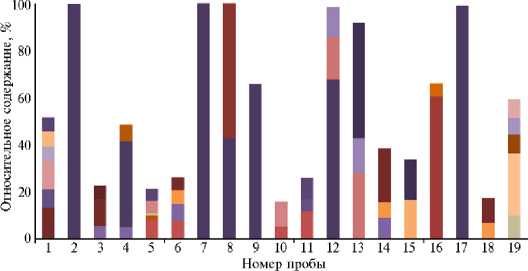
■ Bomboutsia timonensis
■ Cutibacterium acnes
■ Streptococcus agalactiae
■ Peptoniphilus indolicus
■ Histophilus somni
■ Aerococcus viridans
■ Corynebacterium xerosis
Bacteroldes pyogenes
■ Fusobacterium necrophorum
■ Sporohacter termitidis
■ Corynebacterium mans
■ [Eubaeterium] tenue
■ Porphyromonas uenonis
■ Corynebacterium bovis
■ Staphylococcus aureus
■ Eremococcus coleocola
■ Heleococcus ovis
■ SolibaciUus Silvestris
■ Peptostreptococcus anaerobius
■ Facklamia tabacinasalis
■ Psychrobacter pasteurii
■ Aiopobium minutum
■ Bacteroldes heparinolyticus
■ Streptococcus uberis
■ uncStreptophyta
Рис. 3. Относительное содержание преобладающих OTU бактерий в образцах секрета молочной железы у коров ( Bos taurus ) голштинской породы (Свердловская обл., 2021 год) . Группы по оси абсцисс: животные без признаков воспаления молочной железы и репродуктивного тракта (З) (4-19), с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М) (1-16), с признаками воспаления молочной железы и репродуктивного тракта (ЭМ) (3-18), с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э) (2-17). Общее содержание ОТU для некоторых образцов не достигает 100 %, поскольку использованы данные только для ОТU, чей вклад составлял ≥ 5 %; unc — unclassified.
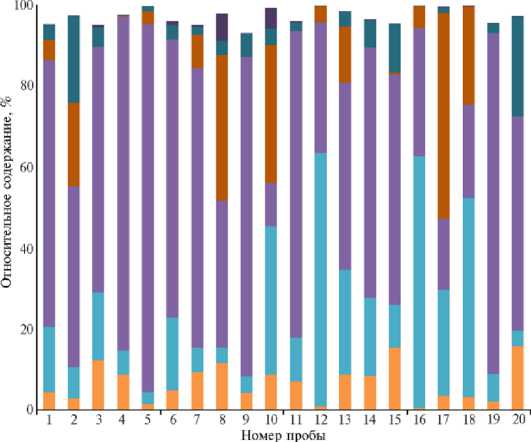
и Aainobacteria и Baaeroidetes ш Flrmlcutes
UFusobacterla и Proteobacteria UTenericutes
Рис. 4. Относительное содержание OTU на уровне филумов бактерий в образцах цервикальных смывов у коров ( Bos taurus) голштинской породы (Свердловская обл., 2021 год). Группы по оси абсцисс: животные без признаков воспаления молочной железы и репродуктивного тракта (З) (4-20), с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М) (1-17), с признаками воспаления молочной железы и репродуктивного тракта (ЭМ) (3-19), с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э) (6-18). Общее содержание ОТU для некоторых образцов не достигает 100 %, поскольку использованы данные только для ОТU, чей вклад составлял ≥ 5 %.
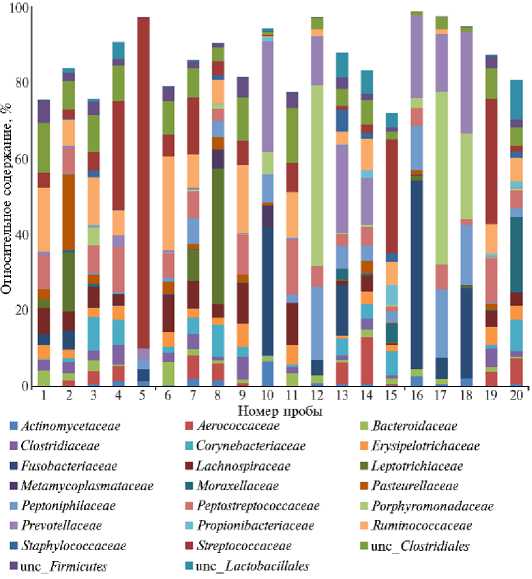
Рис. 5. Относительное содержание OTU по преобладающим бактериальным семействам в образцах цервикальных смывов у коров ( Bos taurus ) голштинской породы (Свердловская обл., 2021 год) . Группы по оси абсцисс: животные без признаков воспаления молочной железы и репродуктивного тракта (З) (4-20), с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М) (1-17), с признаками воспаления молочной железы и репродуктивного тракта (ЭМ) (3-19), с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э) (2-18). Общее содержание ОТU для некоторых образцов не достигает 100 %, поскольку использованы данные только для ОТU, чей вклад составлял > 5 %; unc — unclassified.
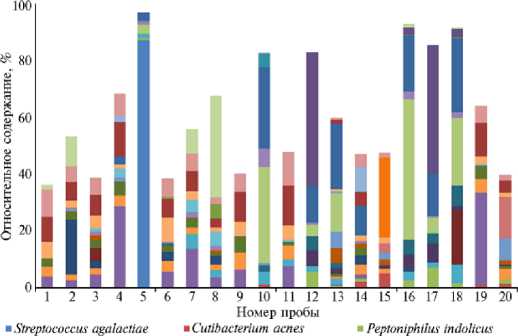
' Peptostreptococcits anaerobius • Parvimonas micro
Helcococcus ovis
Porphyromonas ievi
। Streptococcus pluranlmalium । TTistophilus somni
-
■ Eremococcus coleocola
-
■ Ttueperella pyogenes
-
• Bacteroides heparinolyticus
-
■ Porphyromonas levii
-
■ Vagococcus flusiaiis
। Psychrobacter pasteurii Corynebacterium uropygiale । Romboutsia timonensis
। Mycoplasma canadense [Eubacterium/ tenue
1 Turicibacter sanguinis t Clostridium disporicum । Staphylococcus aureus . Fusobacterium necrophorum Sporohacter termitidis
। Mycoplasma bovigenitalium
। Streptococcus uberis Streptobacillus moniliformis
Рис. 6. Относительное содержание преобладающих OTU бактерий в образцах цервикальных смывов у коров (Bos taurus) голштинской породы (Свердловская обл., 2021 год). Группы по оси абсцисс: животные без признаков воспаления молочной железы и репродуктивного тракта (З) (4-20), с признаками воспаления молочной железы, но без признаков воспаления репродуктивного тракта (М) (1-17), с признаками воспаления молочной железы и репродуктивного тракта (ЭМ) (3-19), с признаками воспаления репродуктивного тракта, но без признаков воспаления молочной железы (Э) (2-18). Общее содержание ОТU для некоторых образцов не достигает 100 %, поскольку использованы данные только для ОТU, чей вклад составлял ≥ 5 %; unc — unclassified.
Для бактериального сообщества по всем пробам цервикальных смывов только для 27 ОТU (1,7 % от общего числа ОТU) вклад в состав микробиоты был ≥ 5 %. Эти ОТU представляли 6 филумов, 8 классов, 12 порядков, 18 семейств и 24 родов бактерий, однако таксономическое разнообразие для всех ОТU намного выше (рис. 6).
Взаимосвязь микробиоты молочной железы и репродуктивного тракта при воспалениях. Для сравнения β-биоразно-образия микробиоты (по данным метагеномного анализа гена 16S rRNA) при патологии воспалительного характера мы использовали коэффициент Жаккарда K J. Среднее значение K J , которое определяли при сравнении пяти хозяйств между собой, составило 0,885. Значение коэффициента Жаккарда (0 ≤ K j ≤ 1) прямо отражает степень различий (чем больше значение коэффициента, тем сильнее различия между пробами) (24). Однако этот индекс учитывает все ОТU, включая те, вклад которых предельно незначителен. Целесообразно учитывать ОТU, имеющие существенный вклад (≥ 1 %) в таксономический состав бактериального сообщества. Поэтому мы оценили различия в составе микробиоты секрета молочной железы и цервикальных смывов по относительному содержанию ОТU, доминирующих по числу (табл.).
Виды микроорганизмов, общие для микробиоты при воспалении молочной железы и репродуктивного тракта, у коров ( Bos taurus ) голштинской породы из пяти обследованных хозяйств (Свердловская обл., 2021 год)
|
¹ 1 |
¹ 2 |
¹ 3 |
¹ 4 |
¹ 5 |
|
Вид Turicibacter sanguinis (A) Helcococcus ovis (D) Helcococcus ovis (D) Turicibacter sanguinis (A) Sporobacter termitidis (B) Peptoniphilus in- Facklamia tabac- Cutibacterium acnes dolicus inasalis Bacteroides tenuis (C) Fusobacterium necro- Staphylococcus au- phorum reus/simiae Clostridium saudiense/disporicum Romboutsia timonensis Butyrivibrio proteoclasticus Примечани е. Буквами (A-D) отмечены совпадающие виды микроорганизмов. |
Peptostreptococcus ana-erobius Sporobacter termitidis (B) Bacteroides tenuis (C) |
|||
При сравнении биологических образцов по ОТU установили, что при воспалении как молочной железы, так и репродуктивного тракта в образцах патологического биоматериала присутствуют Turicibacter sanguinis , Staphylococcus aureus , Sporobacter termitidis , Clostridium saudiense , Peptostrepto-coccus anaerobius , Romboutsia timonensis , Bacteroides tenuis , Butyrivibrio proteo-clasticus , Peptoniphilus indolicus , Helcococcus ovis , Facklamia tabacinasalis , Fuso-bacterium necrophorum , Cutibacterium acnes .
Из бактерий, по которым установлена взаимосвязь по относительному содержанию в секрете молочной железы и цервикальных смывах при воспалении, Turicibacter sanguinis, Staphylococcus aureus, Peptostreptococcus ana-erobius, Peptoniphilus indolicus и Helcococcus ovis описаны как инфекционные агенты, провоцирующие воспалительный процесс (25-28). Fusobacterium nec-rophorum — грамотрицательная неспорообразующая анаэробная палочка, которая обитает в желудочно-кишечном тракте, но при попадании в другие ткани может стать патогенным микроорганизмом, вызывающим некротические заболевания (29). Его роль в рубце заключается в ферментации молочной кислоты и деградации белков корма и эпителия (30). Исследования последних лет показали, что F. necrophorum участвует в возникновении мастита у молочных коров, эндометрита, межпальцевого дерматита и язвы пищевода (29, 31-33). F. necrophorum является одной из преобладающих бактерий, вызывающих внутриутробную инфекцию у коров, и в большем количестве обнаруживается у коров с метритом, чем у здоровых коров. Было показано, что F. necrophorum, выделенный из абсцессов печени крупного рогатого скота, обладает несколькими факторами вирулентности, такими как адгезины, лейкотоксины, эндотоксины, гемолизины, гемагглютинины и протеазы. Однако механизмы, с помощью которых происходит колонизация репродуктивного тракта, остаются неизвестными (29).
Sporobacter termitidis ранее был выявлен в пробах секрета молочной железы как у здоровых, так и у больных маститами (34), также известно, что этот микроорганизм — представитель нормофлоры рубца крупного рогатого скота (35). Romboutsia timonensis , Facklamia tabacinasalis и Bacteroides tenuis, также известный как Eubacterium tenue , выявляли в пробах секрета молочной желез, на коже сосков вымени у коров и в сборном молоке (36, 37).
Butyrivibrio proteoclasticus и Cutibacterium acnes — представители микробиоты рубца, но последний также выделяли из молочных продуктов (38, 39). Clostridium saudiense при помощи секвенирования гена 16S rRNA был обнаружен в желудочно-кишечном тракте свиней, однако требуется дальнейшее изучение его роли в качестве инфекционного агента (40).
Важная роль рубцовой и кишечной микробиоты в репродуктивном здоровье коров отмечается в ряде исследований (41-43). Предполагается, что колонизация половых путей и молочной железы микробиотой желудочно-кишечного тракта влияет на патогенез мастита и эндометрита и можно скорректировать микробиоценоз половых путей, воздействуя на микробиоту рубца (44).
Как в структуре микробных сообществ секрета молочной железы, так и в структуре микробиоценоза цервикальных смывов у крупного рогатого скота преобладающими филумами были Firmicutes , Actinobacteria и Bacteroidetes . Эти данные согласуются с общими представлениями о составе микробиоты репродуктивного тракта коров (45). В некоторых образцах секрета молочной железы и цервикальных смывов, отобранных от животных с одновременным заболеванием маститом и эндометритом (ЭМ), мы отмечали высокое относительное содержание филума Fusobacteria. У большинства животных этот филум доминировал в цервикальных смывах и только у некоторых особей — одновременно и в секрете молочной железы, и в цервикальных смывах. При этом во всех цервикальных смывах, а также в большинстве образцов секрета молочной железы присутствовал филум Proteobacteria . В секрете молочной железы относительное содержание этой группы бактерий по числу ОТU составило 17 %. Здесь протеобактерии были представлены видом Histofilus somni . Histophilus somni — представитель семейства Pasteurellaceae , участвует в развитии воспалительных процессов при различных заболеваниях крупного рогатого скота (тромботический менингоэнцефалит, респираторные заболевания, миокардиты, маститы) (46). В цервикальных смывах обилие протеобактерий (12 % в составе микробиоты при относительном содержании 7,8 % в расчете на число ОТU, для которых вклад в состав микробиоты превышает 1 %) представляли виды Psychrobacter pasteurii (3,3 %), Enterobacteriaceae (2,1 %), Mailhella massiliensis (1,3 %), Oligella ureolytica (1,2 %). По данным британских ученых, виды Psychrobacter редко вызывают заболевания у животных (47). Mailhella massiliensis обладает 336
сульфатредуцирующими свойствами, которые ингибируют процесс образования метана в рубце у крупного рогатого скота (48, 49) , а Oligella ureolytica редко обнаруживается в микробиологических исследованиях как с использованием культуральных методов, так и с помощью секвенирования последовательности гена 16S rRNA (50). Следует отметить, что в известных нам исследованиях развитие репродуктивных заболеваний у коров связывали с высокой относительной численностью бактерий филума Fusobacteria (16, 51), однако подобные данные о Proteobacteria получены нами впервые. Также отметим, что при доминировании основных филумов Firmicutes , Actinobacteria и Bacteroidetes состав микробиоты каждого образца все же различается по наличию и относительному обилию других значимых таксонов, для определения видового состава которых требуется более детальный анализ.
Таким образом, на основании результатов генотипирования микробиоты по 16S rRNA определены таксономические профили образцов секрета молочной железы и цервикальных смывов от здоровых коров и животных с проявлением мастита и эндометрита. В секретах молочной железы идентифицировали представителей 19 бактериальных филумов, в том числе 43 класса, 85 порядков, 165 семейств и 484 рода. Половину бактериальных операционных таксономических единиц составлял филум Firmicutes (680 ОТU, 51,7 %), вторым и третьим филумами по числу ОТU были Actinobacteria и Bacteroidetes (соответственно 14,5 % и 11,3 %). В пробах цервикальных смывов преобладал бактериальный филум Firmicutes (876 ОТU, 55,3 %), вторым и третьим были соответственно Bacteroidetes (13,3 %) и Actinobacteria (11,6 %). Секвенирование 16S rRNA для выявления различий в профиле микробиоты репродуктивного тракта и молочной железы при воспалительном процессе использовано в настоящей работе впервые. Это позволило обнаружить бактерии, которые можно рассматривать как общие этиологические агенты мастита и эндометрита. Полученные нами данные об инфекционных агентах, провоцирующих воспалительный процесс, совпадают с результатами предыдущих исследований. Так, при воспалении обнаружены общие патогены как в секрете молочной железы, так и в цервикальных смывах. Это Turicibacter sanguinis , Staphylococcus aureus , Peptostreptococcus anaerobius , Peptoniphilus indolicus и Helcococcus ovis . Представленные нами результаты полезны для понимания роли бактериальной микробиоты в патогенезе воспалительных заболеваний молочной железы и репродуктивного тракта и для разработки экосистемного подхода к лечебно-профилактическим мероприятиям.
Список литературы Секвенирование фрагментов V3-V4 гена 16S rRNA для определения состава и взаимосвязи микробиоты при воспалении молочной железы и репродуктивного тракта у коров (Bos taurus)
- Owens C.E., Daniels K.M., Ealy A.D., Knowlton K.F., Cockrum R.R. Graduate student litera-ture review: potential mechanisms of interaction between bacteria and the reproductive tract of dairy cattle. Journal of Dairy Science, 2020, 103(11): 10951-10960 (doi: 10.3168/jds.2019-18050).
- Taponen S., McGuinness D., Hiitiö H., Simojoki H., Zadoks R., Pyörälä S. Bovine milk micro-biome: a more complex issue than expected. Veterinary Research, 2019, 50(1): 44 (doi: 10.1186/s13567-019-0662-y).
- Derakhshani H., Fehr K.B., Sepehri S., Francoz D., De Buck J., Barkema H.W., Plaizier J.C., Khafipour E. Invited review: Microbiota of the bovine udder: contributing factors and potential implications for udder health and mastitis susceptibility. Journal of Dairy Science, 2018, 101(12): 10605-10625 (doi: 10.3168/jds.2018-14860).
- Sokolova O.V., Bezborodova N.A., Lysova Y.Y., Pechura E.V. Characteristics of species composition, biochemical and pathogenic nature of the microbiota of mammary gland and the reproductive tract in dairy cows. E3S Web of Conferences, 2021, 282: 03017 (doi: 10.1051/e3sconf/202128203017).
- Vakkamäki J., Taponen S., Heikkilä A-M., Pyörälä S. Bacteriological etiology and treatment of mastitis in Finnish dairy herds. Acta Veterinaria Scandinavica, 2017, 59(1): 33 (doi: 10.1186/s13028-017-0301-4).
- Ault T.B., Clemmons B.A., Reese S.T., Dantas F.G., Franco G.A., Smith T.P.L., Edwards J.L., Myer P.R., Pohler K.G. Bacterial taxonomic composition of the postpartum cow uterus and vagina prior to artificial insemination. Journal of Animal Science, 2019, 97(10): 4305-4313 (doi: 10.1093/jas/skz212).
- Santos T.M., Gilbert R.O., Bicalho R.C. Metagenomic analysis of the uterine bacterial microbiota in healthy and metritic postpartum dairy cows. Journal of Dairy Science, 2011, 94(1): 291-302 (doi: 10.3168/jds.2010-3668).
- Jeon S.J., Vieira-Neto A., Gobikrushanth M., Daetz R., Mingoti R.D., Parize A.C., de Freitas S.L., da Costa A.N., Bicalho R.C., Lima S., Jeong K.C., Galvao K.N. Uterine microbiota progression from calving until establishment of metritis in dairy cows. Applied and Environmental Microbiology, 2015, 81(18): 6324-6332 (doi: 10.1128/AEM.01753-15).
- Bicalho M.L.S., Santin T., Rodrigues M.X., Marques C.E., Lima S.F., Bicalho R.C. Dynamics of the microbiota found in the vaginas of dairy cows during the transition period: associations with uterine diseases and reproductive outcome. Journal of Dairy Science, 2017, 100(4): 3043-3058 (doi: 10.3168/jds.2016-11623).
- Шкуратова И.А., Шилова Е.Н., Соколова О.В., Ряпосова М.В. Программы контроля инфекционных факторов, влияющих на репродуктивную функцию высокопродуктивных молочных коров. Ветеринария и кормление, 2020, 2: 54-57 (doi: 10.30917/ATT-VK-1814-9588-2020-2-13).
- Лаптев Г.Ю., Новикова Н.И., Ильина Л.А., Йылдырым Е.А., Думова В.А., Племяшов К.В., Корочкина Е.А. Сравнительный анализ микробиоценоза влагалища у коров методом realtime PCR. Ветеринария, 2014, 8: 33-37.
- Oikonomou G., Addis M.F., Chassard C., Nader-Macias M.E.F., Grant I., Delbès C., Bogni C.I., Le Loir Y., Even S. Milk microbiota: what are we exactly talking about? Frontiers in Microbiology, 2020, 11: 60 (doi: 10.3389/fmicb.2020.00060).
- Fursova K., Sorokin A., Sokolov S., Dzhelyadin T., Shulcheva I., Shchannikova M., Ni-kanova D., Artemieva O., Zinovieva N., Brovko F. Virulence factors and phylogeny of Staphylo-coccus aureus associated with bovine mastitis in Russia based on genome sequences. Frontiers in Veterinary Science, 2020, 7: 135 (doi: 10.3389/fvets.2020.00135).
- Gruninger R.J., Ribeiro G.O., Cameron A., McAllister T.A. Invited review: application of meta-omics to understand the dynamic nature of the rumen microbiome and how it responds to diet in ruminants. Animal, 2019, 13(9): 1843-1854 (doi: 10.1017/S1751731119000752).
- Gharechahi J., Vahidi M.F., Bahram M., Han J.L., Ding X.Z., Salekdeh G.H. Metagenomic analysis reveals a dynamic microbiome with diversified adaptive functions to utilize high ligno-cellulosic forages in the cattle rumen. The ISME Journal, 2021, 15(4): 1108-1120 (doi: 10.1038/s41396-020-00837-2).
- Ong C.T., Turni C., Blackall P.J., Boe-Hansen G., Hayes B.J., Tabor A.E. Interrogating the bovine reproductive tract metagenomes using culture-independent approaches: a systematic re-view. Animal Microbiome, 2021, 3(1): 41 (doi: 10.1186/s42523-021-00106-3).
- Dahlberg J., Sun L., Persson Waller K., Östensson K., McGuire M., Agenäs S., Dicksved J. Microbiota data from low biomass milk samples is markedly affected by laboratory and reagent contamination. PLoS ONE, 2019, 14(6): e0218257 (doi: 10.1371/journal.pone.0218257).
- Hoque M.N., Istiaq A., Rahman M.S., Islam M.R., Anwar A., Siddiki A.M.A.M.Z., Sultana M., Crandall K.A., Hossain M.A. Microbiome dynamics and genomic determinants of bovine mastitis. Genomics, 2020, 112(6): 5188-5203 (doi: 10.1016/j.ygeno.2020.09.039).
- Ong C.T., Ross E.M., Boe-Hansen G., Turni C., Hayes B.J., Fordyce G., Tabor A.E. Adaptive sampling during sequencing reveals the origins of the bovine reproductive tract microbiome across reproductive stages and sexes. Scientific Reports, 2022, 12(1): 15075 (doi: 10.1038/s41598-022-19022-w).
- Anahtar M.N., Bowman B.A., Kwon D.S. Efficient nucleic acid extraction and 16S rRNA gene sequencing for bacterial community characterization. Journal of Visualized Experiments: JoVE, 2016, (110): 53939 (doi: 10.3791/53939).
- Edgar R.C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998 (doi: 10.1038/nmeth.2604).
- Edgar R.C. SINTAX: a simple non-Bayesian taxonomy classifier for 16S and ITS sequences. bioRxiv, 2016: 074161 (doi: 10.1101/074161).
- Camacho C., Coulouris G., Avagyan V., Ma N., Papadopoulos J., Bealer K., Madden T.L. BLAST+: architecture and applications. BMC Bioinformatics, 2019, 10: 421 (doi: 10.1186/1471-2105-10-421).
- Calle M.L. Statistical analysis of metagenomics data. Genomics & Informatics, 2019, 17(1): e6 (doi: 10.5808/GI.2019.17.1.e6).
- Gryaznova M.V., Syromyatnikov M.Y., Dvoretskaya Y.D., Solodskikh S.A., Klimov N.T., Mi-khalev V.I., Zimnikov V.I., Mikhaylov E.V., Popov V.N. Microbiota of cow’s milk with udder pathologies. Microorganisms, 2021, 9(9): 1974 (doi: 10.3390/microorganisms9091974).
- Liu K., Deng Z., Zhang L., Gu X., Liu G., Liu Y., Chen P., Gao J., Han B., Qu W. Biological characteristics and pathogenicity of Helcococcus ovis isolated from clinical bovine mastitis in a Chi-nese dairy herd. Frontiers in Veterinary Science, 2022, 8: 756438 (doi: 10.3389/fvets.2021.756438).
- Wang M.L., Liu M.C., Xu J., An L.G., Wang J.F., Zhu Y.H. Uterine microbiota of dairy cows with clinical and subclinical endometritis. Frontiers in Microbiology, 2018, 9: 2691 (doi: 10.3389/fmicb.2018.02691).
- Kanoe M., Nouka K., Toda M. Isolation of obligate anaerobic bacteria from bovine abscesses in sites other than the liver. Journal of Medical Microbiology, 1984, 18(3): 365-369 (doi: 10.1099/00222615-18-3-365).
- Francis A.M., Jeon S.J., Cunha F., Jeong K.C., Galvão K.N. Draft genome sequences of two Fusobacterium necrophorum strains isolated from the uterus of dairy cows with metritis. Microbi-ology Resource Announcements, 2019, 8(17): e00201-19 (doi: 10.1128/MRA.00201-19).
- Tadepalli S., Narayanan S.K., Stewart G.C., Chengappa M.M., Nagaraja T.G. Fusobacterium necrophorum: a ruminal bacterium that invades liver to cause abscesses in cattle. Anaerobe, 2009, 15(1-2): 36-43 (doi: 10.1016/j.anaerobe.2008.05.005).
- Polveiro R.C., Vidigal P.M.P., Mendes T.A.O., Yamatogi R.S., Lima M.C., Moreira M.A.S. Effects of enrofloxacin treatment on the bacterial microbiota of milk from goats with persistent mastitis. Scientific Reports, 2020, 10(1): 4421 (doi: 10.1038/s41598-020-61407-2).
- Tosaki K., Kojima H., Akama S., Ootake Y., Inoue K., Katsuda K., Shibahara T. Bovine esoph-ageal and glossal ulceration associated with Pseudomonas aeruginosa and Fusobacterium spp. in a 10-month-old Holstein heifer. The Journal of Veterinary Medical Science, 2018, 80(7): 1174-1178 (doi: 10.1292/jvms.17-0616).
- Staton G.J., Sullivan L.E., Blowey R.W., Carter S.D., Evans N.J. Surveying bovine digital der-matitis and non-healing bovine foot lesions for the presence of Fusobacterium necrophorum, Por-phyromonas endodontalis and Treponema pallidum. The Veterinary Record, 2020, 186(14): 450 (doi: 10.1136/vr.105628).
- Kano R., Kobayashi Y., Nishikawa A., Murata R., Itou T., Ito T., Suzuki K., Kamata H. Next-generation sequencing analysis of bacterial flora in bovine prototheca mastitic milk. Medical My-cology Journal, 2018, 59(3): E41-E46 (doi: 10.3314/mmj.18-00004).
- Mackie R.I., Aminov R.I., Hu W., Klieve A.V., Ouwerkerk D., Sundset M.A., Kamagata Y. Ecology of uncultivated Oscillospira species in the rumen of cattle, sheep, and reindeer as assessed by microscopy and molecular approaches. Applied and Environmental Microbiology, 2003, 69(11): 6808-6815 (doi: 10.1128/AEM.69.11.6808-6815.2003).
- Verdier-Metz I., Delbès C., Bouchon M., Pradel P., Theil S., Rifa E., Corbin A., Chassard C. Influence of post-milking treatment on microbial diversity on the cow teat skin and in milk. Dairy, 2022, 3(2): 262-276 (doi: 10.3390/dairy302002).
- Verdier-Metz I., Gagne G., Bornes S., Monsallier F., Veisseire P., Delbès-Paus C., Montel M.C. Cow teat skin, a potential source of diverse microbial populations for cheese production. Applied and Environmental Microbiology, 2012, 78(2): 326-333 (doi: 10.1128/AEM.06229-11).
- Petri R.M., Vahmani P., Yang H.E., Dugan M.E.R., McAllister T.A. Changes in rumen microbial profiles and subcutaneous fat composition when feeding extruded flaxseed mixed with or before hay. Frontiers in Microbiology, 2018, 9: 1055 (doi: 10.3389/fmicb.2018.01055).
- Dréno B., Pécastaings S., Corvec S., Veraldi S., Khammari A., Roques C. Cutibacterium acnes (Propionibacterium acnes) and acne vulgaris: a brief look at the latest updates. Journal of the Eu-ropean Academy of Dermatology and Venereology: JEADV, 2018, 32(Suppl 2): 5-14 (doi: 10.1111/jdv.15043).
- Kumar H., Jang Y.N., Kim K., Park J., Jung M.W., Park J.E. Compositional and functional characteristics of swine slurry microbes through 16S rRNA metagenomic sequencing approach. Animals, 2020, 10(8): 1372 (doi: 10.3390/ani10081372).
- Jeon S.J., Cunha F., Vieira‐Neto A., Bicalho R.C., Lima S., Bicalho M.L., Galvão K.N. Blood as a route of transmission of uterine pathogens from the gut to the uterus in cows. Microbiome, 2017, 5: 109 (doi: 10.1186/s40168‐017‐0328‐9).
- Khalil A., Batool A., Arif S. Healthy cattle microbiome and dysbiosis in diseased phenotypes. Ruminants, 2022, 2(1): 134-156 (doi: 10.3390/ruminants2010009).
- Deng F., McClure M., Rorie R., Wang X., Chai J., Wei X., Lai S., Zhao J. The vaginal and fecal microbiomes are related to pregnancy status in beef heifers. Journal of Animal Science and Bio-technology, 2019, 10: 92 (doi: 10.1186/s40104-019-0401-2).
- Лаптев Г., Йылдырым Е., Ильина Л. Микробиом рубца — основа здоровья коров. Животноводство России, 2020, 4: 42-45.
- Chen H., Fu K., Pang B., Wang J., Li H., Jiang Z., Feng Y., Tian W., Cao R. Determination of uterine bacterial community in postpartum dairy cows with metritis based on 16S rDNA sequenc-ing. Veterinary and Animal Science, 2020, 10: 100102 (doi: 10.1016/j.vas.2020.100102).
- Ueno Y., Suzuki K., Takamura Y., Hoshinoo K., Takamatsu D., Katsuda K. Antimicrobial re-sistance and associated genetic background of Histophilus somni isolated from clinically affected and healthy cattle. Frontiers in Veterinary Science, 2022, 9: 1040266 (doi: 10.3389/fvets.2022.1040266).
- Maboni G., Blanchard A., Frosth S., Stewart C., Emes R., Tötemeyer S. A distinct bacterial dysbiosis associated skin inflammation in ovine footrot. Scientific Reports, 2017, 7: 45220 (doi: 10.1038/srep45220).
- Jangid A., Fukuda S., Suzuki Y., Taylor T.D., Ohno H., Prakash T. Shotgun metagenomic se-quencing revealed the prebiotic potential of a grain-based diet in mice. Scientific Reports, 2022, 12: 6748 (doi: 10.1038/s41598-022-10762-3).
- Zhao Y., Zhao G. Decreasing ruminal methane production through enhancing the sulfate reduc-tion pathway. Animal Nutrition (Zhongguo xu mu shou yi xue hui), 2022, 9: 320-326 (doi: 10.1016/j.aninu.2022.01.006).
- Horvat R.T., El Atrouni W., Hammoud K., Hawkinson D., Cowden S. Ribosomal RNA sequence analysis of Brucella infection misidentified as Ochrobactrum anthropi infection. Journal of Clinical Microbiology, 2011, 49(3): 1165-1168 (doi: 10.1128/JCM.01131-10).
- Wang Y., Wang J., Li H., Fu K., Pang B., Yang Y., Liu Y., Tian W., Cao R. Characterization of the cervical bacterial community in dairy cows with metritis and during different physiological phases. Theriogenology, 2018, 108: 306-313 (doi: 10.1016/j.theriogenology.2017.12.028).


