Слияние медицинских данных на основе онтологий
Автор: Лебедев С.В., Жукова Н.А.
Журнал: Онтология проектирования @ontology-of-designing
Рубрика: Прикладные онтологии проектирования
Статья в выпуске: 2 (24) т.7, 2017 года.
Бесплатный доступ
В информационных системах медицинских учреждений хранится большой объём данных, относящихся к проводимым лечебно-диагностическим мероприятиям. Собираемые данные представляют собой слабосвязанный и слабоструктурированный массив. Для повышения эффективности информационной поддержки лечения необходимо внедрить в информационные медицинские системы процесс непрерывного слияния данных и знаний. В статье для реализации этого процесса предложен подход, в котором совместно используются модель слияния данных JDL (Joint Directors of Laboratories) и онтологии. Модель определяет состав этапов процесса слияния данных, их функции и взаимосвязь. Онтологии используются для представления и связывания отдельных наборов данных и знаний. Для детализации этапов и проверки подхода в целом рассмотрен упрощённый сценарий слияния данных и знаний. Сценарий разработан по запросу специалистов Северо-Западного федерального медицинского исследовательского центра имени В.А. Алмазова Минздрава России, который является одним из ведущих центров в области кардиологии (Россия, Санкт-Петербург). Сценарий включает несколько этапов, состав которых определён уровнями JDL модели. Предварительно строятся собственные медицинские онтологии. На первом этапе из исходных неструктурированных данных выделяются отдельные объекты - лекарства и диагнозы для каждого пациента. На втором этапе извлечённые объекты связываются с элементами собственных и сторонних онтологий. На третьем этапе на основе установленных связей выявляются пары взаимодействующих лекарств. На четвёртом этапе формируется графическое представление данных. Сценарий позволил подтвердить применимость подхода для слияния медицинских данных, уточнить детали реализации каждого из этапов, определить набор технологий для реализации. В статье предложен новый подход к совместному использованию модели слияния данных и онтологий, позволяющий повысить связанность данных в медицинских информационных системах.
Онтология, модель слияния данных, медицинские информационные системы, открытые связанные данные
Короткий адрес: https://sciup.org/170178748
IDR: 170178748 | УДК: 004.89, | DOI: 10.18287/2223-9537-2017-7-2-145-159
Текст научной статьи Слияние медицинских данных на основе онтологий
В повседневной работе медицинских учреждений накапливается большой объём лечебно-диагностических данных. К ним относятся экспертные заключения профильных врачей, результаты проведения диагностических исследований, дневники лечащих врачей и т.п. Медицинские информационные системы (МИС), которые используется для управления этими данными, обладают рядом недостатков [1]:
-
■ накапливаемые данные слабо структурированы и слабо связаны как между собой, так и с имеющимися базами знаний;
-
■ МИС различных учреждений не связаны между собой.
Для эффективной информационной поддержки лечебно-диагностических мероприятий необходимо внедрить в МИС процесс непрерывного связывания медицинских данных и знаний. На вход процесса могут поступать данные в разных форматах и из различных источников. Результатом должно быть единое представление, которое можно было бы использовать, например, для прогноза течения болезни, планирования лечения и т.п.
Формат представления данных, используемый в таких процессах, должен отвечать следующим требованиям.
-
■ Быть доступным для машинной обработки. Это позволит применять алгоритмы обработки и анализа данных, строить информативные графические представления (графы, таблицы, диаграммы и т.п.), использовать сложные алгоритмы поиска зависимостей и извлечения знаний.
-
■ Быть достаточно выразительным для описания предметной области (ПрО) медицины.
-
■ Позволять связывать разнородные массивы данных и базы знаний.
Таким образом, для решения проблемы необходимо решить две задачи:
-
■ выбрать и адаптировать модель слияния данных;
-
■ выбрать средства для представления и связывания данных и знаний.
1 Текущее состояние области слияния медицинских данных
Рассмотрим существующие решения поставленных задач. В [2] описаны модели процессов слияния данных:
-
■ цикл OODA (O - observe, O - orient, D - decide, A - act);
-
■ модель слияния данных JDL (Joint Directors of Laboratories);
-
■ модель ситуационной осведомленности Эндсли и др.
Для реализации предлагаемого подхода выбрана модель JDL как наиболее общая. Подробное описание модели можно найти в [3, 4]. Модель JDL включает следующие уровни: уровень работы с сырыми данными, уровень извлечения объектных данных, уровень построения ситуации, уровень вычисления последствий и уровень взаимодействия с пользователем. Для каждого уровня описаны функции и способ взаимодействия с соседними уровнями. Модель не ограничивает способы реализации уровней и не привязана к какой-либо ПрО.
Один из вариантов применения JDL модели в области медицины представлен в [5]. Авторы статьи используют модель для объединения методов анализа медицинских данных в единый вычислительный процесс. В отличие от указанной работы в предлагаемом подходе акцент сделан на применение онтологий как основы процесса слияния.
Онтологии – это центральное понятие глобальной семантической сети (semantic web), которое является одним из перспективных направлений развития МИС [6]. Онтологии позволяют формально описать ПрО, связать множество таких описаний друг с другом, использовать описание для аннотирования данных. Таким образом, данные становятся связанными как с общими знаниями, так и с другими данными, обеспечивается интерпретация данных на машинном уровне.
Ключевыми в семантической сети являются модель и схема описания метаданных (RDF/RDFS), язык описания онтологий (OWL), язык запросов к RDF-описаниям (SPARQL). Инструменты семантической сети включают редакторы, хранилища, средства отображения, средства построения систем правил, машины логического вывода и т.п.
Некоторые общие вопросы использования технологий семантической сети в медицине рассмотрены в [7, 8]. В частности, в работе [8] рассматривается само понятие онтологии и применение онтологий в медицине. Авторы обосновывают целесообразность совместного использования онтологий и методов интеллектуального анализа данных (data mining). Приводятся примеры инструментов, позволяющих строить онтологии по текстам на естественном языке.
В целом, задача извлечения знаний в области медицины должна решаться применительно к различным типам данных – числовым, текстовым, графическим. Основное внимание уделяется обработке текстов, т.к. значимые результаты врачебной деятельности представлены именно в таком формате. Один из подходов работы с текстами описан в [9].
Важным условием для построения процесса слияния является наличие открытых медицинских баз знаний, представленных в форматах семантической сети. Одним из проектов по созданию доступных онтологий биомедицины является OBO Foundry1 (Open Biomedical Ontologies). В этот проект входит ряд онтологий, включая онтологию человеческих болезней (Human Disease Ontology), Онтологию взаимодействия лекарств (Drug Interaction and Evidence Ontology) и др. Онтологии OBO Foundry образуют одну из основных частей портала Национального центра биомедицинских онтологий (National Center for Biomedical Ontology, NCBO's BioPortal2). Портал содержит 541 онтологию. Инициатива по созданию и распространению открытых данных в рамках направления семантической сети называется открытыми связанными данными ( Linked Open Data, LOD ) .
В [10] описан облачный сервис, который использует открытые связанные данные для повышения эффективности оказания неотложной помощи. В частности, используется онтология Международной классификации болезней (International Classification of Diseases, ICD). Сервис организован как набор модулей, реализующих извлечение, хранение, преобразование, отображение и связывание данных.
Таким образом, семантическая сеть предоставляет искомые средства для описания и связывания медицинских данных и знаний, позволяет использовать открытые медицинские онтологии. Слияние медицинских данных на основе онтологий можно рассматривать как один из вариантов решения проблемы интеграции системы документооборота медицинского учреждения с существующими базами медицинских знаний. Решение этой проблемы необходимо, например, при автоматизации интеллектуальной деятельности на базе экспертных систем в области медицины [11, 12].
2 Слияние медицинских данных на основе онтологий
Конкретизируем предложенный подход, основанный на реализации JDL модели. Для этого:
-
1) уточним каждый из функциональных уровней JDL модели слияния данных применительно к медицинской области;
-
2) определим место онтологий в процессе слияния данных.
На рисунке 1 представлена уточнённая модель слияния данных. На первом уровне получают слабоструктурированные и слабосвязанные данные: результаты анализов и диагностических процедур, заключения, дневники врачей и т.п. Данные могут поступать из реляционных баз данных, браться из файлов.
На втором уровне из слабоструктурированных данных извлекают отдельные термины и числовые данные. Например, названия назначенных лекарств, поставленный основной диа- гноз, перечень сопутствующих диагнозов, числовые показатели параметров крови. К каждому формату данных должны применяться подходящие методы.
На третьем уровне – построение ситуации – связывают извлечённые термины и данные c понятиями собственных и сторонних онтологий. Например, диагноз пациента можно привязать к соответствующему понятию из Международного классификатора болезней. Под ситуацией будем понимать множество взаимосвязанных данных, ассоциированных с конкретным пациентом.
На четверном уровне полученная ситуация используется для порождения нового знания. Например, используя онтологическое описание воздействия лекарства на течение болезни, можно спрогнозировать последствия приёма лекарства конкретным пациентом. На этом этапе могут быть использованы запросы к онтологиям, логический вывод, продукционные системы.
На пятом уровне отображают результаты двух предыдущих уровней пользователю в форме таблиц, графов, диаграмм, функциональных зависимостей.
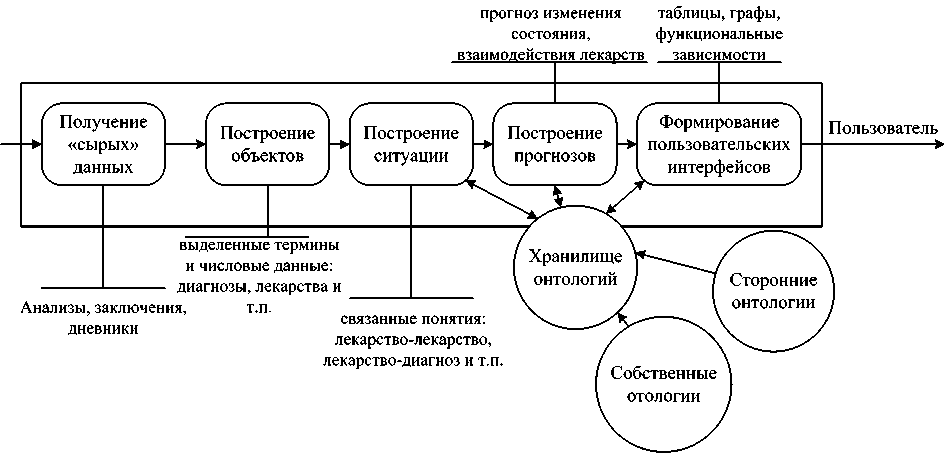
Рисунок 1 – Расширенная модель слияния медицинских данных
В представленной модели онтологии используются, начиная с третьего уровня. Путём аннотирования данные привязываются к элементам онтологии. Существующие между понятиями онтологии связи позволяют связать разрозненные данные. На четвёртом уровне установленные связи используются для получения новых знаний. На последнем уровне на основе онтологий строятся графические преставления.
3 Проверка предложенного подхода
Для проверки подхода подготовлен и реализован упрощённый сценарий поиска взаимодействия лекарств, назначенных пациенту. Сам сценарий подготовлен в интересах СевероЗападного федерального медицинского исследовательского центра имени В. А. Алмазова как демонстрация возможностей технологий семантической сети.
Цель проверки заключается в решении следующих задач:
-
■ определить перечень инструментов и операций для реализации каждого уровня;
-
■ выявить возможные проблемы;
-
■ наметить пути дальнейшего развития подхода.
-
3.1 Описание сценария
Упрощённый сквозной сценарий разделён на этапы, каждый из которых соответствует уровню JDL модели (см. рисунок 2).
На первом этапе получают файл в формате CSV (Comma Separated Values — значения, разделённые запятыми). Файл представляет собой таблицу с заголовком. Каждому пациенту соответствует несколько строк. Строки состоят из нескольких блоков неструктурированного текста. В сценарии используются блок текста истории болезни, содержащий поставленный диагноз, и блок рекомендаций, содержащий перечисление назначенных лекарств.
На втором этапе из блоков сначала извлекаются фрагменты текста, после чего из фрагментов извлекаются интересующие термины – названия лекарств и диагнозы.
На третьем этапе выполняется связывание извлечённых терминов с понятиями онтологий. Используются как собственные, так и сторонние онтологии.
На четвёртом этапе формируется и выполняется SPARQL-запрос на поиск лекарств, которые могут вступить во взаимодействие.
На пятом этапе строится граф взаимодействия лекарств, предписанных одному конкретному пациенту.
Помимо перечисленных этапов введён предварительный этап, на котором строятся собственные онтологии.
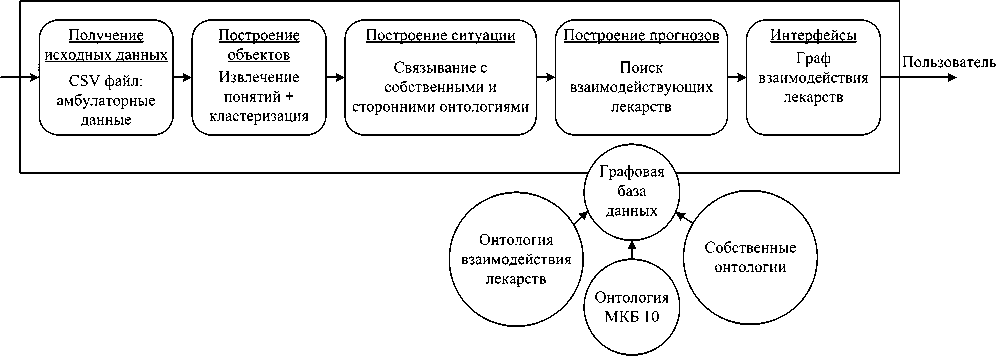
Рисунок 2 – Модель сценария слияния данных
-
3.2 Используемые инструменты
Для построения процесса слияния данных были использованы следующие инструменты:
OpenRefine 3 – приложение для преобразования данных из одного формата в другой. Данные представляются в форме электронных таблиц. Для работы с RDF устанавливается специальное расширение. Расширение позволяет проводить автоматическое связывание данных с элементами выбранной онтологии и экспорт данных в формате RDF. Инструмент используется на втором и третьем этапах.
Blazegraph 4 – графовая база данных. Поддерживает работу с RDF и SPARQL. Позволяет организовать SPARQL-endpoint – точку доступа для удалённого получения данных с помощью SPARQL запросов. Используется на всех этапах, начиная с третьего.
Metaphactory 5 – каркас для построения веб-приложений. Особенностью данного каркаса является формирование HTML-страниц на базе SPARQL-запросов. Каждая страница строится на основе фрагмента кода, включающего запрос и отображение результатов запроса на элементы графического интерфейса. Запросы выполняются к привязанной к каркасу базе Blazegraph через механизм SPARQL-endpoint. Используется на пятом этапе согласно рисунку 2.
TopBraid6 – редактор онтологий на основе платформы Eclipse7. Поддерживает логический вывод, выполнение SPARQL-запросов, совместную разработку онтологий. Используется на предварительном этапе для построения собственных онтологий.
Схема совместного использования инструментов представлена на рисунке 3.
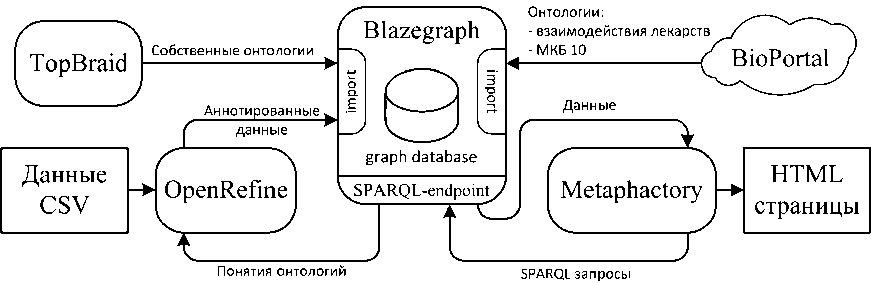
Рисунок 3 – Схема совместного использования инструментов
Перейдем к описанию отдельных этапов сценария.
-
3.3 Онтологии
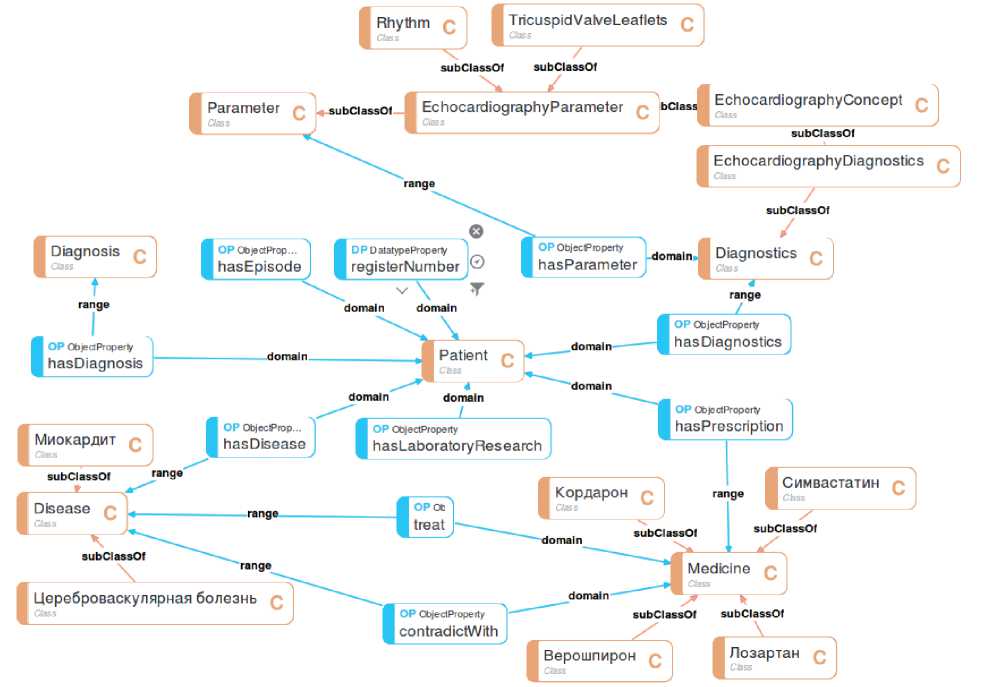
В основе предлагаемого подхода лежит использование онтологий. Используются как собственные онтологии, так и сторонние. Собственные онтологии представляют единую модель, которая позволяет объединить разрозненные данные и отдельные сторонние онтологии. Собственные онтологии организованы в иерархию (см. рисунок 4) и разделены на два множества – метаонтологии и конкретные онтологии. Метаонтологии содержат общие понятия. Конкретные онтологии содержат медицинские данные, аннотированные элементами метаонтологий. Такое разделение позволяет отделить данные от общих понятий и открыто распространять последние.
Онтология Hospital объединяет все метаонтологии и представляет собой онтологию для медицинского учреждения. Онтология Diagnostics содержит понятия для аннотирования результатов различных исследований. В частности, онтология Echocardiography расширяет онтологию Diagnostics понятиями, специфическими для эхокардиографического исследования. Онтология Disease включает понятия для описания болезней. Её расширяет онтология лекарств – MedicineTreatment .
На рисунке 5 представлен фрагмент, объединяющий несколько метаонтологий. Граф визуализирован с помощью средства визуализации онтологий Ontodia8. В качестве источника данных использован SPARQL-endpoint на основе базы Blazegraph. Отметим, что названия конкретных болезней и лекарств были извлечены из конкретных данных и включены в состав метаонтологии, так как они относятся к общим понятиям.
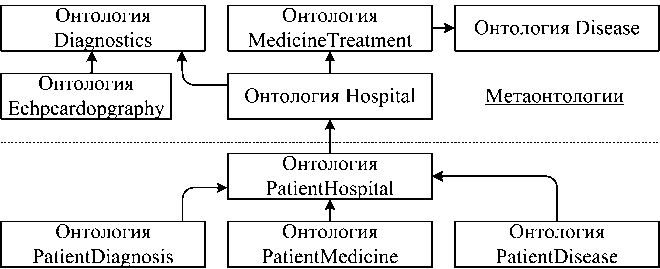
Рисунок 4 – Иерархия онтологий
domain
Patient domain lomaln domain
I TricuspidValveLeaflete p
Class
Rhythm Q
। EchocardiographyParameter p
T Class subClassOf ь Diagnostics q V Class
Я OP ObjectProp... hasEpisode domain domain
I OP ObjectProperty I hasDiagnosis j domain domain
( Кордарон p
Class I
Симвастатин q subClassOf subClassOf subClassOf
( Верошпирон q
Class
OP Ct treat
OP CbjectPrope rty contradictWith
OP CbjectProperty hasPrescription
OP ObjectProp... hasDisease
DP Datatypeproperty registerNumber
OP ObjectProperty has Di agnostics
OP CbjectProperty hasLaboratoryResearch domain I
Disease p
Class
Medicine p
Class
( Diagnosis
C/ass subClassOf subClassOf
( Лозартан q
C6ss subClassOf subClassOf
( Цереброваскулярная болезнь q Class
Parameter ^ subciassot
C/ass
Миокардит q subClassOf bciass EchocardiographyConcept q c/ass subClassOf
I EchocardiographyDiagnostics q Class
OP ObjectProperty
_ . -domalni hasParameter
Рисунок 5 – Фрагмент метаонтологии
В качестве сторонних онтологий были использованы онтология Международной классификации болезней9 в 10 редакции и Онтология взаимодействия лекарств10.
Упомянутые онтологии используется на следующих этапах:
-
■ на третьем уровне для аннотирования извлечённых терминов;
-
■ на четвёртом уровне для поиска возможных взаимодействий лекарств;
-
■ на пятом уровне для подготовки пользовательских представлений.
-
3.4 Извлечение терминов
На рисунке 6 представлены этапы извлечения названий отдельных лекарств из фрагмента текста. Каждый фрагмент содержит рекомендации для одного пациента в части медикаментозного лечения. В результате обработки фрагмента получаем список лекарств, которые ему назначены.
Медикаментозна я^ лечение
Бисопролол 5мг*1 раз утром Индапамид-оетаод 1,5м г”1 раз утром Валсартан (Валз, Валсакор)____ 160мг*2раза утром и веч Капли- аск1 00мг*1паз в день Миксонидин ( Физиотенз'!__ ),4мг*1 раз вечером Амлодипин : 10 мг однократно вечером Аторвастатин ( Аторис ) 10мг*1раз вечером, под контролем АЛТ, ACT, КФК, билирубин общ., холестерин общ, 1 раз в три месяца
а) Исходный фрагмент
Бисопролол Индапамид-ретард Валсартан
Валз
Валсакор Карди-аск Моксонидин Физиотенз
Амлодипин Аторвастатин Аторис
б) Извлеченные названия
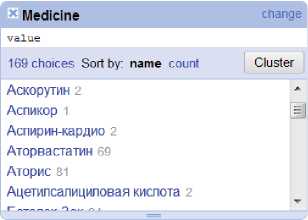
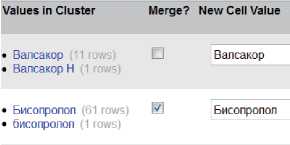
в) Словарь лекарств и унификация названий
Рисунок 6 – Извлечение названий лекарств

О Бисопролол
1 Индапамид-ретард
-
2 Валсартан
-
3 Валз
-
4 Валсакор
-
5 Карди-аск
-
6 Моксонидин
-
7 Физиотенз
-
8 Амлодипин
-
9 Аторвастатин
-
10 Аторис
-
3.5 Связывание извлечённых терминов с понятиями онтологий
г) Нумерация лекарств
На рисунке 6а представлен фрагмент текста, в котором выделяемые названия подчёркнуты красным. Названия извлекаются из текста с помощью операций на основе регулярных выражений, разделения и объединения данных (split, join) [13]. В результате получаем список – рисунок 6б. По извлечённым для всех пациентов названиям строится словарь лекарств – рисунок 6в (верхнее окно). Встроенные в OpenRefine методы кластеризации позволяют провести ограниченную унификацию элементов словаря – рисунок 6в (нижнее окно). В частности, унифицировать названия лекарств, написанных с прописной и строчной буквы. На последнем шаге (рисунок 6г), список назначенных лекарств нумеруется. В дальнейшем это позволит для каждого лекарства сформировать уникальный идентификатор назначения. Помимо названий лекарств из данных также извлекался основной диагноз. На данном этапе используется инструмент OpenRefine. Результатом выполнения операции в OpenRefine является либо модификация исходных столбцов, либо расширение исходной таблицы. Например, пронумерованный список лекарств по каждому пациенту сохранялся в новом столбце.
Уровень связывания данных включает два последовательных шага:
-
1) связывание отдельных понятий с понятиями из собственных и сторонних онтологий;
-
2) аннотирование данных понятиями собственных онтологий и экспорт конкретной онтологии, т.е. онтологии содержащей конкретные данные.
На этом этапе используется OpenRefine. Инструмент предлагает два механизма для связывания данных. Первый механизм – это механизм автоматического связывания (reconciling). Для его использования в OpenRefine регистрируется внешний источник данных (рисунок 7):
-
■ указывается адрес SPARQL-endpoint (онтология, с понятиями которой будут связываться данные, должна быть доступна по этому адресу);
-
■ выбираются и/или задаются свойства онтологии, которые будут просматриваться при связывании.
Add SPARQL-based reconciliation service
Name: D2D Interaction
A human readable name
Endpoint details
Endpoint URL |http://localhost:9999/blazegraph/namespace/drug-dr
Graph URI:
Leave empty to use the default graph
Type: Generic SPARQL (poor performance) ▼
This determines the syntax that will be used for search
Label properties
Select properties that are used to label resources in the endpoint These properties will be used to match resources:
@rdfs: label Eskos:prefLabel П determs: title Cdc:title
-
□ foaf :name
Bother...
Full URIs; separate multiple URIs with whitespace
Рисунок 7 – Регистрация внешнего источника данных
Далее для выбранного столбца данных запускается операция автоматического связывания. В ходе этого процесса инструмент пытается подобрать для каждого элемента столбца элемент из онтологии.
На рисунке 8 представлен фрагмент результата связывания названий лекарств, выделенных на предыдущем этапе, и понятий из Онтологии взаимодействия лекарств. Как видно, из трёх лекарств только для двух было найдено понятие в онтологии.
▼ Medicine name ▼ Medication english name ▼ LinkOnD2D Бисопролол Bisoprolol Choose new match /ObO/CHEBI_3127 Индапамид-ретард Indapamide retard 5 б- Create new topic Search for match Валсартан valsartan http://puri obolibrary org Choose new match /ObO/CHEBI 9927
Рисунок 8 – Фрагмент результата связывания названий лекарств
Для использования Онтологии взаимодействия лекарств русскоязычные названия извлечённых лекарств (точнее, торговых марок) были переведены на английский язык с помощью онлайн-справочника11. Для связывания извлечённых названий лекарств с понятиями онтологии использовались свойства онтологии, которые задают названия действующего вещества (DBname), торговых марок (DBBrand) и синонимы действующих веществ (DBSynonym) – см. рисунок 7, поле Other. Данный механизм также был использован для связывания выделенных диагнозов с элементами онтологии Международной классификации болезней.
Второй механизм позволяет привязывать данные к строго заданным понятиям онтологии. Для этого на основе выделенных терминов составляется SPARQL-запрос к SPARQL-endpoint, в которую предварительно загружается онтология. Полученный от сервиса результат в формате JSON-строки (JavaScript Object Notation) сохраняется в отдельном столбце. Встроенными в OpenRefine средствами синтаксического разбора из неё извлекаются нужные данные и помещаются в новый столбец.
На втором шаге данные аннотируются понятиями собственной онтологии. Для этого средствами OpenRefine строится схема (RDF Skeleton) отображения колонок с данными на понятия выбранной онтологии – рисунок 9. Данная схема используется для сохранения данных в формате RDF, иными словами, для аннотированных данных. Каждая строка такой схемы представляет собой тройку: субъект-предикат-объект. Например, в первой строке рисунка 9 указано, что регистрационный номер пациента « Рег.№ » представляет собой URI, который через свойство rdf:type связан с классом Patient онтологии Hospital .
RDF Skeleton RDF Preview
Available Prefixes: -df °W1 X5d rd£= medicine hospital leaf +add prefix ^manage prefixes
Per.№ URI add rdftype
X>- rdftype-> http://...#Patient .
В В add rdftype В
X>- Medicine index URI hospitakhasPrescriptionS add rdftype
X >-rdfs:label Medication english name cell
-+ В
X>- LinkOnD2D URI owksameAs» Q add rdftype
add properly
X>- http://...#Medicine
В rdfs subClassOfLJ add rdftype


Medicine name URI xowICIass add rdf type add property
Medicine index URI add rdf type
Add another root node
X>- Medicine name URI .
В rdftype □ add rdftype И
► add property
Рисунок 9 – Схема отображения данных на понятия онтологии
Для установления связи с онтологией взаимодействия лекарств формируется три строки схемы отображения (см. рисунок 9). Каждая строка описывает одну тройку. Для всех троек задан один общий субъект – OWL класс, идентификатор для которого строится по русскоязычному названию лекарства (см. рисунок 9-1):
-
1) В первой строке (рисунок 9-2) задаётся тройка, в которой указывается, что созданный класс лекарства является дочерним классом класса Medicine . Множество таких дочерних классов образует словарь лекарств, сформированный на базе конкретных данных. Словарь включается в метаонтологию MedicineTreatment .
-
2) Вторая строка задаёт англоязычное название для класса лекарства (рисунок 9-3). Используется отношение rdfs:label .
-
3) Третьей строкой задаётся тройка, которая привязывает класс лекарства к классам сторонней онтологии через отношение owl:sameAs (рисунок 9-4). Понятия сторонней онтологии после проведения автоматического связывания содержатся в отдельном столбце (см. рисунок 8).
-
3.6 Поиск взаимодействующих лекарств
Отображение формирует собственный словарь лекарств, часть элементов которого связана со сторонней онтологией.
На основе построенной схемы отображения генерируется онтология, которая содержит конкретные данные, проаннотированные понятиями выбранной метаонтологии.
Возможность поиска обеспечена привязкой назначенных пациенту лекарств к элементам Онтологии взаимодействия лекарств. В последней между отдельными лекарствами установлено отношение « may interact with ».
Результат запроса представлен на рисунке 10 в виде графа (регистрационный номер пациента закрашен), построенного с помощью каркаса Metaphactory. Для наглядности граф по- строен для одного пациента.
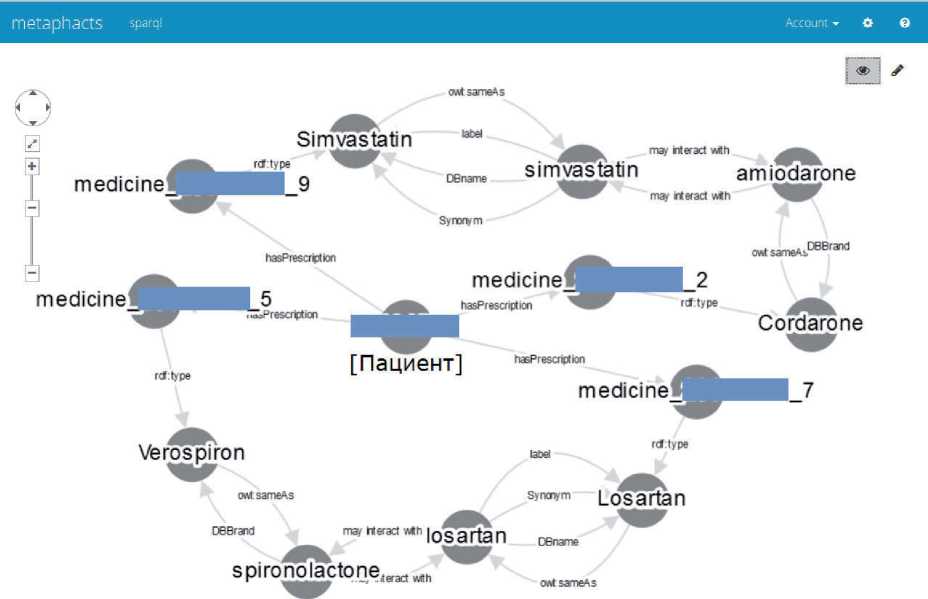
Рисунок 10 - Пример найденного возможного взаимодействия лекарств
Свойством hasPrescription пациент связан с объектом, представляющим собой назначение препарата. Для каждого объекта назначения задан класс лекарственного препарата, взятый из онтологии MedicineTreatment . Класс лекарственного препарата связан свойством owl:sameAs с элементом Онтологии взаимодействия лекарств.
В частности, установлено, что среди прописанных пациенту лекарств могут взаимодействовать две пары: Симвастатин (Simvastatin) – Кордарон (Cordarone) и Верошпирон (Verospiron) – Лозартан (Losartan). Стоит обратить внимание, что взаимодействие Верошпирона (торговая марка) установлено через действующее вещество Спиронолактон (Spironolactone). Как отмечено в п.3.5, это возможно благодаря тому, что в Онтологии взаимодействия лекарств для торговых марок указаны соответствующие действующие вещества.
Для отображения графа использовался Metaphactory. Граф строится с использованием специализированного скриптового языка. Фрагмент кода, описывающий структуру и отображение графа, включает две части:
-
■ SPARQL-запрос для выбора взаимодействующих лекарств, предписанных заданному пациенту;
-
■ описание, по которому результаты запроса отображаются на элементы графического ин
-
3.7 Представление данных пользователю
терфейса.
Кроме представленного в предыдущем разделе графа, возможны и иные графические представления. Например, на рисунке 11 представлена таблица пациентов, где каждому пациенту сопоставлен диагноз из Международного классификатора болезней.
metaphacts sparq Account- 6 О
<9> /
Filter Results
Patient Diagnosis Type Diagnosis Code Diagnosis Description
Patient Диагноз_выписки_основной <> Перенесенный в прошлом инфаркт миокарда Patient Ди а гн оз_уточн ен н ы й_осн овн ой < > Атеросклеротическая болезнь сердца Patient Диагноз_уточненный_основной <> Другие формы стенокардии Patient Диагноз_уточненный_основной <> Другие формы стенокардии Patient Диагноз_уточненный_основной <> Другие формы стенокардии Patient Диагноз_уточненный_основной < ОСМ/115.8> Другая вторичная гипертензия Patient Ди а гн оз_уточн е н н ы й_осн овн ой <> Перенесенный в прошлом инфаркт миокарда Patient Диагноз_уточненный_основной <> Фибрилляция и трепетание предсердий Patient Ди а гн оз_уточн е н н ы й_сопутству ющи й < > Предсердно-желудочковая блокада второй степени Patient Диагноз_уточненный_основной <> Другие формы стенокардии « 1 1 2 3 4 5 6 7 8 9 10 11 >
Рисунок 11 – Таблица диагнозов пациентов
На рисунке 12 представлена сводная диаграмма, отражающая частоту назначения отдельных лекарств в рамках отдельного учреждения (в объеме предоставленных данных).
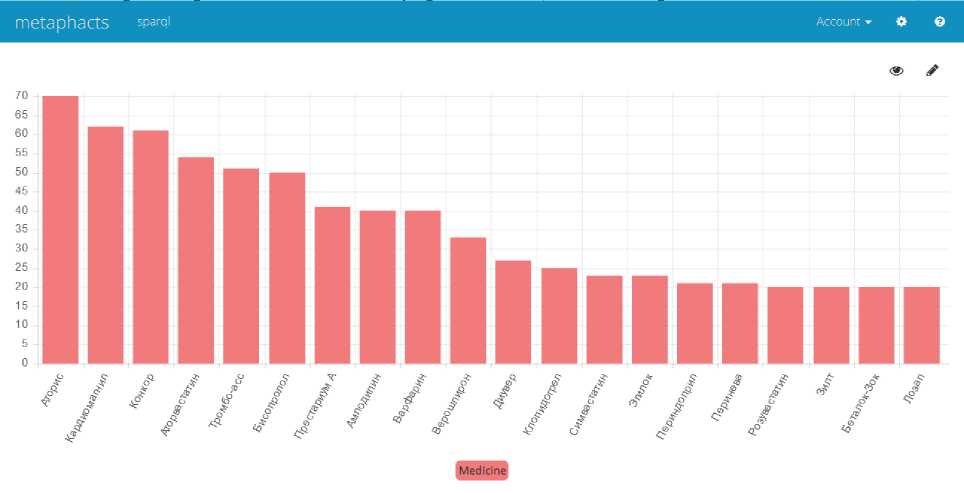
Рисунок 12 – Диаграмма частоты назначения лекарств
Заключение
Представлен подход к совместному использованию онтологий и JDL модели слияния данных. Подход проиллюстрирован на примере сценария поиска взаимодействующих лекарств. Онтологии позволили: формально описать фрагмент ПрО; аннотировать в терминах ПрО медицинские данные; связать данные между собой и со сторонними онтологиями; получить на основе связанных данных дополнительную информацию – пары взаимодействующих лекарств. Модель слияния данных использована в качестве общей схемы, определяющей уровни процесса обработки и их взаимосвязь.
Даны источники открытых медицинских онтологий и представлены варианты использования некоторых из них. Описаны варианты использования инструментов для подготовки данных и онтологий, хранения и отображения онтологий.
При реализации сценария выявлен ряд затруднений. Значительных усилий потребовало выделение названий лекарств, их унификация и перевод на английский язык. Использовать английские названия необходимо для совместимости со сторонними онтологиями. Кроме того, использование русскоязычных идентификаторов онтологических сущностей приводило к ошибкам в работе используемых инструментов.
Развитие представленного сценария может пойти по нескольким направлениям: автоматизация процесса слияния данных, анализ и включение дополнительных сторонних медицинских онтологий, расширение представленного сценария.
Предложенный подход является перспективной основой для реализации процессов слияния данных и знаний в рамках МИС.
Список литературы Слияние медицинских данных на основе онтологий
- Лушнов, А.М. Медицинские информационные системы: многомерный анализ медицинских и экологических данных / А.М. Лушнов, М.С. Лушнов. - Санкт-Петербург, «Геликон Плюс», 2013 - 460 с.
- Foo P.H., Ng G.W. High-level Information Fusion: An Overview // J. Adv. Inf. Fusion. - 2013. - Vol. 8. - №. 1. - С. 33-72.
- Steinberg A.N., Bowman C.L. Revisions to the JDL Data Fusion Model / Llinas J., Hall D.L., Liggins M.E. (ed.). Handbook of Multisensor Data Fusion: Theory and Practice. - CRC Press, 2009. - P. 44-67.
- Bowman C.L., Steinberg A.N. Systems Engineering Approach for Implementing Data Fusion Systems / Llinas J., Hall D. L., Liggins M. E. (ed.). Handbook of Multisensor Data Fusion: Theory and Practice. - CRC Press, 2009. - P. 561-596.
- Lushnov, M. Medical knowledge representation for evaluation of patient's state using complex indicators / M. Lushnov, V. Kudashov, A. Vodyaho, M. Lapaev, N. Zhukova, D. Korobov // International Conference on Knowledge Engineering and the Semantic Web. - Springer International Publishing, 2016. - P. 344-359.


