Сравнительный анализ генных карт Bos taurus L. и Capra hircus L
Автор: Эрнст Л.К., Кленовицкий П.М., Багиров В.А., Иолчиев Б.С., Зиновьева Н.А., Калашников В.В., Фисинин В.И., Жилинский М.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генные карты и молекулярная структура генома
Статья в выпуске: 2 т.48, 2013 года.
Бесплатный доступ
С использованием банка данных по генетическим картам животных и разработанного программного обеспечения проведено сравнение генных порядков у двух представителей семейства Bovidae — Bos taurus L. и Capra hircus L. Показано высокое сходство генных порядков гомеологичных хромосом у сравниваемых видов, за исключением хромосом 9-й и 14-й пар.
Дивергенция, геном, генетические карты, гомеология, кариотип, хромосомы, хромосомные карты
Короткий адрес: https://sciup.org/142133395
IDR: 142133395 | УДК: 636.082.12:599.735.51/.52:575.1:576.3
Текст научной статьи Сравнительный анализ генных карт Bos taurus L. и Capra hircus L
Изучение филогенеза животных невозможно без углубленного использования достижений современной биологии, в частности знаний об особенностях организации и функционирования хромосомного аппарата. В решении вопросов систематики и филогенеза млекопитающих большую роль играют цитогенетические методы (1-3). В последние десятилетия изучение хромосомных наборов применяется и в прикладных исследованиях (4, 5), в том числе в работах по сохранению и рациональному использованию генофонда животных (6-10). В эволюционной генетике нашел широкое приложение анализ тонкой структуры хромосом. Одним из подходов при изучении гомеологии хромосом, а также их отдельных участков у различных видов млекопитающих служит сравнение дифференциальной ис-черченности.
Ряд работ посвящен анализу цитогенетического сходства и эволюции кариотипов (11-13). Как отмечают А.С. Графодатский и Л.С. Битуева (12), гомеологичные участки хромосом идентифицируются по характеру рисунка тем чаще и надежнее, чем меньше сравниваемые кариотипы перестроены относительно друг друга. За редким исключением гомеология хорошо прослеживается при сравнении видов внутри рода или близких родов. Иногда удается с достаточной степенью надежности идентифицировать ее в пределах семейства.
В последнее время для оценки сходства геномов широко используются различные молекулярно-генетические маркеры (14-19). Однако наиболее точным критерием сходства (тождественности) геномов может служить лишь тот факт, что хромосомы или их фрагменты содержат одинаковые нуклеотидные последовательности, расположенные в одном и том же порядке, независимо от аллелизма.
Имеются исследования, посвященные сравнению генного состава отдельных хромосом у разных видов (20-28), также известны работы по анализу филогенеза на основе сопоставления генных карт (29-32). При сравнении дифференциальной структуры хромосом выявлены пути цитогенетической дивергенции у разных видов овец (33-37). Изучение генных порядков у Bos taurus L. и Ovis aries L. позволило более детально проанализировать характер дивергенции их кариотипов (32). Однако во всех этих работах объектом исследованиях были виды с выраженными цитогенети- ческими различиями.
В представляемой работе нами проведено сравнение генных карт у двух родственных видов из семейства Bovidae — Bos taurus L. и Capra hircus L., имеющих сходные хромосомные наборы, с целью поиска различий в организации геномов у этих таксонов на уровне генных порядков.
Методика . Исследования выполняли с привлечением собственного банка данных о хромосомной локализации маркеров у B . taurus L. и C . hircus L., включающего обобщенную информацию из баз данных NAGRP (National Animal Genome Research Program, США) (38), INRA (39, 40) и содержащего сведения о локализации соответственно 28 282 и 708 маркеров. При этом для 249 маркеров известна хромосомная локализация у обоих видов. Кроме того, использовали материалы A. Eggen и R. Fries (41), J.J. Lauvergne с соавт. (42), J. Maurico с соавт. (43) и Bovine genome DataBase (44). Анализ генных карт проводили по алгоритмам, описанным нами ранее (28, 29).
Для цитогенетической характеристики сравниваемых видов из 72часовой культуры периферических лимфоцитов, стимулированной конка-навалином А («Панэко», Россия) в дозе 10 мкг/мл, готовили препараты хромосом. Культивирование лимфоцитов, подготовку и обработку препаратов хромосом проводили по общепринятой методике (45) с внесенными нами модификациями (46).
Результаты исследования документировали с помощью цифровой видеокамеры КС-583С («Digital», Tawan) и пакета программ, совместимых с Windows. Обработку изображений и кариотипирование осуществляли по описанной ранее методике (46).
Результаты . Для видов, образующих семейство Полорогие ( Bovidae ), характерна высокая цитогенетическая вариабельность. Полагают, что исходный кариотип полорогих содержал 60 хромосом, включая 29 пар одноплечих аутосом. Расхождение ветвей, давших начало современным представителям этого семейства, произошло 15-20 млн лет назад. На основании сопоставления хромосомных наборов у различных представителей полорогих принято считать, что эволюция кариотипов в этом семействе происходила главным образом в результате центрических слияний (47). Изменение морфологии половых хромосом у полорогих связывают с перицен-трическими инверсиями.
Из современных полорогих к предковой форме кариологически наиболее близки представители рода Capra , обладающие хромосомной мо-номорфностью (3, 45, 48, 49). Среди них наиболее полно изучен кариотип у C . hirous (50-53). Диплоидное число хромосом у козы равно 60. Кариотип этого вида включает 29 пар акроцентрических аутосом. X-хромосома представляет собой крупный акроцентрик, тогда как Y-хромосома мета-центрична и относится к наиболее мелким в наборе (рис. 1, А). Точная идентификация хромосом, за исключением Y-хромосомы, возможна лишь с применение дифференциального окрашивания. Из одомашненных представителей полорогих по числу и морфологии хромосом к C . hirous наиболее близок B . taurus (см. рис. 1, Б).
Диплоидное число у обоих видов равно 60, а число плеч аутосом — 58. Характер рисунка гомологичных хромосом, исключая 9-ю и 14-ю пары, у обоих видов идентичен (4, 5). У B. taurus и C. hirous для большинства хромосом характерно относительно равномерное чередование светлых и темных блоков по всей длине. Морфологические различия имеют место в организации женских половых хромосом: у B. taurus Х-хромосома — крупный метацентрик. Различие в морфологии женских хромосом у этих видов обусловлено перицентрической инверсией (54).
А

Рис. 1. Кариотип у Capra hircus L. (самец) (А) и Bos taurus L. (самец) (Б). Окраска по Гимзе, увеличение *100.
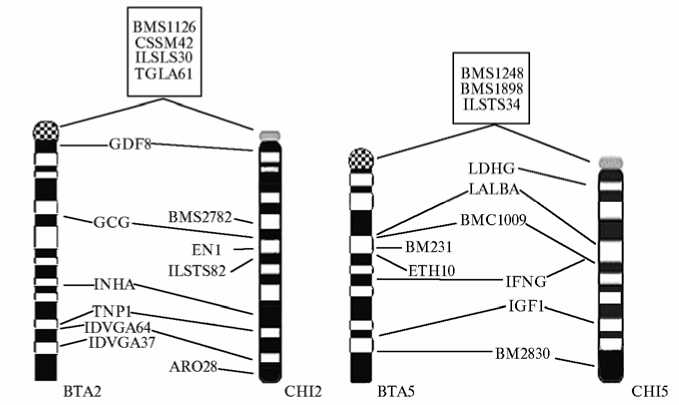
В таблице представлены данные о соответствии генного состава хромосом B. taurus и C. hircus., в частности сведения 95 маркеров первого и 154 маркеров второго типа. Для 22 пар хромосом у анализируемых видов отмечалось полное совпадение их генного состава. В эту группу входят хромосомы 2-5-й, 9-13-й, 15-19-й, 21-25-й, 28-й, 29-й пар и половые хро- мосомы. В качестве примера на рисунке 2, А приведены генные карты 2-й и 5-й пар хромосом крупного рогатого скота и козы.
Соответствие генного состава хромосом у Capra hircus L. (самец) и Bos taurus L. (самец)
|
C . hircus L. |
B . taurus L. |
|
|
№ пары хромосом | |
плечо |
№ пары хромосом и число генов |
|
1-я |
q |
1q (23: 9, 14); 16q (1: 0, 1) |
|
2-я |
q |
2q (14: 6, 8) |
|
3-я |
q |
3q (10: 2, 8) |
|
4-я |
q |
4q (9: 3, 6) |
|
5-я |
q |
5q (11: 4, 7) |
|
6-я |
q |
6q (5: 3, 2) |
|
7-я |
q |
7q (5: 3, 2) |
|
8-я |
q |
8q (7: 2, 5); 11q (1: 0, 1) |
|
9-я |
q |
9q (6: 1, 5) |
|
10-я |
q |
10q (10: 4, 6) |
|
11-я |
q |
11q (16: 5, 11) |
|
12-я |
q |
12q (9: 3, 6) |
|
13-я |
q |
13q (6: 3, 3) |
|
14-я |
q |
9q (3: 0, 3); 14q (8: 3, 5) |
|
15-я |
q |
15q (7: 3, 4) |
|
16-я |
q |
16q (9: 1, 8) |
|
17-я |
q |
17q (7: 2, 5) |
|
18-я |
q |
18q (10: 6, 4) |
|
19-я |
q |
19q (4: 1, 3) |
|
20-я |
q |
1q (1: 1, 0); 9q (1: 0, 1); 20 (6: 0, 6) |
|
21-я |
q |
21q (6: 2, 4) |
|
22-я |
q |
7q (1: 0, 1); 22q (7: 3, 4) |
|
23-я |
q |
23q (5: 2, 3) |
|
24-я |
q |
24q (6: 0, 6) |
|
25-я |
q |
25q (8: 2, 6) |
|
26-я |
q |
14q (1: 0, 1); 26q (4: 1, 3) |
|
27-я |
q |
17q (1: 1, 0); 26q (1: 0, 1); 27q (4: 0, 4) |
|
28-я |
q |
28q (5: 0, 5) |
|
29-я |
q |
29q (4: 0, 4) |
|
X |
X (5: 3, 2) |
|
|
Y |
Y (1: 1, 0) |
|
Примечание. В скобках приведено общее число локусов, известных у C . hircus L., в том числе соответственно структурных генов и ДНК-маркеров.
Необходимо отметить, что для части использованных в представляемой работе генетических маркеров, общих для крупного рогатого скота и козы, точная локализация на хромосомах пока не определена.
По 7 хромосомным парам наблюдали разную степень несоответствия генного состава у C . hircus и B. taurus . Наиболее значительные разли-
А
Б
В
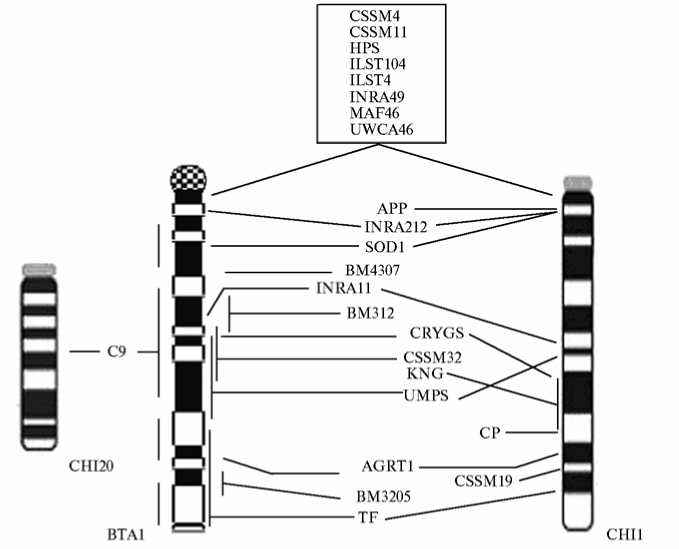
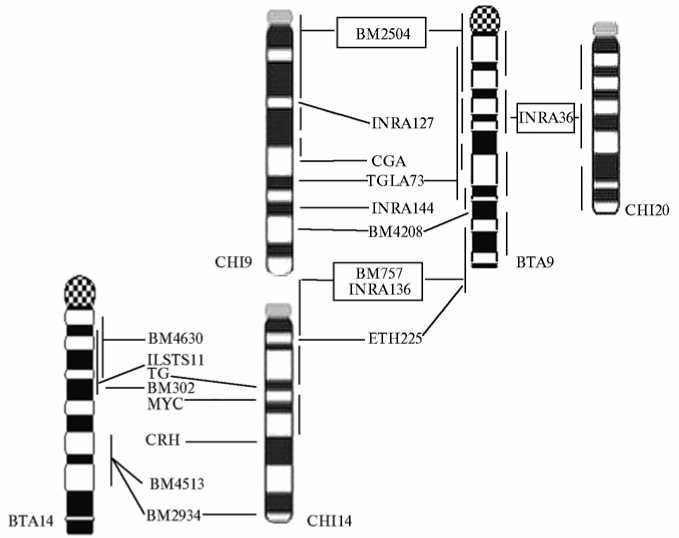
Рис. 2. Сравнение генных карт 2-й и 5-й пар хромосом у Capra hircus L. (CHI2, CHI5) и Bos taurus L. (BTA2, BTA5) (А) , 9-й и 14-й пары у C . hircus (CHI9, CHI14) и B . taurus (BTA9, BTA14) (Б) , а также 1-й пары у C . hircus (CHI1) и B . taurus (BTA1) (В). В рамке приведены обозначения генов, точная локализация которых у обоих видов не известна. Ген С9 и маркер INRA36 локализованы на 20-й хромосоме козы.
чия генных порядков у этих видов выявили между хромосомами 14-й пары (см. рис. 2, Б).
Оказалось, что из 11 генетических маркеров на хромосоме 14-й пары у козы 8 расположены на BTA14 и 3 — на BTA9. У B. taurus хромосо- ма 9-й пары содержит 10 маркеров, локализация которых известна и у C. hircus, из них 6 находятся на CHI9, 3 — на CHI14 и 1 — на CHI20. К сожалению, точная локализация генетических маркеров на BTA9 не выявлена, что пока не позволяет определить характер изменения генных порядков в этих хромосомах. Дополнительно ситуация осложняется тем, что для BTA14 в настоящее время неизвестны реперные гены, локализованные вне CHI14.
Хромосома 1-й пары козы по генному составу соответствует BTA1. Но помимо маркеров, расположенных на CHI1, на BTA1 был выявлен ген 9-го компонента комплемента ( С9 ), находящийся у козы на хромосоме 20-й пары (см. рис. 2, Б).
Хромосомы 8-й и 22-й пар у крупного рогатого скота по большинству маркеров соответствовали CHI8 и CHI22. Но на CHI8 находился также ДНК-маркер HEL13, локализованный у B . taurus на BTA11, а на CHI22 выявили микросателлитный маркер BM7160, сцепленный с BTA7.
По аналогии при сравнении генных порядков в хромосомах 26-й пары было установлено, что CHI26 и BTA26 имеют 4 общих маркера. Помимо этого, на BTA26 присутствовал маркер CSSM43, ассоциированный с CHI27, а на CHI26 — BM4305, сцепленный с BTA14.
Из 8 маркеров, обнаруженных на хромосоме 20-й пары у козы, 6 присутствовали на BTA20, а маркеры C9 и INRA36 имелись соответственно на BTA1 и BTA9. У козы с хромосомой 27-й пары были ассоциированы 6 маркеров, у B . taurus 4 из них локализовались на BTA27, а ген фактора свертываемости 11-F11 и микросателлит CSSM43 — соответственно на хромосомах 17-й и 26-й пар.
Таким образом, у сравниваемых видов организация генных порядков характеризуется высокой степенью сходства. Однако в процессе дивергенции их кариотипов имел место обмен генетическим материалом между негомологичными хромосомами. Классический цитогенетический анализ не позволяет вскрыть все тонкости этого процесса, в связи с чем даже при сравнении родственных видов необходимо изучать организацию их геномов на уровне генных карт.


