Сравнительный анализ последовательностей генов амидаз почвенных актинобактерий рода Rhodococcus
Автор: Павлова Юлия Андреевна, Неустроева Анна Николаевна, Максимов Александр Юрьевич
Журнал: Известия Самарского научного центра Российской академии наук @izvestiya-ssc
Рубрика: Генетика
Статья в выпуске: 5-3 т.13, 2011 года.
Бесплатный доступ
Исследованы почвенные культуры актинобактерий рода Rhodococcus, обладающие термостабильной амидазной активностью. Проведены ПЦР-анализ и секвенирование генов амидаз. Выявлены последовательности, гомологичные известным генам амидаз из R. erythropolis (GenBank, M88614), R. erythropolis (GenBank, E12517) и R. rhodochrous N774.
Короткий адрес: https://sciup.org/148200465
IDR: 148200465 | УДК: 579.26:579.222
Текст научной статьи Сравнительный анализ последовательностей генов амидаз почвенных актинобактерий рода Rhodococcus
Кроме способности к гидролизу амидов амидазы обладают также ацилтрансферазной активностью. Некоторые из этих ферментов проявляют стереоселективность по отношению к хиральным субстратам, например, к производным арилпропионовой кислоты. Бактерии, обладающие высокой амидазной активностью, представляют интерес для биокаталитического получения различных карбоновых кислот, в частности, аммонийных солей акриловой и никотиновой кислот, нестероидных противовоспалительных препаратов [ 0, 0] .
Амидазы, выделенные из различных источников, характеризуются различной субстратной специфичностью [7]. Известные амидазы подразделяются на четыре различные структурные группы [ 0] : I – cигнатурные ферменты, содержащие в первичной структуре GGSS–мотив (глицин-глицин-серин-серин); II - нитрилазы/цианидгидратазы; III – ацил-трансферазы; IV – уреазы [ 0] . Установлено, что даже у бактерий одного и того же вида встречаются структурно не родственные амидазы.
Данная работа посвящена исследованию анализу генов стереоселективных амидаз почвенных микроорганизмов, обладающих высокой амидазной активностью.
МАТЕРИАЛ И МЕТОДЫ
Объекты исследования - выделенные из почв штаммы актинобактерий Rhodococcus erythropolis и R. rhodochrous , активно трансформирующие амиды карбоновых кислот. Культуры изолировали из почвы методом прямого высева и выращивали на минеральной безазотной солевой среде N, содержащей ацетамид в концентрации 10 мМ. Трансформацию амидов и нитрилов с использованием активной биомассы проводили в 1 мл 10 мМ калий-натрий фосфатного буфера, pH 7.2, при начальной концентрации субстрата 2% параллельно при 25 и 50°С в течение 10 мин. Реакцию останавливали добавлением конц. HCl до концентрации 2%. Пробы центрифугировали 5 мин при 12 тыс об/мин. Продукты реакции анализировали методами ГХ (Shimatzu GC-20) и ВЭЖХ (Shimatzu LC-10A). В качестве стандартов использовали растворы чистых нитрилов, амидов и карбоновых кислот. Удельную активность амидазы выражали единицах (Ед), соответствующих 1 мкмоль продукта реакции (кислоты), образуемого за 1 мин, в пересчете на 1 мг веса сухих клеток (мкмоль/мг/мин). Хромосомную ДНК получали фенольным методом, модифицированным для выделения ДНК из акти-номицетов [ 0] . Для полимеразной цепной реакции (ПЦР) генов амидаз и секвенирования соответствующих ПЦР-фрагментов были разработаны праймеры, комплементарные концевым участкам известных последовательностей генов амидаз, депонированных в базе данных GenBank (табл. 1). Амплификацию ДНК проводили с применением термостабильной Taq -SE ДНК-полимеразы (СибЭн-зим, Новосибирск) на термоциклере Т3 (Biometra, Германия). Режим амплификации включал начальный цикл денатурации – 1 мин при 94°С; денатурацию, 94°С – 40 с; отжиг, 55-63° – 30 с; элонгацию, 72°С – 60 с; (35 циклов) и завершающий этап – 60 с при 72°С. Секвенирование последовательностей ПЦР-фрагментов проводили на приборе MagaBace1000 (GE Healthcare) с использованием стандартных наборов в соответствии с инструкцией производителя. Сравнение последовательностей проводили с помощью программы ClustalW.
Таблица 1. Праймеры для амплификации генов амидазы
|
Выявляемый тип гена |
Праймер |
Последовательность |
|
амидаза, гомологичная ферментам из R. erythropolis (E12517) и R. rhodochrous N-774 (X54074) |
AmiRhd-1 AmiRhd-2 |
5’-ATGGCGACAATCCGACCTGACGACA 5’-CTAGGCGGGGCTGAGTTGTGGTGCAGA |
|
амидаза, гомологичная ферменту из R. erythropolis (M88714) |
AmiReR-1 AmiReR-2 |
5’-ATGCGACACGGTGACATCTCCTCGA 5’-TTACGCTTCGACGGTCTTCTCGAC |
|
амидаза, гомологичная ферменту из R. erythropolis (AY026386) |
AmiReX-1 AmiReX-2 |
5’-GTGCGACCCAATCGCCCATTCGGCC 5’-CTACCGCAGCACCGGTGCGCTCGG |
|
амидаза, гомологичная ферментам из R. rhodochrous J1 (S38270) Rhodococcus sp. (BD061400) |
AmiJ1-1 AmiJ1-2 |
5’-ATGTCTTCGTTGACTCCCCCCAATT 5’-TTATGTCAGGGTGCCGGCTGCAGC |
|
AmiBD-2 |
5’-TCAGGACGGCACCGAGGGTCGCGG |
|
|
амидаза, гомологичная ферменту Rhodococcus sp. (A19131) |
AmiRsp-1 AmiRsp-2 |
5’-ATGGGCTTGCATGAACTGACGCTCG 5’-TCAAAGCGGCGCCAGTCGCGGCCA |
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Проведён скрининг образцов подзолистых супесчаных почв, собранных в районах г. Березников, г. Соликамска и г. Перми Пермского края. В результате скрининга нами выделено более 400 культур грамположительных бактерий, активно метаболизирующих амиды и нитрилы, из них 38, обладающих амидазной активностью более 5
мкмоль/мг/мин. Основная часть активных изолятов была представлена актинобактериями, в основном относящимися к роду Rhodococcus . Методами по-лифазной таксономии изоляты идентифицированы как представители рода Rhodococcus : R. erythropolis, R. rhodochrous, R. ruber [ 0] . Их видовая принадлежность была подтверждена методом ПЦР генов 16S рРНК с видоспецифическими праймерами (рис. 1).

Рис. 1. Идентификация штаммов R. erythropolis по генам 16S РНК. R. rhodochrous ИЭГМ7т и R. erythropolis ИЭГМ62т – типовые штаммы соответствующих видов.
Показано, что большинство амидаз изолятов Rhodococcus высокоактивны при повышенной температуре (50°С), что свидетельствует также об их технологической стабильности и является положительным признаком для биотехнологического применения.
Установлено, что ферменты исследуемых культур способны активно трансформировать большой ряд амидосоединений, включая алифатические, разветвленные, непредельные, гидроксилированные и некоторые ароматические амиды [0]. Наилучшими субстратами для ферментов большинства штаммов являются ацетамид и пропионамид (наиболее простые по структуре и легко метаболизируемые субстраты). Однако некоторые штаммы, селекционированные на изобутиронитриле и изо- бутирамиде предпочитали именно эти субстраты с пространственно затрудненным доступом к амидной группе. Известно, что штаммы, выделенные методом обогащенной культуры, часто проявляют высокую конвертирующую способность именно к тому субстрату, который использовался для селекции. Никотинамид и бензамид гидролизовались с меньшей скоростью.
В ходе селекционного процесса при постепенным повышении концентрации субстрата (нитрила) выделены культуры R. erythropolis 4-1-6, 4-1-6 6-21, 11-2, 11-1-3 и R. rhodochrous 11-8, 11-85, проявляющие амидазную активность более 12 Ед. при 25°С и более 30 Ед. при 50°С, а также активность нитрилгидратазы более 10°С.
Проведён молекулярный анализ генов амидаз. Показано, что геномы изолятов Rhodococcus содержат преимущественно последовательности, родственные ранее известным генам амидаз двух
структурных групп: I – энантиоселективные амидазы, как у R. erythropolis (GenBank, E12517), R. rhodochrous N-771, (GenBank, X54074) и II – алифатические амидазы, как у R. erythropolis (GenBank, M88614).
Гены других видов амидаз встречались со значительно меньшей частотой. Последовательности группы I, были обнаружены у отдельных изолятов R. erythropolis и R. rhodochrous. Другие культуры тех же видов содержали другие ферменты. Результаты ПЦР-анализа подтвердили, что наличие амидазы определённого типа не является видоспецифическим признаком.
Большинство штаммов также содержали последовательности, родственные генам α- и β-субъединиц Fe-содержащей нитрилгидратазы штаммов R. rhodochrous N-774, Rhodococcus sp. R312.
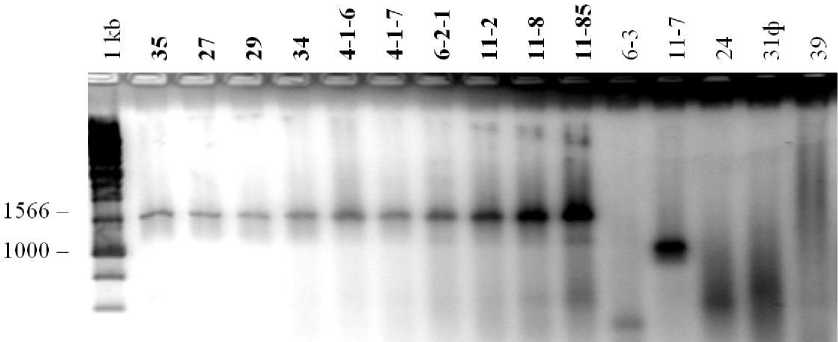
Рис. 2. ПЦР-анализ последовательностей, гомологичных генам амидазы R. erythropolis , GenBank, E12517, у почвенных изолятов Rhodococcus.
Проведено секвенирование полученных ПЦР-фрагментов с помощью ДНК-секвенатирующей системы MegaBase1000. Установлено, что гомоло- гия исследуемых генов почвенных изолятов известной последовательности E12517 составляет от 97,83 до 100% (табл. 2).
Таблица 2. Процент гомологии генов амидаз группы I
|
штамм |
E12517 |
4-1-6 |
4-1-7 |
11-8 |
6-2-1 |
11-1-3/4 |
|
R. erythropolis E12517 |
100,00% |
98,40% |
99,10% |
99,17% |
99,23% |
98,00% |
|
R. erythropolis 4-1-6 |
100,00% |
99,29% |
99,29% |
99,17% |
97,83% |
|
|
R. erythropolis 4-1-7 |
100,00% |
100,00% |
99,90% |
98,53% |
||
|
R. rhodochrous 11-8 |
100,00% |
99,87% |
98,53% |
|||
|
R. erythropolis 6-2-1 |
100,00% |
98,59% |
||||
|
R. erythropolis 11-1-3/4 |
100,00% |
Высокая степень гомологии исследованных последовательностей ДНК из различных источников свидетельствует о высокой консервативности структуры генов и соответствующих ферментов.
При переводе нуклеотидной последовательности в аминокислотную установлено, что у исследуемых штаммов есть ряд нуклеотидных замен по сравнению с ранее известными последовательностями, что, вероятно, обуславливает ранее установ- ленные различия субстратной специфичности ферментов [0, 0].
Притом сходство соответствующих аминокислотных последовательностей составило от 99,7 до 100%, т.е. ряд замен нуклеотидов являются синонимическими, не приводят к заменам аминокислот (рис. 3).
Таким образом, установлено, что среди культур, обладающих высоким уровнем амидазной активно- сти, преобладают представители рода Rhodococcus. Среди почвенных изолятов Rhodococcus, обладающих высокой амидазной активностью, наиболее распространены гены амидаз двух типов, гомологичные последовательностям из R. erythropolis, GenBank, E12517 и R. erythropolis, GenBank, M88614. Гены ранее известного структурного типа энантиоселективных амидаз гомологичных последо- вательности E12517, широко представлены у родо-кокков, обитающих в почве естественной среды. Показано, что гены исследуемого структурного типа амидаз встречаются у изолятов разных видов Rhodococcus и обладают высокой степенью консервативности, но при этом не являются обязательным элементом генома.
Translation_ll-l-34_gene Transi ation_ll-8_gene
Transi ation_62l_gene Translation_4-l-7_gene Ttansi ation_4-l-6_gene Translation_ll-2_gene Translation_E12S17_gene
Усредненная
Transi ation_ll-l-34_gene Translation 11-8 gene
Transi ation_62l_gene2 Translat ion_4-l-7_gene Transi at ion_4-l-6_gene Tr ansi ation_l1-2_gene Translation_E12517_gene
Усредненная
Transi ation_ll-l-34_gene Translation_ll-8_gene Translation 621 gene Translation 4-1-7 gene Translation 4-1-6 gene Translation 11-2 gene
Transi ation_El2517_gene
Усредненная
Transiation_ll-l-34_gene Translation_ll-8_gene Translation_621_gene2 Translation_4-l-7_gene Translation_4-l-6_gene Translation 11-2 gene Translation E12S17 gene
Усредненная
HATIRPDDMAIDTAARHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD HATIRPDDMAIDTAARHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD HATIRPDDMAIDTAARHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD HATIRPDDMAIDTAARHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD HATIRPDDMAIDTAARHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD HATIRPDDMAIDTAARHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD HATIRPDDMAIDTAAKHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD
HATIRPDDMAIDTAARHYGITLDQSARLEWPALIDGALGSYDVVDQLYAD
EATPPTTSREHy1 eatppttsrehR1 eatppttsrehR1 eatrfttsrehS1 EATPPTTSREhK' eatppttsrehJ EATPPTTSREH^
ENPLSAWYVTTSI PPTSDGVLTGRRVAI K* NVT
ENPLSAWYVTTSIPPTSDGVLTGRRVAIK? NVT
gENPLSAWYVTTSIPPTSDGVLTGRRVAIKjNVT gENPLSAWYVTTSIPPTSDGVLTGRRVAIKjNVT LENPLSAWYVTTSIPPTSDGVLTGRRVAIК*Г"~
NVT
5 ENPLSAWYVTTSIPPTSDGVLTGRRVAIK?
NVT
AQENPLSAWYVTTSIPPTSDGVLTGRRVAIКЦИУТ
EATPPTTSREHTVPTASENPLSAWYVTTSIPPTSDGVLTGRRVAIKDNVT
VAGVPHHNGSRTVEGFTPSRDATVVTRLLAAGATVAGKAVCEDLCESGSS VAGVPMMNGSRTVEGFTPSRDATVVTRLLAAGATVAGKAVCEDLCFSGSS VAGVPMMNGSRTVEGFTPSRDATVVTRLLAAGATVAGKAVCEDLCFSGSS VAGVPMMNGSRTVEGFTPSRDATVVTRLLAAGATVAGKAVCEDLCFSGSS VAGVPiniNGSRTVEGFTPSRDATVVTRLLAAGATVAGKAVCEDLCFSGSS VAGVPHHNGSRTVEGFTPSRDATVVTRLLAAGATVAGKAVCEDLCFSGSS VAGVPHMNGSRTVEGFTPSRDATVITRLLAAGATVAGKAVCEDLCFSGSS
VAGVPMMNGSRTVEGFTPSRDATVVTRLLAAGATVAGKAVCEDLCFSGSS
FTPASGPVRMPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA FTPASGPVRMPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA FTPASGPVRNPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA FTPASGPVRMPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA FTPASGPVRMPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA FTPASGPVRNPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA FTPASGPVRNPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA
FTPASGPVRNPWDPQREAGGSSGGSAALVANGDVDFAIGGDQGGSIRIPA
Рис. 3. Сравнение полных белковых последовательностей амидаз Rhodococcus.
Работа поддержана программой Президиума РАН «Молекулярная и клеточная биология», ФЦП «Научные и научно-педагогические кадры инновационной России», 2009–2013 гг., шифр 2009-1.1201-018-001.
-
1. Перцович С.И. и др. Алифатическая амидаза из Rhodococcus rhodochrous – представитель семейства нитрилаз/цианидгидратаз // Биохимия. 2005. Т. 70. С. 1556-1565.
-
2. Максимов А.Ю. и др. Влияние нитрилов и амидов на рост и нитрилгидратазную активность штамма
Rhodococcus sp. gt1 // Прикл. биохимия и микробиология. 2003. Т. 39. № 1. С. 63-68.
-
3. Клонирование ДНК. Методы. М., 1988. 538 с.
-
4. Демаков В.А. и др. Почвенные актинобактерии рода Rhodococcus , обладающие высокой амидазной активностью // Вестник Пермского ун-та. 2009. С. 79-83.
-
5. Кузнецова М.В. и др. Распространение нитрилконвертирующих бактерий в почвах Пермского края // Вестник Пермского ун-та. 2007. Т. 5. С. 96-99.
-
6. Asano Y. Overview of screening for new microbial catalysts and their uses in organic synthesis - selection and optimization of biocatalysts // J. Biotechnol. 2002. V. 94. P. 65-72.
-
7. Banerjee A., Sharma R., Banerjee U.C. The nitriledegrading enzymes: current status and future prospects // Appl. Microbiol. Biotechnol. 2002. V. 60. P. 33-44.
-
8. Cowan D. Biochemistry and biotechnology of mesophilic and thermophilic nitrile metabolizing enzymes // Extremo-philes. 1998. V. 2. № 3. P. 207-216.
-
9. Fornaud D., Arnaud A. Aliphatic and enantioselective amidases: from hydrolysis to acyl transfer activity // Appl. Microbiol. 2001. V. 91. № 9. P. 381-393.
-
10. Kotlova E.K. et al. Isolation and primary characterization of an amidase from Rhodococcus rhodochrous // Biochemistry. 1999. V. 64. N. 4. P. 384-389.
COMPARATIVE ANALYSIS OF GENE SEQUENCES OF AMIDASES OF SOIL ACTINOBACTERIA RHODOCOCCUS
-
1Perm State University, Perm
-
2Institute of Ecology and Genetics of Microorganisms, Ural Branch of RAS, Perm
The soil culture of Rhodococcus actinobacteria with thermostable amidase activity have been investigated. PCR and sequencing of amidase genes were performed. The homologous sequences that are to known genes of the amidase of R. erythropolis (GenBank, M88614), R. erythropolis (GenBank, E12517) and R. rhodochrous N774 were identified.
Список литературы Сравнительный анализ последовательностей генов амидаз почвенных актинобактерий рода Rhodococcus
- Перцович С.И. и др. Алифатическая амидаза из Rhodococcus rhodochrous -представитель семейства нитрилаз/цианидгидратаз//Биохимия. 2005. Т. 70. С. 1556-1565.
- Максимов А.Ю. и др. Влияние нитрилов и амидов на рост и нитрилгидратазную активность штамма Rhodococcus sp. gt1//Прикл. биохимия и микробиология. 2003. Т. 39. № 1. С. 63-68.
- Клонирование ДНК. Методы. М., 1988. 538 с.
- Демаков В.А. и др. Почвенные актинобактерии рода Rhodococcus, обладающие высокой амидазной активностью//Вестник Пермского ун-та. 2009. С. 79-83.
- Кузнецова М.В. и др. Распространение нитрилконвертирующих бактерий в почвах Пермского края//Вестник Пермского ун-та. 2007. Т. 5. С. 96-99.
- Asano Y. Overview of screening for new microbial catalysts and their uses in organic synthesis -selection and optimization of biocatalysts//J. Biotechnol. 2002. V. 94. P. 65-72.
- Banerjee A., Sharma R., Banerjee U.C. The nitriledegrading enzymes: current status and future prospects//Appl. Microbiol. Biotechnol. 2002. V. 60. P. 33-44.
- Cowan D. Biochemistry and biotechnology of mesophilic and thermophilic nitrile metabolizing enzymes//Extremophiles. 1998. V. 2. № 3. P. 207-216.
- Fornaud D., Arnaud A. Aliphatic and enantioselective amidases: from hydrolysis to acyl transfer activity//Appl. Microbiol. 2001. V. 91. № 9. P. 381-393.
- Kotlova E.K. et al. Isolation and primary characterization of an amidase from Rhodococcus rhodochrous//Biochemistry. 1999. V. 64. N. 4. P. 384-389.


