Упрощение трипсинового гидролизата протеома E. coli при поиске пептидов РНК-полимеразы методом масс-спектрометрии с электрораспылительной ионизацией (ES-TOF-MS)
Автор: Корнева Е.С., Верещагин А.Л., Новиков А.В., Грачев М.А.
Журнал: Научное приборостроение @nauchnoe-priborostroenie
Рубрика: Оригинальные статьи
Статья в выпуске: 4 т.17, 2007 года.
Бесплатный доступ
Упрощение трипсинового гидролизата протеома E. coli при поиске пептидов РНК-полимеразы достигнуто путем применения гельпроникающей хроматографии. В результате анализа методом времяпролетной масс-спектрометрии с электрораспылительной ионизацией полученных при хроматографии фракций идентифицированы пептиды, наличие которых свидетельствует о присутствии РНК-полимеразы в исходном суммарном белке.
Короткий адрес: https://sciup.org/14264510
IDR: 14264510 | УДК: 577.112:579.842.11
Текст научной статьи Упрощение трипсинового гидролизата протеома E. coli при поиске пептидов РНК-полимеразы методом масс-спектрометрии с электрораспылительной ионизацией (ES-TOF-MS)
Для поиска индивидуальных белков, возможность присутствия которых в протеомах живых клеток вытекает из структуры их геномов, широко применяются методы протеомики. Целью таких исследований является решение как фундаментальных проблем молекулярной биологии, так и практически важных задач, например задач медицинской диагностики. Наиболее широко применяется сочетание техники двумерного электрофореза — электрофокусировки — с масс-спектрометрией c лазерной ионизацией (MALDI). Применение этой техники имеет два серьезных ограничения. Во-первых, выделение чистых белковых фракций ограничивается недостаточной емкостью пластин двумерных гелей, следствием которой является загрязнение целевых белков мажорными клеточными белками, например тубулинами и актинами. Во-вторых, метод MALDI, как и другие методы масс-спектрометрии пептидов, является принципиально неколичественным, и потому, например, отсутствие искомых сигналов в масс-спектрах никак не говорит об отсутствии соответствующих белков в исследуемых клетках.
В связи с этим в последние годы развивается альтернативный подход, который иногда называют пептидомикой [1], — поиск не самих целевых белков, а индикаторных продуктов их расщепления, например характерных триптических пептидов. Для разделения пептидов можно применять не гель-электрофорез, а жидкостную хроматографию, динамический диапазон допустимой нагрузки для которой гораздо более широк по сравнению с гель-электрофорезом. Для реализации техники пептидомики необходимо выделить из клеток суммарный белок и подвергнуть его химической модификации для разрыва дисульфидных связей и их последующей защиты (обычно путем алкилирования йодуксусной кислотой). Необходимо также принять меры для предотвращения неконтролируемого расщепления белков клеточными протеазами. Далее необходимо упростить подвергаемую масс-спектрометрии сложнейшую смесь пептидов для того, чтобы обеспечить ионизацию целевых пептидов и их надежную детекцию в масс-спектрах.
Целью настоящей работы было исследование возможности идентификации в протеоме E. coli РНК-полимеразы — белка с умеренным уровнем экспрессии (7000 копий на клетку [2]) путем трипсинолиза, гель-проникающей хроматографии и времяпролетной масс-спектрометрии с электро-распылительной ионизацией.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использовали концентрированную культуру E. coli , ацетонитрил ("Криохром", Санкт-Петербург), йодуксусную кислоту, трифторуксусную кислоту фирмы "Merk" (Германия), PMSF ("Bio Rad", США), меркаптоэтанол ("Хеликон", Москва), азид натрия ("Лабтех", Москва), ацетон ("Экос-1"), дигидроортофосфат калия, тиомочевину, додецилсульфат натрия (SDS) фирмы "Реахим" (Москва), Tris и мочевину фирмы "Sigma" (Германия), TPCK трипсин ("Sigma" T 1426, Германия).
Лизаты клеток E. coli готовили, экстрагируя биомассу Tris-буфером, содержащим мочевину, тиомочевину, SDS. Полученный лизат последовательно карбоксиметилировали 0.05 М йодуксусной кислотой, восстановили 0.7 М меркаптоэтанолом и карбоксиметилировали 0.075 М йодуксусной кислотой. Белок из смеси осаждали ацетоном [3]. Последующий трипсинолиз выполняли
Номер фракции
Объем, мкл
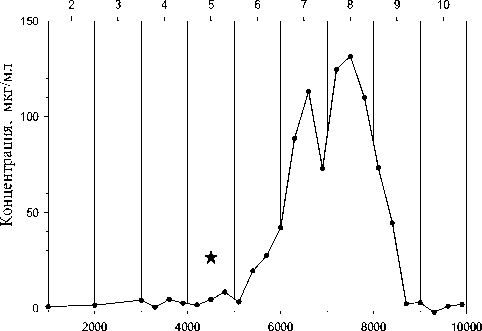
Рис. 1. Профиль гель-фильтрации триптических пептидов E. coli по стандартной методике [3, 5], время инкубирования 6 ч.
Гель-фильтрацию проводили на колонке 20×0.5 см с сефадексом G-25. Элюирование — 0.2 М фосфатным буфером (рН = 6.5) с 0.02 % NаN3, собирая фракции по 1 мл. Фракции концентрировали и короткое время диализовали на холоде.
Хроматограммы записывали на микроколоноч-ном жидкостном хроматографе с многоволновой фотометрической детекцией "Милихром А-02" (ЗАО "ЭкоНова", Новосибирск, Россия). Масс-спектрометрический анализ проводили на приборе MX 5303, оборудованном электрораспылительным источником ионов (ESI) и времяпролетным масс-анализатором (TOF) в режиме регистрации положительных ионов (Институт аналитического приборостроения РАН, Санкт-Петербург, Россия). Объем анализируемой пробы 10 мкл. Скорость подачи образца 1–2 мкл/мин. Анализ фракций производился в режиме "жидкостной хроматограф—масс-спектрометр" в режиме прямой стыковки.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Известно, что в геноме E. coli закодировано около 4800 белков, дающих при трипсинолизе 37 000 пептидов. РНК-полимераза E. coli состоит из пяти субъединиц: β' (165 кДа), β (155 кДа), двух α (35 кДа) и σ (70 кДа). Прогнозируемая по геному смесь пептидов РНК-полимеразы E. coli представляет собой набор из приблизительно 450 компонентов. Из них основную часть составляют пептиды длиной менее 15 аминокислотных остатков (390 штук), а пептиды, содержащие более чем 20 аминокислот, имеются в количестве лишь 37 штук. Мы предположили, что, поделив сумму триптических пептидов E. coli с помощью гель-проникающей хроматографии, можно снизить ее сложность в области крупных молекул до степени, достаточной для регистрации индивидуальных пептидов РНК-полимеразы в масс-спектре. Основное внимание было уделено изучению фракций, содержащих длинные пептиды.
Суммарный белок E. coli выделяли из супернатанта, полученного путем центрифугирования гомогената бактериальной массы в смеси c 7 M мочевиной, 2 М тиомочевиной, 0.1 % SDS и 0.04 M Tris.OH (рН 8.5). Супернатант последовательно подвергали алкилированию, восстановлению и вновь алкилированию. Белок осаждали ацетоном, растворяли в 0.1 М растворе Tris.OH и гидролизовали трипсином до получения постоянного профиля обращенно-фазовой жидкостной хроматографии.
Полученную смесь триптических пептидов E. coli разделяли методом гель-проникающей хроматографии на сефадексе G-25 (рис. 1). Масс-спектрометрии было подвергнуто 7 фракций. Судя по выходной кривой гель-проникающей хроматографии, короткие пептиды были отсеяны на 95 %.
Отобранные для дальнейшего анализа фракции короткое время диализовали против воды и концентрировали упариванием в вакууме, а затем доводили до объема 40 мкл. В полученных образцах методом времяпролетной масс-спектрометрии были обнаружены пептиды, распределившиеся в соответствии с размерами (табл., рис. 2).
Пептиды РНК-полимеразы, обнаруженные в трипсинолизате биомассы E. coli
|
Аминокислотный участок |
Аминокислотная последовательность |
Субъединица |
М. в. |
№ фракции |
|
1356–1369 |
LIPAGTGYAYHQDR |
B' |
1560.7685 |
6 |
|
1264–1284 |
ATIVNAGSSDFLEGEQVEYSR |
B' |
2270.952 |
5 |
|
912–933 |
GEAIGVIAAQSIGEPGTQLTMR |
B' |
2198.226 |
5 |
1100.6
1136.98
1100.11
1101.1
1101.59
1136.48
1137.48
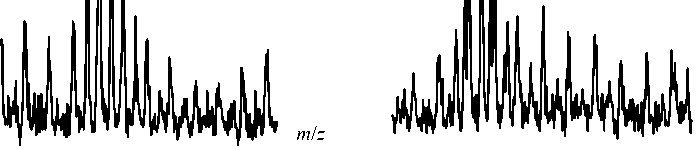
m / z
GEAIGVIAAQSIGEPGTQLTMR
ATIVNAGSSDFLEGEQVEYSR
1080 1100 1120 1140 1160
Рис. 2. Фрагмент масс-спектра фракции 5, содержащий сигналы пептидов GEAIGVIAAQSIGEPGTQLTMR, ATIVNAGSSDFLEGEQVEYSR субъединицы B′
РНК полимеразы E.coli . На врезке показаны в увеличенном масштабе участки ( ★ )
В группе крупных пептидов (длиннее 20 аминокислотных остатков) два из интересовавших нас сигнала были отчетливо видны на фоне сигналов фрагментов, принадлежащих другим клеточным белкам (рис. 2). Массы, предположительно соот ветствующие двухзарядным катионам пептидов
GEAIGVIAAQSIGEPGTQLTMR, ATIVNAGSSDFLEGEQVEYSR,
781.38
б
781.98
782 39
776 778 780 782 784 786 788 790
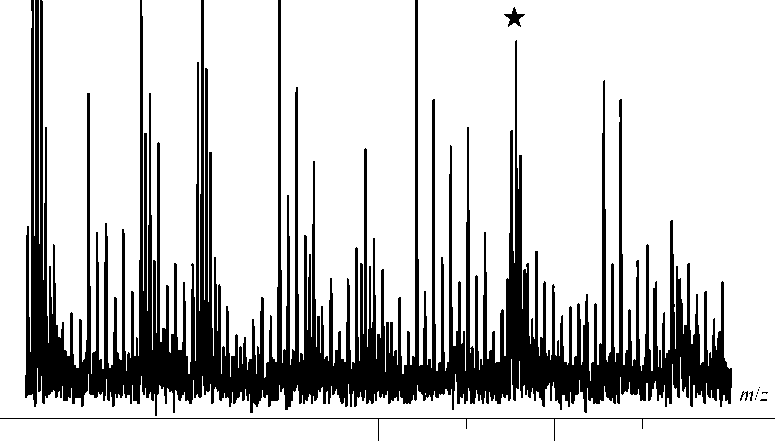
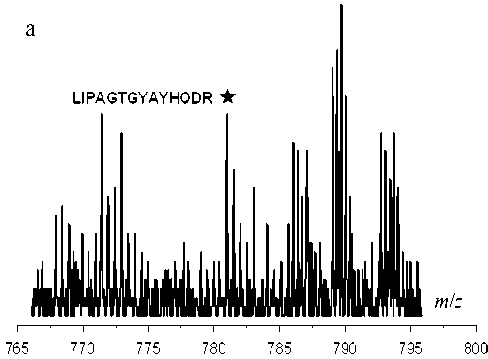
Рис. 3. Фрагмент (а) масс-спектра фракции 6, содержащий сигнал пептида LIPAGTGYAYHQDR субъединицы B′ РНК полимеразы E. Coli . Участок спектра ( ★ ) показан в увеличении на (б)
исходном белке присутствовала РНК-полимераза.
Принято считать, что трипсиновый гидролизат протеома живой клетки, состоящий из нескольких тысяч пептидов, представляет большую сложность для обнаружения в нем конкретных пептидов из-за трудоемких операций по их выделению или обогащению. Нами показано, что методом, включающим лишь одну стадию упрощения полного трипсинового гидролизата суммарного белка, можно установить наличие в нем протеина, не являющегося мажорным.
Работа выполнена в рамках программы фундаментальных исследований Российской академии наук "Физико-химическая биология" (проект 10.3) и гранта РФФИ 04-04-48669.


